Method Article
Microarrays de DNA: Controle de Qualidade da amostra, Hibridação Array e Digitalização
Neste Artigo
Resumo
Nós demonstramos o uso de microarrays de DNA para perfis de expressão do sistema nervoso. Descrevemos RNA controle de qualidade, rotulagem da amostra, e hibridização da matriz e de digitalização.
Resumo
Microarray perfil de expressão do sistema nervoso fornece uma abordagem poderosa para a identificação de atividades gene em diferentes estágios de desenvolvimento, diferentes estados fisiológicos ou patológicos, resposta à terapia, e, em geral, qualquer condição que está sendo testada experimentalmente 1. Perfil de expressão de tecidos neurais exige isolamento de RNA de alta qualidade, a amplificação do RNA isolado e hibridização de microarranjos de DNA. Neste artigo vamos descrever protocolos de experimentos de microarray reprodutíveis a partir do tecido de tumor cerebral 2. Vamos começar por realizar uma análise de controle de qualidade das amostras de RNA isolado com 2100 da Agilent Bioanalyzer "lab-on-a-chip" de tecnologia. Amostras de RNA de alta qualidade são essenciais para o sucesso de qualquer experimento de microarray, e os Bioanalyzer 2100 fornece uma medida rápida e quantitativa da qualidade da amostra. Amostras de RNA são amplificados e rotulado através da realização de transcrição reversa para a obtenção de cDNA, seguido por transcrição in vitro na presença de nucleotídeos marcados para produzir cRNA rotulados. Usando um kit rotulagem dual-cores, vamos rotular nossa amostra experimental com Cy3 e uma amostra de referência com Cy5. Ambas as amostras serão então combinadas e hibridizado com arrays 4x44 da Agilent K. Duas cores matrizes oferecem a vantagem de uma comparação direta entre duas amostras de RNA, aumentando a precisão das medições, em especial para pequenas mudanças nos níveis de expressão, porque as duas amostras de RNA são cruzados competitivamente a um microarray único. As matrizes serão verificados nos dois comprimentos de onda correspondentes, ea proporção de Cy3 para Cy5 sinal para cada recurso será usado como uma medida direta da abundância relativa do mRNA correspondente. Esta análise identifica genes que são diferencialmente expressos em resposta às condições experimentais sendo testados.
Protocolo
1. Análise de controle de qualidade da amostra de RNA com 2100 Bioanalyzer
Antes de começar,
- Desnaturar a escada e suas amostras a 70 ° C por 10 min. Frio imediatamente em gelo.
- Limpe o instrumento com eletrodos RNaseZAP por 1 min, seguido por RNase água por 10 seg. Permitir que os eletrodos para secar ao ar.
- Prepare a matriz de gel por pipetagem 550 mL da matriz de gel em um filtro de spin fornecido com o kit. Centrifugar a 1500 RCF por 10 min em temperatura ambiente. Alíquota do filtrado gel em 65 mL alíquotas e armazenar a 4 ° C por até 1 mês.
- Para preparar a estação de priming chip,
- Inserir uma seringa de 1 ml por meio do clipe e no adaptador luer lock.
- Ajuste a placa de base para a posição C, soltando o parafuso sob a base, o levantamento da placa e reapertar o parafuso.
- Ajustar o clip da seringa na posição superior.
- Equilibrar a concentração de corantes à temperatura ambiente. Vortex por 10 segundos. Adicionar 1 ml de tintura para uma alíquota de 65 mL de gel filtrada. Vortex bem e centrifugar a 13,000 rcf durante 10 min à temperatura ambiente. Use dentro de um dia.
Quando estiver pronto para executar suas amostras, - Posição de um chip na estação de priming. Neste protocolo estamos usando RNA 6.000 fichas nano. Para carregar o gel,
- Pipetar 9 mL da mistura de gel-dye na bem marcado com um branco "G" contra um fundo preto. Verifique se o seu ponta é posicionada na parte inferior do poço.
- A posição do êmbolo da seringa de 1 ml. Perto da estação de priming. Certifique-se de um clique-locks.
- Pressione o êmbolo lenta mas firmemente até que seja realizada pelo clipe.
- Aguarde 30 segundos. Lançamento do clipe.
- Deixe o êmbolo subir por si só. Depois que ele pára de se mover, espere alguns segundos e puxar o êmbolo de volta à posição de 1 ml. Abrir a estação de priming.
- Pipetar 9 mL da mistura de gel-dye em cada um dos dois poços marcados "G".
- Pipetar 5 mL do marcador em cada um dos 12 poços de amostra e na escada bem.
- Pipetar 1 mL da escada em escada bem preparado.
- Pipetar 1 mL de cada amostra nas cavidades da amostra.
- Pipetar 1 mL do marcador em cada amostra não utilizada também.
- Vortex do chip de 1 min a 2.400 rpm.
- Para executar o chip,
- Iniciar o Software Especialista 2100.
- A posição do chip e feche a tampa. Se o instrumento está on-line, um ícone irá indicar se a tampa é aberta ou fechada e que tipo de chip é inserido. Verifique se a porta correta está selecionada.
- Executar o ensaio dentro de 5 min de carga das amostras para evitar a evaporação.
2. Amplificação e rotulagem
Para se preparar para análise microarray, amostras de RNA são amplificados e rotulado, geralmente em uma reação com base em T7 RNA polimerase 3. CDNA dupla fita é gerado pela transcrição reversa. cDNA é usada em uma reação de transcrição in vitro para gerar o que é conhecido como cRNA. Esta reação é realizada na presença de ribonucleotídeos rotulados, produzindo quantidades micrograma de RNA marcado para a hibridização matriz. A escolha de amplificação / rotulagem métodos depende da plataforma de microarray subseqüente a ser utilizado. Nesta seção, descrevemos a geração de RNA fluorescente etiquetado utilizando kit da Agilent rotulagem rápida Amp.
- Pipeta ng 50-5 mg de RNA em um tubo de 1,5 ml. O volume não deve exceder 8,3 mL. Se necessário, concentrar a sua amostra de RNA por centrifugação a vácuo ou precipitação com álcool.
- Adicionar 1,2 mL de primer T7.
- Trazer para um volume final de 11,5 mL com RNase água.
- Incubar a 65 ° C por 10 min para desnaturar o RNA. Recomendamos o uso de um termociclador com uma tampa quente para evitar a condensação no topo do tubo.
- Durante a incubação de 10 min, Buffer Strand quente 5X Primeiro a 80 ° C por 5 min. Vortex e spin down. Mantenha em temperatura ambiente até que esteja pronto para uso.
- Após a incubação de 10 min, esfriar rapidamente as amostras de RNA desnaturado no gelo.
- Prepare a mistura mestre cDNA. Por reação, adicione o seguinte:
- 4 mL de buffer Strand 5X First
- 2 l 0,1 M DTT
- 1 ml 10 mM dNTPs
- Uma enzima MMLV-RT mL
- 0,5 mL Out RNase.
- Misturar os componentes invertendo o tubo 4 vezes. Não vortex. Girar para coletar o conteúdo na parte inferior do tubo.
- Adicionar 8,5 mL de master mix por amostra. Misture por pipetagem cima e para baixo.
- Incubar por 2 horas a 40 ° C, seguido por 10 min a 65 ° C. Recomendamos o uso de um termociclador com uma tampa aquecida.
- Transferir os tubos para o gelo por 5 min.
- Prepare a mistura de mestre de transcrição. Por reação, adicione o seguinte:
- 15,3 de água livre de nuclease mL
- 20 lTampão de transcrição 4X
- 6 mL 0,1 M DTT
- 8 mL PNT
- 6,4 mL de PEG 50% (PEG deve ser pré-aquecido a 40 ° C e agitadas para garantir a ressuspensão)
- 0,5 mL de RNase Out
- 0,6 mL pirofosfatase inorgânicos
- 0,8 mL T7 RNA polimerase
- 2,4 mL Cy3 ou Cy5 CTP
- Brevemente girar a amostras a recolher o conteúdo na parte inferior do tubo.
- Adicionar 60 mL da mistura de Transcrição Mestre por amostra.
- Incubar a 40 ° C por 2 horas.
- Adicionar 20 mL de RNase água.
- Purificar o cRNA rotulados para remover nucleotídeos sem personalidade jurídica. Recomendamos o uso de colunas RNeasy Qiagen mini.
- Quantificar o cRNA rotulados. Recomendamos a utilização de um espectrofotômetro NanoDrop2000 em Microarray modo de medição. Certifique-se de selecionar RNA-40 como o tipo de amostra.
- Concentração registro de exemplo, o rendimento (calculado como a concentração multiplicado pelo volume) e atividade específica (calculado como 1000 multiplicado pela ração de concentração de corante sobre a concentração cRNA). Para a hibridização microarray, é necessário para alcançar um rendimento de pelo menos 825 ng e uma atividade específica de pelo menos 8 pmol / mg.
3. Hibridização de microarranjos
- Ligue a estação de hibridação e regulado a 65 ° C, pelo menos, duas horas antes de hibridização começa.
- Para preparar a amostra de hibridização, misture o seguinte em um tubo de 1,5 ml:
- 825 ng Cy3-cRNA (isto é normalmente o "tratado" amostra)
- 825 ng Cy5-cRNA (geralmente é a sua "referência" da amostra)
- 11 mL 10X agente de bloqueio.
- Adicionar RNase água para um volume final de 52,8 mL
- Adicionar 2,2 mL de buffer fragmentação.
- Vortex em baixa velocidade para misturar.
- Incubar a 60 ° C por exatamente 30 min. Este é o passo cRNA fragmentação, que gera fragmentos rotulados para a hibridização matriz. É muito importante para incubar exatamente como descrito. Recomendamos o uso de um termociclador com uma tampa aquecida. É muito importante para incubar exatamente como descrito para gerar sondas do comprimento médio correto. Fragmentação excessiva ou insuficiente resultará em falsos negativos ou falsos positivos.
- Adicionar 55 mL de tampão de hibridação 2X de parar de fragmentação. Mix por pipetagem, tendo especial cuidado para não introduzir bolhas.
- Centrifugar por 1 min em velocidade máxima para coletar o conteúdo na parte inferior do tubo. Se as bolhas são observadas, centrífuga mais tempo para removê-los.
- Colocar o tubo no gelo, protegidas da luz. Carga da amostra sobre a matriz o mais rapidamente possível.
- Para carregar a matriz, primeiro prepare a câmara de hibridização. Este protocolo descreve o uso de matrizes da Agilent 4x44K com o sistema de hibridação Roche-NimbleGen e câmaras misturador A4.
- Coloque uma lâmina de microarray no lado de código de barras de montagem / desmontagem ferramenta, em primeiro lugar.
- Abra um mixer A4 e expor o adesivo. Segurando a matriz e montagem / desmontagem ferramenta para evitar qualquer movimento, coloque o misturador A4 no slide, a partir da extremidade mais distante. Verifique se ele está corretamente alinhado à ferramenta. Vara do misturador ao longo de toda a lâmina matriz.
- Puxe a partir do final do misturador para remover o slide / montagem mixer.
- Usando a ferramenta de zurrar, pressione ao longo de toda a junta adesiva para ter certeza que está bem colado à theslide. Pequenas bolhas de ar presas entre o adesivo ea lâmina pode ser visto contra um fundo escuro. Tire um tempo extra para remover essas bolhas com a ferramenta de zurrar. Sua matriz está agora pronto para ser carregado.
- Use uma pipeta de deslocamento positivo para carregar 100 ml da amostra de hibridação. Empurre a ponta firmemente dentro do buraco da porta, então dispensar lenta e suavemente de modo que o ar não fica preso na área da matriz. Nunca tente sugar a amostra de volta. Uma vez que toda a amostra foi dispensado, manter a ponta no buraco da porta sem soltar o êmbolo. Quando a amostra cobre todo o conjunto e líquido começa a sair do porto de ventilação, remover rapidamente a pipeta. Apenas liberte o êmbolo uma vez a dica é longe do slide.
- Dab delicadamente o líquido em excesso em ambas as portas. Certifique-se de não chamar a amostra para fora da câmara em si.
- Cobrir os buracos da porta com etiquetas, usando uma pinça.
- Coloque o slide na estação de hibridização, e verificar que os buracos da bexiga são corretamente posicionado sobre o O-rings. Isso irá garantir boa mistura durante a hibridação.
- Feche a tampa deslizante ea tampa da estação. Conjunto da estação de mistura modo B.
- Hibridizar a matriz a 65 ° C por 17 horas.
- Prepare Wash Buffer 2 pela adição de Triton X-102 a uma concentração final de 0,005%. Mantenha a 37 ° C durante a noite.
4. Lavar Microarray
- Adicionar Triton X-102 a Wash Buffer 1 para uma concentração final de 0,005%. Mantê-la em sala de temperature.
- Quando terminar de hibridação, encher um copo prato "A" com Wash Buffer 1. O prato deve ser grande o suficiente para manter a ferramenta de montagem / desmontagem e permitir que algumas manobras também. Todas as lavagens devem ser feitas em vidro. Evite pratos de plástico, uma vez que eles tendem a lixiviar compostos, resultando em alta de fundo na matriz.
- Colocar um porta-lâminas e uma barra de agitação em um "B" coloração prato. Preencher com Tampão de lavagem 1, certificando-se que cobre o rack. Coloque em um prato agitar à temperatura ambiente.
- Colocar uma barra de agitação em um prato coloração vazio "C" e coloque em um prato misture.
- Remover a matriz e colocá-lo na ferramenta de montagem / desmontagem. Submergir o conjunto inteiro no prato "A".
- Enquanto segura a ferramenta de montagem / desmontagem eo slide de lados opostos com uma mão, com cuidado retire o misturador A4. Certifique-se de não arranhar a superfície da matriz.
- Colocar rapidamente o slide no rack na coloração prato "B". A partir de agora, a exposição ao ar deve ser minimizado desde o corante é sensível à Cy5 ozônio. Não lave mais de 8 matrizes de cada vez.
- Lavar por 1 minuto mexendo na velocidade média.
- Preencha coloração prato "C", com pré-aquecido tampão de lavagem 2. Transferir as lâminas e lavar por 1 minuto.
- Muito lentamente, remova a porta-lâminas de coloração prato "C" para minimizar gotículas deixadas para trás no slide.
- Seca o slide, girando por 2 minutos. Se uma centrífuga compatível não estiver disponível, centrifugar o slide em um tubo de 50 ml cônico ou golpe gás argônio sobre ele.
- Colocar as lâminas em um tubo cônico de 50 ml, e preenchê-lo com gás argônio. Digitalizar imediatamente para evitar perda de sinal. Recomendamos a utilização do scanner 4000B GenePix de dispositivos moleculares.
5. Resultados representativos:
Boa qualidade total de amostras de RNA deve produzir apenas dois picos principais, quando executado em um Bioanalyzer para controle de qualidade, correspondente ao 2 principais espécies de RNA ribossômico. Alguma degradação RNA vai mostrar como uma mancha antes do pico RNA ribossomal primeiro. Severamente degradadas, RNA de baixa qualidade vai mostrar um pico amplo ou uma série de picos em tempos de retenção baixa, enquanto os dois picos ribossomal RNA será de intensidade muito baixa ou não identificáveis em tudo. Para exemplos da saída Bioanalyzer 2100, veja a figura 1.
O software 2100 de peritos irá calcular um número de integridade do RNA, ou RIN, como uma medida quantitativa da qualidade do RNA. Amostras de alta qualidade, com valores RIN superior a 9, são obviamente melhores para aplicações de microarray. No entanto, temos utilizado amostras com valores RIN tão baixo quanto 5,2 em geração de dados de microarray de qualidade razoável.
Matrizes de boa qualidade deve produzir sinal de alta em valores relativamente baixos PMT. Em nosso experimento, a maior parte das transcrições devem estar presentes em níveis semelhantes na amostra experimental e de referência; maciça, amplas mudanças na expressão genética provavelmente não têm qualquer significado biológico. Por isso, a maioria do array deve olhar amarelo, em vez de verde ou vermelho. Sinais de boa qualidade também deve estar em uma faixa dinâmica de tal forma que os histogramas sinal completamente sobrepõem. Por exemplo, veja a figura 2.
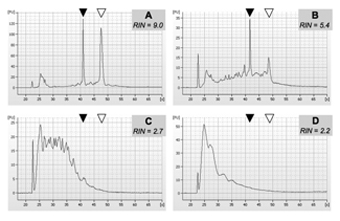
Figura 1. Exemplo de quatro amostras de RNA isoladas de tecido do cérebro humano tumor. RNA foi isolado utilizando o protocolo apresentado em 3.1.1. Qualidade da amostra foi avaliada através do Bioanalyzer 2100 em um chip 6000 RNA como descrito em 3.1.3. As amostras são representativas de quatro qualidades distintas RNA: A, muito boa qualidade RNA (RIN> 9); B, RNA parcialmente degradadas (RIN 5-6, observe o pico significativamente mais baixos para 28S rRNA); C, RNA altamente degradadas (RIN < 3); D, quase completamente degradadas RNA (RIN = 2). Temos utilizado com sucesso amostras com qualidades semelhantes ou melhores do que B (RIN> 5,2) para aplicações de microarray a jusante. Arrowheads sólida e aberta indicar a posição do 18S rRNA 28S e espécies, respectivamente.
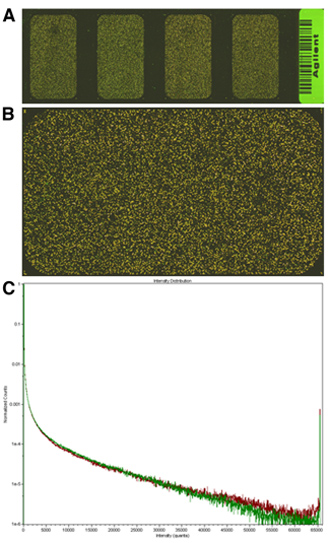
Figura 2. Exemplos de imagens da matriz e gráficos de dispersão. A, visualização de slide todo, mostrando as quatro matrizes no formato Agilent 4X44K; B, varredura de alta resolução de uma das áreas de matriz, note que nenhum dos sinais (vermelho ou verde) é predominante; C, gráfico de dispersão da imagem em B, note que os sinais se sobrepõem totalmente e não mais de 1x10 -6 características são a intensidade de saturação.
Discussão
Perfil de expressão com microarrays de DNA oferece uma abordagem simples para identificar genes diferencialmente expressos entre duas amostras biológicas. Experiências bem sucedidas de perfis de expressão exigir amostras de RNA de alta qualidade, etiquetagem robusta e hibridização. Em nossa experiência, as matrizes comerciais e kits de rotulagem Agilent fornecem dados de alta qualidade a um custo razoável. Enquanto Agilent também oferece sua própria hibridização e máquinas de varredura, nós somos a favor do sistema de hibridação de Roche-NimbleGen eo GenePix 4000B scanner microarray de dispositivos moleculares. Note que a unidade de hibridização Roche-NimbleGen era conhecido anteriormente como o sistema de hibridização MAUI. Após a recente aquisição empresarial pela Roche, o mixers A4 foram descontinuados. A partir do momento da redação deste texto, eles ainda podem ser encontrados através de outros vendedores, como Kreatech Diagnostics ( www.kreatech.com ; este site também oferece uma ferramenta conveniente compatibilidade array), mas a longo prazo disponibilidade de estoque é incerto. Sistemas de hibridação estão disponíveis (por exemplo, de Agilent), no entanto, se mixers compatíveis podem ser encontradas para a matriz a ser utilizado, recomendamos o sistema Roche-NimbleGen para a sua qualidade e reprodutibilidade.
Divulgações
Agradecimentos
Este trabalho foi financiado por doações de ED da S. James McDonnell Fundação 21 de Century Award Program, eo Prêmio Diretor do NIH Innovator Novo. GAB foi apoiado em parte por uma bolsa de pós-doutorado do Instituto de Medicina Regenerativa da Califórnia.
Materiais
| Name | Company | Catalog Number | Comments |
| 2100 Bioanalyzer | Agilent Technologies | G2943CA | Includes chip priming station, vortexer, software and laptop computer |
| 2100 Bioanalyzer electrophoresis set | Agilent Technologies | G2947CA | |
| RNA 6000 Nano kit | Agilent Technologies | 5067-1511 | includes size ladder, marker, gel matrix, dye, electrode cleaners, spin filters and nanochips |
| RNase Zap | Applied Biosystems | AM9780M | |
| Quick Amp labeling kit, two-color | Agilent Technologies | 5190-0444 | |
| RNeasy mini kit | Qiagen | 74104 | |
| Gene Expression Hybridization Kit | Agilent Technologies | 5188-542 | Includes Blocking agent, Fragmentation buffer and Hybridization buffer |
| Gene Expression Wash Buffer Kit | Agilent Technologies | 5188-5327 | Includes Wash Buffers 1 and 2 and Triton X-102 |
| Hybridization Station | Roche Group | 05223652001 | 4-slide model |
| Hybridization System Accesory Kit | Roche Group | 05327695001 | Contains verification assembly for testing mixing, replacement O-rings, disassembly tool, forceps and brayer |
| A4 mixer hybridization chambers | |||
| Whole Human Genome (4x44) Oligo Microarray | Agilent Technologies | G4112F | |
| Positive displacement pipette | Gilson, Inc. | F148504 | |
| Capillary pistons for positive displacement pipette | Gilson, Inc. | F148415 | |
| Microarray dryer | Nimblegen | 05223636001 | |
| Microarray fluorescent scanner, Axon GenePix 4000B | Molecular Devices | GENEPIX4000B | |
| GenePix Pro 6.0 software | Molecular Devices |
Referências
- Diaz, E. One Decade Later: What has Gene Expression Profiling Told us About Neuronal Cell Types, Brain Function and Disease. Curr Genomics. 10, 318-318 (2009).
- Barisone, G. A. Expression profiling in Neuroscience, edited by Ioannis Karamanos. , SPRINGER SCIENCE+BUSINESS MEDIA, LLC. New York. (2010).
- Gelder, R. N. V. an Amplified RNA synthesized from limited quantities of heterogeneous cDNA. Proc Natl Acad Sci U S A. 87, 1663-1663 (1990).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados