Method Article
DNA microarray: Esempio di controllo della qualità, ibridazione e scansione di array
In questo articolo
Riepilogo
Dimostriamo l'uso di DNA microarray per profili di espressione del sistema nervoso. Noi descriviamo il controllo di qualità RNA, l'etichettatura del campione, e l'ibridazione di array e la scansione.
Abstract
Profilo di espressione Microarray del sistema nervoso fornisce un approccio efficace per individuare le attività di geni in diversi stadi di sviluppo, diversi stati fisiologici o patologici, la risposta alla terapia, e, in generale, qualsiasi condizione che viene testato sperimentalmente 1. Profili di espressione dei tessuti neurali richiede l'isolamento di RNA di alta qualità, amplificazione del RNA isolato e ibridazione di DNA microarray. In questo articolo si descrivono i protocolli per gli esperimenti microarray riproducibili dal tessuto cerebrale tumore 2. Inizieremo con l'esecuzione di un'analisi di controllo di qualità di campioni di RNA isolato con Agilent 2100 Bioanalyzer "lab-on-a-chip" tecnologia. Campioni di RNA di alta qualità sono fondamentali per il successo di qualsiasi esperimento microarray, e il 2100 Bioanalyzer fornisce una rapida misurazione quantitativa della qualità del campione. Campioni di RNA vengono poi amplificati ed etichettato eseguendo la trascrizione inversa di ottenere cDNA, seguita da trascrizione in vitro in presenza di nucleotidi etichettati in modo da produrre etichettati cRNA. Di un doppio-colore kit etichettatura, ci etichetta nostro campione sperimentale con Cy3 e un campione di riferimento con Cy5. Entrambi i campioni saranno poi combinati e ibridato ad array di Agilent K 4x44. A due colori array offrono il vantaggio di un confronto diretto tra due campioni di RNA, aumentando così la precisione delle misure, in particolare per le piccole variazioni nei livelli di espressione, perché i due campioni di RNA sono ibridate competitivo ad un singolo microarray. Gli array saranno esaminati a due lunghezze d'onda corrispondenti, e il rapporto tra Cy3 per Cy5 segnale per ciascuna funzione sarà utilizzata come una misura diretta della relativa abbondanza di mRNA corrispondente. Questa analisi identifica i geni che sono differenzialmente espressi in risposta alle condizioni sperimentali in fase di test.
Protocollo
1. Analisi della qualità controllo del campione di RNA con 2100 Bioanalyzer
Prima di iniziare,
- Denaturare la scala ed i campioni a 70 ° C per 10 min. Freddo immediatamente in ghiaccio.
- Pulire gli elettrodi strumento con RNaseZAP per 1 minuto, seguita da RNase-free acqua per 10 sec. Lasciare gli elettrodi asciugare all'aria.
- Preparare la matrice del gel da pipeting 550 microlitri della matrice del gel in un filtro rotativo fornito con il kit. Centrifugare a 1.500 RCF per 10 minuti a temperatura ambiente. Aliquota del filtrato gel in 65 microlitri aliquote e conservare a 4 ° C per un massimo di 1 mese.
- Per preparare la stazione di chip di priming,
- Inserire una siringa da 1 ml con la clip e all'adattatore luer lock.
- Regolare la piastra di base alla posizione C svitando la vite sotto la base, sollevando il piatto e riavvitare la vite.
- Regolare la clip siringa per la prima posizione.
- Equilibrare il concentrato tintura a temperatura ambiente. Vortex per 10 sec. Aggiungere 1 ml di tintura di un'aliquota di 65 ml di gel filtrato. Vortex bene e centrifugare a 13.000 RCF per 10 minuti a temperatura ambiente. Utilizzare entro un giorno.
Quando si è pronti per eseguire i vostri campioni, - Posizione di un chip sulla stazione di adescamento. In questo protocollo che stiamo usando RNA 6000 chip nano. Per caricare il gel,
- Pipettare 9 ml di gel colorante-mix nel pozzo contrassegnati con un bianco "G" su uno sfondo nero. Assicurati che il tuo suggerimento è posizionato al fondo del pozzo.
- Posizionare lo stantuffo della siringa a 1 ml. Chiudere la stazione di adescamento. Assicurarsi che click-serrature.
- Premere il pistone verso il basso lentamente ma costantemente fino a quando non è in possesso della clip.
- Attendere 30 sec. Rilasciare il fermaglio.
- Lasciate che il pistone salire da solo. Dopo che smette di muoversi, attendere qualche secondo e tirare lo stantuffo per la posizione 1 ml. Aprire la stazione di adescamento.
- Pipettare 9 ml di gel colorante-mix in ciascuna delle 2 pozzi marcata "G".
- Pipettare 5 ml di marker in ciascuno dei 12 pozzetti del campione e nella scala bene.
- Pipettare 1 ml di scala in scaletta preparata bene.
- Pipettare 1 ml di ciascun campione nei pozzetti del campione.
- Pipettare 1 ml di marker in ogni campione non utilizzate bene.
- Vortex il chip per 1 min a 2.400 giri al minuto.
- Per eseguire il chip,
- Avviare il software di Expert 2100.
- La posizione del chip e chiudere il coperchio. Se lo strumento è on-line, verrà mostrata un'icona se il coperchio è aperto o chiuso e che tipo di chip è inserito. Assicurarsi che la porta sia selezionata.
- Eseguire il test entro 5 minuti di caricare i campioni per evitare l'evaporazione.
2. Amplificazione ed etichettatura
Per preparare per l'analisi microarray, campioni di RNA sono amplificati ed etichettato, di solito in una reazione basata su RNA polimerasi di T7 3. Doppia elica del DNA è generata da trascrizione inversa. cDNA viene utilizzato in un reazione di trascrizione in vitro per generare ciò che è noto come cRNA. Questa reazione avviene in presenza di ribonucleotidi etichettati, producendo quantità microgrammi di RNA marcato per l'ibridazione di array. La scelta di amplificazione / etichettatura metodi dipende dalla piattaforma microarray successive per essere utilizzato. In questa sezione, si descrive la generazione di fluorescente utilizzando RNA di Agilent rapida kit etichettatura Amp.
- Pipettare 50 ng a 5 mg di RNA in una provetta da 1,5 ml. Il volume non deve superare i 8,3 microlitri. Se necessario, concentrare il campione di RNA mediante centrifugazione sotto vuoto o di precipitazione con alcool.
- Aggiungere 1,2 l di fondo T7.
- Portare ad un volume finale di 11,5 microlitri con RNase-free acqua.
- Incubare a 65 ° C per 10 minuti per denaturare l'RNA. È consigliabile utilizzare un termociclatore con un coperchio caldo per evitare la condensa nella parte superiore del tubo.
- Durante l'incubazione 10 min, Buffer caldo Strand 5X prima a 80 ° C per 5 min. Vortex e spin giù. Tenere a temperatura ambiente fino al momento dell'uso.
- Dopo l'incubazione 10 minuti, raffreddare rapidamente i campioni di RNA denaturato sul ghiaccio.
- Preparare l'impasto cDNA master. Per reazione, aggiungere il seguente:
- 4 microlitri tampone 5X First Strand
- 2 microlitri 0,1 M DTT
- 1 ml 10 mM dNTP
- 1 ml MMLV-RT enzima
- Fuori 0,5 microlitri RNAsi.
- Miscelare i componenti mediante inversione della provetta 4 volte. Non vortice. Spin down per raccogliere il contenuto sul fondo della provetta.
- Aggiungere 8,5 ml di master mix per ogni campione. Mescolare pipeting su e giù.
- Incubare per 2 ore a 40 ° C, seguita da 10 minuti a 65 ° C. È consigliabile utilizzare un termociclatore con un coperchio riscaldato.
- Trasferire i tubi in ghiaccio per 5 min.
- Preparare la master mix trascrizione. Per reazione, aggiungere il seguente:
- 15,3 microlitri acqua priva di nucleasi
- 20 lTrascrizione buffer di 4X
- 6 microlitri 0,1 M DTT
- 8 NTP microlitri
- 6,4 microlitri PEG 50% (PEG dovrebbe essere preriscaldata a 40 ° C e in agitazione per garantire la risospensione)
- 0,5 microlitri RNasi Out
- 0,6 microlitri pirofosfatasi inorganici
- 0,8 microlitri T7 RNA polimerasi
- 2,4 microlitri Cy3 o Cy5 CTP
- In breve giro i campioni per raccogliere il contenuto sul fondo della provetta.
- Aggiungere 60 ml di Master Mix Trascrizione per campione.
- Incubare a 40 ° C per 2 ore.
- Aggiungere 20 ml di acqua RNase-free.
- Purificare l'cRNA etichettati in modo da rimuovere nucleotidi prive di personalità giuridica. Si consiglia di utilizzare le colonne Qiagen RNeasy mini.
- Quantificare il cRNA etichettati. È consigliabile utilizzare una NanoDrop2000 spettrofotometro in modalità di misurazione Microarray. Accertarsi di selezionare l'RNA-40 come il tipo di campione.
- Record concentrazione del campione, il rendimento (calcolato come la concentrazione moltiplicato per il volume) e l'attività specifica (calcolato a 1000 moltiplicato per la razione di concentrazione di colorante sulla concentrazione cRNA). Per l'ibridazione microarray, è necessario per ottenere una resa di almeno 825 ng e un'attività specifica di almeno 8 pmol / mg.
3. Microarray di ibridazione
- Accendere la stazione di ibridazione e impostare a 65 ° C almeno 2 ore prima di ibridazione comincia.
- Per preparare il campione di ibridazione, mescolare le seguenti in un tubo da 1,5 ml:
- 825 ng Cy3-cRNA (questo di solito è il tuo campione "trattati")
- 825 ng Cy5-cRNA (questo di solito è il tuo campione "di riferimento")
- 11 microlitri agente 10X blocco.
- Aggiungi RNasi-free acqua ad un volume finale di 52,8 microlitri
- Aggiungere 2,2 microlitri di buffer frammentazione.
- Vortex a bassa velocità per mescolare.
- Incubare a 60 ° C per esattamente 30 minuti. Questo è il passo frammentazione cRNA, che genera frammenti etichettati per l'ibridazione di array. E 'molto importante per incubare esattamente come descritto. Si consiglia l'uso di un termociclatore con un coperchio riscaldato. E 'molto importante per incubare esattamente come descritto per generare sonde della lunghezza media corretta. Frammentazione eccessiva o insufficiente si tradurrà in falsi negativi e falsi positivi.
- Aggiungere 55 ml di buffer di ibridazione 2X per fermare la frammentazione. Mescolare pipettando, prestando particolare attenzione a non introdurre bolle.
- Centrifugare per 1 minuto alla massima velocità per raccogliere il contenuto sul fondo della provetta. Se le bolle sono osservati, centrifuga più tempo per rimuoverli.
- Posizionare la provetta in ghiaccio, al riparo dalla luce. Caricare il campione sulla matrice il più presto possibile.
- Per caricare l'array, prima preparare la camera di ibridazione. Questo protocollo descrive l'uso di array Agilent 4x44K con la Roche-NimbleGen sistema di ibridazione e camere mixer A4.
- Posizionare un vetrino microarray nel fianco codice a barre di montaggio / smontaggio strumento, in primo luogo.
- Aprire un mixer A4 ed esporre l'adesivo. Tenendo l'array e il montaggio / smontaggio strumento per evitare qualsiasi movimento, posizionare il mixer A4 sulla diapositiva, a partire in fondo. Assicurarsi che sia correttamente allineato allo strumento. Bastone del miscelatore tutta la diapositiva array.
- Tirare a partire dalla fine del mixer per rimuovere il vetrino / gruppo miscelatore.
- Utilizzando lo strumento ragli, premere lungo tutta la guarnizione adesiva per assicurarsi che sia saldamente incollato alla theslide. Piccole bolle d'aria intrappolate tra l'adesivo e la diapositiva può essere visto su uno sfondo scuro. Prendere tempo per rimuovere queste bolle con lo strumento ragliare. L'array è ora pronto per essere caricato.
- Utilizzare una pipetta positivo spostamento di carico 100 l di campione ibridazione. Spingere la punta saldamente all'interno del foro porta, poi dispensare lentamente e dolcemente in modo che l'aria non è intrappolata nella zona di array. Non tentare mai di succhiare il campione backup. Una volta che l'intero campione è stato erogato, mantenere la punta sul buco porta senza rilasciare lo stantuffo. Quando il campione copre l'intera matrice e liquido comincia a venire fuori alla porta di sfiato, rimuovere velocemente la pipetta. Rilasciare solo il pistone una volta che la punta è lontano dalla diapositiva.
- Tamponare delicatamente il liquido in eccesso a entrambe le porte. Assicurati di non trarre esempio dalla camera stessa.
- Coprire i fori porta con adesivi utilizzando una pinzetta.
- Mettere il vetrino della stazione ibridazione, e controllare che i fori della vescica sono correttamente posizionato sopra l'O-ring. Questo assicurerà corretta miscelazione durante ibridazione.
- Chiudere il coperchio e far scorrere il coperchio della stazione. Impostare la stazione di miscelazione modalità B.
- Ibridare l'array a 65 ° C per 17 ore.
- Preparare il tampone di lavaggio 2, aggiungendo Triton X-102 ad una concentrazione finale di 0,005%. Mantenere a 37 ° C durante la notte.
4. Microarray lavaggio
- Aggiungi Triton X-102 di tampone di lavaggio da 1 a una concentrazione finale di 0,005%. Conservare a Temperatura ambientee.
- Quando l'ibridazione è finito, riempire un bicchiere piatto "A" con il tampone di lavaggio 1. Il piatto dovrebbe essere abbastanza grande per tenere il montaggio / smontaggio strumento e consentire alcune manovre pure. Tutti i lavaggi dovrebbe essere fatto in vetro. Evitare piatti di plastica, in quanto tendono a percolare composti nel fondo di alta dell'array.
- Mettere un rack scivolo e una ancoretta in una colorazione piatto "B". Riempire con tampone di lavaggio 1, facendo attenzione che copre il rack. Mettere su un piatto mescolate a temperatura ambiente.
- Mettere un ancoretta su un piatto colorazione vuota "C" e posto su un piatto mescolare.
- Rimuovere l'array e metterlo nel montaggio / smontaggio utensili. Immergere tutta l'assemblea nel piatto "A".
- Tenendo premuto il montaggio / smontaggio utensili e il vetrino da lati opposti con una mano, con cura staccare il mixer A4. Fai attenzione a non graffiare la superficie dell'array.
- Rapidamente posto il vetrino nel rack in colorazione piatto "B". D'ora in poi, l'esposizione all'aria deve essere ridotto al minimo in quanto il Cy5 colorante è sensibile all'ozono. Non lavare più di 8 array per volta.
- Lavare mescolando per 1 minuto a velocità media.
- Compila colorazione piatto "C" con pre-riscaldato tampone di lavaggio 2. Trasferire i vetrini e lavare per 1 minuto.
- Molto lentamente rimuovere il rack diapositiva colorazione piatto "C" per ridurre al minimo le goccioline lasciato sul vetrino.
- Asciugare il vetrino facendo girare per 2 minuti. Se una centrifuga compatibile non è disponibile, centrifugare il vetrino in un tubo da 50 ml conica o colpo di gas argon su di esso.
- Collocare i vetrini in 50 ml di tubo conico e riempirlo con gas argon. Scansione immediatamente per evitare la perdita di segnale. Si consiglia di utilizzare lo scanner GenePix 4000B da Molecular Devices.
5. Rappresentante dei risultati:
Buona qualità dei campioni di RNA totale dovrebbe produrre solo due picchi principali quando viene eseguito in un Bioanalyzer per il controllo qualità, corrispondenti ai 2 principali specie di RNA ribosomiale. Alcuni degradazione dell'RNA mostrerà come uno striscio prima del primo picco di RNA ribosomiale. Pesantemente degradato, l'RNA di bassa qualità mostrerà un picco largo o una serie di picchi a tempi di ritenzione bassa, mentre i 2 picchi di RNA ribosomiale sarà di intensità molto bassa o non identificabili a tutti. Per gli esempi dell'output 2100 Bioanalyzer, vedi figura 1.
L'Esperto 2100 software calcola un numero di integrità di RNA, o RIN, come misura quantitativa della qualità RNA. Campioni di alta qualità, con valori RIN superiore a 9, sono ovviamente migliori per le applicazioni di microarray. Tuttavia, abbiamo usato i campioni con valori di RIN più basso 5,2 nella generazione dei dati di microarray di qualità ragionevole.
Array di buona qualità dovrebbe produrre segnale alto a valori relativamente bassi PMT. Nel nostro esperimento, la maggior parte delle trascrizioni sono tenuti ad essere presenti a livelli simili nel campione sperimentale e di riferimento; massiccia, i cambiamenti nell'espressione genica diffusa probabilmente prive di qualsiasi significato biologico. Pertanto, la maggior parte del vettore dovrebbe apparire giallo piuttosto che verde o rosso. Segnali di buona qualità deve essere in una gamma dinamica in modo che il segnale istogrammi si sovrappongono completamente. Ad esempio, vedere figura 2.
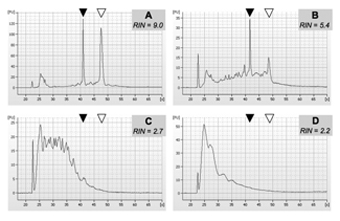
Figura 1. Esempio di quattro campioni di RNA isolato da tessuti umani tumore al cervello. RNA è stato isolato utilizzando il protocollo presentato in 3.1.1. Qualità del campione è stata valutata utilizzando il 2100 Bioanalyzer su un chip 6000 RNA come descritto al punto 3.1.3. I campioni sono rappresentativi di 4 distinte qualità RNA: A, ottime RNA (RIN> 9); B, parzialmente degradate RNA (RIN 5-6, nota il picco significativamente più basso per rRNA 28S), C, altamente degradata RNA (RIN < 3); D, quasi completamente degradato l'RNA (RIN = 2). Abbiamo utilizzato con successo i campioni con caratteristiche simili o migliori di quelle B (RIN> 5.2) per le applicazioni di microarray a valle. Punte di freccia solide e aprire indicano la posizione del 18S e 28S rRNA specie, rispettivamente.
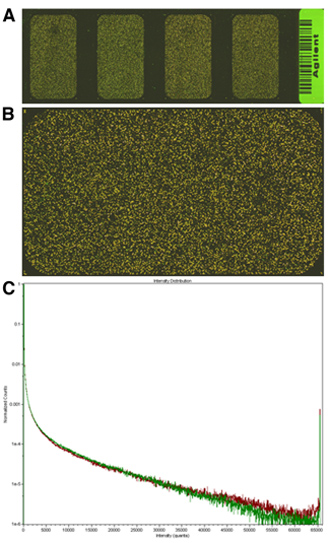
Figura 2. Esempi di immagini array e grafici a dispersione. A, anteprima scivolo, con indicazione della quattro array nel formato 4X44K Agilent; B, la scansione ad alta risoluzione di una delle zone array, nota che nessuno dei due segnali (rosso o verde) è predominante, C, grafico a dispersione dell'immagine in B, si noti che i segnali si sovrappongono completamente e non superiore a 1x10 -6 caratteristiche sono ad intensità saturando.
Discussione
Profili di espressione con DNA microarray fornisce un metodo semplice per identificare i geni differenzialmente espressi tra due campioni biologici. Esperimenti di successo profili di espressione richiedono campioni di RNA di alta qualità, etichettatura robusto e ibridazione. Nella nostra esperienza, gli array commerciale e kit di etichettatura di Agilent fornire dati di alta qualità ad un costo ragionevole. Mentre Agilent offre anche la loro ibridazione propri e dei macchinari di scansione, siamo favorevoli al sistema di ibridazione da Roche-NimbleGen e la GenePix 4000B scanner microarray da Molecular Devices. Si noti che la Roche-NimbleGen unità di ibridazione è stato precedentemente noto come il sistema di ibridazione MAUI. Dopo la recente acquisizione aziendale da Roche, i mixer A4 sono stati interrotti. A partire dal momento in cui scriviamo, possono ancora essere trovati attraverso altri venditori come diagnostica KREATECH ( www.kreatech.com , questo sito offre anche un comodo strumento di compatibilità array), ma a lungo termine la disponibilità di magazzino è incerta. Sistemi di ibridazione altri sono disponibili (ad esempio, da Agilent), ma se mixer compatibili è disponibile per l'array in uso, si consiglia il sistema di Roche-NimbleGen per la sua qualità e riproducibilità.
Divulgazioni
Riconoscimenti
Questo lavoro è stato sostenuto dalle concessioni alla disfunzione erettile dal S. James McDonnell Foundation Award Program 21 ° secolo, e Innovator Award nuovo un direttore NIH. GAB è stato sostenuto in parte da una borsa di studio post-dottorato presso l'Istituto di Medicina Rigenerativa della California.
Materiali
| Name | Company | Catalog Number | Comments |
| 2100 Bioanalyzer | Agilent Technologies | G2943CA | Includes chip priming station, vortexer, software and laptop computer |
| 2100 Bioanalyzer electrophoresis set | Agilent Technologies | G2947CA | |
| RNA 6000 Nano kit | Agilent Technologies | 5067-1511 | includes size ladder, marker, gel matrix, dye, electrode cleaners, spin filters and nanochips |
| RNase Zap | Applied Biosystems | AM9780M | |
| Quick Amp labeling kit, two-color | Agilent Technologies | 5190-0444 | |
| RNeasy mini kit | Qiagen | 74104 | |
| Gene Expression Hybridization Kit | Agilent Technologies | 5188-542 | Includes Blocking agent, Fragmentation buffer and Hybridization buffer |
| Gene Expression Wash Buffer Kit | Agilent Technologies | 5188-5327 | Includes Wash Buffers 1 and 2 and Triton X-102 |
| Hybridization Station | Roche Group | 05223652001 | 4-slide model |
| Hybridization System Accesory Kit | Roche Group | 05327695001 | Contains verification assembly for testing mixing, replacement O-rings, disassembly tool, forceps and brayer |
| A4 mixer hybridization chambers | |||
| Whole Human Genome (4x44) Oligo Microarray | Agilent Technologies | G4112F | |
| Positive displacement pipette | Gilson, Inc. | F148504 | |
| Capillary pistons for positive displacement pipette | Gilson, Inc. | F148415 | |
| Microarray dryer | Nimblegen | 05223636001 | |
| Microarray fluorescent scanner, Axon GenePix 4000B | Molecular Devices | GENEPIX4000B | |
| GenePix Pro 6.0 software | Molecular Devices |
Riferimenti
- Diaz, E. One Decade Later: What has Gene Expression Profiling Told us About Neuronal Cell Types, Brain Function and Disease. Curr Genomics. 10, 318-318 (2009).
- Barisone, G. A. Expression profiling in Neuroscience, edited by Ioannis Karamanos. , SPRINGER SCIENCE+BUSINESS MEDIA, LLC. New York. (2010).
- Gelder, R. N. V. an Amplified RNA synthesized from limited quantities of heterogeneous cDNA. Proc Natl Acad Sci U S A. 87, 1663-1663 (1990).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon