Method Article
Cellules souches: le contrôle qualité des échantillons, hybridation Array et balayage
Dans cet article
Résumé
Nous démontrons l'utilisation de puces à ADN pour le profilage de l'expression du système nerveux. Nous décrivons le contrôle qualité de l'ARN, l'étiquetage de l'échantillon, et l'hybridation de tableau et de numérisation.
Résumé
Profilage de l'expression Microarray du système nerveux fournit une approche puissante pour identifier les activités de gènes dans différents stades de développement, les différents états physiologiques ou pathologiques, la réponse au traitement, et, en général, toute condition qui est testée expérimentalement, 1. Profilage de l'expression des tissus neuronaux nécessite l'isolement de l'ARN de haute qualité, l'amplification de l'ARN isolé et l'hybridation de puces à ADN. Dans cet article, nous décrivons les protocoles des expériences biopuces reproductibles à partir de tissus tumeur au cerveau 2. Nous allons commencer par réaliser une analyse de contrôle de qualité des échantillons d'ARN isolés avec Agilent 2100 Bioanalyzer «lab-on-a-chip" de la technologie. Haute qualité des échantillons d'ARN sont essentiels pour la réussite de toute expérience microarray, et le bioanalyseur 2100 fournit un moyen rapide, mesure quantitative de la qualité de l'échantillon. Les échantillons d'ARN sont ensuite amplifiés et marqués en effectuant la transcription inverse pour obtenir l'ADNc, suivi par transcription in vitro en présence de nucléotides marqués à produire étiquetés ARNc. En utilisant un kit de marquage bicolore, nous marquerons notre échantillon expérimental avec Cy3 et un échantillon de référence avec Cy5. Les deux échantillons seront ensuite combinées et hybridé à des tableaux d'Agilent K 4x44. Double-couleurs des tableaux offrent l'avantage d'une comparaison directe entre deux échantillons d'ARN, augmentant ainsi la précision des mesures, en particulier pour de petits changements dans les niveaux d'expression, parce que les deux échantillons d'ARN sont hybridés de manière compétitive aux puces une seule. Les tableaux seront analysés lors des deux longueurs d'onde correspondant, et le ratio de Cy3 au Cy5 signal pour chaque fonctionnalité sera utilisée comme une mesure directe de l'abondance relative de l'ARNm correspondant. Cette analyse identifie des gènes qui sont différentiellement exprimés en réponse à des conditions expérimentales testées.
Protocole
1. Analyse de contrôle de la qualité de l'échantillon d'ARN avec 2100 bioanalyseur
Avant de commencer,
- Dénaturer l'échelle et vos échantillons à 70 ° C pendant 10 min. Réfrigérer immédiatement sur la glace.
- Nettoyer les électrodes avec des instruments RNaseZAP pendant 1 min, suivie par l'eau sans RNase pendant 10 sec. Autoriser les électrodes sécher à l'air.
- Préparer la matrice de gel par pipetage 550 pl de la matrice de gel dans un filtre rotatif fourni avec le kit. Centrifuger à 1500 RCF pendant 10 min à température ambiante. Aliquoter filtré sur gel en 65 aliquotes et stocker à 4 ° C pour un maximum de 1 mois.
- Afin de préparer la station d'amorçage à puce,
- Insérer une seringue de 1 ml dans le clip et dans l'adaptateur luer lock.
- Ajustez la plaque de base à la position C en desserrant la vis sous la base, en soulevant la plaque et en resserrant la vis.
- Ajustez le clip de la seringue pour la première position.
- Equilibrer le concentré de teinture à la température ambiante. Vortex pendant 10 sec. Ajouter 1 l de teinture à une aliquote de 65 pl de gel filtré. Vortex bien et centrifuger à 13 000 RCF pendant 10 min à température ambiante. Utiliser moins d'un jour.
Lorsque vous êtes prêt à exécuter vos échantillons, - Position d'une puce sur la station d'amorçage. Dans ce protocole, nous utilisons des puces ARN 6000 nano. Pour charger le gel,
- Pipette 9 ul du mélange gel-dye dans le bien marqués avec un blanc "G" sur un fond noir. Assurez-vous que votre astuce est positionné au bas du puits.
- Positionner le piston de la seringue à 1 ml. Fermer la station d'amorçage. Assurez-vous qu'il cliquez-écluses.
- Appuyez sur le piston vers le bas, lentement mais sûrement jusqu'à ce qu'elle soit tenue par le clip.
- Attendre 30 sec. Libérez le clip.
- Laissez le piston monte en soi. Après qu'il cesse de bouger, d'attendre quelques secondes et tirez sur le piston à la position de 1 ml. Ouvrez la station d'amorçage.
- Pipette 9 ul du mélange gel-dye dans chacun des 2 puits marqué "G".
- Pipeter 5 pl du marqueur dans chacun des 12 puits d'échantillon et de l'échelle bien.
- Pipeter 1 pl de l'échelle préparés échelle bien.
- Pipeter 1 ul de chaque échantillon dans le puits d'échantillon.
- Pipeter 1 pl de la marque dans chaque échantillon inutilisée bien.
- Vortex de la puce pendant 1 min à 2400 rpm.
- Pour exécuter la puce,
- Démarrez le logiciel 2100 Expert.
- Position de la puce et fermer le couvercle. Si l'instrument est en ligne, une icône apparaîtra si le couvercle est ouvert ou fermé et quel type de puce est insérée. Assurez-vous que le port correct est sélectionné.
- Dosage Exécutez sein d'embarquement 5 mn les échantillons pour prévenir évaporation.
2. L'amplification et l'étiquetage
Pour se préparer à l'analyse des microréseaux, des échantillons d'ARN sont amplifiés et marqués, habituellement dans une réaction basée sur l'ARN polymérase T7 3. ADNc double brin est généré par transcription inverse. L'ADNc est utilisé dans une réaction de transcription in vitro pour générer ce qui est connu comme ARNc. Cette réaction est effectuée en présence de ribonucléotides étiquetés, produisant des quantités de microgramme d'ARN marqués d'hybridation tableau. Le choix de l'amplification / étiquetage des méthodes dépend de la plateforme microarray postérieurement à être utilisé. Dans cette section, nous décrivons la génération d'ARN marqué par fluorescence à l'aide d'Agilent kit rapide étiquetage Amp.
- Pipeter 50 ng à 5 ug d'ARN dans un tube de 1,5 ml. Le volume ne doit pas dépasser 8,3 pi. Si nécessaire, concentrer votre échantillon d'ARN par centrifugation sous vide ou par précipitation avec de l'alcool.
- Ajouter 1,2 ul d'apprêt T7.
- Porter à un volume final de 11,5 ul avec l'eau sans RNase.
- Incuber à 65 ° C pendant 10 min pour dénaturer l'ARN. Nous vous recommandons d'utiliser un thermocycleur avec un couvercle chaud pour éviter la condensation dans le haut du tube.
- Pendant les 10 minutes d'incubation, Buffer 5X First Strand chaude à 80 ° C pendant 5 min. Vortex et spin bas. Gardez à température ambiante jusqu'au moment de servir.
- Après l'incubation de 10 min, refroidir rapidement les échantillons d'ARN dénaturé sur la glace.
- Préparer le mélange maître d'ADNc. Par réaction, ajoutez la ligne suivante:
- 4 pl de tampon 5X First Strand
- 2 pl 0,1 M TNT
- 1 pl 10 mM de dNTPs
- 1 pl MMLV-RT enzymatiques
- 0,5 sur ul RNase.
- Mélanger les composants en inversant le tube 4 fois. Ne pas vortex. Isoler pour recueillir le contenu au fond du tube.
- Ajouter 8,5 ul de mélange maître par échantillon. Mélanger par pipetage de haut en bas.
- Incuber pendant 2 heures à 40 ° C, suivi de 10 min à 65 ° C. Nous vous recommandons d'utiliser un thermocycleur avec un couvercle chauffé.
- Transférer les tubes à la glace pendant 5 min.
- Préparer le mélange maître de transcription. Par réaction, ajoutez la ligne suivante:
- 15,3 ul eau sans nucléase
- 20 plTampon de transcription 4X
- 6 pl 0,1 M TNT
- 8 PNT ul
- 6,4 ul de PEG 50% (PEG doit être préchauffée à 40 ° C et un vortex pour assurer la remise en suspension)
- 0,5 ul RNase Out
- 0,6 ul pyrophosphatase inorganique
- 0,8 ul T7 RNA polymérase
- 2,4 ul-Cy3 ou Cy5 CTP
- Centrifuger brièvement les échantillons pour recueillir le contenu au fond du tube.
- Ajouter 60 ul de Mix Master transcription par échantillon.
- Incuber à 40 ° C pendant 2 heures.
- Ajouter 20 ul d'eau sans RNase.
- Purifier l'ARNc marqué pour enlever nucléotides non incorporés. Nous recommandons l'utilisation de Qiagen RNeasy mini colonnes.
- Quantifier les ARNc marqué. Nous recommandons l'utilisation d'un spectrophotomètre NanoDrop2000 en mode de mesure biopuces. Assurez-vous que vous sélectionnez l'ARN-40 comme le type d'échantillon.
- Concentration de l'échantillon record, le rendement (calculé comme la concentration, multipliée par le volume) et l'activité spécifique (calculée comme 1000, multiplié par la ration de concentration du colorant sur la concentration ARNc). Pour l'hybridation puces, il est nécessaire d'atteindre un rendement d'au moins 825 ng et une activité spécifique d'au moins 8 pmol / mg.
3. L'hybridation Microarray
- Tournez sur la station d'hybridation et réglé à 65 ° C au moins 2 heures avant l'hybridation commence.
- Pour préparer l'échantillon hybridation, mélanger les suivants dans un tube de 1,5 ml:
- 825 ng Cy3 ARNc (ce qui est généralement votre "traité" de l'échantillon)
- 825 ng Cy5-ARNc (ce qui est généralement votre «référence» de l'échantillon)
- 11 ul d'agent de blocage 10X.
- Ajouter l'eau sans RNase à un volume final de 52,8 ul
- Ajouter 2,2 ul de tampon de fragmentation.
- Vortex à basse vitesse pour mélanger.
- Incuber à 60 ° C pendant exactement 30 minutes. Cette étape est la fragmentation des ARNc, qui génère des fragments marqués d'hybridation tableau. Il est très important pour incuber exactement comme décrit. Nous recommandons l'utilisation d'un thermocycleur avec un couvercle chauffé. Il est très important pour incuber exactement comme décrit pour générer des sondes de la durée moyenne correcte. Une fragmentation excessive ou insuffisante se traduira par de faux négatifs ou faux positifs.
- Ajouter 55 ul de tampon d'hybridation 2X pour arrêter la fragmentation. Mélanger à la pipette, en prenant soin de ne pas introduire de bulles.
- Centrifuger 1 min à vitesse maximale pour recueillir le contenu au fond du tube. Si des bulles sont observées, centrifugeuse plus de temps à les supprimer.
- Placer le tube sur la glace, protégé de la lumière. Chargez l'échantillon sur le tableau dès que possible.
- Pour charger le tableau, d'abord préparer la chambre d'hybridation. Ce protocole décrit l'utilisation de tableaux 4x44K Agilent avec le système d'hybridation et de la Roche-Nimblegen chambres mélangeur A4.
- Placez une diapositive dans la puce le code à barres de montage / démontage d'outils, d'abord.
- Ouvrir un mélangeur A4 et exposer l'adhésif. Tenir le tableau et l'assemblage / désassemblage outil pour empêcher tout mouvement, placer le mélangeur A4 sur la diapositive, en commençant à l'extrémité. Assurez-vous qu'il est correctement aligné sur l'outil. Bâton mélangeur tout au long de la glissière de tableau.
- Tirez sur la fin de la table de mixage pour enlever la lame / montage mélangeur.
- Utilisation de l'outil braiment, la presse tout au long du joint adhésif pour s'assurer qu'il est solidement collé à coulisseau. Petites bulles d'air piégées entre l'adhésif et la lame peut être vu sur un fond sombre. Prenez le temps supplémentaire pour éliminer ces bulles avec l'outil braiement. Votre tableau est maintenant prêt à être chargé.
- Utiliser une pipette à déplacement positif à une charge de 100 pl de l'échantillon d'hybridation. Poussez le bout serré dans le trou du port, puis passer lentement et en douceur pour que l'air n'est pas emprisonné dans la zone de tableau. Ne jamais essayer d'aspirer l'échantillon sauvegarder. Une fois l'ensemble de l'échantillon a été distribuée, garder la pointe sur le hublot, sans relâcher le piston. Lorsque l'échantillon couvre la totalité du tableau et le liquide commence à sortir de l'évent, enlever rapidement la pipette. Seul le piston communiqué une fois que la pointe est loin de la glissière.
- Tamponnez délicatement l'excès de liquide dans les deux ports. Assurez vous de ne pas tirer l'échantillon hors de la chambre elle-même.
- Recouvrez les trous du port avec des autocollants en utilisant une pince à épiler.
- Mettez la lame sur la station d'hybridation, et vérifiez que les trous vessie sont correctement positionnés sur les joints toriques. Cela permettra d'assurer un bon mélange lors de l'hybridation.
- Fermez le couvercle coulissant et le couvercle station. Réglez la station de mélange en mode B.
- Hybrider le tableau à 65 ° C pendant 17 heures.
- Préparer le tampon de lavage 2 en ajoutant de Triton X-102 à une concentration finale de 0,005%. Gardez à 37 ° C pendant la nuit.
4. Laver Microarray
- Ajouter Triton X-102 à Wash Buffer 1 à une concentration finale de 0,005%. Conserver à températur ambiantee.
- Lorsque l'hybridation est terminé, remplir un verre plat "A" avec le tampon de lavage 1. Le plat doit être assez grand pour tenir l'outil de montage / démontage et de permettre des manœuvres aussi bien. Tous les lavages doit être fait dans de la verrerie. Eviter les plats en plastique, car ils ont tendance à lessiver les composés résultant de fond élevé dans le tableau.
- Mettez un rack de diapositives et un barreau dans un plat à coloration «B». Remplir avec le tampon de lavage 1, faire en sorte qu'il recouvre la baie. Placer sur une plaque d'agitation à température ambiante.
- Mettez un barreau sur un plat de coloration vide "C" et le placer sur une plaque d'agitation.
- Retirez le tableau et le mettre dans l'outil de montage / démontage. Plonger l'ensemble dans le plat "A".
- Tout en maintenant l'outil de montage / démontage et la diapositive à partir des côtés opposés d'une seule main, soigneusement décoller le mélangeur A4. Assurez vous de ne pas rayer la zone de tableau.
- Vite placer la lame dans le rack de coloration plat "B". A partir de maintenant, l'exposition à l'air doit être minimisée car le colorant Cy5 est sensible à l'ozone. Ne lavez pas plus de 8 tableaux à la fois.
- Laver pendant 1 minute en remuant à vitesse moyenne.
- Remplissez coloration plat "C" avec le tampon de lavage préchauffé 2. Placer les lames et laver pendant 1 minute.
- Très lentement, retirez le porte-diapositive de coloration plat "C" pour minimiser les gouttelettes laissées sur la diapositive.
- Sécher le glisser en faisant tourner pendant 2 minutes. Si une centrifugeuse compatibles n'est pas disponible, centrifuger les glisser dans un tube conique de 50 ml ou un coup de gaz argon sur elle.
- Placer les lames dans un tube conique de 50 ml et le remplir avec du gaz argon. Balayage immédiatement pour éviter la perte de signal. Nous recommandons d'utiliser le scanner GenePix 4000B de Molecular Devices.
5. Les résultats représentatifs:
Bonne qualité totale échantillons d'ARN devrait produire seulement deux pics majeurs lorsqu'il est exécuté dans un bioanalyseur de contrôle de qualité, correspondant aux deux principales espèces d'ARN ribosomal. Certains dégradation de l'ARN se montrer comme un frottis avant le pic premier ARN ribosomal. Fortement dégradées, l'ARN de faible qualité montrera un large pic ou d'une série de pics au temps de rétention faible, tandis que les 2 pics ARN ribosomique seront de très faible intensité ou pas identifiables à tout. Pour des exemples de la sortie 2100 bioanalyseur, voir la figure 1.
L'Expert 2100 logiciel calcule un numéro intégrité de l'ARN, ou RIN, comme une mesure quantitative de la qualité de l'ARN. Échantillons de haute qualité, avec des valeurs RIN supérieur à 9, sont évidemment mieux pour les applications de biopuces. Cependant, nous avons utilisé des échantillons avec des valeurs RIN aussi bas que 5,2 pour générer données de biopuces de qualité raisonnable.
Tableaux de bonne qualité devrait produire un signal à des valeurs élevées PMT relativement faible. Dans notre expérience, la plupart des transcriptions devraient être présents à des niveaux similaires dans l'échantillon expérimental et de référence; massive, de vastes changements dans l'expression génique sera probablement n'ont aucune signification biologique. Par conséquent, la plupart du tableau devrait ressembler plutôt jaune que verte ou rouge. Signaux de bonne qualité devrait également être dans une plage dynamique tels que les histogrammes signal entièrement chevauchent. Pour des exemples, voir la figure 2.
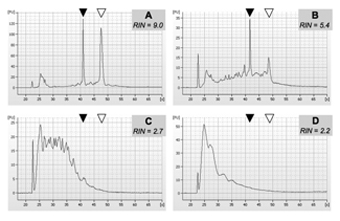
Figure 1. Exemple de quatre échantillons d'ARN isolés à partir de tissus de tumeurs cérébrales humaines. L'ARN a été isolé en utilisant le protocole présenté en 3.1.1. La qualité des échantillons a été évaluée en utilisant le bioanalyseur 2100 sur une puce 6000 ARN tel que décrit au paragraphe 3.1.3. Les échantillons sont représentatifs de quatre qualités ARN distincts: A, de très bonne qualité d'ARN (RIN> 9); B, partiellement dégradées ARN (RIN 5-6, noter le pic de nettement plus faible pour l'ARNr 28S); C, fortement dégradées ARN (RIN < 3); D, presque complètement dégradé ARN (RIN = 2). Nous avons utilisé avec succès des échantillons avec des qualités similaires ou supérieures à B (RIN> 5,2) pour les applications de biopuces en aval. Des pointes de flèches solides et ouverts indiquent la position de la 18S et 28S ARNr des espèces, respectivement.
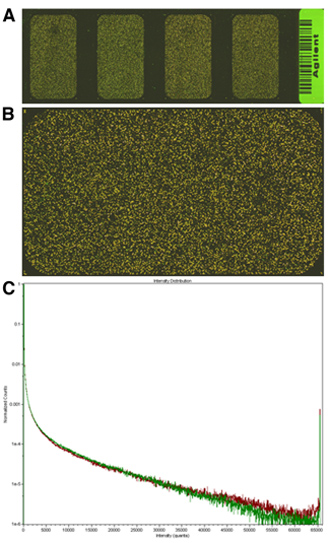
Figure 2. Exemples d'images de tableau et des diagrammes de dispersion. Un aperçu, faites glisser l'ensemble, montrant les quatre tableaux au format 4X44K Agilent; B, numérisation haute résolution de l'une des zones de tableau, noter que ni des signaux (vert ou rouge) est prédominante; C, diagramme de dispersion de l'image dans B, note que les signaux totalement le chevauchement et pas plus de 1x10 -6 fonctionnalités sont à l'intensité de saturation.
Discussion
Profilage de l'expression avec des puces à ADN offre une approche simple pour identifier les gènes différentiellement exprimés entre deux échantillons biologiques. Le succès des expériences de profilage d'expression de haute qualité nécessitent des échantillons d'ARN, l'étiquetage robuste et d'hybridation. Dans notre expérience, les tableaux et les kits commerciaux d'Agilent étiquetage fournissent des données de haute qualité à un coût raisonnable. Bien Agilent propose également de leurs propres machines et l'hybridation de numérisation, nous privilégions le système d'hybridation de Roche-Nimblegen et le GenePix 4000B biopuces scanner de Molecular Devices. Notez que l'unité d'hybridation Roche-Nimblegen était anciennement connu sous le système d'hybridation Maui. Après la récente acquisition d'entreprise par Roche, les mélangeurs A4 ont été abandonnées. Dès l'instant où j'écris ces lignes, ils peuvent encore être trouvés par d'autres vendeurs tels que Diagnostics Kreatech ( www.kreatech.com ; ce site propose également un outil pratique éventail de compatibilité), mais la disponibilité du stock à long terme est incertaine. Autres systèmes d'hybridation sont disponibles (par exemple, d'Agilent), mais si mélangeurs compatible peut être trouvé pour le tableau utilisé, nous recommandons le système de la Roche-NimbleGen pour sa qualité et de reproductibilité.
Déclarations de divulgation
Remerciements
Ce travail a été soutenu par des subventions à l'ED de la James S. McDonnell 21 Programme er Prix siècle, et le Prix du directeur des NIH Innovateur Nouvelle. AGE a été soutenu en partie par une bourse postdoctorale du California Institute of Regenerative Medicine.
matériels
| Name | Company | Catalog Number | Comments |
| 2100 Bioanalyzer | Agilent Technologies | G2943CA | Includes chip priming station, vortexer, software and laptop computer |
| 2100 Bioanalyzer electrophoresis set | Agilent Technologies | G2947CA | |
| RNA 6000 Nano kit | Agilent Technologies | 5067-1511 | includes size ladder, marker, gel matrix, dye, electrode cleaners, spin filters and nanochips |
| RNase Zap | Applied Biosystems | AM9780M | |
| Quick Amp labeling kit, two-color | Agilent Technologies | 5190-0444 | |
| RNeasy mini kit | Qiagen | 74104 | |
| Gene Expression Hybridization Kit | Agilent Technologies | 5188-542 | Includes Blocking agent, Fragmentation buffer and Hybridization buffer |
| Gene Expression Wash Buffer Kit | Agilent Technologies | 5188-5327 | Includes Wash Buffers 1 and 2 and Triton X-102 |
| Hybridization Station | Roche Group | 05223652001 | 4-slide model |
| Hybridization System Accesory Kit | Roche Group | 05327695001 | Contains verification assembly for testing mixing, replacement O-rings, disassembly tool, forceps and brayer |
| A4 mixer hybridization chambers | |||
| Whole Human Genome (4x44) Oligo Microarray | Agilent Technologies | G4112F | |
| Positive displacement pipette | Gilson, Inc. | F148504 | |
| Capillary pistons for positive displacement pipette | Gilson, Inc. | F148415 | |
| Microarray dryer | Nimblegen | 05223636001 | |
| Microarray fluorescent scanner, Axon GenePix 4000B | Molecular Devices | GENEPIX4000B | |
| GenePix Pro 6.0 software | Molecular Devices |
Références
- Diaz, E. One Decade Later: What has Gene Expression Profiling Told us About Neuronal Cell Types, Brain Function and Disease. Curr Genomics. 10, 318-318 (2009).
- Barisone, G. A. . Expression profiling in Neuroscience, edited by Ioannis Karamanos. , (2010).
- Gelder, R. N. V. a. n. Amplified RNA synthesized from limited quantities of heterogeneous cDNA. Proc Natl Acad Sci U S A. 87, 1663-1663 (1990).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon