Method Article
DNA Microarrays: Sample Quality Control, Array Hybridisierung und Scannen
In diesem Artikel
Zusammenfassung
Wir zeigen die Verwendung von DNA-Microarrays für die Expression Profiling des Nervensystems. Wir beschreiben RNA Qualitätskontrolle, Probe Kennzeichnung und Array-Hybridisierung und Scannen.
Zusammenfassung
Microarray Expression Profiling des Nervensystems bietet eine leistungsstarke Ansatz zur Identifizierung von Gen-Aktivitäten in verschiedenen Stadien der Entwicklung, verschiedene physiologische oder pathologische Zustände, Ansprechen auf die Therapie und in der Regel keine Bedingung, dass experimentell ist 1 getestet. Expression Profiling von Nervengewebe erfordert die Isolierung von hochwertigen RNA, Amplifikation der isolierten RNA und Hybridisierung an DNA-Mikroarrays. In diesem Artikel beschreiben wir Protokolle für reproduzierbare Microarray-Experimente aus dem Gehirn Tumorgewebe 2. Wir werden von der Durchführung einer Qualitätskontrolle Analyse der isolierten RNA-Proben mit der Agilent 2100 Bioanalyzer "lab-on-a-chip"-Technologie zu beginnen. Hochwertige RNA-Proben sind entscheidend für den Erfolg eines jeden Microarray-Experiment, und die 2100 Bioanalyzer bietet eine schnelle, quantitative Messung der Qualität der Proben. RNA-Proben werden dann verstärkt und gekennzeichnet durch die Durchführung der reversen Transkription zu erhalten cDNA, gefolgt von in vitro-Transkription in Gegenwart von markierten Nukleotiden an markierte cRNA produzieren. Durch die Verwendung einer Zwei-Farben-Labeling Kit, werden wir unsere experimentellen Probe mit Cy3-und eine Referenzprobe mit Cy5-Label. Beide Proben werden dann kombiniert und gekreuzt werden, um 4x44 K Agilent Arrays. Dual-Farb-Arrays bieten den Vorteil, dass ein direkter Vergleich zwischen zwei RNA-Proben, wodurch die Genauigkeit der Messungen, insbesondere für kleine Änderungen in Expression, weil die beiden RNA-Proben kompetitiv an einem einzigen Microarray hybridisiert sind. Die Arrays werden an den beiden entsprechenden Wellenlängen gescannt werden, und das Verhältnis von Cy3 zu Cy5-Signal für jede Funktion wird als eine direkte Messung der relativen Häufigkeit der entsprechenden mRNA verwendet werden. Diese Analyse identifiziert Gene, die in Reaktion auf die experimentellen Bedingungen getestet werden ausgedrückt.
Protokoll
1. Qualitätskontrolle Analyse der RNA-Probe mit 2100 Bioanalyzer
Bevor Sie beginnen,
- Denaturieren die Leiter und Ihre Proben bei 70 ° C für 10 min. Gefühlt sofort auf Eis.
- Reinigen Sie das Instrument Elektroden mit RNaseZAP für 1 min, durch RNase-freies Wasser für 10 sec folgte. Lassen Sie die Elektroden an der Luft trocknen.
- Bereiten Sie die Gel-Matrix durch Pipettieren 550 ul der Gel-Matrix in einen Spin Filter mit dem Kit bei. Zentrifuge bei 1.500 RCF für 10 min bei Raumtemperatur. Aliquot der gefilterten Gel in 65 ul Aliquots und lagern bei 4 ° C für bis zu 1 Monat.
- Zur Vorbereitung der Chip Priming Station,
- Legen Sie eine 1-ml-Spritze durch den Clip und in den Luer-Lock-Adapter.
- Stellen Sie die Grundplatte an C durch Lösen der Schraube unter der Basis, die Aufhebung der Platte und Nachziehen der Schrauben Position.
- Passen Sie die Spritze Clip in die obere Position.
- Äquilibrieren der Farbstoffkonzentrat auf Raumtemperatur. Vortex für 10 Sekunden. Add 1 ul Farbstoff ein 65 ul-Aliquot der gefilterten Gel. Vortex gut und Zentrifuge bei 13.000 RCF für 10 min bei Raumtemperatur. Verwenden Sie innerhalb eines Tages.
Wenn Sie bereit sind Ihre Proben ausgeführt werden, - Position ein Chip auf der Grundierung entfernt. In diesem Protokoll sind wir mit RNA 6000 Nano-Chips. Um das Gel laden,
- Pipette 9 ul des Gel-Farbstoff-Mix in den Brunnen mit einem weißen "G" vor einem schwarzen Hintergrund markiert. Achten Sie darauf, Ihren Tipp ganz am Ende der gut positioniert ist.
- Position der Spritze mit 1 ml. Schließen Sie die Grundierung Station. Stellen Sie sicher, Klick-Schlösser.
- Drücken Sie den Kolben langsam, aber stetig, bis sie durch den Clip gehalten wird.
- Warten Sie 30 Sekunden. Lassen Sie die Clips.
- Lassen Sie den Kolben nach oben von allein. Nachdem er aufhört sich zu bewegen, für ein paar Sekunden warten, und ziehen Sie den Kolben zurück, um die 1 ml Position. Öffnen Sie die Grundierung Station.
- Pipette 9 ul des Gel-Farbstoff-Mix in jedem der 2 Brunnen mit der Aufschrift "G".
- Je 5 ul des Markers in jeder der 12 Proben Brunnen und in die Leiter gut.
- Pipette 1 ul der vorbereiteten Leiter in Ladder gut.
- Pipette 1 ul jeder Probe in der Probe Brunnen.
- Pipette 1 ul der Marker in jeder ungenutzten Probe gut.
- Vortex den Chip für 1 min bei 2.400 Umdrehungen pro Minute.
- Um den Chip laufen,
- Anfang der 2100 Expert Software.
- Positionieren Sie den Chip und den Deckel schließen. Wenn das Gerät online ist, wird ein Symbol zeigen, ob der Deckel offen oder geschlossen ist und welche Art von Chip eingesetzt wird. Stellen Sie sicher, den richtigen Anschluss gewählt ist.
- Führen Sie den Test innerhalb von 5 min von dem Laden der Proben, um Verdunstung zu verhindern.
2. Amplifikation und Markierung
Zur Vorbereitung für die Microarray-Analyse werden RNA-Proben amplifiziert und beschriftet, in der Regel in einer Reaktion auf T7-RNA-Polymerase 3. Doppelsträngigen cDNA durch reverse Transkription erzeugt. cDNA wird in einem in vitro Transkriptions-Reaktion eingesetzt zu generieren, was als cRNA bekannt. Diese Reaktion wird in Gegenwart von markiertem Ribonukleotiden durchgeführt, Herstellung Mikrogramm-Mengen von markierten RNA für Array-Hybridisierung. Die Wahl der Verstärkung / Kennzeichnung Methoden hängt von den nachfolgenden Microarray-Plattform verwendet werden. In diesem Abschnitt beschreiben wir die Erzeugung von fluoreszenzmarkierten RNA mit Hilfe von Agilent Quick-Amp Labeling Kit.
- Je 50 ng bis 5 ug RNA in ein 1,5 ml-Tube. Das Volumen sollte nicht mehr als 8,3 pl. Wenn nötig, konzentrieren Sie Ihre RNA Probe durch Vakuumzentrifugation oder Fällung mit Alkohol.
- Add 1,2 ul der T7-Primer.
- Bringen Sie auf ein Endvolumen von 11,5 ul mit RNase-freiem Wasser.
- Inkubation bei 65 ° C für 10 min auf die RNA zu denaturieren. Wir empfehlen die Verwendung eines Thermocycler mit einem heißen Deckel, um Kondensation in der Spitze des Rohres zu verhindern.
- Während der 10 min Inkubation, warm 5X First Strand Buffer zu 80 ° C für 5 min. Vortex-und Spin-Down. Halten Sie bei RT bis zur Verwendung.
- Nach der 10 min Inkubation schnell die denaturierte RNA-Proben auf Eis abkühlen.
- Bereiten Sie die cDNA-Mastermix. Per Reaktion, fügen Sie die folgenden:
- 4 ul 5X First Strand Buffer
- 2 ul 0,1 M DTT
- 1 ul 10 mM dNTPs
- 1 ul MMLV-RT-Enzym
- 0,5 ul RNase Out.
- Mix-Komponenten durch Invertieren des Röhrchens 4 mal. Nicht vortexen. Spin down, um den Inhalt auf den Boden des Röhrchens zu sammeln.
- Add 8,5 ul Mastermix pro Probe. Mix durch Pipettieren rauf und runter.
- Inkubieren für 2 h bei 40 ° C, gefolgt von 10 min bei 65 ° C. Wir empfehlen die Verwendung eines Thermocycler mit beheiztem Deckel.
- Übertragen Sie die Rohre auf Eis für 5 min.
- Bereiten Sie die Transkription Master-Mix. Per Reaktion, fügen Sie die folgenden:
- 15,3 ul Nuklease-freies Wasser
- 20 pl4X Transkriptionspuffer
- 6 ul 0,1 M DTT
- 8 ul NTPs
- 6,4 ul 50% PEG (PEG soll auf 40 ° C vorgewärmt werden und verwirbelt, um sicherzustellen, Resuspension)
- 0,5 ul RNase Out
- 0,6 ul anorganischer Pyrophosphatase
- 0,8 ul T7-RNA-Polymerase
- 2,4 ul Cy3-oder Cy5-CTP
- Kurz Spin der Proben, um den Inhalt auf den Boden des Röhrchens zu sammeln.
- Add 60 ul Transkription Master Mix pro Probe.
- Inkubation bei 40 ° C für 2 Stunden.
- Add 20 ul RNase-freiem Wasser.
- Purify der markierten cRNA auf unincorporated Nukleotide zu entfernen. Wir empfehlen die Verwendung von Qiagen RNeasy Mini-Säulen.
- Quantifizierung der markierten cRNA. Wir empfehlen die Verwendung eines NanoDrop2000 Spektralphotometer in Microarray Messmodus. Wählen Sie unbedingt RNA-40 als die Art der Probe.
- Rekord Probenkonzentration, Ausbeute (berechnet als die Konzentration durch das Volumen multipliziert) und spezifische Aktivität (berechnet als 1000 durch das Verhältnis von Farbstoff-Konzentration über cRNA Konzentration multipliziert). Für Microarray-Hybridisierung, ist es notwendig, eine Ausbeute von einem Mindestgewicht von 825 ng und eine spezifische Aktivität von mindestens 8 pmol / ug zu erreichen.
3. Microarray-Hybridisierung
- Schalten Sie den Hybridisierungs-Station und stellen bei 65 ° C mindestens 2 Stunden vor der Hybridisierung beginnt.
- Zur Vorbereitung der Hybridisierung Probe, mischen Sie die folgenden in einem 1,5 ml-Tube:
- 825 ng Cy3-cRNA (dies ist in der Regel Ihre "behandelt" sample)
- 825 ng Cy5-cRNA (dies ist in der Regel Ihre "Referenz"-Probe)
- 11 ul 10x Blocker.
- Add RNase-freiem Wasser auf ein Endvolumen von 52,8 ul
- Add 2,2 ul Fragmentierung Puffer.
- Vortex bei niedriger Geschwindigkeit zu mischen.
- Inkubation bei 60 ° C für genau 30 min. Dies ist die cRNA Fragmentierung Schritt, der markierten Fragmente erzeugt für Array-Hybridisierung. Es ist sehr wichtig, um inkubieren genau wie beschrieben. Wir empfehlen die Verwendung von einem Thermocycler mit beheiztem Deckel. Es ist sehr wichtig, um inkubieren genau so, wie beschrieben, um Sonden der richtige durchschnittliche Länge zu generieren. Übermäßiges oder ungenügendes Fragmentierung wird in falsch-negativen oder falsch positiven Ergebnis.
- Add 55 ul 2X Hybridisierungspuffer, um die Fragmentierung zu stoppen. Mix durch Pipettieren, wobei besondere Sorgfalt nicht zu Blasen führen.
- Säule für 1 min bei maximaler Geschwindigkeit, um den Inhalt auf den Boden des Röhrchens zu sammeln. Wenn Blasen beobachtet werden, mehr Zentrifuge, um sie zu entfernen.
- Das Röhrchen auf Eis, aus dem Licht geschützt. Legen Sie die Probe auf dem Array so schnell wie möglich.
- So laden Sie das Array, bereiten Sie zunächst die Hybridisierung Kammer. Dieses Protokoll beschreibt die Verwendung von Agilent 4x44K Arrays mit dem Roche-NimbleGen Hybridisierung und A4 Mischer Kammern.
- Legen Sie eine Microarray-Folie in der Montage / Demontage-Werkzeug, Barcode-Seite zuerst.
- Öffnen Sie ein A4-Mixer und die Klebefläche freizulegen. Halten Sie die Array-und Montage / Demontage-Werkzeug, um jede Bewegung zu vermeiden, legen Sie das A4-Mixer auf der Folie, beginnend am hinteren Ende. Stellen Sie sicher, es richtig ist, um das Werkzeug ausgerichtet. Kleben Sie den Mixer auf der ganzen Array schieben.
- Ziehen Sie aus dem Ende des Mischers, um die Folie / Mischgruppe entfernen.
- Mit dem Geschrei Werkzeug, drücken Sie die ganze Zeit den Klebstoff Dichtung um sicherzustellen, dass es eng wird theslide geklebt. Kleine Luftblasen zwischen dem Kleber und die Folie eingeschlossene Luft kann vor einem dunklen Hintergrund gesehen werden. Nehmen Sie zusätzliche Zeit, um diese Blasen mit dem Geschrei Werkzeug zu entfernen. Ihr Array ist nun geladen werden.
- Verwenden Sie eine positive Verschiebung Pipette auf 100 ul der Hybridisierung Probe zu laden. Schieben Sie die Spitze fest im Hafen Loch, dann langsam und gleichmäßig so zu verzichten, dass keine Luft in dem Array-Bereich eingeschlossen. Versuchen Sie niemals, das Sample bis zu saugen. Sobald die gesamte Probe verzichtet hat, halten Sie die Spitze auf den Hafen Loch ohne Loslassen des Kolbens. Wenn die Probe das gesamte Array deckt und Flüssigkeit beginnt zu kommen die Entlüftungsöffnung, schnell zu entfernen, die Pipette. Nur lassen Sie die Kolben einmal die Spitze weg von der Folie.
- Vorsichtig abtupfen die überschüssige Flüssigkeit an beiden Ports. Achten Sie darauf, nicht zu ziehen Probe aus der Kammer selber.
- Decken Sie die Bullaugen mit Aufklebern mit einer Pinzette.
- Setzen Sie den Schieber auf der Hybridisierung Station, und überprüfen Sie, ob die Blase Löcher korrekt über die O-Ringe positioniert. Dadurch wird sichergestellt, gute Durchmischung während der Hybridisierung.
- Schließen Sie die Schieber und die Station Deckel. Stellen Sie die Station Mischen-Modus B.
- Hybridisieren das Array bei 65 ° C für 17 Stunden.
- Bereiten Waschpuffer 2 durch Zugabe von Triton X-102 zu einer Endkonzentration von 0,005%. Halten bei 37 ° C über Nacht.
4. Microarray Waschen
- Add Triton X-102, um Buffer 1 bis zu einer Endkonzentration von 0,005% Wash. Halten Sie bei Zimmertemperatur Temperature.
- Wenn Hybridisierung abgeschlossen ist, füllen Sie eine Glasschale "A" mit Waschpuffer 1. Das Gericht sollte groß genug sein, um die Montage / Demontage-Werkzeug zu halten und damit einige Manöver als auch. Alle Wäschen sollte in Gläser getan werden. Vermeiden Sie Plastikgeschirr, da sie auslaugen Verbindungen was zu hohen Hintergrund im Array neigen.
- Legen Sie ein Dia-Rack und einem Rührstab in einer Färbung Gericht "B". Füllen mit Waschpuffer 1, sicherstellen, dass es deckt die Rack. Legen Sie auf einer Rührplatte bei Raumtemperatur.
- Setzen Sie einen Rührstab auf einem leeren Flecken dish "C" und auf eine Rührplatte.
- Entfernen Sie das Array und steckte es in die Montage / Demontage-Werkzeug. Tauchen Sie die ganze Versammlung in der Schüssel "A".
- Halten Sie die Montage / Demontage-Werkzeug und die Folie von gegenüberliegenden Seiten mit einer Hand, sorgfältig schälen die A4-Mixer. Achten Sie darauf, nicht zu verkratzen das Array-Bereich.
- Raschen Platzierung der Folie in das Rack in Färbung Gericht "B". Von nun an sollte sich an der Luft minimiert, da die Cy5-Farbstoff ist empfindlich gegen Ozon. Waschen Sie nicht mehr als 8-Arrays zu einem Zeitpunkt.
- Wash für 1 Minute unter Rühren bei mittlerer Geschwindigkeit.
- Füllen Färbung dish "C" mit vorgewärmten Waschpuffer 2. Die Objektträger und waschen für 1 Minute.
- Sehr langsam entfernen Sie die Folie Rack von Färbung dish "C" zu Tröpfchen hinter auf der Folie links zu minimieren.
- Trocknen Sie die Folie durch Spinnen für 2 Minuten. Wenn ein kompatibles Zentrifuge nicht verfügbar ist, Zentrifuge die Folie in einem 50-ml konische Röhre oder Schlag Argon darüber.
- Die Objektträger in einer 50-ml konischen Rohr und füllen Sie ihn mit dem Edelgas Argon. Scan sofort zu signalisieren, zu vermeiden. Wir empfehlen die Verwendung des GenePix 4000B-Scanner von Molecular Devices.
5. Repräsentative Ergebnisse:
Gute Qualität der Gesamt-RNA-Proben sollten produzieren nur zwei große Spitzen, wenn in einem Bioanalyzer für die Qualitätskontrolle laufen, entsprechend den 2 großen ribosomalen RNA-Spezies. Einige RNA-Abbau wird als ein Abstrich vor der ersten ribosomale RNA Peak zeigen. Erheblich beeinträchtigt, wird geringer Qualität RNA zeigen einen breiten Peak oder eine Reihe von Peaks bei niedrigen Retentionszeiten, während die 2 ribosomale RNA Gipfel sehr geringer Intensität oder nicht identifizierbare überhaupt sein wird. Beispiele für die 2100 Bioanalyzer Ausgang, siehe Abbildung 1.
Die Expert 2100 Software berechnet eine RNA Integrity Number, oder RIN als eine quantitative Messung der RNA-Qualität. Hochwertige Proben mit RIN Werte größer als 9, sind offensichtlich am besten für Mikroarray-Anwendungen. Allerdings haben wir Proben mit RIN Werte so niedrig wie 5.2 in Erzeugung von Microarray-Daten von angemessener Qualität verwendet.
Gute Qualität Arrays sollten High-Signal bei relativ niedrigen PMT Werte erzeugen. In unserem Experiment sind die meisten der Transkripte voraussichtlich auf ähnlichem Niveau in der experimentellen und Referenzprobe vorhanden; massive, breite Veränderungen in der Genexpression wird wahrscheinlich fehlt jede biologische Bedeutung. Daher sollten die meisten der Array suchen, anstatt grün oder rot, gelb. Gute Qualität Signale sollten auch in einem dynamischen Bereich, so dass das Signal Histogramme vollständig überlappen. Für Beispiele siehe Abb. 2.
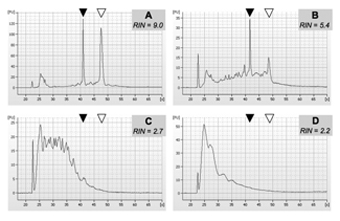
Abbildung 1. Beispiel von vier RNA-Proben aus menschlichem Gehirn Tumorgewebe isoliert. RNA wurde unter Verwendung des Protokolls in 3.1.1 vorgestellt. Beispiel Qualität wurde anhand der 2100 Bioanalyzer auf einem RNA 6000-Chip wie in 3.1.3 beschrieben. Die Proben sind repräsentativ für 4 verschiedene RNA Qualitäten: A, sehr gute RNA-Qualität (RIN> 9); B, partiell abgebauten RNA (RIN 5-6, beachten Sie die deutlich niedrigeren Peak für 28S rRNA); C, stark degradierte RNA (RIN < 3); D, fast vollständig abgebaut RNA (RIN = 2). Wir haben erfolgreich Proben mit ähnlichen Eigenschaften wie oder besser als B (RIN> 5,2) für nachgeschaltete Microarray-Anwendungen eingesetzt. Solide und offene Pfeilspitzen zeigen die Position der 18S und 28S rRNA Tierarten.
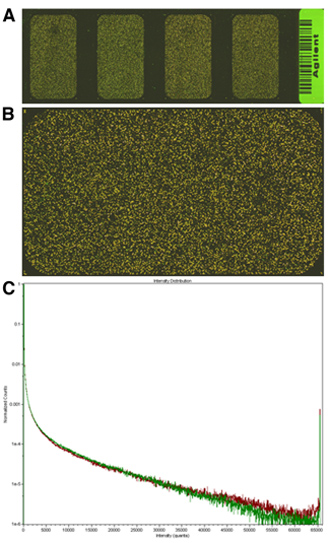
Abbildung 2. Beispiele für Array von Bildern und Streudiagramme. A, ganze Dia-Vorschau und zeigt die vier Arrays in der 4X44K Agilent-Format; B, hochauflösende Scans von einem der Array-Bereiche zu beachten, dass keines der Signale (rot oder grün) überwiegt; C, Streudiagramm des Bildes in B, beachten Sie, dass die Signale vollständig überlappen und nicht mehr als 1x10 -6 Features sind bei Sättigung Intensität.
Diskussion
Expression Profiling mit DNA Microarrays bietet eine einfache Lösung, um differentiell exprimierte Gene zwischen zwei biologischen Proben zu identifizieren. Erfolgreiche Expression Profiling Experimente erfordern qualitativ hochwertige RNA-Proben, robust Kennzeichnung und Hybridisierung. In unserer Erfahrung, bieten die kommerziellen Arrays und Kennzeichnung Kits von Agilent qualitativ hochwertige Daten zu einem vernünftigen Preis. Während Agilent bietet auch eigene Hybridisierung und Scannen Maschinen, favorisieren wir die Hybridisierung von Roche NimbleGen-und GenePix 4000B Microarray-Scanner von Molecular Devices. Beachten Sie, dass die Roche-NimbleGen Hybridisierung Einheit früher als MAUI Hybridisierung System bekannt. Nach der jüngsten Firmenübernahme von Roche haben die A4 Mischer eingestellt worden. Ab dem Zeitpunkt des Schreibens dieses Artikels, können sie immer noch durch andere Anbietern wie KREATECH Diagnostics (gefunden werden www.kreatech.com ; dieser Website bietet auch eine bequeme Array-Kompatibilität-Tool), aber langfristige Lieferfähigkeit ist ungewiss. Andere Hybridisierung Systeme zur Verfügung stehen (z. B. von Agilent), aber wenn kompatiblen Mixer zu finden für das Array verwendet werden, empfehlen wir die Roche-NimbleGen Systems für seine Qualität und Reproduzierbarkeit.
Offenlegungen
Danksagungen
Diese Arbeit wurde durch Stipendien an ED aus der James S. McDonnell Foundation 21 st Century Award Program und ein NIH Director des New Innovator Award unterstützt. GAB wurde zum Teil durch ein Postdoc-Stipendium am California Institute of Regenerative Medicine unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 2100 Bioanalyzer | Agilent Technologies | G2943CA | Includes chip priming station, vortexer, software and laptop computer |
| 2100 Bioanalyzer electrophoresis set | Agilent Technologies | G2947CA | |
| RNA 6000 Nano kit | Agilent Technologies | 5067-1511 | includes size ladder, marker, gel matrix, dye, electrode cleaners, spin filters and nanochips |
| RNase Zap | Applied Biosystems | AM9780M | |
| Quick Amp labeling kit, two-color | Agilent Technologies | 5190-0444 | |
| RNeasy mini kit | Qiagen | 74104 | |
| Gene Expression Hybridization Kit | Agilent Technologies | 5188-542 | Includes Blocking agent, Fragmentation buffer and Hybridization buffer |
| Gene Expression Wash Buffer Kit | Agilent Technologies | 5188-5327 | Includes Wash Buffers 1 and 2 and Triton X-102 |
| Hybridization Station | Roche Group | 05223652001 | 4-slide model |
| Hybridization System Accesory Kit | Roche Group | 05327695001 | Contains verification assembly for testing mixing, replacement O-rings, disassembly tool, forceps and brayer |
| A4 mixer hybridization chambers | |||
| Whole Human Genome (4x44) Oligo Microarray | Agilent Technologies | G4112F | |
| Positive displacement pipette | Gilson, Inc. | F148504 | |
| Capillary pistons for positive displacement pipette | Gilson, Inc. | F148415 | |
| Microarray dryer | Nimblegen | 05223636001 | |
| Microarray fluorescent scanner, Axon GenePix 4000B | Molecular Devices | GENEPIX4000B | |
| GenePix Pro 6.0 software | Molecular Devices |
Referenzen
- Diaz, E. One Decade Later: What has Gene Expression Profiling Told us About Neuronal Cell Types, Brain Function and Disease. Curr Genomics. 10, 318-318 (2009).
- Barisone, G. A. . Expression profiling in Neuroscience, edited by Ioannis Karamanos. , (2010).
- Gelder, R. N. V. a. n. Amplified RNA synthesized from limited quantities of heterogeneous cDNA. Proc Natl Acad Sci U S A. 87, 1663-1663 (1990).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten