Method Article
Perfiles de transcripción de los organismos unicelulares de los cardiomiocitos de ratón adulto
En este artículo
Resumen
Perfiles de expresión de células individuales permite el análisis detallado de la expresión génica de células individuales. Se describen los métodos para el aislamiento de los cardiomiocitos, y la preparación de los lisados resultantes, ya sea para toda microarrays transcriptoma o qPCR de objetivos específicos.
Resumen
Si bien numerosos estudios han examinado los cambios de expresión génica de los homogeneizados de tejido cardíaco, lo que impide el estudio de la variación estocástica inherente entre las células en un tejido. El aislamiento de poblaciones puras de cardiomiocitos a través de una perfusión de colagenasa de los corazones del ratón facilita la generación de microarrays de células única de toda la expresión de genes transcriptoma, o qPCR de objetivos específicos con matrices nanofluídicos. Se describe aquí un procedimiento para examinar las células individuales perfiles de expresión génica de los cardiomiocitos aislados desde el corazón. Este paradigma permite la evaluación de los parámetros de interés que no dependen de la media (por ejemplo, la variación entre las células en un tejido) que no es posible cuando se utilizan los flujos de trabajo convencionales, todo el tejido para la evaluación de la expresión génica (Figura 1). Hemos logrado la amplificación de la robusto microgramos transcriptoma de células individuales rendimiento de cDNA de doble cadena que facilita el uso de microarrays de icélulas INDIVIDUALES. En el procedimiento se describe el uso de las matrices de NimbleGen, que fueron seleccionados por su facilidad de uso y capacidad de personalizar su diseño. Por otra parte, una transcriptasa inversa - amplificación específicos (RT-STA) de reacción, permite qPCR de cientos de objetivos por nanofluídicos PCR. Con cualquiera de estos métodos, es posible examinar la variabilidad de la expresión entre las células, así como examinar los perfiles de expresión de los tipos de células poco comunes dentro de un pañuelo de papel. En general, el único gen de células enfoque de expresión permite la generación de datos que potencialmente pueden identificar los perfiles de expresión peculiar que suelen promediar la hora de examinar la expresión de millones de células a partir de homogeneizados de tejidos típicos generados a partir de su conjunto.
Protocolo
1. Paso 1 - aislamiento de los cardiomiocitos
- Preparar el tampón de digestión de la siguiente manera:
10 x tampón de digestión (hecho en ultra-pura H 2 O)Conc. final. g / L g/500 ml 10x solución g / L NaCl 130 mM 7.597 3.3 75.97 KCl 5 mM 0.4 0.2 4.0 Ácido pirúvico 3 nM 0.33 0.165 3.30 HEPES 25 mM 5.96 2.85 59.6 MgCl2 0,5 mM 0.101 0.05 1.01 NaH 2PO 4monobasic 0,33 mM 0.04 0.02 0.40 Dextrosa 22 mM 3.96 1.98 39.6
pH a 7,4 con NaOH - Utilizando el tampón de digestión 10x, hacer las siguientes cuatro soluciones para cada corazón que se perfundidos (hecho en ultra-pura H 2 O):
Tampón de digestión A (Haga tres alícuotas de esta solución)
- 50 ml - 1x tampón de digestión que contiene:
- L 75 - solución EGTA (100 mM EGTA)
Digestión Buffer B (hacer uno de estos)
- 25 ml - 1x tampón de digestión que contiene:
- 1,25 l - solución de CaCl2 (1 M)
- 1 mg - la proteasa
- 25 mg - colagenasa (can también utilizan el 75% de esta cantidad = 18,75 mg) **
Colección de búfer (hacer uno de estos)
- 2,5 ml - 1x tampón de digestión que contiene:
- 1,25 l - solución de CaCl2 (1 M)
- 0,5 mg - la proteasa
- 15 mg - colagenasa (75% = 11,25 mg) **
- 125 mg - BSA
La neutralización de tampón de lavado (hacer uno de estos)
- 25 ml - 1x tampón de digestión que contiene:
- 6,25 l - solución de CaCl2 (1 M)
- 250 mg de BSA
** Dependiendo de la fuente y el lote de la colagenasa, la actividad de las enzimas puede variar. Puede que sea necesario para optimizar la cantidad de enzima añadido a este búfer para evitar el exceso de la digestión.
- Establecer el sistema de aislamiento como se ha descrito anteriormente en dVENTA AL 1,2 con tres vasos de 15 mL de tampón de digestión A en el hielo, un tubo de 50 ml de tampón de digestión A y la B 25 ml digestión Buffer listo para perfusión (tampón de digestión A deben ser lavados a través de la línea, por lo menos de 5 ml ejecuta a través del sistema), Buffer Colección calentado en un baño de agua para elevar la temperatura a 37 ° C, y el tampón de lavado de neutralización a temperatura ambiente.
- Llenar la cánula de perfusión y el plato de la disección en la que se limpia el corazón y atada en la punta de la perfusión con A. frío tampón de digestión sin apretar el nudo algunos de sutura (4-O a 6-O tamaño) alrededor de la parte superior de la cánula. Esta tarde se utiliza para mantener la aorta para evitar que el corazón se salga de la cánula.
- Terminales sedar al ratón con una inyección IP de agente anestésico (0,25 ml solución de sodio pentobarbital) y asegurarse de que el ratón está inconsciente y no reacciona a los estímulos de dolor. Abrir la cavidad torácica con cuidado a lo largo del margen dorsal cortando el diafragma(Figura 2). Corte por arriba hacia arriba y reflejan la caja torácica anterior (de estas incisiones se denominan B y A, respectivamente, en la Figura 2) Crítica -. El ratón todavía tiene que ser la respiración antes de estas incisiones.
- Usando una pipeta de plástico cortadas a la medida, con cuidado aspirar el corazón en la pipeta. Eliminar de un plumazo de la cavidad torácica, teniendo cuidado de dejar suficiente de la aorta en el corazón para identificar claramente la rama aorta (Figura 3). Con una pipeta se utiliza, ya que causa un mínimo daño al corazón.
- Suelta el corazón en un vaso de precipitados de tampón de digestión fría y un enjuague la sangre de distancia. Después de 15 a 45 segundos el lugar del corazón en el segundo vaso de tampón de digestión fría para enjuagar un nuevo brevemente. Transferir el corazón en la placa de Petri también está lleno de frío A. tampón de digestión
- Ver el corazón en el plato de un tampón de digestión en el ámbito de disección y cuidadosamente aislar la aorta y sus ramas. Con cuidado, corte justo por debajo de los últimos Branch (Figura 4).
- Coloque la punta de la cánula en la aorta y ligar en el lugar con el 4-O a 6-O sutura mediante la configuración del escenario bajo un microscopio de disección. Asegúrese de que la cánula no se sienta muy bajo dentro de la aorta y se mantiene por encima de las arterias coronarias (Figura 4).
- Con tampón de digestión A fluye a través de la configuración de la perfusión, coloque la punta de la perfusión a la instalación y comenzar a perfundir el corazón hasta que la mayoría de la sangre se lava con el corazón y el líquido que sale del corazón es evidente. Crítica - la temperatura del líquido de perfusión debe ser lo más cercano posible a 37 ° C cuando se trata de la sonda. Si la solución es demasiado caliente o demasiado fría después de la perfusión va a matar el tejido. El tiempo transcurrido hasta la perfusión del corazón también es fundamental. La ventana de tiempo debe ser inferior a 5-10 minutos de la extracción del corazón hasta que comienza la perfusión. El corazón debe ser removido bajo anestesia, no CO 2 afijación o dislocación cervical.
- Una vez que la sangre se fuehed desde el corazón, vaya a la B tampón de digestión de la A. tampón de digestión Asegúrese de que el B tampón de digestión es que ahora fluye a través del corazón. La solución comenzará a tener un tinte amarillo. Espere 2-3 minutos hasta que el corazón muestra signos de perder su forma y comienza a verse redondeada que es una indicación de que la digestión está funcionando. El miocardio debería aparecer más claro a medida que avanza la perfusión.
- Cortar los ventrículos con unas tijeras y colocarlo en un tubo cónico de 50 ml que contiene 15 ml de tampón de la colección a 37 ° C. Cortan cuidadosamente el ventrículo en trozos pequeños, mientras que la colocación de los tejidos en el búfer de la colección. Mezclar suavemente con una pipeta de plástico para disolver las adherencias célula-célula y hacer una mezcla de células de cardiomiocitos sola.
- Cuando la mayoría de los tejidos que se disuelva, (menos de 1 min) que las piezas de tejido más grande hunden hasta el fondo. Retire y guarde el sobrenadante a un tubo cónico de 15 ml. Deje que la gravedad tire de una pastilla a partir del sobrenadante de 10 a 20 min (15min preferido) a temperatura ambiente.
- Retire y deseche el sobrenadante. Lavar pellet añadiendo 5 ml de la solución de lavado de neutralización de la pastilla y mezcle suavemente las células para volver a suspender. En este punto, las células están inmediatamente disponibles para ser seleccionados para el análisis de células individuales (vaya al paso 2).
2. Paso 2 - Aislamiento de células individuales
- Prepare un micromanipulador con una llama y un tubo capilar de vidrio que se utilizan para manipular las células. El micromanipulador es simplemente un tubo capilar muy fino, que entonces se une a una larga pieza de tubo flexible que se puede mover o recoger las células cuando se aplica succión.
- Proceder de inmediato a la selección de las células individuales bajo un microscopio (Nikon Eclipse TS100, 20x) con un micromanipulador que fue creado con una pipeta y sacó una bolsa de aire para evitar que el material se transfiere a la célula de la muestra muestras. Asegúrese de que la población de células está sano(70% viable) y no daña el proceso de aislamiento.
- Diluir las células en una pequeña placa de Petri, para que puedan ser seleccionados individualmente. Cuando las células recogiendo asegúrese de capturar las células en un volumen pequeño (menos de 2 l). Seleccione sólo las células sanas que tienen la morfología característica de cardiomiocitos normal (Figura 5).
- Coloque las células individuales en la parte inferior de cada uno de los tubos de PCR y congelar inmediatamente en hielo seco. Estas células se almacenan a -80 ° C hasta que se van a utilizar para el análisis de expresión génica.
3. Paso 3A - Single qPCR células de la expresión génica
Para llevar a cabo qPCR en las células de un solo emplean un protocolo adaptado de uno desarrollado para la expresión de un solo gen de la célula por la Corporación Fluidigm para sus matrices Biomark nanofluídicos qPCR (Fluidigm - Protocolo de avanzar en el desarrollo # 30) 3,4.
- La preparación de la transcripción inversa - Ampliación específicos (RT-STA). Para serginebra, volver a suspender todas las parejas de cebadores (hemos usado hasta 125 pares de cebadores con éxito) de los genes de interés en 100 mM en tampón 1x suspensión de ADN (10 mM Tris, pH 8,0, 0,1 mM EDTA). Combinar y diluir aún más los pares de cebadores de avance y retroceso a 20 M para cada ensayo. Por último, diluir todos los pares de cebadores en una solución con una concentración final de 200 nM para cada ensayo que se amplifica en la reacción RT-STA. Para ello, toma cada par de cebadores y diluir a 1:100. Por ejemplo, si usted tiene 30 pares de cebadores de ensayo a 20 micras, que tendrá que añadir una L de cada par (30 en total l), y luego hacer el resto del volumen de 70 l de tampón de ADN de suspensión para llegar a la final dilución volumen de 100 mL.
- Ahora que las soluciones de imprimación están preparados, montar la mezcla de reacción RT-STA principal. Prepare suficiente mezcla maestra para cada una de las células individuales que desee ampliar.
Tabla 1. RT-STA maestro de la mezclaReactivo Volumen en l (por reacción) CellsDirect mezcla de reacción 2x 5.0 Superíndice III RT mezcla de platino Taq 0.2 RT-STA Primer mezcla (200 nM cada ensayo) 2.5 Nucleasa libre H 2 O (o tampón 1x suspensión de ADN) 1.3 Total 9.0 - Quitarse de inmediato las células congeladas y centrifugar a máxima RCF de 30 segundos para asegurar que las células se colocan en la parte inferior del tubo. Añadir 9 l de RT-STA mezcla en el tubo y proceder a la reacción en un termociclador.
- 50 ° C - 15 minutos
- 95 ° C - 2 minutos
- 18 ciclos de
- 95 ° C durante 15 segundos
- 60° C durante 4 minutos
- Mantener a 4 ° C
- Retire los tubos de la máquina de PCR y añadir 4 l de ExoSAP-IT para la reacción. ExoSAP-IT es un reactivo de limpieza de PCR, lo que eliminará el exceso de cebadores y la enzima de la reacción RT-STA. Ejecutar los tubos a través de la siguiente reacción en una máquina de PCR.
- 37 ° C - 15 minutos
- 80 ° C - 15 minutos
- Mantener a 4 ° C
- Cuando el tratamiento ExoSAP-Es completo, diluir la muestra 1:05 con tampón suspensión de ADN. Las muestras se preparan ahora para el análisis de qPCR. Utilizamos este material junto con matrices de Fluidigm Gene qPCR expresión (48.48, 96.96 o placas). También es posible utilizar este método con más instrumentos qPCR tradicionales (96 o 384 formatos también). Sin embargo, estos instrumentos suelen requerir mucho más de entrada de la muestra, que restringe el número de ensayos que pueden ser performed de una sola célula.
3. Paso 3B - amplificación transcriptoma enteros y microarrays de células individuales
Extracción y de amplificación
- Use WTA2 Sigma kit (Cat. # WTA2) para amplificar cDNA de doble cadena de ambas pastillas y las células individuales. Las células individuales se "extrae" a través del primer paso del protocolo de Sigma WTA2 de acuerdo a las instrucciones del fabricante. Durante esta primera etapa de la incubación con el tampón de la biblioteca de síntesis a 37 ° C durante 5 minutos rompe la membrana de las células individuales y "extrae" el mensaje para la amplificación.
- Amplificar las células individuales en dos rondas, siguiendo las instrucciones del kit de WTA2 la Sigma-Aldrich. Realizar la etapa de preparación de la biblioteca de acuerdo con el protocolo del fabricante, a continuación, agregar 1 / 5 de la muestra de la biblioteca a 70 l de la amplificación WTA2
Master Mix. Amplificar las muestras durante 25 ciclos con los parámetros recomendados en bicicleta. - Purificar el usi muestrasng Qiagen QIAquick PCR kit de purificación (Cat # 28104) y el proceso de Qiacube Qiagen, utilizando el "Liberador de espacio en las reacciones de amplificación de PCR QIAquick estándar V4" protocolo.
- Concentrar las muestras purificadas a 5 l, con un vacío de velocidad, y poner a través de una segunda ronda de amplificación de 17 ciclos (WTA2 protocolo de amplificación y los parámetros de ciclos repetidos).
- Purificar las muestras utilizando el kit QIAquick PCR Purificación y control de calidad utilizando un espectrofotómetro NanoDrop y Bioanalyzer Agilent 7500 kit de ADN, según el protocolo del fabricante.
Etiquetado y matrices NimbleGen la expresión génica
- Etiqueta 2 g de doble cadena de cDNA de cada muestra de células amplificado utilizando un kit de marcaje de NimbleGen color (Cat # 05223555001) según el protocolo del fabricante.
- Hibridar 5 g de Cy3 muestra la etiqueta de Mus musculus 12x135k, NimbleGen arrays de expresión génica (Cat # 05543797001) segúnOrding el protocolo del fabricante. Después de la hibridación de las muestras de las matrices, las diapositivas se deben lavar y secar antes de que se pueden obtener imágenes en un escáner láser.
- Exploración despliegue de expresiones genéticas mediante un escáner láser. Aquí, se utiliza un escáner Molecular Devices GenePix 4200A con la configuración de 100POW, y 300 350PMT. A continuación, generar los archivos de par para cada serie con el software NimbleScan NimbleGen y, posteriormente, analizar la expresión del transcriptoma conjunto de cada matriz de células individuales (una tabla de típica estable expresado transcripciones de los cardiomiocitos se incluye como un suplemento, el cuadro S1).
Si la perfusión del corazón funcionaba bien debe haber un alto porcentaje de células saludables para el corazón que conservan su morfología rectangular típica del aislamiento. Si la perfusión no procedió bien, entonces habrá un gran porcentaje de células muertas (ver imágenes en la Figura 5). Si las células están correctamente amplificado utilizando los métodos WTA2 o RT STA-lan de los productos finales deben pasar las pruebas de control de calidad posterior a la calidad de las muestras de ARNm por NanoDrop y Bioanalyser (Figura 6). Para el flujo de trabajo de microarrays, que se termine esto después de la amplificación WTA, donde se analiza el ARNm por tanto NanoDrop y Bioanalyzer. Representante de los resultados positivos de este análisis se muestran en la Figura 6. El WTA2 muestras deben mostrar una amplificación fuerte tanto con la lectura del espectrofotómetro NanoDrop, así como la lectura de la electroferograma Bioanalyzer (BA) chip (Figura 6). Una tabla de los genes que se han detectado de forma estable a través de microarrays de células individuales se incluye como un ejemplo (Tabla S1). Para el proceso de qPCR, la calidad de los datos pueden ser evaluados mediante la realización de un paso se funden a la final de la reacción de PCR para asegurar que el primer sets están amplificando el producto deseado (Figura 7). Si es correcto, este paso de fusión debe generar un pico de fusión específico. Para ilustrar este punto, un subconjunto de nanofluídicos qPCR datos se muestran en un mapa de calor de fusión pico temperatura (Figura 8) 8. Es posible evaluar la calidad de un producto de PCR por su temperatura de fusión para garantizar la ausencia de productos no específicos 5. Cada fila de este mapa representa una muestra de células (S1-S40) pusieron a prueba en 43 ensayos de qPCR independiente (A1-A43). En esta figura, está claro que algunas pruebas son muy variables y son probablemente menos confiables que los ensayos más estable con una mayor especificidad de la PCR.
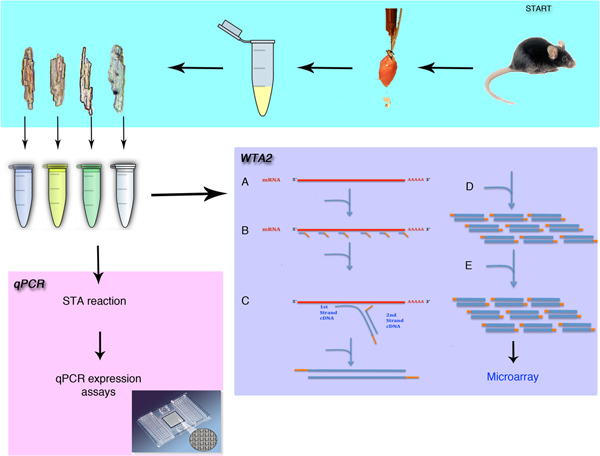
Figura 1. Visión general de la aislamiento de células individuales y el análisis de la expresión génica. El procedimiento se muestran en la extirpación quirúrgica del corazón del ratón y el aislamiento de los cardiomiocitos individuales. Los métodos descritos en este procedimiento son los métodos, ya sea para qPCR análisis de microarrays o después de la amplificación transcriptoma conjunto (WTA). El procedimiento comienza con el WTA de lisis (A) y la unión de los cebadores de Sigma universal (B) al ARNmpiscina. La extensión de estos cebadores (C) y la fase de amplificación (D) crear microgramos de material amplificado. Esta amplificación se repite (E) para generar suficiente material para el procedimiento de microarrays.

Figura 2. Representación de la ubicación de las incisiones para extraer el corazón del ratón. Corte a lo largo de la pared lateral de la caja torácica (A), luego se corta a lo largo del borde inferior de la caja torácica (B). Cortar los vasos encima y por debajo del corazón a la remoción dejando al mismo tiempo suficiente de la aorta para canalizar el corazón. Imágenes adaptadas de MJ Cook, 6.
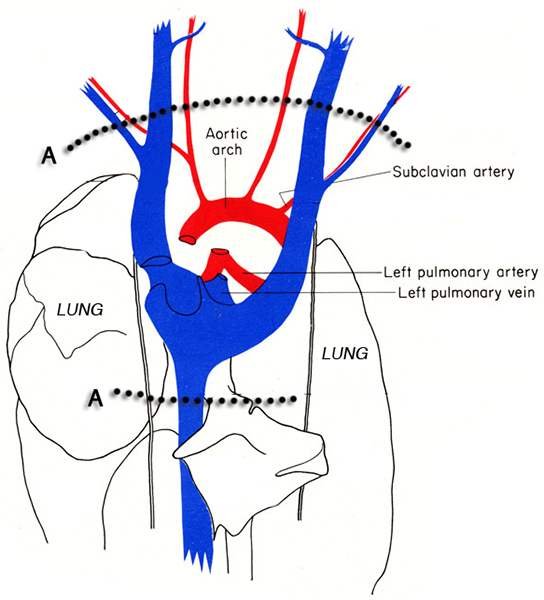
Figura 3. Diagrama del tórax del ratón. Las líneas punteadas indican la ubicación de las incisiones (A) para extirpar el corazón de la cavidad torácica sin dañar el h eart tejido. Imágenes adaptadas de MJ Cook, 6.
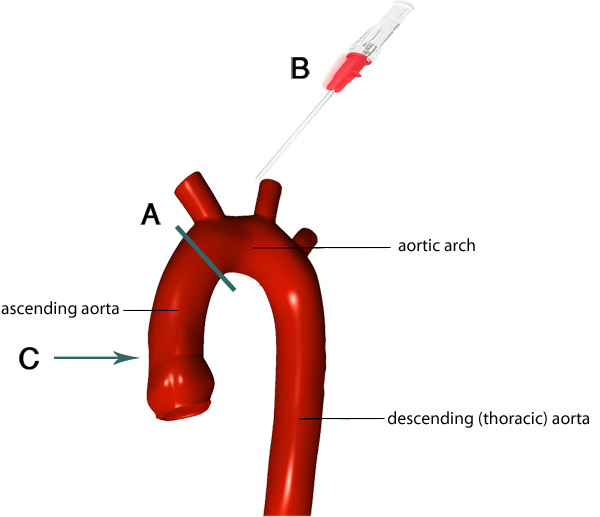
Figura 4. Imagen del arco aórtico y sus ramas. La línea gris indica el lugar ideal de donde recortar la aorta (A). La aorta que permanezcan unidas al corazón es una cánula para que la punta de la cánula (B) pasa al nivel apropiado dentro de la aorta (C). Imágenes adaptadas de F. Gaillard 7.

Figura 5. Selección de celdas individuales para el análisis de expresión génica. Cada panel muestra los cardiomiocitos imágenes en el microscopio óptico que muestra la morfología típica de los cardiomiocitos. Cardiomiocitos sanos son indicados por las flechas verdes. Las células que están muertos o moribundos se muestran con flechas rojas. Estas células muertas no se debe utilizar en el análisis.
iles/ftp_upload/3302/3302fig6.jpg "alt =" Figura 6 "/>
Figura 6. Control de calidad los resultados WTA para la amplificación de células individuales. (A) Imagen de un espectrofotómetro de lectura NanoDrop que sea apropiado para las transcripciones de amplificación de la reacción de la WTA. (B) La lectura de un chip Bioanalyzer muestra los resultados de tres células amplificadas.
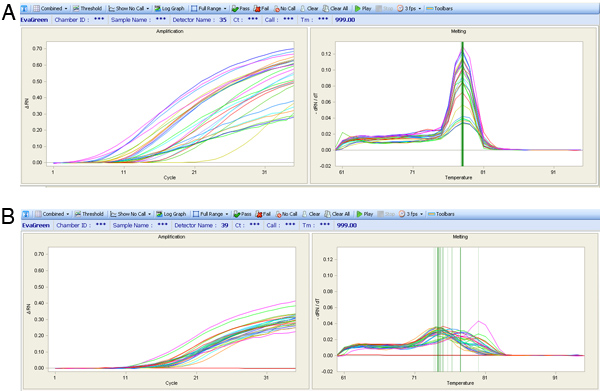
Figura 7. QPCR curvas de amplificación (izquierda) y el pico de fusión curvas de temperatura (derecha). (A) Los resultados de la expresión de una prueba de calidad se muestran con un solo pico fusión a pesar de las curvas de amplificación diferentes. (B) Derretir curvas de un primer set pobre, que tiene picos de fusión muy variable que no es ideal para el análisis de la expresión.
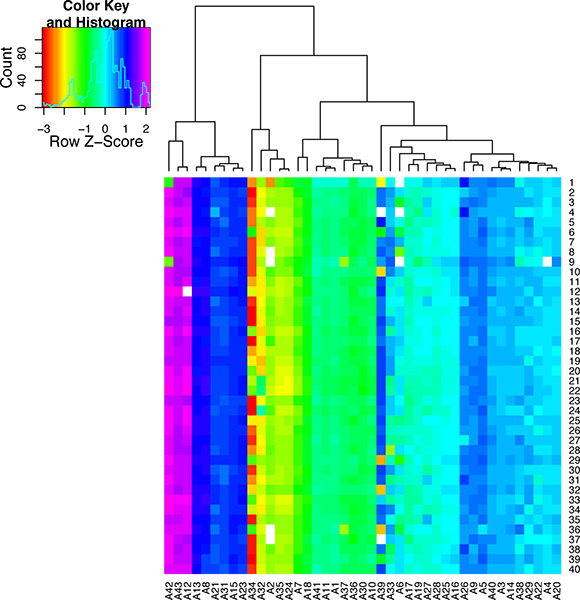
Figura 8. Los resultados de control de calidad de la única célula qPCR reaccionariones. Este mapa de calor se ha creado para mostrar las temperaturas de fusión como una escala de colores. La temperatura de fusión del pico de cada ensayo qPCR (A1-A43) se muestra por 40 celdas individuales (S1-40). Los ensayos se agruparon de acuerdo a su temperatura de fusión. Al observar la columna para cada ensayo, es posible ver la variación de fusión a través de todas las muestras. Esta cifra demuestra que algunos ensayos de PCR cuantitativa son muy específicos, mientras que otros son muy variables y por lo tanto no son adecuados para el análisis de células individuales.
Tabla S1. mesa de consulta de datos de microarrays. Estos genes se enumeran de acuerdo a la robustez en el que se detectan en los cardiomiocitos a través de una sola gran base de datos. Esta lista de genes indica la sensibilidad de la detección de una serie de genes que se expresan típicamente en cardiomiocitos individuales.
Discusión
Este método tiene la posibilidad de generar cardiomiocitos para un número de funcionales, así como estudios de expresión génica. El examen de las células individuales es un área floreciente en el análisis de expresión génica. La ventaja de estudiar los niveles de la transcripción a nivel de la célula es que permite el examen de una población celular pura, que no es posible a partir de preparaciones de tejidos conjunto. Además, el análisis de células individuales permite el examen de la variación estocástica de los niveles de mRNA en las células individuales para definir las poblaciones de células que se creía que ser homogénea 8,9. Además de identificar los genes que son potencialmente estocástico en su expresión génica 10,11,12, el método también permite la identificación de las poblaciones celulares raras definidas por sus perfiles de expresión génica.
Aunque este método ofrece una serie de interesantes aplicaciones potenciales en el análisis de expresión génica, hay somadvertencias e y consideraciones en el uso de células individuales. La principal limitación para el análisis de células individuales, es la recolección de suficientes células en el experimento para alcanzar la significación estadística en cuanto a la métrica de interés, por ejemplo, la varianza. En los casos en que el número de células aisladas de tejido de interés no es una limitación, se puede examinar cientos de células por grupo, utilizando métodos como la secuenciación de próxima generación, o matrices nanofluídicos. Sin embargo, en algunos casos, puede haber dificultades para obtener suficientes células de los tejidos de interés. Esto puede en parte ser causado por procedimientos tiempo idiosincrasia intensiva para la recolección del tipo de células específicas. Sin embargo, una cuidadosa planificación y preparación antes de proceder con la colección única célula puede todavía permitir un análisis riguroso sola célula con un error técnico limitado. Al examinar los resultados debe tenerse en cuenta, que aunque este procedimiento para aislar las células se ha utilizado en numerosas Studies (incluida la microscopía, la electrofisiología, etc), el propio aislamiento potencialmente podría afectar a la expresión génica. Al igual que con cualquier otro método que manipula las muestras biológicas, los resultados de sus hallazgos deben ser cuidadosamente validados para asegurar la expresión de células individuales es representativa del propio tejido y el sesgo de no técnicos. Métodos como la hibridación in situ puede ser útil para verificar estos resultados en el tejido intacto. Por último, es fundamental para asegurar que los datos generados es cuidadosamente revisada para el control de calidad. Los datos mostrados en la Figura 8 demuestra que los ensayos de qPCR puede ser muy fuerte en su especificidad, como el ensayo N º 13 (A13) o tener un alto nivel de variabilidad que puede llevar a la variación de técnicas, como prueba de # 34 (A34).
Divulgaciones
Acceso Libre y la producción de este de este artículo es patrocinado por Sigma-Aldrich.
Agradecimientos
Los autores desean agradecer la asistencia técnica de la C. Zambataro durante el rodaje de este protocolo. Agradecemos el apoyo de la Fundación Glenn de Investigación Médica (SM), la Fundación Hillblom, y los Institutos Nacionales de Salud para el premio Centro de Nathan Shock (P30AG025708) y PO1AG025901. JMF fue apoyada por T32AG000266 otorgado al Instituto Buck de Investigación sobre el Envejecimiento.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
| NaCl | Sigma-Aldrich | 71378 | |
| KCl | Sigma-Aldrich | 60128 | |
| Ácido pirúvico | Sigma-Aldrich | P4562 | |
| Hepes | Sigma-Aldrich | 54457 | |
| MgCl2 | Sigma-Aldrich | M1020 | |
| NaH 2 PO 4 monobásico | Sigma-Aldrich | P9791 | |
| Dextrosa | Sigma-Aldrich | G6721 | |
| NaOH | Sigma-Aldrich | 72068 | |
| EGTA | Sigma-Aldrich | O3777 | |
| CaCl2 | Sigma-Aldrich | 21115 | |
| Proteasa, Tipo XIV | Sigma-Aldrich | P5147 | |
| Colladeshidrogenasa, Tipo I | Worthington | C9891 | |
| Suspensión de ADN 1x Buffer (10 mM Tris, pH 8,0, 0,1 mM EDTA) | TEKnova, | PN T0221 | |
| BSA | Sigma-Aldrich | B8667 | |
| Pentobarbital sódico | Henry Schein Vet. * Fuente de | Contactar con proveedor para ordenar | * Deben ser adquiridos a través de un veterinario o un laboratorio profesional de animales |
| ExoSAP IT | Affymetrix / USB | 78201 | |
| CellsDirect 2x Mix Rxn | Invitrogen | 11737-030 | |
| Superíndice III RT Platinum Taq Mix | Invitrogen | 10928-042 | |
| RT-STA Primer Mix | IDT | Costumbre | |
| Nucleasa libre de agua | Sigma-Aldrich | W4502 | |
| WTA2 | Sigma-Aldrich | WTA2-50rxn | |
| QIAquick PCR kit de purificación | Qiagen | 28104 | |
| Qiacube | Qiagen | 9001292 | |
| NanoDrop Espectrofotómetro | NanoDrop | 2000c | |
| Bioanalyzer 7500 kit de ADN | Agilent | 7500 kit | |
| Un color kit de etiquetado | Roche-NimbleGen | 5223555001 | |
| Mus musculus 12x135k serie | Roche-NimbleGen | 5543797001 | |
| GenePix 4200A Escáner | Molecular Devices | 4200A | |
| TransPlex completa transcriptoma enteros Kit de amplificación (WTA2 Kit) | Sigma-Aldrich | WTA2-50RXN |
Referencias
- Wolska, B. M., Solaro, R. J. Method for isolation of adult mouse cardiac myocytes for studies of contraction and microfluorimetry. Am. J. Physiol. 271 (3 Pt 2), H1250-H1255 (1996).
- Shioya, T. A simple technique for isolating healthy heart cells from mouse models. J. Physiol. Sci. 57 (6), 327-335 (2007).
- Guo, G., Huss, M., Tong, G. Q., Wang, C., Li Sun, L., Clarke, N. D., Robson, P. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Dev. Cell. 18 (4), 675-685 (2010).
- Narsinh, K. H., Sun, N., Sanchez-Freire, V., Lee, A. S., Almeida, P., Hu, S., Jan, T., Wilson, K. D., Leong, D., Rosenberg, J., Yao, M., Robbins, R. C., Wu, J. C. Single cell transcriptional profiling reveals heterogeneity of human induced pluripotent stem cells. J. Clin. Invest. 121 (3), 1217-1221 (2011).
- D'haene, B., Vandesompele, J., Hellemans, J. Accurate and objective copy number profiling using real-time quantitative PCR. Methods. 50 (4), 262-270 (2010).
- Cook, M. J. The Anatomy of the Laboratory Mouse. Informatics. , (1965).
- Gaillard, F. Aorta. Radiopaedia. , (2008).
- Ståhlberg, A., Andersson, D., Aurelius, J., Faiz, M., Pekna, M., Kubista, M., Pekny, M. Defining cell populations with single-cell gene expression profiling: correlations and identification of astrocyte subpopulations. Nucleic Acids Res. 39 (4), e24-e24 (2011).
- Zhong, J. F., Chen, Y., Marcus, J. S., Scherer, A., Quake, S. R., Taylor, C. R., Weiner, L. P. A microfluidic processor for gene expression profiling of single human embryonic stem cells. Lab Chip. 8 (1), 68-74 (2008).
- Bahar, R., Hartmann, C. H., Rodriguez, K. A., Denny, A. D., Busuttil, R. A., Dollé, M. E., Calder, R. B., Chisholm, G. B., Pollock, B. H., Klein, C. A., Vijg, J. Increased cell-to-cell variation in gene expression in ageing mouse heart. Nature. 441 (7096), 1011-1014 (2006).
- Raj, A., van Oudenaarden, A. Nature, nurture, or chance: stochastic gene expression and its consequences. Cell. 135 (2), 216-226 (2008).
- Elowitz, M., Levine, A., Siggia, E., Swain, P. Stochastic gene expression in a single cell. Science. 297 (5584), 1183-1186 (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados