Method Article
성인 마우스 Cardiomyocytes의 단세포 Transcriptional 프로파일
요약
단세포 표현 프로 파일링 개별 세포의 자세한 유전자 발현 분석을 수 있습니다. 우리는 cardiomyocytes의 격리를 위해 방법을 설명하고, 구체적인 목표의 전체 transcriptome의 microarray 또는 qPCR 중 하나에 대한 결과 lysates를 준비.
초록
수많은 연구 심장 조직의 homogenates에서 유전자 발현 변화를 조사하고 있지만, 이것은 조직 내의 세포 사이의 고유의 확률적 변화를 공부하고 방지할 수 있습니다. 마우스 마음 collagenase 재관류를 통해 순수한 cardiomyocyte 인구의 격리는 전체 transcriptome 유전자 발현에 대한 단세포 microarrays의 생성, 또는 nanofluidic 배열을 사용하여 특정 대상의 qPCR을 용이하게합니다. 우리는 여기서 마음에서 격리 cardiomyocytes의 단일 세포 유전자 발현 프로파일을 조사하는 절차를 설명합니다. 이 패러다임은 유전자 발현의 평가 (그림 1) 종래의 전체 조직 워크플로를 사용 가능한 경우는 아니지만 (조직 내의 세포 사이의 예제 분산을위한) 의미에 의존하지 않습니다 흥미 통계 평가 수 있습니다. 우리는 전에 microarrays의 사용을 용이하게 이중 좌초 cDNA의 단일 세포 transcriptome 항복 마이크로 그램의 강력한 증폭을 달성ndividual 세포. 절차에서는 사용하고 자신의 디자인을 사용자 정의할 수있는 기능들의 편리를 위해 선택되었다 NimbleGen 배열의 사용을 설명합니다. 또는 반대 transcriptase - 구체적인 목표 증폭 (RT - STA) 반응, nanofluidic PCR에 의한 대상의 수백 qPCR을 허용합니다. 어느 이러한 방식을 사용하여, 그것은 세포 사이의 표현의 다양성뿐만 아니라, 조직 내에서 드문 세포 유형의 표현 프로필을 검토을 검토하는 것이 가능합니다. 전반적으로, 단일 세포 유전자 표현 방식은 잠재적으로 전체 조직에서 생성된 전형적인 homogenates의 세포 수백만의 표현을 조사했을 때 일반적으로 밖으로 평균 아르 특이한 표현 프로파일을 식별할 수있는 데이터의 생성을 허용합니다.
프로토콜
1. 1 단계 - Cardiomyocyte 격리
- 다음과 같이 소화 버퍼를 준비 :
10 X의 소화 버퍼 (울트라 순수한 H 2 O에서 만든)최종 Conc. G / L g/500 ML 10X 솔루션 G / L NaCl 130 MM 7.597 3.3 75.97 KCl 5 MM 0.4 0.2 4.0 Pyruvic 산 3 NM 0.33 0.165 3.30 HEPES 25 MM 5.96 2.85 59.6 MgCl 2 0.5 MM 0.101 0.05 1.01 아니이PO 4monobasic 0.33 MM 0.04 0.02 0.40 우선당 22 MM 3.96 1.98 39.6
NaOH로 pH를 7.4 버퍼에 - 10X 소화 버퍼를 사용하여, (울트라 순수한 H 2 O에서 만든) perfused됩니다 각각의 마음에 대해 다음 네 가지 솔루션을 확인하십시오
소화의 버퍼 (세명이 솔루션의 aliquots 확인)를
- 50 ML - 1X 소화 버퍼가 포함 :
- μL 75 - EGTA 용액 (100 MM EGTA)
소화 버퍼 B (다음 중 하나를 확인하십시오)
- 25 ML - 1X 소화 버퍼가 포함 :
- 1.25 μL - CaCl 2 솔루션 (1 M)
- 한 MG - 프로 테아제
- 25 MG - Collagenase (CAN **도이 금액 = 18.75 MG의 75 %)를 사용
수집 버퍼 (이 중 하나를합니다)
- 2.5 ML - 1X 소화 버퍼가 포함 :
- 1.25 μL - CaCl 2 솔루션 (1 M)
- 0.5 MG - 프로 테아제
- 15 MG - Collagenase (75 % = 11.25 MG) **
- 125 MG - BSA
중화 워시 버퍼 (이 중 하나를 확인하십시오)
- 25 ML - 1X 소화 버퍼가 포함 :
- 6.25 μL - CaCl 2 솔루션 (1 M)
- 250 MG BSA
** 소스 및 collagenase의 배치에 따라 효소 활동은 다를 수 있습니다. 그것은 오버 소화를 방지하기 위해이 버퍼에 추가 효소의 양을 최적화 할 수 있습니다.
- 로 이전에 D에서 설명하는 격리 시스템을 설정소화 버퍼 세 15 ML 비커와 1,2를 etail 얼음, 소화 버퍼 중 하나가 50 ML 튜브 및 (소화 버퍼가 5 ML의 최소한 라인을 통해 플러시해야 재관류를위한 준비 25 ML의 소화 버퍼 B ) 시스템을 통해 실행 컬렉션 버퍼 ° C, 그리고 중화 워시 버퍼 RT에서 37로 온도를 올릴 물을 욕조에 기까지 했어요.
- 심장은 정맥의 상단 부분 주위에 매듭을 대충 감기 소화 버퍼 A. 몇 가지 봉합 (4 - O 6 - O 크기)를 청소하고 재관류 팁에 묶여 될 재관류의 정맥과 절개의 접시를 채우십시오. 이것은 나중에 정맥을 늑대의 심장을 방지하기 위해 대동맥을 보유하는 데 사용됩니다.
- 말기 진정 마취 에이전트 (0.25 ML 나트륨 Pentobarbital 솔루션)의 IP 주사로 마우스와 마우스를 보장 의식과 통증 자극에 응답합니다. 다이어프램을 절단하여 흉부 여백을 따라 조심스럽게 흉강을 열고 절단(그림 2). 위쪽 머리에 잘라 (이 incisions는 그림 2에서 각각 B와 A를 지정) 앞쪽에 ribcage을 반영 중요 -. 마우스가 여전히 이러한 incisions하기 전에 호흡을해야합니다.
- 크기로 플라스틱 피펫 컷을 사용 부드럽게 피펫으로 마음을 빨아. 명확하게 대동맥 지점 (그림 3)을 식별하기 위해 심장에 충분히 대동맥의를 떠날주의되는 흉강의 심장을 도려내. 그것은 심장에 최소한의 손상을 초래 이후 피펫이 사용됩니다.
- 얼음 차가운 소화 버퍼 중 하나를 비커에 마음을 내려 거리에 피를 씻어. 15 일 이후 - 얼음 차가운 소화 버퍼의 두 번째 비커에 45 초 장소 심장 간단히 다시 린스합니다. 또한 감기에 소화 버퍼 A. 가득 페트리 접시에 마음을 전송
- 절개 범위에 따라 소화 버퍼의 접시에 마음을 확인하고주의 깊게 대동맥과 그 가지를 분리. 조심스럽게 마지막 branc 단 열등한 트림H (그림 4).
- 대동맥에 정맥 팁을 맞춤과 해부 현미경하에 무대 설치를 사용하여 6 - O의 치료 4 - O와 장소에서 ligate. 정맥은 대동맥 내에 너무 낮은 앉아 관상 동맥 (그림 4) 이상 유지하지 않도록.
- 소화 버퍼가 재관류 설정을 통해 흐르는와 설치에 재관류 팁을 배치하고, 혈액의 대부분은 마음이 맑은 떠나는 마음과 유체에서 씻었 때까지 마음을 perfuse 시작합니다. 중요 - 재관류 유체의 온도, 37 최대한해야합니다 ° C는 카테 테르 나올때. 솔루션이 너무 뜨겁거나 너무 차가운 경우 다음 재관류가 조직을 죽일 것입니다. 심장의 재관류 때까지 시간도 중요합니다. 재관류가 시작 때까지 시간 창이 심장의 제거 5-10 분 미만이어야합니다. 심장은 마취하에 제거이 첨부 또는 자궁 전위를 CO하지 있어야합니다.
- 일단 피가였다마음에서 쓰인는 소화 버퍼 B는 이제 심장을 통해 흐르는 있는지 확인 소화 버퍼 A.에서 소화 버퍼 B로 전환합니다. 솔루션은 노란 색조를 가지고 시작합니다. 심장의 모양을 잃을 조짐을 보이고 있으며 소화가 작동하는지 표시하는 둥근 모양을 시작할 때까지 2-3 분 정도 기다립니다. myocardium는 재관류가 진행되는 동안 라이터 나타납니다.
- 37 컬렉션 버퍼 15 ML을 포함한 50 ML 원뿔 튜브에 가위와 장소 심실을 잘라 ° C. 컬렉션 버퍼에 조직을 배치하는 동안 부드럽게 작은 조각으로 잘라 뇌실. 세포 - 세포 adhesions를 해산하고 단일 셀 cardiomyocyte 혼합물을 만들기 위해 플라스틱 피펫로 부드럽게 섞는다.
- 대부분의 조직이 해산하는 경우 (이하 1 분)는 큰 조직 조각이 바닥에 싱크하자. 15 ML 원뿔 튜브에 뜨는를 제거하고 저장합니다. 중력은 10 분에서 20 분 (15 이상 뜨는에서 펠릿를 끌어 오자분 상온에서) 선호.
- 제거하고 뜨는 폐기하십시오. 펠렛에 중화 워시 버퍼의 5 ML를 추가하고 부드럽게 그들을 resuspend 수있는 세포를 혼합하여 펠렛 씻으십시오. 이 시점에서 세포는 즉시 단세포 분석 (2 단계로 진행)을 선택하실 준비가 된 것입니다.
2. 2 단계 - 단일 세포의 분리
- 불꽃과 세포를 조작하는 데 사용됩니다 유리 모세관을 사용하여 micromanipulator를 준비합니다. micromanipulator은 단순히 다음 이동하거나 흡입이 적용되었을 때 세포를 데리러 수있는 유연한 튜브의 긴 조각에 연결된 매우 훌륭한 모세관 튜브입니다.
- 즉시 뽑아 피펫 및 단일 세포 샘플 샘플로 전송되는 자료를 막기 위해 출입구로 만들어진 micromanipulator를 사용하여 현미경 (니콘 이클립스 TS100, 20x)에서 개별 세포의 선택에 진행합니다. 셀 인구 건강 있는지 확인(70 % 가능한)과 격리 과정에서 손상되지 않습니다.
- 그들이 개별적으로 선택할 수 수 있도록 작은 배양 접시에있는 세포를 희석. 따기 세포가 작은 볼륨 (이하 2 μL)에 세포를 캡처해야하는 경우. 독특한 정상 cardiomyocyte의 형태 (그림 5)만을 건강한 세포를 선택합니다.
- 각 PCR 튜브의 바닥에 각각의 세포를 놓고 드라이 아이스에 즉시 그들을 동결. 그들은 유전자 표현 분석을 위해 사용됩니다 ° C까지 이러한 세포는 다음 -80에 저장됩니다.
3. 단계 3A - 단일 셀 qPCR 유전자 발현
(- 사전 개발 프로토콜 제 30 호 플루) 3,4 단일 세포에서 qPCR을 수행하기 위해서는 자신의 바이오 마크 Nanofluidic qPCR 배열에 대한 플루 Corporation에서 단일 세포 유전자 발현 위해 개발된 하나에서 적응 프로토콜을 사용합니다.
- 특정 대상 증폭 (RT - STA) - 역방향 전사를 준비. 수진은 1X DNA 서스펜션 버퍼 100 μm의 (10 MM 트리스, 산도 8.0, 0.1 MM EDTA (에틸렌 다이아 민 테트라 초산))에서 관심있는 유전자에 대한 (우리가 성공적으로 125 프라이머 쌍까지 사용한) 모든 프라이머 쌍을 resuspend. 결합하고 더 각각의 분석 20 μm의에 순방향 및 역방향 프라이머 쌍을 희석. 마지막으로, RT - STA 반응에서 증폭됩니다 각 분석 200 NM의 최종 농도 하나의 솔루션으로 모든 프라이머 쌍을 희석. 이렇게하려면 각 프라이머 쌍을 가지고 1:100 그들을 희석. 당신이 20 μm의 30 분석 프라이머 쌍을 가지고있다면 예를 들어, 각 쌍 (30 μL 합계) 1 μL를 추가해야합니다 후 최종 도달 DNA 서스펜션 버퍼의 70 μL와 볼륨의 나머지 부분을 구성하고 100 μL의 희석 볼륨.
- 뇌관 솔루션이 준비 이제, RT - STA 반응 마스터 믹스를 조립. 정도가 증폭하고자하는 각각의 세포 각각에 대해 마스터 믹스를 준비합니다.
표 1. RT - STA 마스터 믹스시약 μL의 볼륨 (사람마다 반응) 2X 반응 믹스를 CellsDirect 5.0 윗첨자 III RT Plantinum DNA 형성 촉매 믹스 0.2 RT - STA의 프라이머 믹스 (200 nm의 각각의 분석) 2.5 Nuclease 무료 H 2 O (또는 1X DNA 서스펜션 버퍼) 1.3 합계 9.0 - 즉시 세포가 튜브의 하단에 위치되는 것을 보장하기 위해 30 초 동안 최대 RCF에 냉동 세포와 원심 분리기를 제거합니다. 튜브에 RT - STA 믹스 9 μL를 추가하고 thermocycler의 반응로 진행합니다.
- 50 ° C - 15 분
- 95 ° C - 2 분
- 18주기
- 15 초 동안 95 ° C
- 604 분간 ° C
- 4 잡고 ° C
- PCR 기계에서 튜브를 제거하고 반응 ExoSAP - IT 4 μL를 추가합니다. ExoSAP - IT는 RT - STA 반응에서 초과 primers와 효소를 제거하는 PCR 정리 시약입니다. PCR 기계에 다음과 같은 반응을 통해 튜브를 실행합니다.
- 37 ° C - 15 분
- 80 ° C - 15 분
- 4 잡고 ° C
- ExoSAP - IT 치료가 완료되면, DNA 서스펜션 버퍼 샘플 1:5 희석. 샘플은 현재 qPCR 분석 준비하고 있습니다. 우리는 플루의 유전자 표현 qPCR 배열 (48.48 또는 96.96 플레이트)과 함께이 자료를 사용합니다. 그것은 전통적인 qPCR 장비 (96 자 또는 384 자 형식)와 함께이 방법을 사용할 수도 있습니다. 그러나 이러한 악기는 일반적으로 P 수 assays의 수를 제한하는 많은 샘플 입력을 요구하나의 세포에서 erformed.
3. 3B 단계 - 단일 세포의 전체 transcriptome 증폭 및 microarray를
추출 및 증폭
- 알약 및 단일 세포 모두에서 더블 좌초 cDNA를 증폭하기 위해 시그마의 WTA2 키트 (CAT # WTA2)를 사용합니다. 단일 세포는 제조 업체의 지침에 따라 시그마의 WTA2 프로토콜의 첫 번째 단계를 통해 "추출"입니다. 이 첫 번째 단계에서 37 도서관 합성 버퍼로 부화 ° C 5 분 단일 세포의 막과 "추출"증폭에 대한 메시지를 방해하십시오.
- 시그마 - 올드 리치의 WTA2 키트의 지시 사항을 사용하여 두 라운드에서 단일 세포를 증폭. 제조 업체의 프로토콜에 따라 도서관 준비 단계를 수행, 다음 WTA2 증폭의 70 μL에 라이브러리 샘플의 1 / 5에 추가
마스터 믹스. 권장되는 자전거 매개 변수를 사용하여 25주기 위해 샘플을 확대. - 샘플 usi 정화NG Qiagen의 PCR QIAquick 정화 키트 (CAT # 28104)과 "정리 QIAquick PCR 증폭 반응 표준 V4"프로토콜을 사용하여 Qiagen의 Qiacube을 처리합니다.
- 속도 진공을 사용하여 5 μL로 정화 샘플을 집중, 17 사이클 (WTA2 증폭 프로토콜과 사이클링 매개 변수 반복)에 대한 증폭의 두 번째 라운드를 통해 넣어.
- QIAquick PCR 정화 키트를 사용하여 샘플을 정화하고 제조 업체의 프로토콜에 따라 NanoDrop 분광 광도계와 애질런트의 BioAnalyzer DNA 7500 키트를 사용하여 품질을 확인합니다.
라벨 및 NimbleGen의 유전자 발현 배열
- 이중의 라벨이 μg는 제조 업체의 프로토콜에 따라 NimbleGen의 한 컬러 라벨링 키트 (CAT # 05223555001)를 사용하여 각 증폭 세포 샘플의 cDNA 좌초.
- 뮤스 musculus 12x135k, NimbleGen 유전자 발현 배열 (CAT # 05543797001) ACC로 Cy3 레이블 샘플의 5 μg을 잡종제조 업체의 프로토콜에 ording. 배열에 대한 샘플의 하이브 리다이 제이션 후, 슬라이드는 다음 씻어과 그들이 레이저 스캐너에 몇 군데하기 전에 건조해야합니다.
- 레이저 스캐너를 사용하여 스캔 유전자 발현 배열. 여기, 우리는 분자 장치 GenePix 4200A의 100POW의 설정 스캐너, 300 - 350PMT를 사용합니다. 다음 NimbleGen의 NimbleScan 소프트웨어를 사용하여 각 배열에 대해 쌍 파일을 생성하고 이후 각 단일 셀 어레이 (전형적인 안정 표현 cardiomyocyte 성적의 테이블이 보충으로 포함되며 테이블 S1을)에서 전체 transcriptome 표현을위한 분석.
심장 재관류가 잘 작동하면 격리시 자신의 전형적인 직사각형 형태를 유지 건강한 심장 세포의 높은 비율이있을 것입니다. 재관류가 잘 진행되지 않은 경우 다음 죽은 세포의 큰 비율 (그림 5에 이미지 참조)가있을 것입니다. 전지가 올바르게 중 WTA2 또는 RT - STA 방법에게를 사용하여 증폭하는 경우N 최종 제품은 NanoDrop 및 BioAnalyser (그림 6)에 의해 mRNA 샘플의 품질에 대한 후속 품질 관리 시험을 통과해야합니다. microarray 작업 흐름을 위해, 이것은 mRNA가 NanoDrop과 Bioanalyzer 모두에서 테스트 WTA 증폭 후 완료됩니다. 이 분석을위한 대표적인 긍정적인 결과는 그림 6에 표시됩니다. WTA2 샘플은 분광 광도계 NanoDrop 읽기뿐만 아니라 Bioanalyzer (BA) 칩 (그림 6)에서 electropherogram 판독 모두 강력한 증폭을 표시해야합니다. 안정 단일 세포의 microarray를 통해 발견되었습니다 유전자의 테이블은 예를 들어 (표 S1)로 포함되어 있습니다. qPCR 프로세스, 데이터의 품질 뇌관 세트가 원하는 제품 (그림 7) 확장되는 것을 보장하기 위해 PCR 반응의 끝에 용융 단계를 수행하여 평가하실 수 있습니다. 올바른 경우이 용융 단계는 특정한 용해 피크를 생성해야합니다. 이 지점을 설명하기 위해 nanofluidic qPCR 데이터의 하위 집합이 최고 녹기 t의 히트맵에 표시됩니다emperature (그림 8) 8. 이외의 특정 제품 5 부재를 보장하기 위해 용융 온도에 의한 PCR 제품의 품질을 평가하는 것이 가능합니다. 이지도의 각 행에 43 별도 qPCR의 assays (A1 - A43)에서 테스트 한 셀 샘플 (S1 - S40)를 나타냅니다. 이 그림에서, 그것은 일부 assays 매우 변수이며, 아마도 덜 신뢰성 높은 PCR의 특이성과 함께보다 안정적인 assays 이상의 것을 분명하다.
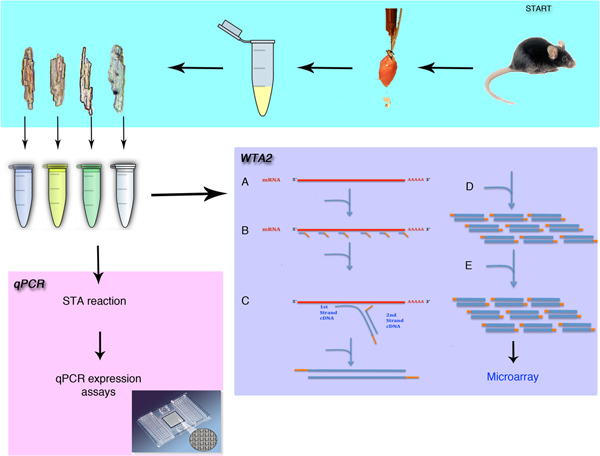
그림 1. 단세포 격리 및 유전자 발현 분석의 개요. 절차는 마우스 심장의 외과 제거 및 개별 cardiomyocytes의 고립을 설명합니다. 이 절차에서 설명하는 방법은 전체 transcriptome 증폭 (WTA) 이후 qPCR 또는 microarray 분석 중 하나에 대한 방법이 포함됩니다. WTA 절차는 다음 시그마의 유니버셜 primers의 바인딩 (B) mRNA에 용해 (A)로 시작수영장. 이러한 primers (C)와 증폭 (D) 단계의 확장 증폭 소재의 마이크로 그램을 만듭니다. 이 증폭은 다음 (E) microarray 절차에 대한 충분한 자료를 생성하는 반복됩니다.

그림 2. 마우스의 마음을 제거 incisions의 위치를 표현. 흉곽의 측면 벽 (A)을 따라 잘라 후 흉곽의 하부 여백 (B)를 따라 했네요. 여전히 마음을 cannulate 정도로 대동맥의를 떠나면서 위와 심장 아래의 혈관을 절단하면 제거하실 수 있습니다. 이미지 MJ 쿡 6 적응.
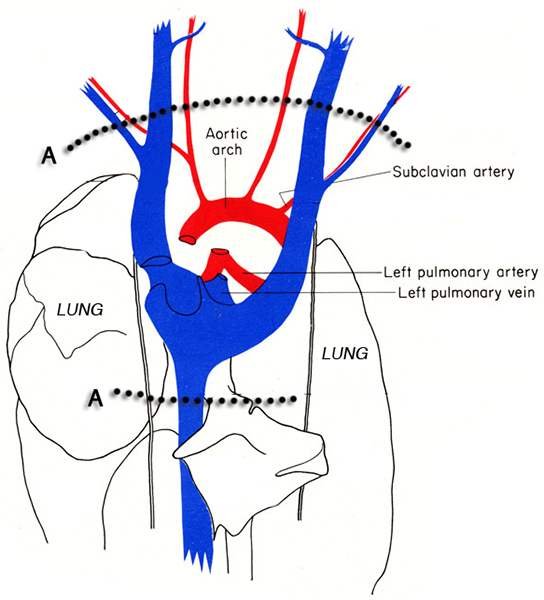
그림 3. 마우스의 흉부의 다이어그램. 점선은 H를 손상하지 않고 흉강의 심장을 절개하는 incisions (A)의 위치를 나타냅니다 eart 조직. 이미지 MJ 쿡 6 적응.
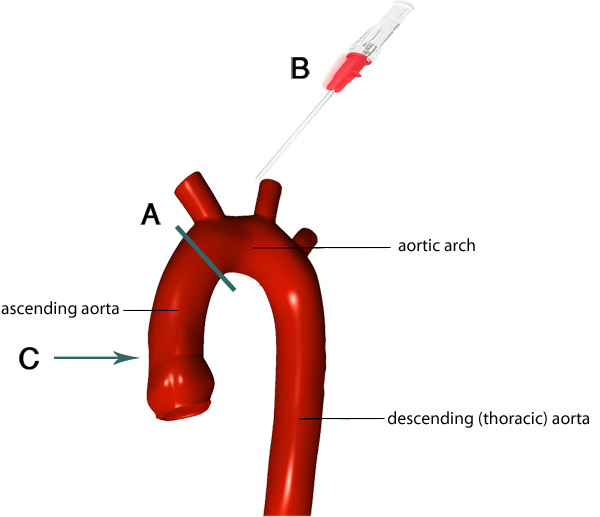
그림 4. 대동맥 아치와 가지의 이미지. 회색 라인은 대동맥 (A)를 잘라 어디에 이상적인 위치를 나타냅니다. 심장에 연결된 대동맥 나머지는 정맥 (B)의 끝이 대동맥 내에서 적절한 수준 (C)로 이동 있도록 cannulated입니다. 이미지는 F. 가일 라드 7 적응.

그림 5. 유전자 발현 분석을위한 단일 세포의 선택. 각 패널은 cardiomyocytes의 전형적인 형태를 보여주는 빛을 현미경으로 몇 군데 cardiomyocytes를 보여줍니다. 건강 cardiomyocytes은 녹색 화살표로 표시됩니다. 죽었거나 죽어 가고 있는데 전지는 빨간색 화살표로 표시됩니다. 이러한 죽은 세포는 분석에 사용할 수 없습니다.
iles/ftp_upload/3302/3302fig6.jpg "고도는 ="그림 6 "/>
그림 6. 단세포 증폭을위한 품질 관리 WTA 결과. WTA 반응에서 증폭 성적에 적합 NanoDrop 분광 광도계 판독의 (A) 이미지. (B) BioAnalyzer 칩의 판독 세 증폭 세포에서 결과를 보여줍니다.
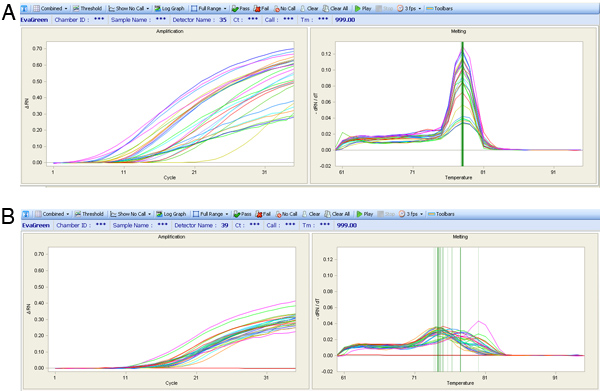
그림 7. qPCR의 증폭 곡선 (왼쪽 차트)와 피크 온도 곡선 (오른쪽 도표)를 용융. (A) 품질 분석에서 표현식 결과는 서로 다른 증폭 곡선에도 불구하고 하나의 용해 피크로 표시됩니다. (B) 표현 분석에 이상적되지 않습니다 높은 변수 용해의 봉우리를 가진 가난한 프라이머 세트에 대한 곡선을 용융.
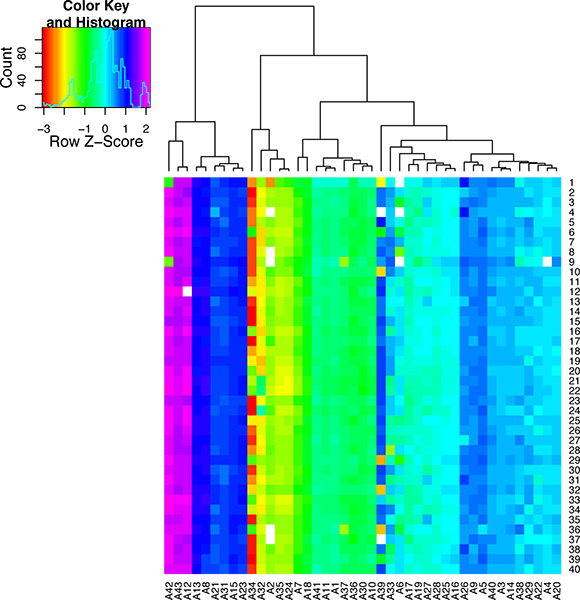
단일 셀 qPCR 반응에 대한 그림 8. 품질 관리 결과이온. 이 열지도는 색상 규모로 용해 온도를 표시하기 위해 만들어졌습니다. 각 qPCR 분석에 대한 피크 용융 온도 (A1 - A43은) 40 개인 세포 (S1 - 40)에 표시됩니다. assays들은 용융 온도에 따라 클러스터했다. 각 분석에 대한 칼럼을보고 이것은 변화가 모든 샘플에 걸쳐 용해 볼 수 있습니다. 이 그림은 다른 사람들이 높은 변수이며, 따라서 단일 세포 분석에 적합하지 동안 일부 qPCR의 assays는 매우 구체적인 것을 보여줍니다.
테이블 S1. microarray 데이터의 보충 테이블. 이들 유전자들은이 큰 데이터 세트에 걸쳐 단일 cardiomyocytes에서 감지되는 견고에 따라 나열되어 있습니다. 이 유전자 목록은 일반적으로 단일 cardiomyocytes로 표현됩니다 유전자의 여러 검출의 감도를 나타냅니다.
토론
이 메서드는 기능의 숫자뿐만 아니라 유전자 발현 연구에 대한 cardiomyocytes를 생성할 수있는 가능성이 있습니다. 단일 세포의 검사는 유전자 발현 분석에 급성장 지역입니다. 단일 세포 수준에서 transcriptional 수준을 검토의 장점은 전체 조직의 준비에서 가능하지 않은 순수한 세포 인구의 시험을 허용한다는 것입니다. 또한, 단일 세포 분석은 이전에 균질 8,9 것으로 생각되었다 세포 인구를 정의하는 각각의 세포에서 mRNA 수준의 확률적 변화의 시험을 수 있습니다. 자신의 유전자 표현 10,11,12에 잠재적으로 확률적 아르 유전자를 식별하는 이외에 방법은 또한 유전자 발현 프로파일에 의해 정의된 희귀 세포 집단의 식별을 허용합니다.
이 방법은 유전자 발현 분석에 흥미로운 가능성이 사용하는 다수의를 제공하고 있지만, som있다단일 세포의 사용에 전자주의 사항 및 고려 사항. 단일 세포 분석에 기본 제한은, 예를 들어 분산에 대한 관심의 메트 릭에 관련하여 통계적 중요성을 달성하기 위해 실험에서 충분한 세포의 모음입니다. 관심의 조직으로부터 격리 세포의 숫자가 제한되지 않습니다 경우에, 하나는 같은 차세대 시퀀싱, 또는 nanofluidic 배열과 같은 접근법을 사용하여 그룹 당 세포의 수백을 확인하실 수 있습니다. 하지만 일부 경우에, 관심의 조직에서 충분한 세포를 얻기에 어려움이있을 수 있습니다. 이 부분에서 대상 셀 형식의 컬렉션에 대한 특이한 시간 집중 과정으로 인해 발생할 수 있습니다. 그러나, 단일 셀 컬렉션을 진행하기 위해 신중 계획과 사전 준비는 여전히 제한된 기술 오류와 함께 엄격한 단일 세포 분석을 위해 허용할 수 있습니다. 그것이 고려되어야 그 결과를 검토하면, 세포 분리에 대해이 절차는 여러 studie에 사용되었습니다 비록 그s이 (현미경, 전기 생리학, 등), 격리 자체는 잠재적으로 유전자 발현에 영향을 수 있습니다. 생물 학적 샘플을 조작하는 방법과 마찬가지로, 당신의 결과의 결과를주의 깊게 단세포 표현은 조직 자체 기술하지 편견 대표하기 위해 검증해야합니다. 이러한 현장 하이브 리다이 제이션에서와 같이 방법은 손상 조직에서 이러한 결과를 확인하기 위해 유용합니다. 마지막으로, 그것은 생성된 데이터가 신중하게 품질 관리를위한 체크되어 있는지 확인하는 것이 중요합니다. 그림 8에 표시된 데이터는 qPCR의 assays 이러한 분석 # 13 (A13)와 같은, 그들의 특이성이 매우 강력한하거나 그러한 분석 # 34 (A34)와 같은 기술적인 변화로 이어질 수있는 다양성의 높은 수준을 가지고 수 있습니다 보여줍니다.
공개
무료 접속이 생산은 시그마 - 올드 리치를 후원합니다.
감사의 말
저자는이 프로토콜의 촬영 기간 동안 C. Zambataro의 기술 지원을 인정하고 싶습니다. 우리는 기꺼이 글렌 네이선 충격 센터 상을 의학 연구 (SM), Hillblom 재단과 국립 보건원에 대한 재단 (P30AG025708)와 PO1AG025901의 지원을 인정합니다. JMF는 노화 연구에 대한 책임 연구원에게 수여 T32AG000266에 의해 지원되었다.
자료
| Name | Company | Catalog Number | Comments |
| 시약의 이름 | 회사 | 카탈로그 번호 | 댓글 |
| NaCl | 시그마 - 올드 리치 | 71,378 | |
| KCl | 시그마 - 올드 리치 | 60,128 | |
| Pyruvic 산 | 시그마 - 올드 리치 | P4562 | |
| Hepes | 시그마 - 올드 리치 | 54,457 | |
| MgCl 2 | 시그마 - 올드 리치 | M1020 | |
| 아니 2 PO 4 일염기의 | 시그마 - 올드 리치 | P9791 | |
| 우선당 | 시그마 - 올드 리치 | G6721 | |
| NaOH | 시그마 - 올드 리치 | 72,068 | |
| EGTA | 시그마 - 올드 리치 | O3777 | |
| CaCl 2 | 시그마 - 올드 리치 | 21,115 | |
| 단백 분해 효소, 유형 XIV | 시그마 - 올드 리치 | P5147 | |
| Collagenase, 전을 입력합니다 | 워싱턴 | C9891 | |
| 1X의 DNA 서스펜션 버퍼 (10 MM 트리스, 산도 8.0, 0.1 MM EDTA (에틸렌 다이아 민 테트라 초산)) | TEKnova, | PN T0221 | |
| BSA | 시그마 - 올드 리치 | B8667 | |
| Pentobarbital 나트륨 | 헨리 Schein의 투어뇌려. 공급 * | 주문에 대한 공급 업체에 문의하십시오 | *는 수의사 또는 동물 전문 연구소를 통해 procured되어야합니다 |
| IT를 ExoSAP | Affymetrix / USB | 78,201 | |
| 2X Rxn 믹스를 CellsDirect | Invitrogen | 11737-030 | |
| 윗첨자 III RT 플래티넘 DNA 형성 촉매 믹스 | Invitrogen | 10928-042 | |
| RT - STA의 뇌관 믹스 | IDT | 사용자 정의 | |
| Nuclease 무료 물 | 시그마 - 올드 리치 | W4502 | |
| WTA2 | 시그마 - 올드 리치 | WTA2 - 50rxn | |
| Qiaquick PCR 정화 키트 | Qiagen | 28,104 | |
| Qiacube | Qiagen | 9,001,292 | |
| NanoDrop 분광 광도계 | NanoDrop | 2000c | |
| BioAnalyzer DNA 7500 키트 | 애질런트 | 7500 키트 | |
| 한 컬러 라벨링 키트 | 호슈 - NimbleGen | 5223555001 | |
| 뮤스 Musculus 12x135k 어레이 | 호슈 - NimbleGen | 5543797001 | |
| GenePix 4200A 스캐너 | 분자 장치 | 4200A | |
| TransPlex 완전한 전체 Transcriptome 증폭 키트 (WTA2 키트) | 시그마 - 올드 리치 | WTA2 - 50RXN |
참고문헌
- Wolska, B. M., Solaro, R. J. Method for isolation of adult mouse cardiac myocytes for studies of contraction and microfluorimetry. Am. J. Physiol. 271 (3 Pt 2), H1250-H1255 (1996).
- Shioya, T. A simple technique for isolating healthy heart cells from mouse models. J. Physiol. Sci. 57 (6), 327-335 (2007).
- Guo, G., Huss, M., Tong, G. Q., Wang, C., Li Sun, L., Clarke, N. D., Robson, P. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Dev. Cell. 18 (4), 675-685 (2010).
- Narsinh, K. H., Sun, N., Sanchez-Freire, V., Lee, A. S., Almeida, P., Hu, S., Jan, T., Wilson, K. D., Leong, D., Rosenberg, J., Yao, M., Robbins, R. C., Wu, J. C. Single cell transcriptional profiling reveals heterogeneity of human induced pluripotent stem cells. J. Clin. Invest. 121 (3), 1217-1221 (2011).
- D'haene, B., Vandesompele, J., Hellemans, J. Accurate and objective copy number profiling using real-time quantitative PCR. Methods. 50 (4), 262-270 (2010).
- Cook, M. J. The Anatomy of the Laboratory Mouse. Informatics. , (1965).
- Gaillard, F. Aorta. Radiopaedia. , (2008).
- Ståhlberg, A., Andersson, D., Aurelius, J., Faiz, M., Pekna, M., Kubista, M., Pekny, M. Defining cell populations with single-cell gene expression profiling: correlations and identification of astrocyte subpopulations. Nucleic Acids Res. 39 (4), e24-e24 (2011).
- Zhong, J. F., Chen, Y., Marcus, J. S., Scherer, A., Quake, S. R., Taylor, C. R., Weiner, L. P. A microfluidic processor for gene expression profiling of single human embryonic stem cells. Lab Chip. 8 (1), 68-74 (2008).
- Bahar, R., Hartmann, C. H., Rodriguez, K. A., Denny, A. D., Busuttil, R. A., Dollé, M. E., Calder, R. B., Chisholm, G. B., Pollock, B. H., Klein, C. A., Vijg, J. Increased cell-to-cell variation in gene expression in ageing mouse heart. Nature. 441 (7096), 1011-1014 (2006).
- Raj, A., van Oudenaarden, A. Nature, nurture, or chance: stochastic gene expression and its consequences. Cell. 135 (2), 216-226 (2008).
- Elowitz, M., Levine, A., Siggia, E., Swain, P. Stochastic gene expression in a single cell. Science. 297 (5584), 1183-1186 (2002).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유