Method Article
Profilage Single Cell transcriptionnelle des cardiomyocytes de souris adultes
Dans cet article
Résumé
Simple cellule de profilage de l'expression génique permet l'analyse détaillée d'expression des cellules individuelles. Nous décrivons des méthodes pour l'isolement des cardiomyocytes, et la préparation des lysats résultant soit pour tout ou biopuces transcriptome qPCR d'objectifs spécifiques.
Résumé
Alors que de nombreuses études ont examiné les changements d'expression génique à partir des homogénats de tissu cardiaque, ce qui empêche l'étude de la variation stochastique inhérente entre les cellules dans un tissu. L'isolement des populations pures cardiomyocyte à travers une perfusion de collagénase des cœurs de souris facilite la génération de puces à cellule unique pour l'expression génique toute transcriptome, ou qPCR d'objectifs spécifiques en utilisant des tableaux nanofluidique. Nous décrivons ici une procédure pour examiner les profils de simples cellules d'expression génétique des cardiomyocytes isolés du coeur. Ce paradigme permet l'évaluation des mesures d'intérêt qui ne sont pas dépendants de la moyenne (par exemple pour la variance entre les cellules dans un tissu) qui n'est pas possible lors de l'utilisation traditionnelle des workflows tissu entier pour l'évaluation de l'expression génique (figure 1). Nous avons réalisé l'amplification robuste du transcriptome des cellules microgrammes seule cession d'ADNc double brin qui facilite l'utilisation de puces sur les iertaines cellules. Dans la procédure que nous décrivons l'utilisation de tableaux de NimbleGen, qui ont été sélectionnés pour leur facilité d'utilisation et de possibilité de personnaliser leur conception. Alternativement, une transcriptase inverse - amplification de la cible spécifiques (RT-STA) de réaction, permet de qPCR de centaines de cibles en nanofluidique PCR. En utilisant une de ces approches, il est possible d'examiner la variabilité d'expression entre les cellules, ainsi que l'examen des profils d'expression de types de cellules rares à partir d'un tissu. Globalement, l'approche cellule unique permet l'expression des gènes à la génération de données qui peuvent permettre de définir des profils d'expression idiosyncrasique qui sont généralement en moyenne à l'examen d'expression de millions de cellules à partir d'homogénats typiques générés à partir des tissus ensemble.
Protocole
1. Étape 1 - L'isolement des cardiomyocytes
- Préparer le tampon de digestion comme suit:
10 x tampon de digestion (made in ultra-pure de H 2 O)Conc Finale. g / L g/500 ml Solution de 10x g / L NaCl 130 mM 7,597 3.3 75,97 KCl 5 mM 0.4 0.2 4,0 L'acide pyruvique 3 nM 0,33 0,165 3,30 HEPES 25 mM 5,96 2,85 59,6 MgCl 2 0,5 mM 0,101 0,05 1,01 NaH 2PO 4monobasic 0,33 mM 0,04 0,02 0,40 Dextrose 22 mM 3,96 1,98 39,6
tampon pH à 7,4 avec NaOH - Utiliser le tampon de digestion 10x, faire les quatre solutions suivantes pour chaque cœur qui sera perfusé (made in ultra-pure H 2 O):
Un tampon de digestion (Faire trois aliquotes de cette solution)
- 50 ml - 1x tampon de digestion contenant:
- 75 uL - solution de l'EGTA (100 mM EGTA)
Tampon de digestion B (faire un de ces)
- 25 ml - 1x tampon de digestion contenant:
- 1,25 uL - solution de CaCl 2 (1 M)
- 1 mg - Protéase
- 25 mg - collagénase (can utilisent aussi 75% de ce montant = 18,75 mg) **
Collection Tampon (faire un de ces)
- 2,5 ml - 1x tampon de digestion contenant:
- 1,25 uL - solution de CaCl 2 (1 M)
- 0,5 mg - Protéase
- 15 mg - collagénase (75% = 11,25 mg) **
- 125 mg - BSA
Tampon de lavage de neutralisation (faire un de ces)
- 25 ml - 1x tampon de digestion contenant:
- 6,25 uL - solution de CaCl 2 (1 M)
- 250 mg de BSA
** Selon la source et le lot de la collagénase, l'activité des enzymes peut varier. Il peut être nécessaire pour optimiser la quantité d'enzyme ajoutée à cette zone tampon pour empêcher une digestion.
- Mettre en place le système d'isolation tel que précédemment décrit dans Détail 1,2 avec trois béchers 15 ml de tampon de digestion A sur la glace, un tube de 50 ml de tampon de digestion A et le B 25 ml de tampon de digestion prêt pour perfusion (Un tampon de digestion doit être rincé à travers la ligne, au minimum de 5 ml passent par le système), Buffer Collection réchauffé dans un bain d'eau pour élever la température à 37 ° C, et le tampon de lavage de neutralisation à la température ambiante.
- Remplissez la canule de perfusion et le plat de dissection où le cœur sera nettoyé et attaché sur la pointe de perfusion avec A. Digestion tampon froid Librement noeud des sutures (4-O à la taille de la 6-O) autour de la partie supérieure de la canule. Ce sera ensuite utilisée pour maintenir l'aorte afin de prévenir le cœur glisser la canule.
- Terminale endormir la souris avec une injection IP de l'agent anesthésiant (0,25 ml solution de sodium pentobarbital) et s'assurer que la souris est inconsciente et insensible aux stimuli douloureux. Couper la cavité thoracique soigneusement le long de la marge thoraciques en coupant le diaphragme(Figure 2). Coupez le haut et supérieurement refléter la cage thoracique antérieure (ces incisions sont désignés B et A, respectivement à la Figure 2) Critique -. La souris doit être encore respirer avant que ces incisions.
- Utiliser une coupe de pipette en plastique à la taille, doucement sucer le cœur dans la pipette. Couper le coeur de la cavité thoracique en prenant soin de laisser suffisamment de l'aorte dans le coeur d'identifier clairement la branche aortique (figure 3). Une pipette est utilisée car elle provoque des dommages minimes au cœur.
- Laissez tomber le cœur dans un bécher de tampon de digestion de la glace froide et rincez A le sang loin. Après 15 à 45 sec placer au cœur dans le second bécher de tampon de digestion glacée Un brièvement rincer de nouveau. Transfert du coeur dans la boîte de Pétri aussi rempli avec A. Digestion Tampon à froid
- Voir le coeur dans le plat de tampon de digestion A sous le champ de dissection et soigneusement isoler les aorte et ses branches. Soigneusement garniture peine inférieure à la dernière Branch (figure 4).
- Monter le extrémité de la canule dans l'aorte et ligaturer en place avec la 4-O à la 6-O de suture à l'aide de la configuration scénique sous un microscope de dissection. Assurez-vous que la canule ne pas s'asseoir trop bas dans l'aorte et reste au-dessus des artères coronaires (figure 4).
- Avec un tampon de digestion qui coule à travers la mise en place de perfusion, placez la pointe de perfusion à l'installation, et commencer à perfuser le cœur jusqu'à la majorité du sang est lavé par le cœur et fluide sortant du coeur est clair. Critique - la température du liquide de perfusion doit être aussi proche que possible de 37 ° C quand il sort du cathéter. Si la solution est trop chaude ou trop froide, puis la perfusion va tuer le tissu. Le temps jusqu'à ce que la perfusion du cœur est aussi critique. La fenêtre de temps doit être inférieure à 5-10 min de l'ablation du cœur jusqu'à la perfusion commence. Le cœur doit être retiré sous anesthésie, pas de CO 2 apposition ou la dislocation cervicale.
- Une fois que le sang est ahed du cœur, passez à la B tampon de digestion de l'A. tampon de digestion S'assurer que le tampon de digestion B est désormais circule dans le coeur. La solution va commencer à avoir une teinte jaune. Attendre 2-3 min jusqu'à ce que le coeur donne des signes de perdre sa forme et commence à regarder arrondie qui est une indication que la digestion est efficace. Le myocarde devrait apparaître plus léger que le produit de perfusion.
- Couper les ventricules avec des ciseaux et placer dans un tube conique de 50 ml contenant 15 ml de tampon Collection à 37 ° C. Doucement coupé le ventricule en petits morceaux tout en plaçant le tissu dans le tampon Collection. Mélanger doucement avec une pipette en plastique à dissoudre les adhérences cellule-cellule et faire un mélange seule cellule cardiomyocytes.
- Quand la plupart des tissus est dissoute, (moins de 1 min) laisser les morceaux de tissu plus couler au fond. Retirez et conservez le surnageant dans un tube de 15 ml conique. Laissez la gravité tirez une pastille à partir du surnageant de 10 à 20 min (15min de préférence) à température ambiante.
- Retirer et jeter le surnageant. Laver le culot en ajoutant 5 ml de tampon de lavage de neutralisation au culot et mélanger délicatement les cellules pour les remettre en suspension. A ce stade, les cellules sont immédiatement prêts à être sélectionnés pour l'analyse cellulaire unique (passez à l'étape 2).
2. Étape 2 - L'isolement des cellules individuelles
- Préparer un micromanipulateur l'aide d'une flamme et un tube capillaire en verre qui sera utilisé pour manipuler des cellules. Le micromanipulateur est tout simplement un tube très fin capillaire qui est alors attaché à un long morceau de tuyau flexible qui peut se déplacer ou ramasser les cellules lorsque l'aspiration est appliquée.
- Procéder immédiatement à la sélection des cellules individuelles au microscope (Nikon Eclipse TS100, 20x) à l'aide d'un micromanipulateur qui a été créé avec une pipette tiré et un sas pour empêcher les matériaux d'être transféré à l'échantillon de cellules échantillons individuels. Assurez-vous que la population cellulaire est sain(70% viable) et non endommagé par le processus d'isolement.
- Diluer les cellules dans une petite boîte de Petri afin qu'ils puissent être sélectionnés individuellement. Lorsque les cellules picking être sûr de capter les cellules dans un petit volume (moins de 2 uL). Sélectionner uniquement les cellules saines qui ont la morphologie caractéristique normale des cardiomyocytes (figure 5).
- Placer des cellules individuelles dans le fond de tubes de PCR individuels et les congeler immédiatement sur la glace sèche. Ces cellules sont ensuite stockés à -80 ° C jusqu'à ce qu'ils soient utilisés pour l'analyse d'expression génique.
3. Étape 3A - Simple d'expression génique de cellules qPCR
Pour effectuer qPCR sur les cellules individuelles employer un protocole adapté de celui développé pour l'expression génique des cellules unique par Fluidigm Corporation pour leur tableaux Biomark qPCR nanofluidique (Fluidigm - Protocole développement Advance # 30) 3,4.
- Préparation de la transcription inverse - amplification de la cible spécifique (RT-STA). Pour êtrele gin, la suspension de toutes les paires d'amorces (nous avons utilisé jusqu'à 125 paires d'amorces avec succès) pour les gènes d'intérêt à 100 uM dans un tampon de l'ADN de suspension 1x (10 mM Tris, pH 8,0, EDTA 0,1 mM). Mélanger et diluer les paires d'amorces avant et arrière à 20 pM pour chaque dosage. Enfin, diluer toutes les paires d'amorces dans une solution avec une concentration finale de 200 nM pour chaque dosage qui sera amplifié dans la réaction de RT-STA. Pour ce faire, prendre chaque paire d'amorces et de les diluer 1:100. Par exemple, si vous avez 30 paires d'amorces de dosage à 20 uM, vous devrez ajouter 1 ul de chaque paire (30 au total uL), et ensuite faire le reste du volume avec 70 ul de tampon de suspension d'ADN pour atteindre la finale le volume de dilution de 100 mL.
- Maintenant que les solutions d'amorces sont préparées, assembler le mix RT-STA réaction maître. Préparez assez master mix pour chacune des cellules individuelles que vous souhaitez amplifier.
Tableau 1. RT-STA master mixRéactif Volume de ul (par réaction) CellsDirect mélange réactionnel 2x 5,0 SuperScript III RT Plantinum Taq mélangez 0.2 Mélange d'amorces de RT-STA (200 nM de chaque dosage) 2.5 Nucléase libre H 2 O (ou 1x tampon de suspension ADN) 1.3 Total des 9,0 - Retirer immédiatement les cellules congelées et centrifuger au maximum RCF pendant 30 secondes pour s'assurer que les cellules sont placées au fond du tube. Ajouter 9 uL de RT-STA mélange pour le tube et de procéder à la réaction dans un thermocycleur.
- 50 ° C - 15 minutes
- 95 ° C - 2 minutes
- 18 cycles de
- 95 ° C pendant 15 secondes
- 60° C pendant 4 minutes
- Tenir à 4 ° C
- Retirer les tubes de l'appareil PCR et ajouter 4 ul de ExoSAP-IT pour la réaction. ExoSAP-IT est un réactif nettoyage de PCR qui permettra d'éliminer l'excès et amorces enzyme de la réaction de RT-STA. Exécuter les tubes à travers la réaction suivante dans une machine à PCR.
- 37 ° C - 15 minutes
- 80 ° C - 15 minutes
- Tenir à 4 ° C
- Lorsque le traitement ExoSAP-IT est complète; diluer les échantillons avec un tampon de 01h05 suspension de l'ADN. Les échantillons sont maintenant prêts pour analyse qPCR. Nous utilisons ce matériel en conjonction avec les tableaux Gene Fluidigm qPCR expression (48.48, 96.96 ou plaques). Il est également possible d'utiliser cette approche avec une instrumentation plus traditionnelle qPCR (96 puits ou 384 formats bien). Toutefois, ces instruments nécessitent généralement l'entrée d'échantillon beaucoup plus ce qui limite le nombre de tests qui peuvent être performed d'une seule cellule.
3. Étape 3B - amplification du transcriptome entier et microarray des cellules individuelles
Extraction et l'amplification
- Utilisez Sigma WTA2 kit (Cat # WTA2) pour amplifier ADNc double brin à la fois de boulettes et des cellules individuelles. Les cellules individuelles sont "extraites" via la première étape du protocole Sigma WTA2 conformément aux instructions du fabricant. Durant cette première étape de l'incubation avec le tampon de synthèse de bibliothèques à 37 ° C pendant 5 minutes perturbe la membrane de la cellule individuelle et de "extraits" du message pour l'amplification.
- Amplifier des cellules individuelles en deux tours, en utilisant les instructions du kit WTA2 Sigma-Aldrich. Effectuez l'étape de préparation bibliothèque selon le protocole du fabricant, puis ajouter 1 / 5 de l'échantillon bibliothèque à 70 pi de l'amplification WTA2
Mix Master. Amplifier les échantillons pendant 25 cycles en utilisant les paramètres de cycle recommandée. - Purifier l'USI échantillonsng Qiagen QIAquick PCR Purification (Cat # 28104) et processus sur QIAcube Qiagen en utilisant le "nettoyage Réactions QIAquick amplification par PCR standard V4" protocole.
- Concentrer les échantillons purifiés à 5 ul, en utilisant un aspirateur vitesse, et soumis à un second tour de l'amplification de 17 cycles (WTA2 protocole d'amplification et de paramètres de cycles répétés).
- Purifier échantillons en utilisant le kit QIAquick PCR Purification et vérifier la qualité de l'aide d'un spectrophotomètre NanoDrop et Agilent BioAnalyzer DNA kit 7500 conformément au protocole du fabricant.
Étiquetage et tableaux NimbleGen l'expression des gènes
- Etiquette 2 pg d'ADNc double brin de chaque échantillon de cellules amplifiées en utilisant NimbleGen Un kit de marquage de couleur (Cat # 05223555001) selon le protocole du fabricant.
- Hybrider 5 ug d'échantillon marqué au Cy3 Mus musculus 12x135k, tableaux NimbleGen l'expression des gènes (Cat # 05543797001) selonOrding au protocole du fabricant. Après l'hybridation des échantillons à des tableaux, les lames doivent ensuite être lavés et séchés avant qu'ils puissent être reproduits sur un scanner laser.
- Les tableaux de numérisation expression des gènes en utilisant un scanner laser. Ici, nous utilisons une Molecular Devices GenePix 4200A Scanner avec des réglages de 100POW, et 300-350PMT. Puis générer les fichiers de paire pour chaque tableau en utilisant le logiciel NimbleScan NimbleGen et ensuite d'analyser l'expression du transcriptome l'ensemble de chaque tableau, seule cellule (un tableau de type exprimée de façon stable les transcriptions des cardiomyocytes est inclus en tant que supplément; tableau S1).
Si la perfusion cardiaque fonctionnait bien il devrait y avoir un pourcentage élevé de cellules cardiaques saines qui conservent leur morphologie typique rectangulaire sur l'isolement. Si la perfusion ne procède pas ainsi alors il y aura un grand pourcentage de cellules mortes (voir les images dans la Figure 5). Si les cellules sont correctement amplifié en utilisant soit des méthodes WTA2 ou RT-STA dun les produits finis devrait passer des tests ultérieurs de contrôle qualité pour la qualité des échantillons et de l'ARNm par NanoDrop Bioanalyser (figure 6). Pour le workflow microréseau, cela est terminé après amplification WTA où l'ARNm est testé par deux NanoDrop et bioanalyseur. Représentant des résultats positifs pour cette analyse sont présentés dans la figure 6. Le WTA2 échantillons devraient montrer une amplification à la fois robuste avec la lecture spectrophotomètre NanoDrop, ainsi que la lecture de l'électrophérogramme Bioanalyzer (BA) à puce (figure 6). Une table de gènes qui ont été détectés par stablement microréseau de cellules uniques est inclus comme un exemple (tableau S1). Pour le processus de qPCR, la qualité des données peut être évaluée en effectuant une étape de fusion à la fin de la réaction de PCR pour s'assurer que les ensembles d'amorces amplifient le produit désiré (figure 7). Si elle est correcte, cette étape de fusion devrait générer un pic spécifique fondre. Pour illustrer ce point, un sous-ensemble de données nanofluidique qPCR est représenté dans une heatmap du pic de fusion température (figure 8) 8. Il est possible d'évaluer la qualité d'un produit de PCR par sa température de fusion afin d'assurer l'absence de produits non spécifiques 5. Chaque ligne de cette carte représente un échantillon de cellules (S1-S40) testés dans 43 essais qPCR séparé (A1-A43). Dans cette figure, il est clair que certains dosages sont très variables et sont probablement moins fiables que les tests plus stable avec une spécificité plus élevée PCR.
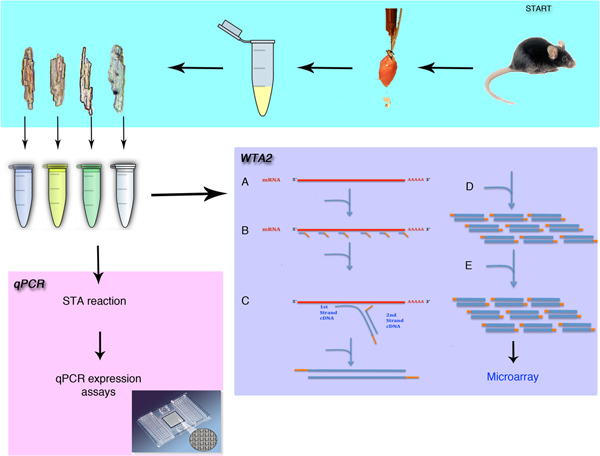
Figure 1. Vue d'ensemble de l'isolement cellulaire unique et d'analyse d'expression génique. La procédure sera la preuve de l'ablation chirurgicale du cœur de la souris et l'isolement des cardiomyocytes individuels. Les méthodes présentées dans cette procédure sont les méthodes de qPCR ou soit l'analyse des microréseaux après amplification du transcriptome entier (WTA). La procédure commence par la WTA lyse (A), puis la liaison des amorces universelles de Sigma (B) à l'ARNmpiscine. L'extension de ces amorces (C) et l'amplification (D) phase de création microgrammes de matériel amplifié. Cette amplification est ensuite répété (e) pour générer suffisamment de matériel pour la procédure de biopuces.

Figure 2. Représentation de l'emplacement de l'incision pour enlever le cœur de la souris. Couper le long de la paroi latérale de la cage thoracique (A), puis coupez le long de la marge inférieure de la cage thoracique (B). Couper les vaisseaux au-dessus et en dessous du coeur permettent de retrait, tout en laissant assez de l'aorte à cathétériser le cœur. Images adaptées de MJ Cook, 6.
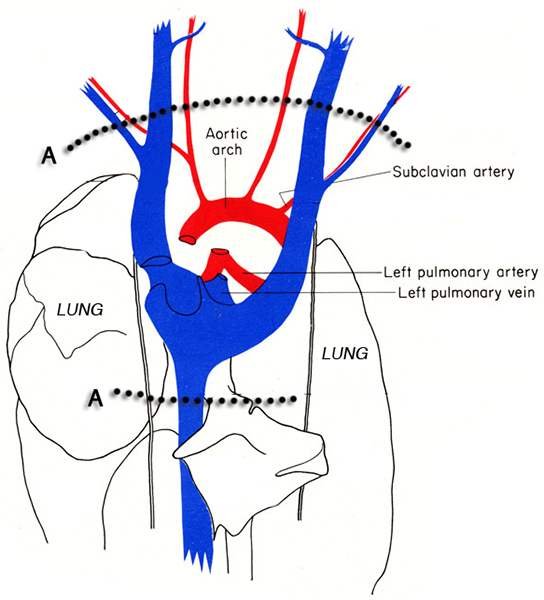
Figure 3. Schéma du thorax de la souris. Les lignes pointillées indiquent les emplacements des incisions (A) pour exciser le cœur de la cavité thoracique, sans endommager l'h tissus eart. Images adaptées de MJ Cook, 6.
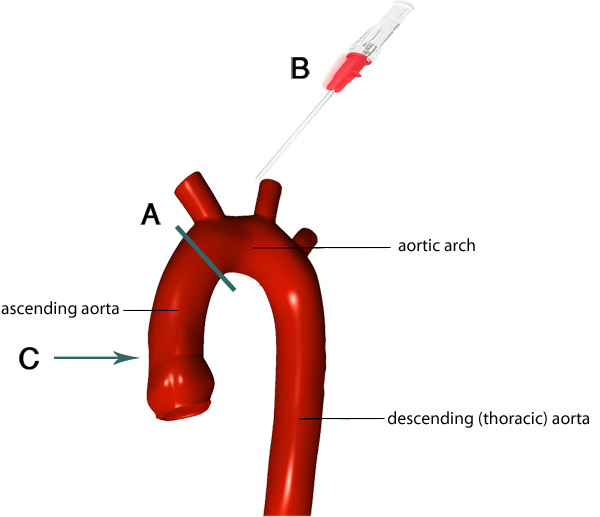
Figure 4. Image de l'arc aortique et de ses branches. La ligne grise indique l'emplacement idéal d'où couper l'aorte (A). L'aorte en restant attaché à cœur est canulée de sorte que l'extrémité de la canule (B) passe au niveau approprié dans l'aorte (C). Images adapté de F. Gaillard 7.

Figure 5. Sélection de cellules uniques pour l'analyse de l'expression génique. Chaque panneau montre cardiomyocytes imagé au microscope optique montrant la morphologie typique des cardiomyocytes. Cardiomyocytes saines sont indiquées par les flèches vertes. Les cellules qui sont morts ou mourants sont indiquées par des flèches rouges. Ces cellules mortes ne doivent pas être utilisées dans l'analyse.
iles/ftp_upload/3302/3302fig6.jpg "alt =" Figure 6 "/>
Figure 6. Contrôle de la qualité des résultats WTA pour l'amplification seule cellule. (A) Image d'un spectrophotomètre NanoDrop lecture qui est approprié pour les transcriptions amplifié à partir de la réaction du circuit WTA. (B) La lecture de la puce BioAnalyzer montre les résultats de trois cellules amplifiées.
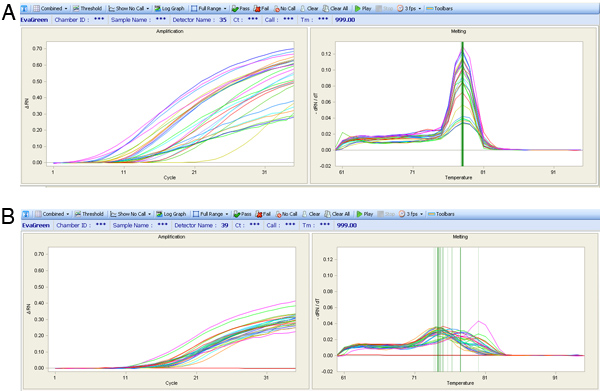
Figure 7. Courbes d'amplification PCR quantitative (graphique de gauche) et pic de fusion des courbes de température (graphique de droite). (A) Les résultats d'expression à partir d'un test de qualité sont présentés avec un seul pic fondre en dépit des courbes d'amplification différents. (B) Faites fondre les courbes d'un ensemble d'amorces pauvres, ce qui a des pics fondre très variable qui n'est pas idéal pour l'analyse de l'expression.
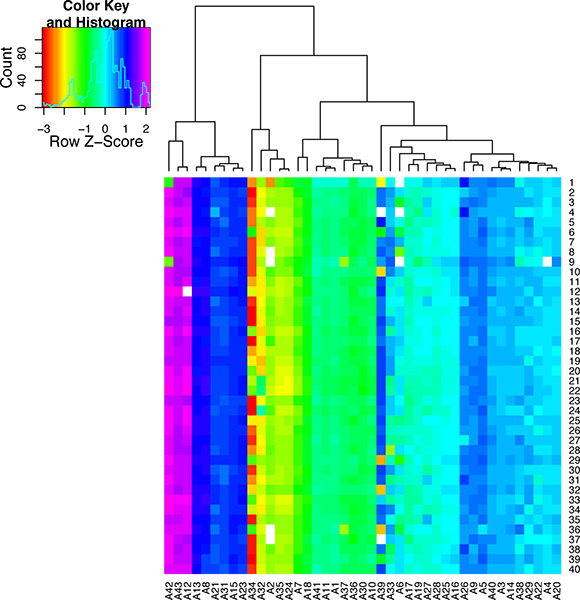
Résultats de la figure 8. Contrôle de la qualité pour la seule cellule de qPCR réagissentions. Cette carte de la chaleur a été créé pour afficher les températures de fusion comme une échelle de couleurs. La température de fusion de pointe pour chaque dosage qPCR (A1-A43) est indiqué pour 40 cellules individuelles (S1-40). Les tests ont été regroupés en fonction de leur température de fusion. En regardant vers le bas de la colonne pour chaque dosage, il est possible de voir la variation de fondre sur l'ensemble des échantillons. Ce chiffre démontre que certains tests qPCR sont très spécifiques tandis que d'autres sont très variables et ne sont donc pas adapté à l'analyse unicellulaire.
Tableau S1. Tableau complémentaire des données de biopuces. Ces gènes sont répertoriés en fonction de la robustesse dans lequel ils sont détectés dans les cardiomyocytes unique à travers une vaste base de données. Cette liste de gènes indique la sensibilité de détection pour un certain nombre de gènes qui sont généralement exprimés en cardiomyocytes unique.
Discussion
Cette méthode a la possibilité de générer des cardiomyocytes pour un certain nombre de fonctions ainsi que les études d'expression génique. L'examen des cellules individuelles est une région en plein essor dans l'analyse de l'expression génique. L'avantage de l'examen de niveau transcriptionnel au niveau de la cellule unique est qu'il permet l'examen d'une population cellulaire pure, qui n'est pas possible à partir des préparations de tissus ensemble. De plus, l'analyse seule cellule permet à l'examen de la variation stochastique des taux d'ARNm dans des cellules individuelles pour définir les populations de cellules qui étaient auparavant considérés comme homogènes 8,9. En plus d'identifier les gènes qui sont potentiellement stochastique dans leur expression des gènes 10,11,12, la méthode permet aussi l'identification des populations de cellules rares définies par leurs profils d'expression génique.
Bien que cette méthode fournit un certain nombre de passionnantes utilisations potentielles dans l'analyse de l'expression des gènes, il ya des some mises en garde et considérations dans l'utilisation des cellules individuelles. La principale limitation à l'analyse unicellulaire, est la collecte de suffisamment de cellules dans l'expérience pour atteindre une signification statistique en ce qui concerne la métrique d'intérêt, de la variance par exemple. Dans le cas où le nombre de cellules isolées à partir du tissu d'intérêt n'est pas une limitation, on peut examiner des centaines de cellules par groupe, en utilisant des approches telles que séquençage de nouvelle génération, ou des tableaux nanofluidique. Toutefois, dans certains cas, il peut y avoir des difficultés à obtenir suffisamment de cellules du tissu d'intérêt. Cela peut être en partie causée par le temps des procédures idiosyncratiques intensif pour la collection du type de cellules ciblées. Cependant, une planification minutieuse et la préparation avant de procéder à la collecte seule cellule peut encore permettre une analyse rigoureuse de cellule unique avec une erreur technique limitée. Lorsque l'on examine les résultats, il doit être considéré que, même si cette procédure pour isoler les cellules a été utilisé dans de nombreux Studies (y compris la microscopie, l'électrophysiologie, etc), l'isolement se pourrait potentiellement effet l'expression des gènes. Comme avec n'importe quelle méthode qui manipule des échantillons biologiques, les résultats de vos résultats devraient être soigneusement validé pour garantir l'expression cellule unique est représentatif du tissu lui-même et un biais non pas technique. Des méthodes telles que l'hybridation in situ peut s'avérer utile de vérifier ces résultats dans le tissu intact. Enfin, il est essentiel de s'assurer que les données générées sont soigneusement vérifiées pour le contrôle qualité. Les données présentées dans la figure 8 montre que les dosages qPCR peut être très robustes dans leur spécificité, comme test n ° 13 (A13) ou ont un niveau élevé de variabilité qui peut conduire à la variance techniques telles que le dosage n ° 34 (A34).
Déclarations de divulgation
Accès libre et la production de cette de cet article est sponsorisé Sigma-Aldrich.
Remerciements
Les auteurs tiennent à remercier l'assistance technique de C. Zambataro pendant le tournage de ce protocole. Nous remercions chaleureusement le soutien de la Fondation Glenn pour la recherche médicale (SM), la fondation Hillblom, et le National Institutes of Health pour un prix de Nathan Centre Choc (P30AG025708) et PO1AG025901. JMF a été soutenue par T32AG000266 décerné à l'Institut Buck pour la recherche sur le vieillissement.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Société | Numéro de catalogue | Commentaires |
| NaCl | Sigma-Aldrich | 71378 | |
| KCl | Sigma-Aldrich | 60128 | |
| L'acide pyruvique | Sigma-Aldrich | P4562 | |
| Hepes | Sigma-Aldrich | 54457 | |
| MgCl 2 | Sigma-Aldrich | M1020 | |
| NaH 2 PO 4 monobasique | Sigma-Aldrich | P9791 | |
| Dextrose | Sigma-Aldrich | G6721 | |
| NaOH | Sigma-Aldrich | 72068 | |
| EGTA | Sigma-Aldrich | O3777 | |
| CaCl 2 | Sigma-Aldrich | 21115 | |
| Protéase, type XIV | Sigma-Aldrich | P5147 | |
| Collaoxygénase, Type I | Worthington | C9891 | |
| Tampon 1x suspension l'ADN (10 mM Tris, pH 8,0, EDTA 0,1 mM) | TEKnova, | PN T0221 | |
| BSA | Sigma-Aldrich | B8667 | |
| Pentobarbital sodique | Henry Schein Vet. * Alimentation | Contacter avec le fournisseur pour commander | * Doit être procuré par un vétérinaire ou un laboratoire animalier professionnel |
| IT ExoSAP | Affymetrix / USB | 78201 | |
| CellsDirect 2x Mix Rxn | Invitrogen | 11737-030 | |
| SuperScript III Platinum Taq Mix RT | Invitrogen | 10928-042 | |
| Mélange d'amorces de RT-STA | IDT | Personnalisé | |
| Nucléase l'eau libre | Sigma-Aldrich | W4502 | |
| WTA2 | Sigma-Aldrich | WTA2-50rxn | |
| Kit QIAquick PCR Purification | Qiagen | 28104 | |
| QIAcube | Qiagen | 9001292 | |
| Spectrophotomètre NanoDrop | NanoDrop | 2000c | |
| BioAnalyzer DNA kit 7500 | Agilent | 7500 Kit | |
| Un kit de marquage couleur | Roche-NimbleGen | 5223555001 | |
| Mus musculus 12x135k array | Roche-NimbleGen | 5543797001 | |
| GenePix 4200A Scanner | Molecular Devices | 4200A | |
| Transplex Complete kit complet d'amplification du transcriptome (WTA2 Kit) | Sigma-Aldrich | WTA2-50RXN |
Références
- Wolska, B. M., Solaro, R. J. Method for isolation of adult mouse cardiac myocytes for studies of contraction and microfluorimetry. Am. J. Physiol. 271 (3 Pt 2), H1250-H1255 (1996).
- Shioya, T. A simple technique for isolating healthy heart cells from mouse models. J. Physiol. Sci. 57 (6), 327-335 (2007).
- Guo, G., Huss, M., Tong, G. Q., Wang, C., Li Sun, L., Clarke, N. D., Robson, P. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Dev. Cell. 18 (4), 675-685 (2010).
- Narsinh, K. H., Sun, N., Sanchez-Freire, V., Lee, A. S., Almeida, P., Hu, S., Jan, T., Wilson, K. D., Leong, D., Rosenberg, J., Yao, M., Robbins, R. C., Wu, J. C. Single cell transcriptional profiling reveals heterogeneity of human induced pluripotent stem cells. J. Clin. Invest. 121 (3), 1217-1221 (2011).
- D'haene, B., Vandesompele, J., Hellemans, J. Accurate and objective copy number profiling using real-time quantitative PCR. Methods. 50 (4), 262-270 (2010).
- Cook, M. J. The Anatomy of the Laboratory Mouse. Informatics. , (1965).
- Gaillard, F. Aorta. Radiopaedia. , (2008).
- Ståhlberg, A., Andersson, D., Aurelius, J., Faiz, M., Pekna, M., Kubista, M., Pekny, M. Defining cell populations with single-cell gene expression profiling: correlations and identification of astrocyte subpopulations. Nucleic Acids Res. 39 (4), e24-e24 (2011).
- Zhong, J. F., Chen, Y., Marcus, J. S., Scherer, A., Quake, S. R., Taylor, C. R., Weiner, L. P. A microfluidic processor for gene expression profiling of single human embryonic stem cells. Lab Chip. 8 (1), 68-74 (2008).
- Bahar, R., Hartmann, C. H., Rodriguez, K. A., Denny, A. D., Busuttil, R. A., Dollé, M. E., Calder, R. B., Chisholm, G. B., Pollock, B. H., Klein, C. A., Vijg, J. Increased cell-to-cell variation in gene expression in ageing mouse heart. Nature. 441 (7096), 1011-1014 (2006).
- Raj, A., van Oudenaarden, A. Nature, nurture, or chance: stochastic gene expression and its consequences. Cell. 135 (2), 216-226 (2008).
- Elowitz, M., Levine, A., Siggia, E., Swain, P. Stochastic gene expression in a single cell. Science. 297 (5584), 1183-1186 (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon