Method Article
Single Cell Transcriptional Profiling der erwachsenen Maus Kardiomyozyten
In diesem Artikel
Zusammenfassung
Einzel-Zell-Expression Profiling ermöglicht die detaillierte Genexpressionsanalyse einzelner Zellen. Wir beschreiben Methoden zur Isolierung von Kardiomyozyten, und die Vorbereitung der daraus resultierenden Lysate entweder für ganze Transkriptom-Mikroarray oder qPCR der spezifischen Ziele.
Zusammenfassung
Während zahlreiche Studien Veränderungen der Genexpression aus Homogenaten von Herzgewebe untersucht haben, verhindert dies die Untersuchung der inhärenten stochastischen Variation zwischen den Zellen innerhalb eines Gewebes. Isolierung von reinen Kardiomyozyten Populationen durch eine Kollagenase Perfusion von Mäuseherzen vereinfacht die Erstellung von einzelnen Zelle Microarrays für die ganze Transkriptom-Genexpression oder qPCR spezifische Ziele mit nanofluidischen Arrays. Wir beschreiben hier ein Verfahren zur einzelnen Zelle Genexpressionsprofile von Kardiomyozyten aus dem Herzen isoliert zu untersuchen. Dieses Paradigma ermöglicht die Auswertung der Messdaten von Interesse, die nicht auf den Mittelwert angewiesen (zB Varianz zwischen den Zellen innerhalb eines Gewebes), die nicht möglich ist, wenn mit konventionellen ganze Gewebe Workflows für die Auswertung der Gen-Expression (Abbildung 1). Wir haben robuste Verstärkung der einzelnen Zelle Transkriptom Nachgeben Mikrogramm doppelsträngigen cDNA, dass der Einsatz von Microarrays auf i erleichtert erreichtndividuelle Zellen. In das Verfahren beschreiben wir die Verwendung von NimbleGen Arrays, die für ihre Benutzerfreundlichkeit und die Fähigkeit, ihr Design anpassen ausgewählt wurden. Alternativ kann ein Reverse-Transkriptase - ermöglicht spezifische Target-Amplifikation (RT-STA) Reaktion, für die qPCR von Hunderten von Zielen durch nanofluidischen PCR. Mit diesen beiden Ansätzen ist es möglich, die Variabilität des Ausdrucks zwischen den Zellen, sowie die Prüfung Expressionsprofile von seltenen Zelltypen innerhalb eines Gewebes zu untersuchen. Insgesamt ermöglicht die einzelne Zelle Genexpression Ansatz für die Generierung von Daten, die möglicherweise identifizieren können eigenwilligen Ausdruck Profile, die typischerweise sind bei der Prüfung Ausdruck von Millionen von Zellen, die aus typischen Homogenaten von ganzen Geweben erzeugt werden gemittelt.
Protokoll
1. Schritt 1 - Cardiomyocyte Isolation
- Bereiten Sie die Verdauung Puffer wie folgt:
10 x Digestion Buffer (made in ultra-reinem H 2 O)Finale Conc. g / L g/500 ml 10x Lösung g / L NaCl 130 mM 7,597 3,3 75,97 KCl 5 mM 0,4 0,2 4,0 Brenztraubensäure 3 nM 0,33 0,165 3,30 HEPES 25 mM 5,96 2,85 59,6 MgCl 2 0,5 mM 0,101 0,05 1,01 NaH 2PO 4monobasic 0,33 mm 0,04 0,02 0,40 Traubenzucker 22 mM 3,96 1,98 39,6
pH-Puffer mit NaOH auf 7,4 - Mit dem 10x Digestion Buffer, die folgenden vier Lösungen für jedes Herz, das perfundiert (made in ultra-reinem H 2 O) werden sein:
Digestion Buffer A (Machen Sie drei Aliquots dieser Lösungen)
- 50 ml - 1x Digestion Buffer enthält:
- 75 &mgr; l - EGTA-Lösung (100 mM EGTA)
Digestion Buffer B (Machen Sie einen von diesen)
- 25 ml - 1x Digestion Buffer enthält:
- 1,25 ul - CaCl 2-Lösung (1 M)
- 1 mg - Protease
- 25 mg - Collagenase (can auch 75% von diesem Betrag = 18.75 mg) **
Sammlung Buffer (Machen Sie eine der folgenden)
- 2,5 ml - 1x Digestion Buffer enthält:
- 1,25 ul - CaCl 2-Lösung (1 M)
- 0,5 mg - Protease
- 15 mg - Collagenase (75% = 11,25 mg) **
- 125 mg - BSA
Neutralisation Waschpuffer (Machen Sie eine der folgenden)
- 25 ml - 1x Digestion Buffer enthält:
- 6,25 ul - CaCl 2-Lösung (1 M)
- 250 mg BSA
** Je nach Quelle und Batch von Kollagenase, kann die Enzymaktivität variieren. Es kann notwendig sein, um die Menge an Enzym hinzugefügt, um diesen Puffer über-Verdauung zu verhindern optimieren.
- Richten Sie die Isolation System wie zuvor in d beschriebenetail 1,2 mit drei 15-ml-Becher Digestion Buffer A auf Eis, einem 50-ml-Tube Digestion Buffer A und 25 ml Digestion Buffer B bereit für die Perfusion (Digestion Buffer A sollte durch die Leitung gespült werden, um mindestens 5 mL laufen durch das System), erwärmt Sammlung Buffer in einem Wasserbad auf die Temperatur auf 37 ° C, und die Neutralisation Waschpuffer bei RT zu erhöhen.
- Füllen Sie den Perfusionskanüle und der Dissektion Gericht, wo das Herz zu reinigen und werden gebunden an die Perfusion Spitze mit kalten Digestion Buffer A. lose Knoten einige Naht (4-O bis 6-O-Größe) um den oberen Teil der Kanüle. Dies wird später verwendet, um die Aorta halten, um das Herz ein Abrutschen der Kanüle zu verhindern.
- Terminally sedate der Maus mit einer IP-Injektion von Anästhetikum (0,25 ml Natrium-Pentobarbital-Lösung) und sorgen für die Maus ist bewusstlos und reagiert nicht auf Schmerzreize. Schneiden Sie die Brusthöhle vorsichtig entlang der Brust-Marge, indem das Zwerchfell(Abbildung 2). Cut souverän nach oben und spiegeln die vorderen Brustkorb (diese Anschnitte sind B und A bezeichnet sind in Abbildung 2) Kritisch -. Die Maus muss noch vor der Atmung, um diese Einschnitte.
- Mit einem Kunststoff-Pipette auf Maß geschnitten, sanft saugen das Herz in die Pipette. Schneiden Sie das Herz aus der Brusthöhle darauf achten, genug der Aorta auf das Herz zu verlassen, um eindeutig die Aorta Zweig (Abbildung 3). Eine Pipette wird verwendet, da es eine minimale Schädigung des Herzens führt.
- Drop das Herz in einem Becherglas mit eiskaltem Digestion Buffer A und spülen das Blut weg. Nach 15 bis 45 sec Platz das Herz in die zweite Becherglas mit eiskaltem Digestion Buffer A nochmals kurz abspülen. Übertragen Sie die Herzen in der Petrischale auch mit kalten Digestion Buffer A. gefüllt
- Sehen Sie sich die Herzen in die Schüssel der Verdauung Puffer A unter der Dissektion Umfang und sorgfältig zu isolieren die Aorta und ihrer Äste. Vorsichtig schneiden unmittelbar unterhalb des letzten Branch (Abb. 4).
- Fit die Spitze der Kanüle in die Aorta und ligieren statt mit dem 4-O bis 6-O Naht mit dem Bühnenaufbau unter einem Dissektionsmikroskop. Stellen Sie sicher, dass die Kanüle nicht sitzen zu tief in die Aorta und bleibt oberhalb der Koronararterien (Abbildung 4).
- Mit Digestion Buffer A fließt durch die Perfusion Setup, legen Sie die Perfusion Spitze auf, um das Setup, und beginnen, die Herzen perfuse, bis die Mehrheit der Blut aus dem Herzen und Flüssigkeit verlässt das Herz ist klar, gewaschen wird. Kritisch - die Temperatur des Perfusionsflüssigkeit muss so nah wie möglich an 37 ° C, wenn es rauskommt des Katheters. Wenn die Lösung ist zu heiß oder zu kalt dann die Durchblutung wird das Gewebe zu töten. Die Zeit bis zur Perfusion des Herzens ist ebenfalls entscheidend. Das Zeitfenster muss kleiner sein als 5-10 min von der Entfernung des Herzens bis Perfusion beginnt. Das Herz muss unter Narkose entfernt werden, nicht CO 2 Anbringung oder Genickbruch.
- Sobald das Blut wurdehed aus dem Herzen, um die Verdauung Buffer B Schalter von der Verdauung Buffer A. Stellen Sie sicher, dass die Verdauung Buffer B fließt jetzt durch das Herz. Die Lösung beginnt, eine gelbe Tönung haben. Warten Sie 2-3 min, bis das Herz zeigt Anzeichen von seine Form verliert und beginnt, schauen abgerundet was ein Indiz dafür, dass die Verdauung funktioniert. Der Herzmuskel ist heller als die Perfusion erfolgt.
- Schneiden Sie die Ventrikel mit einer Schere und in einen 50 ml konischen Röhrchen mit 15 ml Sammlung Buffer bei 37 ° C. Vorsichtig schneiden die Ventrikel in kleine Stücke beim Platzieren des Gewebes in der Sammlung Buffer. Vorsichtig mischen mit einem Kunststoff-Pipette auf die Zell-Zell-Adhäsionen lösen und eine einzelne Zelle Kardiomyozyten Mischung.
- Wenn die meisten der Gewebe gelöst ist, sei (weniger als 1 min) die größere Gewebestücke zu Boden sinken. Entfernen Sie und speichern Sie den Überstand in ein 15 mL konischen Rohr. Lassen Sie die Schwerkraft ziehen ein Pellet aus dem Überstand über 10 bis 20 min (15min bevorzugt) bei Raumtemperatur.
- Entfernen und den Überstand verwerfen. Wash Pellet durch Zugabe von 5 mL der Neutralisation Waschpuffer auf die Pellets und vorsichtig mischen die Zellen, um sie zu resuspendieren. An dieser Stelle werden die Zellen sofort bereit, für einzelne Zelle Analyse (fahren Sie mit Schritt 2) ausgewählt werden.
2. Schritt 2 - Isolierung von Einzelzellen
- Bereiten Sie einen Mikromanipulator mit einer Flamme und einem Glas Kapillare verwendet, um Zellen zu manipulieren. Der Mikromanipulator ist einfach eine sehr feine Kapillare, die dann auf ein langes Stück Schlauch, die sich bewegen oder abholen können Zellen, wenn Saug angewendet wird, ist angebracht.
- Unmittelbar nach Auswahl der einzelnen Zellen unter einem Mikroskop (Nikon Eclipse TS100, 20x) mit einem Mikromanipulator, die mit einer Pipette gezogen und eine Luftschleuse, um Material von Wesen, die einzelne Zelle Proben Probe übertragen werden dürfen erstellt wurde fortfahren. Stellen Sie sicher, dass die Zell-Population gesund ist(70% lebensfähig) und nicht aus der Isolierung beschädigt wird.
- Verdünnen Sie die Zellen, die in einer kleinen Petrischale, so dass sie einzeln ausgewählt werden können. Bei der Kommissionierung Zellen sicher, dass die Zellen in einem kleinen Volumen (weniger als 2 ul) zu erfassen. Wählen Sie nur gesunde Zellen, die die Unterscheidungskraft normalen Kardiomyozyten Morphologie (Abbildung 5).
- Legen Sie einzelne Zellen in die Unterseite des einzelnen PCR-Röhrchen und frieren sie sofort auf Trockeneis. Diese Zellen werden dann bei -80 ° C gelagert, bis sie zur Genexpressionsanalyse eingesetzt werden sollen.
3. Schritt 3A - Single Zelle qPCR Genexpression
Um qPCR an einzelnen Zellen beschäftigen ein Protokoll aus einer einzigen Zelle für die Genexpression von Fluidigm Corporation entwickelt für ihre BioMark nanofluidischen qPCR-Arrays angepasst (Fluidigm - Vorentwicklung Protokoll Nr. 30) 3,4.
- Vorbereiten des Reverse Transkription - spezifische Targets Amplification (RT-STA). UmGin, resuspendieren alle Primerpaare (wir haben bis zu 125 Primerpaare erfolgreich eingesetzt) für die Gene von Interesse bei 100 pM in 1x DNA Suspensionspuffer (10 mM Tris, pH 8,0, 0,1 mM EDTA). Kombinieren und weiteren Verdünnung der Vorwärts-und Rückwärts-Primer-Paare bis 20 pM für jeden Test. Schließlich verdünnen alle Primerpaare in einer Lösung mit einer Endkonzentration von 200 nM für jeden Test, der in der RT-STA-Reaktion amplifiziert werden. Um dies zu tun, nehmen jedes Primerpaar und verdünnt sie 1:100. Zum Beispiel, wenn Sie 30-Assay Primerpaare bei 20 uM haben, müssen Sie 1 ul jedes Paares (30 ul insgesamt) hinzufügen, und dann den Rest der Band mit 70 ul DNA Suspensionspuffer das Finale zu erreichen Verdünnung Volumen von 100 ul.
- Nun, da die Primer-Lösungen bereit sind, montieren die RT-STA Reaktion Master-Mix. Bereiten Sie genügend Master-Mix für jedes der einzelnen Zellen, die Sie möchten, zu verstärken.
Tabelle 1. RT-STA Master-MixReagens Volume in ul (pro Reaktion) CellsDirect 2x Reaction Mix 5,0 SuperScript III RT Plantinum Taq-Mix 0,2 RT-STA Primergemisch (200 nM jedes Assay) 2,5 Nuclease freie H 2 O (oder 1x DNA Suspensionspuffer) 1,3 Gesamt 9,0 - Umgehend entfernen eingefrorenen Zellen und Zentrifuge bei maximaler RCF für 30 Sekunden, um sicherzustellen, dass die Zellen am Boden der Röhre positioniert sind. Add 9 ul RT-STA-Mix auf die Tube und fahren Sie mit der Reaktion in einem Thermocycler.
- 50 ° C - 15 Minuten
- 95 ° C - 2 Minuten
- 18 Zyklen von
- 95 ° C für 15 Sekunden
- 60° C für 4 Minuten
- Halten bei 4 ° C
- Entfernen Sie die Schläuche von der PCR-Maschine und fügen 4 ul ExoSAP-IT, um die Reaktion. ExoSAP-IT ist ein PCR Cleanup Reagenz, das überschüssige Primer und Enzym aus dem RT-STA Reaktion zu entfernen. Führen Sie die Röhren durch die folgende Reaktion in einer PCR-Maschine.
- 37 ° C - 15 Minuten
- 80 ° C - 15 Minuten
- Halten bei 4 ° C
- Wenn die ExoSAP-IT-Behandlung abgeschlossen ist, verdünnt die Proben 1:5 mit DNA Suspensionspuffer. Die Proben werden nun für die qPCR-Analyse vorbereitet. Wir verarbeiten diesen Werkstoff in Verbindung mit Fluidigms Genexpression qPCR-Arrays (48,48 oder 96,96 Platten). Es ist auch möglich, diesen Ansatz mit eher traditionellen qPCR Instrumentierung (96 well oder 384-Well-Format) verwenden. Allerdings sind diese Instrumente erfordern in der Regel viel mehr Probe-Eingang, der die Anzahl der Tests, p kann beschränkterformed aus einer einzigen Zelle.
3. Schritt 3B - Whole Transkriptom-Amplifikation und Mikroarray von Einzelzellen
Extraktion und Amplifikation
- Verwenden Sigma WTA2 Kit (Cat # WTA2) zu doppelsträngigen cDNA aus beiden Pellets und einzelnen Zellen zu verstärken. Einzelne Zellen sind "extrahiert" über den ersten Schritt der Sigma WTA2 Protokoll gemäß den Anweisungen des Herstellers. Während dieser erste Schritt der Inkubation mit Bibliothek Synthese-Puffer bei 37 ° C für 5 Minuten unterbricht der Membran der einzelnen Zellen und "Auszüge" die Nachricht für die Amplifikation.
- Amplify einzelne Zellen in zwei Runden, mit den Richtungen von WTA2 Kit der Firma Sigma-Aldrich ist. Führen Sie die Bibliothek prep Schritt nach dem Protokoll des Herstellers, dann fügen Sie 1 / 5 der Bibliothek Probe zu 70 ul der WTA2 Verstärkung
Master Mix. Amplify die Proben für 25 Zyklen mit der empfohlenen Radtouren Parameter. - Purify die Proben using Qiagen QIAquick PCR Purification Kit (Kat. Nr. 28104) und Prozess zu QIAGENs QIAcube mit dem "Cleanup QIAquick PCR-Amplifikation Standard-V4"-Protokoll.
- Konzentrieren Sie sich die gereinigten Proben zu 5 ul, mit einer Geschwindigkeit Vakuum, und setzte durch eine zweite Runde der Amplifikation für 17 Zyklen (WTA2 Amplifikationsprotokoll und Radfahren Parameter wiederholt).
- Purify Proben mit dem QIAquick PCR Purification Kit und prüfen Sie die Qualität mit einem NanoDrop Spektralphotometer und Agilent BioAnalyzer DNA 7500 Kit nach Herstellerangaben Protokoll.
Labeling und NimbleGen Gene Expression Arrays
- Label 2 pg doppelsträngigen cDNA aus jeder Zelle verstärkt Probe mit NimbleGen One Color Labeling Kit (Kat. Nr. 05223555001) nach dem Protokoll des Herstellers.
- Hybridisieren 5 ug Cy3 markierte Probe zu Mus musculus 12x135k, NimbleGen Gene Expression Arrays (Kat. Nr. 05543797001) nachording dem Protokoll des Herstellers. Nach der Hybridisierung der Proben zu den Arrays, sollten die Folien dann gewaschen und getrocknet werden, bevor sie auf einem Laser-Scanner abgebildet werden können.
- Scan Gene Expression Arrays mit einem Laser-Scanner. Hier verwenden wir eine Molecular Devices GenePix 4200A Scanner mit Einstellungen 100POW, und 300-350PMT. Dann erzeugen die beiden Dateien für jedes Array mit NimbleGen NimbleScan Software und damit auch für ganze Transkriptom-Expression zu analysieren aus jeder einzelnen Zelle array (eine Tabelle mit typischen stabil exprimiert Kardiomyozyten Transkripte ist als Ergänzung enthalten sind; Tabelle S1).
Wenn das Herz Perfusion gut funktioniert sollte es einen hohen Anteil an gesunden Herzen Zellen, die ihre typischen rechteckigen Morphologie behalten nach Isolation. Wenn die Durchblutung nicht gut gehen dann gibt es einen großen Prozentsatz der toten Zellen (siehe Bilder in Abbildung 5). Wenn die Zellen richtig verstärkt entweder mit dem WTA2 oder RT-STA-Methoden dern der Endprodukte sollte anschließenden Qualitätstests für die Qualität der mRNA-Proben von NanoDrop und Bioanalyzer (Abbildung 6) übergeben. Für die Microarray-Workflow, ist dies nach WTA-Amplifikation, wo die mRNA von beiden NanoDrop und Bioanalyzer getestet abgeschlossen ist. Vertreter positive Ergebnisse für diese Analyse sind in Abbildung 6 dargestellt. Die WTA2 Proben sollten eine robuste Amplifikation mit beiden NanoDrop Spektralphotometer Lesen, sowie die Elektropherogramm Auslesen aus dem Bioanalyzer (BA)-Chip (Abbildung 6) zeigen. Eine Tabelle von Genen, die stabil über Microarray einzelner Zellen nachgewiesen wurden als Beispiel (Tabelle S1) enthalten. Für die qPCR-Prozess kann die Qualität der Daten, indem eine Schmelze Schritt am Ende der PCR-Reaktion, um sicherzustellen, dass die Primer-Sets sind verstärkt das gewünschte Produkt (Abbildung 7) beurteilt werden. Wenn das stimmt, sollte diese Schmelze Schritt erzeugen eine spezifische Schmelzpeak. Um dies zu verdeutlichen, ist eine Teilmenge von nanofluidischen qPCR-Daten in einer Heatmap der Spitze schmelzen t gezeigtemperatur (Abbildung 8) 8. Es ist möglich, die Qualität eines PCR-Produkt durch seine Schmelztemperatur zu bewerten, um die Abwesenheit von nicht-spezifische Produkte 5 zu gewährleisten. Jede Zeile dieser Karte stellt eine Zellprobe (S1-S40) in 43 separaten qPCR-Assays (A1-A43) getestet. In dieser Figur wird deutlich, dass einige Tests sehr variabel sind und wahrscheinlich weniger zuverlässig als die stabilere Assays mit höherer Spezifität PCR.
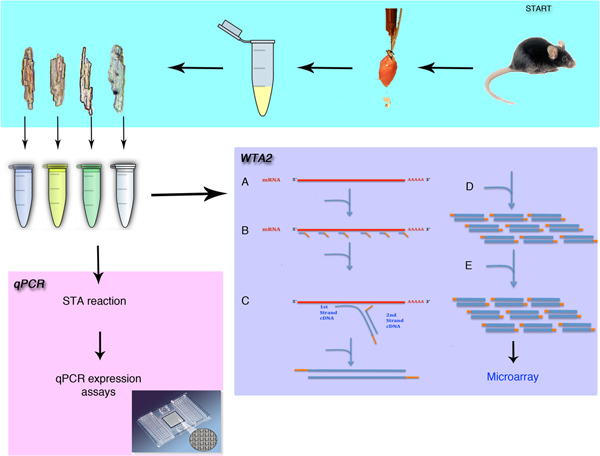
Abbildung 1. Übersicht der einzelnen Zelle isoliert und Genexpressionsanalyse. Das Verfahren wird zeigen, die chirurgische Entfernung der Maus Herz und die Isolierung der einzelnen Herzmuskelzellen. Die Methoden in diesem Verfahren erörtert die Verfahren für eine entweder qPCR oder Mikroarray-Analyse nach ganzen Transkriptom-Amplifikation (WTA). Die WTA Verfahren beginnt mit der Lyse (A), dann die Bindung von Sigma die universelle Primer (B) auf die mRNAPool. Die Verlängerung dieser Primer (C) und die Verstärkung (D)-Phase erstellen Mikrogramm amplifizierten Materials. Diese Verstärkung wird dann wiederholt (E), um genügend Material für die Microarray-Verfahren zu erzeugen.

Abbildung 2. Darstellung der Lage der Einschnitte in das Herz von der Maus zu entfernen. Schneiden Sie entlang der Seitenwand des Brustkorbes (A), dann entlang der untere Rand des Brustkorbs (B) geschnitten. Schneiden Sie die Schiffe oberhalb und unterhalb des Herzens ermöglichen Entfernung und lässt noch genug von der Aorta in das Herz kanülieren. Bilder von MJ Koch 6 angepasst.
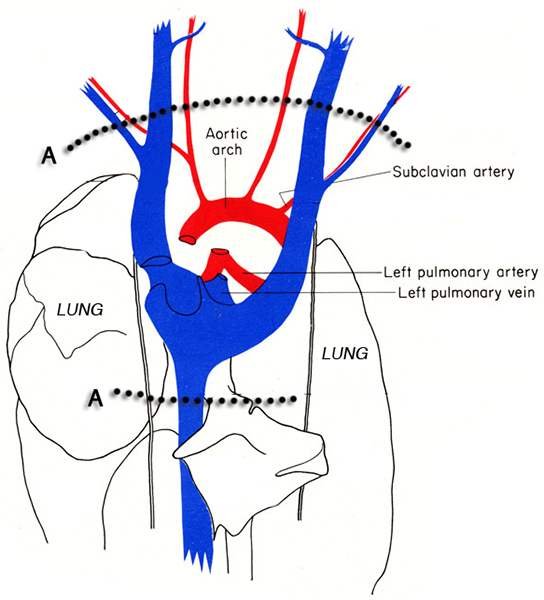
Abbildung 3. Schematische Darstellung des Thorax der Maus. Die gestrichelten Linien zeigen die Positionen der Schnitte (A) für Ausschneiden des Herzens aus der Brusthöhle, ohne die h eart Gewebe. Bilder von MJ Koch 6 angepasst.
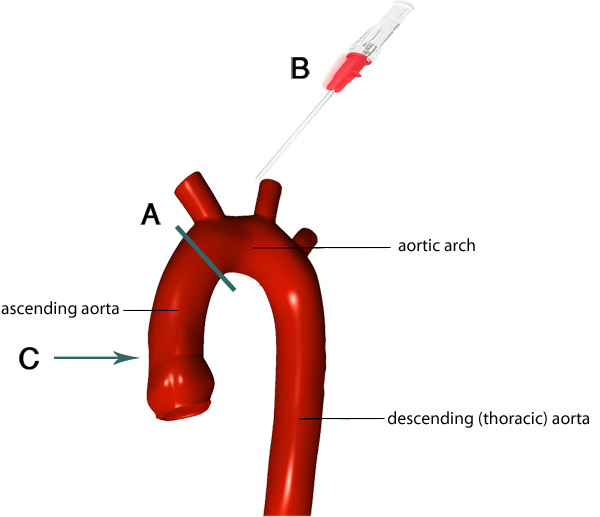
Abbildung 4. Das Bild des Aortenbogens und seiner Äste. Die graue Linie zeigt den idealen Ort, an dem der Aorta (A) zu trimmen. Die restlichen Aorta an das Herz durchbohrt, so dass die Spitze der Kanüle (B) auf der entsprechenden Ebene in der Aorta (C) geht. Bilder von F. Gaillard 7 angepasst.

Abbildung 5. Auswahl der einzelnen Zellen zur Genexpressionsanalyse. Jedes Panel zeigt Kardiomyozyten unter dem Lichtmikroskop zeigt typische Morphologie von Kardiomyozyten abgebildet. Gesunde Kardiomyozyten durch die grünen Pfeile angedeutet. Zellen, die tot oder sterbend sind mit roten Pfeilen dargestellt. Diese toten Zellen sollten nicht in die Analyse verwendet werden.
iles/ftp_upload/3302/3302fig6.jpg "alt =" Bild 6 "/>
Abbildung 6. Qualitätskontrolle WTA Ergebnisse für die einzelne Zelle Verstärkung. (A) Bild eines NanoDrop Spektralphotometer ausgelesen, die für das verstärkte Transkripte aus dem WTA Reaktion ist. (B) Das Auslesen von BioAnalyzer Chip zeigt die Ergebnisse von drei verstärkt Zellen.
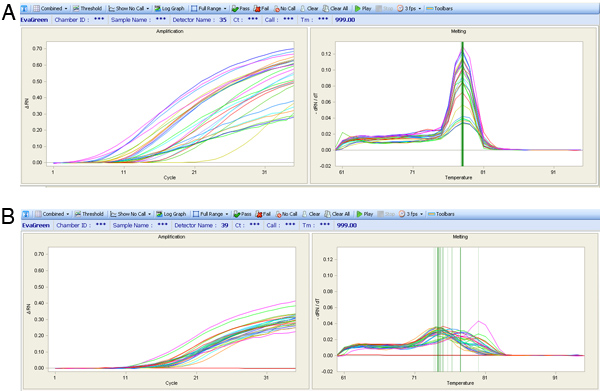
Abbildung 7. QPCR Amplifikationskurven (Left-Chart) und die maximale Temperatur der Schmelze Kurven (rechte Skala). (A) Der Ausdruck ergibt sich aus einer Qualität Assay mit einem einzigen Schmelzpeak trotz unterschiedlicher Amplifikationskurven gezeigt. (B) Melt-Kurven für eine arme Primer-Set, das sehr variabel Schmelzpeaks was nicht gerade ideal für die Expressionsanalyse hat.
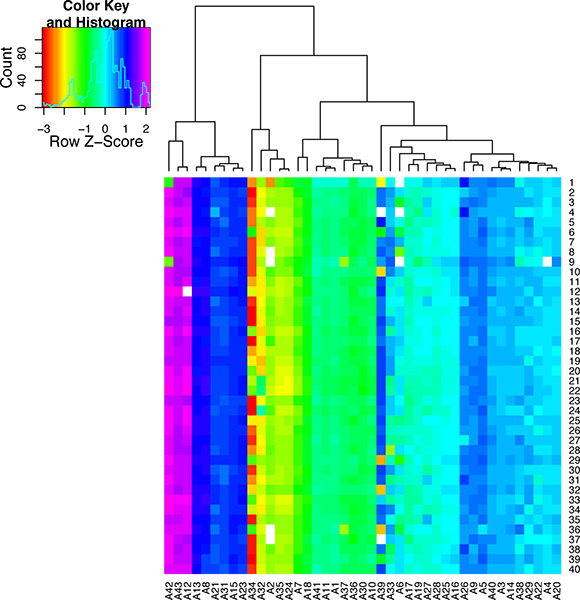
Abbildung 8. Qualitätssicherung für die einzelne Zelle qPCR reagierenIonen. Diese Wärme Karte wurde geschaffen, um die Temperaturen der Schmelze als Farbskala angezeigt werden. Die Peak-Schmelztemperatur für jedes qPCR-Assays (A1-A43) ist für 40 einzelne Zellen (S1-40) gezeigt. Die Assays wurden nach ihrer Schmelztemperatur geclustert. Dazu suchen Sie unten in der Spalte für jeden Assay ist es möglich zu sehen, die Unterschiede zwischen all den Proben zu schmelzen. Diese Zahl zeigt, dass einige qPCR-Assays sehr spezifisch sind, während andere sehr variabel sind und somit nicht geeignet für einzelne Zelle Analyse.
Tabelle S1. Supplemental Tabelle der Microarray-Daten. Diese Gene sind nach der Robustheit, in denen sie in einzelnen Herzmuskelzellen über eine große Datenmenge erfasst werden aufgelistet. Dieses Gen Liste zeigt die Empfindlichkeit des Nachweises für eine Reihe von Genen, die typischerweise in einzelnen Kardiomyozyten exprimiert werden.
Diskussion
Diese Methode hat die Möglichkeit, Kardiomyozyten für eine Reihe von funktionalen als auch Genexpressionsstudien generieren. Die Untersuchung der einzelnen Zellen ist ein aktuelles Gebiet in der Genexpressionsanalyse. Der Vorteil der Untersuchung transkriptioneller Ebene auf der Ebene der einzelnen Zelle ist, dass sie die Prüfung eines reinen Zellpopulation, die nicht aus ganzen Gewebes Vorbereitungen möglich erlaubt. Darüber hinaus ermöglicht einzelne Zelle Analyse für die Prüfung der stochastischen Variation der mRNA-Spiegel in einzelnen Zellen auf Zellpopulationen, die bisher glaubten, eine homogene 8,9 definieren. Neben der Identifizierung von Genen, die möglicherweise stochastische in ihrer Genexpression 10,11,12 sind, die Methode ermöglicht es auch für die Identifizierung von seltenen Zellpopulationen durch ihre Genexpressionsprofile definiert.
Während diese Methode bietet eine Reihe von spannenden Einsatzmöglichkeiten in der Genexpressionsanalyse, gibt es some Einschränkungen und Überlegungen in die Verwendung von einzelnen Zellen. Der primäre Beschränkung auf einzelne Zelle Analyse, ist die Sammlung von genügend Zellen in dem Experiment um eine statistische Signifikanz in Bezug auf die Metrik von Interesse zu erreichen, zum Beispiel Varianz. In Fällen, in denen die Zahl der Zellen aus dem Gewebe von Interesse isoliert ist keine Einschränkung, kann man untersuchen, Hunderte von Zellen pro Gruppe, wobei Ansätze wie der nächsten Generation Sequenzierung, oder nanofluidischen Arrays. In einigen Fällen kann es zu Schwierigkeiten bei der Beschaffung genügend Zellen aus dem Gewebe von Interesse sein. Dies kann zum Teil durch eigenwillige zeitintensive Verfahren für die Erhebung der gezielten Zelltyp verursacht werden. Allerdings kann eine sorgfältige Planung und Vorbereitung vor mit einzelnen Zelle Kollektion Verfahren noch einer strengen einzelne Zelle Analyse mit begrenzten technischen Fehler erlauben. Bei der Prüfung der Ergebnisse davon ausgegangen werden muss, dass, obwohl dieses Verfahren zur Isolierung von Zellen in zahlreichen studie verwendet wurdes (einschließlich Mikroskopie, Elektrophysiologie, etc.), könnte die Isolierung selbst potenziell Wirkung Genexpression. Wie bei jeder Methode, die biologischen Proben manipuliert, sollten Sie die Ergebnisse Ihrer Ergebnisse sorgfältig überprüft werden, um sicherzustellen, dass die einzelne Zelle Ausdruck steht stellvertretend für die Gewebe selbst und nicht technische Bias. Methoden wie in-situ-Hybridisierung kann nützlich sein, um diese Ergebnisse in der intakten Gewebe zu überprüfen. Schließlich ist es entscheidend, um sicherzustellen, dass die generierten Daten sorgfältig für die Qualitätskontrolle überprüft. Die Daten in Abbildung 8 zeigt, dass qPCR-Assays können als sehr robust in ihrer Spezifität, wie z. B. Test Nr. 13 (A13) oder haben ein hohes Maß an Variabilität, die technische Varianz wie Assay # 34 (A34) führen kann.
Offenlegungen
Freier Zugang und Produktion dieses dieses Artikels ist Sigma-Aldrich gesponsert.
Danksagungen
Die Autoren bedanken sich bei der technischen Unterstützung von C. Zambataro während der Dreharbeiten zu diesem Protokoll zu bestätigen. Wir danken für die Unterstützung des Glenn Foundation for Medical Research (SM), Die Hillblom Fundament, und die National Institutes of Health für eine Nathan Shock Center Award (P30AG025708) und PO1AG025901. JMF wurde von T32AG000266 verliehen, die Buck Institute for Research on Aging unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| NaCl | Sigma-Aldrich | 71378 | |
| KCl | Sigma-Aldrich | 60128 | |
| Brenztraubensäure | Sigma-Aldrich | P4562 | |
| Hepes | Sigma-Aldrich | 54457 | |
| MgCl 2 | Sigma-Aldrich | M1020 | |
| NaH 2 PO 4 einbasischen | Sigma-Aldrich | P9791 | |
| Traubenzucker | Sigma-Aldrich | G6721 | |
| NaOH | Sigma-Aldrich | 72068 | |
| EGTA | Sigma-Aldrich | O3777 | |
| CaCl 2 | Sigma-Aldrich | 21115 | |
| Protease, Type XIV | Sigma-Aldrich | P5147 | |
| Collagenase, Type I | Worthington | C9891 | |
| 1x DNA Suspension Buffer (10 mM Tris, pH 8,0, 0,1 mM EDTA) | TEKnova, | PN T0221 | |
| BSA | Sigma-Aldrich | B8667 | |
| Natrium-Pentobarbital | Henry Schein Vet. Versorgung * | Kontaktlieferant für die Bestellung | * Muss durch einen Tierarzt oder Versuchstier professionelle beschafft werden |
| ExoSAP IT | Affymetrix / USB | 78201 | |
| CellsDirect 2x Rxn Mix | Invitrogen | 11737-030 | |
| SuperScript III RT Platinum Taq Mix | Invitrogen | 10928-042 | |
| RT-STA Primermischung | IDT | Brauch | |
| Nuclease freies Wasser | Sigma-Aldrich | W4502 | |
| WTA2 | Sigma-Aldrich | WTA2-50rxn | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| QIAcube | Qiagen | 9001292 | |
| NanoDrop Spektralphotometer | NanoDrop | 2000c | |
| BioAnalyzer DNA 7500 Kit | Agilent | 7500 Kit | |
| Eine Farbe Labeling Kit | Roche NimbleGen- | 5223555001 | |
| Mus Musculus 12x135k Array | Roche NimbleGen- | 5543797001 | |
| GenePix 4200A Scanner | Molecular Devices | 4200A | |
| TransPlex Ganzes Transkriptom Amplification Kit (WTA2 Kit) | Sigma-Aldrich | WTA2-50RXN |
Referenzen
- Wolska, B. M., Solaro, R. J. Method for isolation of adult mouse cardiac myocytes for studies of contraction and microfluorimetry. Am. J. Physiol. 271 (3 Pt 2), H1250-H1255 (1996).
- Shioya, T. A simple technique for isolating healthy heart cells from mouse models. J. Physiol. Sci. 57 (6), 327-335 (2007).
- Guo, G., Huss, M., Tong, G. Q., Wang, C., Li Sun, L., Clarke, N. D., Robson, P. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Dev. Cell. 18 (4), 675-685 (2010).
- Narsinh, K. H., Sun, N., Sanchez-Freire, V., Lee, A. S., Almeida, P., Hu, S., Jan, T., Wilson, K. D., Leong, D., Rosenberg, J., Yao, M., Robbins, R. C., Wu, J. C. Single cell transcriptional profiling reveals heterogeneity of human induced pluripotent stem cells. J. Clin. Invest. 121 (3), 1217-1221 (2011).
- D'haene, B., Vandesompele, J., Hellemans, J. Accurate and objective copy number profiling using real-time quantitative PCR. Methods. 50 (4), 262-270 (2010).
- Cook, M. J. The Anatomy of the Laboratory Mouse. Informatics. , (1965).
- Gaillard, F. Aorta. Radiopaedia. , (2008).
- Ståhlberg, A., Andersson, D., Aurelius, J., Faiz, M., Pekna, M., Kubista, M., Pekny, M. Defining cell populations with single-cell gene expression profiling: correlations and identification of astrocyte subpopulations. Nucleic Acids Res. 39 (4), e24-e24 (2011).
- Zhong, J. F., Chen, Y., Marcus, J. S., Scherer, A., Quake, S. R., Taylor, C. R., Weiner, L. P. A microfluidic processor for gene expression profiling of single human embryonic stem cells. Lab Chip. 8 (1), 68-74 (2008).
- Bahar, R., Hartmann, C. H., Rodriguez, K. A., Denny, A. D., Busuttil, R. A., Dollé, M. E., Calder, R. B., Chisholm, G. B., Pollock, B. H., Klein, C. A., Vijg, J. Increased cell-to-cell variation in gene expression in ageing mouse heart. Nature. 441 (7096), 1011-1014 (2006).
- Raj, A., van Oudenaarden, A. Nature, nurture, or chance: stochastic gene expression and its consequences. Cell. 135 (2), 216-226 (2008).
- Elowitz, M., Levine, A., Siggia, E., Swain, P. Stochastic gene expression in a single cell. Science. 297 (5584), 1183-1186 (2002).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten