Method Article
Profiling única célula transcricional dos cardiomiócitos de rato adulto
Neste Artigo
Resumo
Perfil de expressão única célula permite a análise detalhada de expressão gênica de células individuais. Descrevemos métodos para o isolamento dos cardiomiócitos, e preparar a lisados resultando tanto para microarray transcriptoma todo ou qPCR de metas específicas.
Resumo
Enquanto numerosos estudos têm examinado as mudanças de expressão gênica de homogenatos de tecido do coração, isso impede de estudar a variação estocástica inerente entre as células dentro de um tecido. Isolamento de populações puras de cardiomiócitos através de uma perfusão colagenase de corações de camundongos facilita a geração de microarrays única célula para a expressão do gene toda transcriptoma, ou qPCR de metas específicas usando arrays nanofluidic. Descrevemos aqui um procedimento para examinar única célula perfis de expressão gênica dos cardiomiócitos isolados do coração. Este paradigma permite a avaliação das métricas de interesse que não são dependentes da média (por exemplo, entre variância células dentro de um tecido) que não é possível ao usar fluxos de trabalho convencionais tecido inteiro para a avaliação da expressão gênica (Figura 1). Temos conseguido amplificação robusta do microgramas única célula transcriptoma rendendo cDNA de dupla fita que facilita o uso de microarrays na icélulas INDIVIDUAIS. No procedimento que descrevem o uso de matrizes NimbleGen que foram escolhidos devido a sua facilidade de uso e capacidade de personalizar o seu design. Alternativamente, uma transcriptase reversa - amplificação do alvo específico (RT-STA) reação, permite qPCR de centenas de alvos por nanofluidic PCR. Usando um desses enfoques, é possível examinar a variabilidade de expressão entre as células, bem como examinar perfis de expressão de tipos de células raras a partir de um tecido. No geral, o único celular abordagem a expressão do gene permite a geração de dados que podem potencialmente identificar perfis de expressão idiossincrática que normalmente são em média para fora quando examinar expressão de milhões de células de homogenatos típicos gerados a partir de tecidos todo.
Protocolo
1. Passo 1 - isolamento dos cardiomiócitos
- Preparar o tampão de digestão da seguinte forma:
10 x tampão de digestão (feito em ultra-pura H 2 O)Conc final. g / L g/500 mL 10x solução g / L NaCl 130 mM 7,597 3,3 75,97 KCl 5 mM 0,4 0,2 4,0 Ácido pirúvico 3 nM 0,33 0,165 3,30 HEPES 25 mM 5,96 2,85 59,6 MgCl 2 0,5 mM 0,101 0,05 1,01 NaH 2PO 4monobasic 0,33 mM 0,04 0,02 0,40 Dextrose 22 mM 3,96 1,98 39,6
pH para 7,4 com NaOH - Usando o tampão de digestão 10x, faça as seguintes quatro soluções para cada coração que será perfundidos (feito em ultra-pura H 2 O):
Tampão de digestão A (Faça três alíquotas deste soluções)
- 50 mL - 1x tampão de digestão contendo:
- 75 mL - solução de EGTA (100 mM EGTA)
Tampão de digestão B (Faça um desses)
- 25 mL - 1x tampão de digestão contendo:
- 1,25 mL - 2 Solution CaCl (1 M)
- 1 mg - Protease
- 25 mg - colagenase (can também usar 75% desse montante = 18,75 mg) **
Coleta de Buffer (Faça um desses)
- 2,5 ml - 1x tampão de digestão contendo:
- 1,25 mL - 2 Solution CaCl (1 M)
- 0,5 mg - Protease
- 15 mg - colagenase (75% = 11,25 mg) **
- 125 mg - BSA
Tampão de Neutralização Wash (Faça um desses)
- 25 mL - 1x tampão de digestão contendo:
- 6,25 mL - 2 Solution CaCl (1 M)
- 250 mg BSA
** Dependendo da fonte e lote de colagenase, a atividade enzimática pode variar. Pode ser necessário para otimizar a quantidade de enzima adicionado a este tampão para impedir que mais de digestão.
- Configure o sistema de isolamento, como descrito anteriormente em detail 1,2 com três copos de 15 mL de tampão de digestão A no gelo, um tubo de 50 mL de tampão de digestão A e B do 25 mL tampão de digestão pronto para perfusão (tampão de digestão A deve ser lavada através da linha, no mínimo de 5 mL executado através do sistema), Buffer Colecção aquecido em banho-maria para elevar a temperatura a 37 ° C, eo tampão de lavagem de Neutralização de RT.
- Preencher a cânula de perfusão eo prato dissecção onde o coração vai ser limpo e amarrado na ponta de perfusão com frio A. Digestão buffer Loosely nó alguns sutura (4-O para 6-O tamanho) ao redor da porção superior da cânula. Este será mais tarde utilizado para manter a aorta para evitar que o coração deslizar fora da cânula.
- Terminal sedar o mouse com uma injeção de anestésico IP (0,25 mL de solução de sódio Pentobarbital) e garantir o mouse estiver inconsciente e não responde a estímulos de dor. Corte aberto da cavidade torácica com cuidado ao longo da margem torácica cortando o diafragma(Figura 2). Cortadas superiormente para cima e para refletir a caixa torácica anterior (estas incisões são designados B e A, respectivamente, na Figura 2) Crítica -. O mouse ainda deve ser a respiração antes de estas incisões.
- Usando uma pipeta de plástico cortados ao tamanho, sugar delicadamente o coração na pipeta. Cortar o coração para fora da cavidade torácica, tendo o cuidado de deixar o suficiente da aorta no coração para identificar claramente o ramo aorta (Figura 3). A pipeta é usado, uma vez que provoca danos mínimos para o coração.
- Soltar o coração em um copo de gelo tampão de digestão fria A e lavar o sangue para fora. Depois de 15-45 seg coração o lugar para o copo de gelo segundo tampão de digestão fria Uma brevemente enxágüe novamente. Transferir o coração para a placa de Petri também cheio de frio A. Digestão buffer
- Ver o coração no prato de um tampão de digestão no âmbito dissecção e cuidadosamente isolar a aorta e seus ramos. Aparar cuidadosamente apenas inferior à última branch (Figura 4).
- Coloque a ponta da cânula na aorta e ligadura no lugar com o 4-O para 6-O sutura utilizando a configuração do palco sob um microscópio de dissecação. Garantir que a cânula não se senta muito baixo dentro da aorta e permanece acima das artérias coronárias (Figura 4).
- Com tampão de digestão A flui através da instalação de perfusão, coloque a ponta de perfusão para a instalação e começar a perfundir o coração até que a maioria de sangue é lavado com o coração e fluido que sai do coração é claro. Crítica - a temperatura do líquido de perfusão deve ser o mais próximo possível de 37 ° C quando ele sai do cateter. Se a solução é muito quente ou muito frio, então a perfusão vai matar o tecido. O tempo até a perfusão do coração também é crítica. A janela de tempo deve ser inferior a 5-10 minutos da remoção do coração até perfusão começa. O coração deve ser removida sob anestesia, não CO 2 afixação ou deslocamento cervical.
- Quando o sangue é erahed do coração, mude para o tampão de digestão B do tampão de digestão A. Verifique se o tampão de digestão B agora está fluindo através do coração. A solução começará a ter uma coloração amarela. Esperar 2-3 min até que o coração dá sinais de perder sua forma e começa a parecer arredondada que é uma indicação de que a digestão está funcionando. O miocárdio deve aparecer mais leves como os proventos de perfusão.
- Cortar os ventrículos off com uma tesoura e coloque em um tubo cônico de 50 mL contendo 15 mL de tampão de Coleta a 37 ° C. Gentilmente cortar o ventrículo em pequenos pedaços ao colocar o tecido para o buffer Collection. Misture delicadamente com uma pipeta de plástico para dissolver as adesões célula-célula e fazer uma mistura única de cardiomiócitos de células.
- Quando a maioria do tecido é dissolvido, (menos de 1 min) deixe os pedaços maiores de tecido afundar até o fundo. Remova e guarde o sobrenadante para um tubo cônico de 15 mL. Deixe a gravidade puxa uma pelota do sobrenadante durante 10 a 20 min (15min de preferência) em temperatura ambiente.
- Remova e descarte o sobrenadante. Lavar pellet adicionando 5 mL do tampão de lavagem de neutralização para o pellet e misture delicadamente as células para ressuspender-los. Neste ponto, as células são imediatamente pronto para ser selecionado para análise única célula (vá para o passo 2).
2. Passo 2 - Isolamento de células individuais
- Prepare um micromanipulador usando uma chama e um tubo capilar de vidro que será utilizado para manipular células. O micromanipulador é simplesmente um tubo muito fino capilar que é então ligado a um longo pedaço de tubo flexível que pode mover ou pegar células quando a sucção é aplicada.
- Imediatamente proceder à seleção das células individuais sob um microscópio (Nikon Eclipse TS100, 20x) usando um micromanipulador, que foi criado com uma pipeta puxado e uma câmara para evitar que o material de ser transferido para a única amostra de amostras de células. Garantir que a população de células é saudável(70% viáveis) e não danificado a partir do processo de isolamento.
- Diluir as células para baixo em uma pequena placa de Petri, para que possam ser selecionados individualmente. Quando as células não se esqueça de escolher captar as células em um volume pequeno (menos de 2 mL). Seleccionar apenas as células saudáveis que têm a morfologia dos cardiomiócitos distintivo normal (Figura 5).
- Coloque células individuais na parte inferior dos tubos individuais PCR e congelá-los imediatamente em gelo seco. Estas células são então armazenados a -80 ° C até que estejam a ser utilizado para análise de expressão gênica.
3. Etapa 3A - Single celular expressão gênica qPCR
Para executar qPCR em células individuais empregar um protocolo adaptado de um desenvolvido para única expressão génica em células pela Corporação Fluidigm para suas matrizes Biomark nanofluidic qPCR (Fluidigm - Protocolo de Desenvolvimento Avanço # 30) 3,4.
- Preparando a transcrição reversa - Amplificação-alvo específicos (RT-STA). Para sergin, ressuspender todos os pares de primers (temos usado até 125 pares de primers com sucesso) para os genes de interesse a 100 mM em tampão 1x DNA de suspensão (10 mM Tris, pH 8,0, 0,1 mM EDTA). Combinar e ainda diluir a pares de primers para a frente e para trás a 20 M para cada ensaio. Por fim, diluir todos os pares de primers em uma solução com uma concentração final de 200 nM de cada ensaio que serão amplificados na reação de RT-STA. Para fazer isso, tome cada par de primer e diluí-los 1:100. Por exemplo, se você tem 30 pares de ensaio primário em 20 mM, você vai precisar adicionar 1 mL de cada par (30 no total mL), e depois fazer o restante do volume com 70 mL de tampão suspensão DNA para chegar à final volume de diluição de 100 mL.
- Agora que as soluções cartilha são preparados, montar o RT-STA master mix de reação. Prepare o suficiente master mix para cada uma das células individuais que deseja ampliar.
Tabela 1. STA RT-Mix MasterReagente Volume em mL (reação per) CellsDirect mix Reação 2x 5,0 SuperScript III RT Plantinum Taq mix 0,2 RT-STA mix Primer (200 nM cada ensaio) 2,5 Nuclease grátis H 2 O (ou tampão suspensão 1x DNA) 1,3 Total 9 - Remover imediatamente as células congeladas e centrifugar a RCF máximo por 30 segundos para garantir que as células estão posicionados na parte inferior do tubo. Adicionar 9 mL de RT-STA mistura para o tubo e prosseguir com a reação em um termociclador.
- 50 ° C - 15 minutos
- 95 ° C - 2 minutos
- 18 ciclos de
- 95 ° C por 15 segundos
- 60° C por 4 minutos
- Segure a 4 ° C
- Remover os tubos da máquina de PCR e adicionar 4 mL de ExoSAP-IT para a reação. ExoSAP-IT é um reagente de limpeza PCR que irá remover o excesso primers e enzima a partir da reação RT-STA. Executar os tubos através da reação em uma máquina de PCR.
- 37 ° C - 15 minutos
- 80 ° C - 15 minutos
- Segure a 4 ° C
- Quando o tratamento ExoSAP-IT é completa; diluir a 1:05 amostras com DNA de buffer suspensão. As amostras estão agora preparados para análise qPCR. Nós usamos este material em conjunto com Gene Fluidigm Arrays expressão qPCR (48,48, ou 96,96 placas). Também é possível usar essa abordagem com instrumentação mais tradicional qPCR (96 ou 384 formatos bem assim). No entanto, esses instrumentos geralmente requerem entrada de amostra muito mais o que restringe o número de ensaios que podem ser performed de uma única célula.
3. Etapa 3B - amplificação transcriptoma inteiros e microarray de células individuais
Extração e amplificação
- Use WTA2 Sigma kit (Cat # WTA2) para amplificar cDNA dupla fita de ambos os pellets e células individuais. As células individuais são "extraídos" através da primeira etapa do protocolo Sigma WTA2 de acordo com instruções do fabricante. Durante esta primeira etapa da incubação com tampão de síntese biblioteca a 37 ° C por 5 minutos rompe a membrana das células individuais e "extrai" a mensagem para a amplificação.
- Amplificar uma única célula em dois turnos, utilizando as orientações do kit WTA2 da Sigma-Aldrich. Realizar a etapa preparatória biblioteca de acordo com o protocolo do fabricante; em seguida, adicione 1 / 5 da amostra a 70 mL biblioteca da amplificação WTA2
Mix Master. Ampliar as amostras para 25 ciclos usando os parâmetros recomendados ciclismo. - Purificar o usi amostrasQIAquick ng Kit da Qiagen PCR Purification (Cat # 28104) e processo em Qiacube Qiagen, utilizando a "Limpeza de QIAquick reações de amplificação PCR padrão V4" protocolo.
- Concentrar as amostras purificadas a 5 mL, utilizando-se um vácuo, velocidade e submetido a uma segunda rodada de amplificação por 17 ciclos (WTA2 protocolo de amplificação repetida e parâmetros de bicicleta).
- Purificar amostras usando o PCR Purification Kit QIAquick e verificar a qualidade usando um espectrofotômetro NanoDrop e Bioanalyzer DNA da Agilent 7500 kit acordo com o protocolo do fabricante.
Rotulagem e Arrays NimbleGen Expressão Gênica
- G rótulo 2 de dupla fita de cDNA de cada amostra de células amplificado com um Kit de NimbleGen Labeling Color (Cat # 05223555001), de acordo com o protocolo do fabricante.
- Hibridizam 5 mg de amostra Cy3 rotulados para Mus musculus 12x135k, Arrays NimbleGen Expressão Gênica (Cat # 05543797001) according o protocolo do fabricante. Após a hibridização das amostras para as matrizes, os slides devem ser lavadas e secas antes que eles possam ser visualizados em um scanner laser.
- Gene Expression scan Arrays usando um scanner a laser. Aqui, nós usamos um Molecular Devices GenePix Scanner 4200A com configurações de 100POW, e 300-350PMT. Em seguida, gerar os arquivos par para cada matriz usando software NimbleScan NimbleGen e, posteriormente, analisar a expressão transcriptoma completo de cada matriz única célula (a tabela de forma estável típico expressa transcrições dos cardiomiócitos é incluído como um suplemento; S1 Tabela).
Se a perfusão do coração funcionava bem deve haver uma elevada percentagem de células cardíacas saudáveis que mantêm a sua morfologia típica retangular no isolamento. Se a perfusão não proceder bem, então vai haver uma grande percentagem de células mortas (veja imagens na Figura 5). Se as células estão corretamente amplificada usando os métodos WTA2 ou RT-STA on o produto final deve passar por testes de qualidade para controle posterior da qualidade de amostras de mRNA por NanoDrop e BioAnalyser (Figura 6). Para o fluxo de trabalho microarray, este é concluída após WTA de amplificação onde o mRNA é posta à prova com NanoDrop e Bioanalyzer. Representante resultados positivos para esta análise são mostrados na Figura 6. O WTA2 amostras deve mostrar uma amplificação robusta, com tanto a leitura do espectrofotômetro NanoDrop, bem como a leitura do chip electrofograma Bioanalyzer (BA) (Figura 6). Uma tabela de genes que foram detectados de forma estável através de microarray de uma única célula é incluído como um exemplo (Tabela S1). Para o processo de qPCR, a qualidade dos dados pode ser avaliada através da realização de uma etapa de fusão, no final da reação de PCR para garantir que os conjuntos de primers estão ampliando o produto desejado (Figura 7). Se correto, esta etapa de fusão deve gerar um pico específico derreter. Para ilustrar este ponto, um subconjunto de dados nanofluidic qPCR é mostrado em um mapa de calor de derreter pico temperatura (Figura 8) 8. É possível avaliar a qualidade de um produto de PCR pela sua temperatura de fusão para garantir a ausência de produtos não específicos 5. Cada linha deste mapa representa uma amostra de células (S1-S40) testaram em 43 ensaios de qPCR separado (A1-A43). Nesta figura, é claro que alguns ensaios são bastante variáveis e são provavelmente menos confiável do que os ensaios mais estável com maior especificidade da PCR.
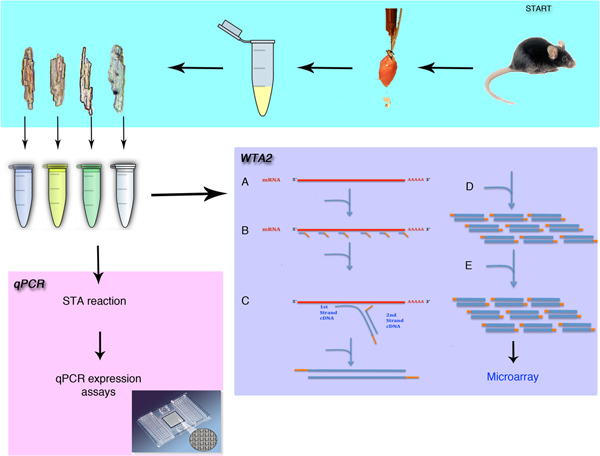
Figura 1. Visão geral do isolamento de células individuais e análise de expressão gênica. O procedimento irá demonstrar a remoção cirúrgica do coração do rato e do isolamento dos cardiomiócitos individual. Os métodos discutidos neste processo incluem métodos para qualquer qPCR ou análise de microarray após amplificação transcriptoma completo (WTA). O procedimento começa com o WTA de lise (A), em seguida, a ligação dos primers Sigma é universal (B) para o mRNApiscina. A extensão destes primers (C) ea ampliação de fase (D) criar microgramas de material amplificado. Esta amplificação é então repetido (E) para gerar material suficiente para o procedimento de microarray.

Figura 2. Representação da localização das incisões para remover o coração do mouse. Corte ao longo da parede lateral da caixa torácica (A), em seguida, corte ao longo da margem inferior da caixa torácica (B). Cortar os vasos acima e abaixo do coração permitir a remoção ao mesmo tempo deixando o suficiente da aorta para canulação do coração. Imagens adaptado do MJ Cozinhe 6.
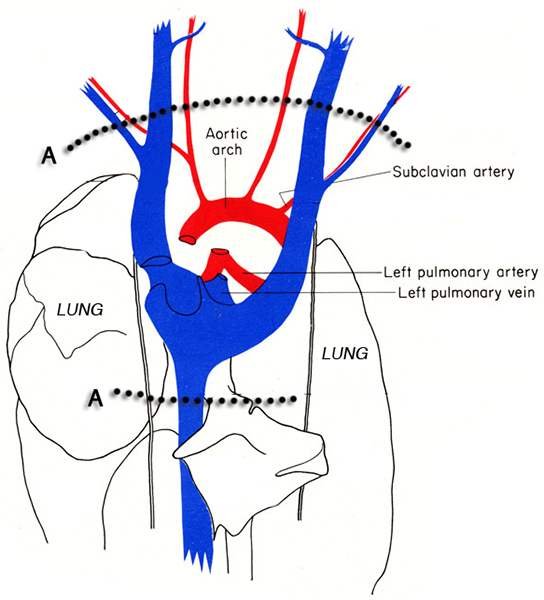
Figura 3. Diagrama do tórax do mouse. As linhas pontilhadas indicam os locais das incisões (A) para excisão do coração da cavidade torácica sem danificar a h tecido eart. Imagens adaptado do MJ Cozinhe 6.
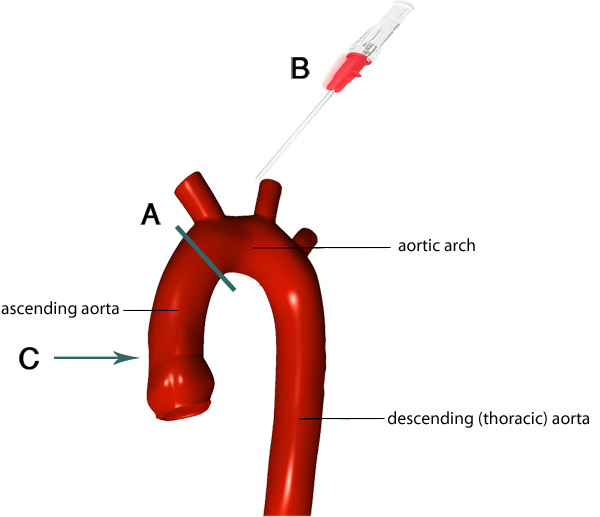
Figura 4. Imagem do arco aórtico e seus ramos. A linha cinza indica a localização ideal de onde se pode cortar a aorta (A). Restante da aorta junto ao próprio coração é canulados de modo que a ponta da cânula (B) vai para o nível apropriado dentro da aorta (C). Imagens adaptado de F. Gaillard 7.

Figura 5. Seleção de células individuais para análise de expressão gênica. Cada painel mostra cardiomiócitos fotografada ao microscópio óptico mostrando a morfologia típica de cardiomiócitos. Cardiomiócitos saudáveis são indicados pelas setas verdes. Células que são mortas ou a morrer são mostradas com setas vermelhas. Essas células mortas não devem ser utilizados na análise.
iles/ftp_upload/3302/3302fig6.jpg "alt =" Figura 6 "/>
Figura 6. Resultados de controle de qualidade WTA para a amplificação única célula. (A) Imagem de um espectrofotômetro de leitura NanoDrop que é apropriado para as transcrições amplificado a partir da reação WTA. (B) A leitura do chip de Bioanalyzer mostra os resultados de três células amplificado.
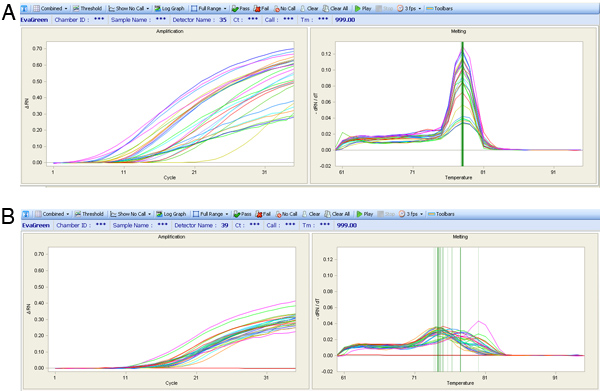
Figura 7. Curvas de amplificação de qPCR (gráfico à esquerda) e pico de derreter curvas de temperatura (gráfico à direita). (A) A expressão resulta de um ensaio de qualidade são mostrados com um único pico de fusão, apesar de curvas de amplificação diferente. (B) Melt curvas para um conjunto de primers pobres, que tem picos de fusão altamente variável que não é ideal para a análise de expressão.
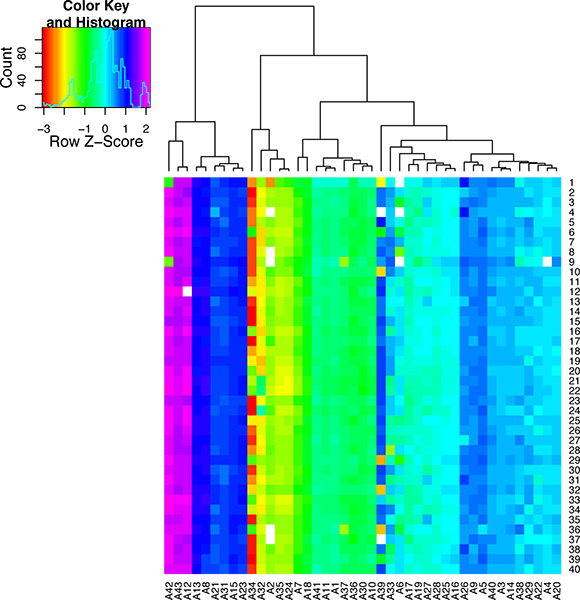
Figura 8. Qualidade resultados do controlo para o único celular qPCR reagiríons. Este mapa de calor foi criado para mostrar as temperaturas derretem como uma escala de cores. A temperatura de fusão de pico para cada ensaio qPCR (A1-A43) é mostrada por 40 células individuais (S1-40). Os ensaios foram agrupados de acordo com sua temperatura de fusão. Ao olhar para baixo na coluna para cada ensaio, é possível ver a variação derreter em todas as amostras. Este número demonstra que alguns ensaios qPCR são muito específicos, enquanto outros são altamente variáveis e, portanto, não são adequados para a análise única célula.
S1 mesa. Suplementar tabela de dados de microarrays. Estes genes são listados de acordo com a robustez em que são detectados em cardiomiócitos única através de um grande conjunto de dados. Esta lista gene indica a sensibilidade de detecção para um número de genes que normalmente são expressos em cardiomiócitos único.
Discussão
Este método tem a possibilidade de gerar cardiomiócitos para um número de funcionais, bem como estudos de expressão gênica. O exame de células individuais é uma área crescente na análise de expressão gênica. A vantagem de examinar os níveis de transcrição ao nível da célula é que ela permite o exame de uma população celular pura, que não é possível a partir de preparações de tecido inteiro. Além disso, a análise única célula permite a análise de variação estocástica dos níveis de mRNA em células individuais para definir populações de células que foram previamente pensado para ser 8,9 homogênea. Além de identificar genes que são potencialmente estocásticos em sua expressão genética 10,11,12, o método também permite a identificação de populações de células raras definidos por seus perfis de expressão gênica.
Embora este método oferece uma série de emocionantes usos potenciais em análise de expressão gênica, há somressalvas e considerações e na utilização de células individuais. A principal limitação para a análise única célula, é a coleção de células suficientes no experimento para alcançar significância estatística em relação à métrica de interesse, por exemplo variância. Nos casos em que o número de células isoladas do tecido de interesse não é uma limitação, pode-se examinar centenas de células por grupo, utilizando abordagens como a próxima geração de sequenciamento, ou matrizes nanofluidic. No entanto, em alguns casos, pode haver dificuldade na obtenção de células suficientes a partir do tecido de interesse. Isso pode em parte ser causada por procedimentos intensivos tempo idiossincrática para a recolha do tipo de célula-alvo. No entanto, um planejamento cuidadoso e preparação antes de prosseguir com a coleta de células individuais podem ainda permitir a análise de células rigorosa único com erro técnico limitado. Ao examinar os resultados deve-se considerar, que, apesar de este procedimento para isolar células tem sido utilizado em inúmeros Studies (incluindo microscopia, eletrofisiologia, etc), o isolamento em si poderia potencialmente efeito a expressão do gene. Como acontece com qualquer método que manipula as amostras biológicas, os resultados de suas descobertas devem ser cuidadosamente validada para garantir a expressão única célula é representativa do próprio tecido e viés não técnicos. Métodos, tais como a hibridização in situ pode ser útil para verificar esses resultados no tecido intacto. Por último, é fundamental para garantir que os dados gerados é cuidadosamente verificado para controle de qualidade. Os dados mostrados na Figura 8 demonstra que as análises de qPCR pode ser muito robusto em sua especificidade, como o ensaio # 13 (A13) ou ter um alto nível de variabilidade que pode levar a variância técnicas, tais como ensaio # 34 (A34).
Divulgações
Acesso livre e produção deste deste artigo é patrocinado Sigma-Aldrich.
Agradecimentos
Os autores gostariam de agradecer a assistência técnica da C. Zambataro durante as filmagens do presente protocolo. Agradecemos o apoio da Fundação Glenn de Pesquisa Médica (SM), a Fundação Hillblom, e os Institutos Nacionais de Saúde para um prêmio Centro de Choque Nathan (P30AG025708) e PO1AG025901. JMF foi apoiada por T32AG000266 adjudicado ao Instituto Buck de Pesquisa sobre o Envelhecimento.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| NaCl | Sigma-Aldrich | 71378 | |
| KCl | Sigma-Aldrich | 60128 | |
| Ácido pirúvico | Sigma-Aldrich | P4562 | |
| Hepes | Sigma-Aldrich | 54457 | |
| MgCl 2 | Sigma-Aldrich | M1020 | |
| NaH 2 PO 4 monobásico | Sigma-Aldrich | P9791 | |
| Dextrose | Sigma-Aldrich | G6721 | |
| NaOH | Sigma-Aldrich | 72068 | |
| EGTA | Sigma-Aldrich | O3777 | |
| CaCl 2 | Sigma-Aldrich | 21115 | |
| Protease, Tipo XIV | Sigma-Aldrich | P5147 | |
| Collaoxigenase, Tipo I | Worthington | C9891 | |
| 1x tampão Suspensão DNA (10 mM Tris, pH 8,0, 0,1 mM EDTA) | TEKnova, | PN T0221 | |
| BSA | Sigma-Aldrich | B8667 | |
| Pentobarbital sódico | Henry Schein Vet. * Oferta | Contactar com fornecedor para encomendar | * Devem ser adquiridos através de um veterinário ou animais de laboratório profissional |
| ExoSAP TI | Affymetrix / USB | 78201 | |
| CellsDirect Mix Rxn 2x | Invitrogen | 11737-030 | |
| SuperScript III RT Platinum Taq Mix | Invitrogen | 10928-042 | |
| RT-Mix STA Primer | IDT | Personalizado | |
| Água livre de nuclease | Sigma-Aldrich | W4502 | |
| WTA2 | Sigma-Aldrich | WTA2-50rxn | |
| QIAquick kit de purificação de PCR | Qiagen | 28104 | |
| Qiacube | Qiagen | 9001292 | |
| Espectrofotômetro NanoDrop | NanoDrop | 2000c | |
| Bioanalyzer DNA kit 7500 | Agilent | 7500 kit | |
| Um kit Labeling Cor | Roche-NimbleGen | 5223555001 | |
| Mus musculus série 12x135k | Roche-NimbleGen | 5543797001 | |
| GenePix 4200A Scanner | Dispositivos moleculares | 4200A | |
| TransPlex Kit Completo Amplification Whole Transcriptoma (WTA2 Kit) | Sigma-Aldrich | WTA2-50RXN |
Referências
- Wolska, B. M., Solaro, R. J. Method for isolation of adult mouse cardiac myocytes for studies of contraction and microfluorimetry. Am. J. Physiol. 271 (3 Pt 2), H1250-H1255 (1996).
- Shioya, T. A simple technique for isolating healthy heart cells from mouse models. J. Physiol. Sci. 57 (6), 327-335 (2007).
- Guo, G., Huss, M., Tong, G. Q., Wang, C., Li Sun, L., Clarke, N. D., Robson, P. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Dev. Cell. 18 (4), 675-685 (2010).
- Narsinh, K. H., Sun, N., Sanchez-Freire, V., Lee, A. S., Almeida, P., Hu, S., Jan, T., Wilson, K. D., Leong, D., Rosenberg, J., Yao, M., Robbins, R. C., Wu, J. C. Single cell transcriptional profiling reveals heterogeneity of human induced pluripotent stem cells. J. Clin. Invest. 121 (3), 1217-1221 (2011).
- D'haene, B., Vandesompele, J., Hellemans, J. Accurate and objective copy number profiling using real-time quantitative PCR. Methods. 50 (4), 262-270 (2010).
- Cook, M. J. The Anatomy of the Laboratory Mouse. Informatics. , (1965).
- Gaillard, F. Aorta. Radiopaedia. , (2008).
- Ståhlberg, A., Andersson, D., Aurelius, J., Faiz, M., Pekna, M., Kubista, M., Pekny, M. Defining cell populations with single-cell gene expression profiling: correlations and identification of astrocyte subpopulations. Nucleic Acids Res. 39 (4), e24-e24 (2011).
- Zhong, J. F., Chen, Y., Marcus, J. S., Scherer, A., Quake, S. R., Taylor, C. R., Weiner, L. P. A microfluidic processor for gene expression profiling of single human embryonic stem cells. Lab Chip. 8 (1), 68-74 (2008).
- Bahar, R., Hartmann, C. H., Rodriguez, K. A., Denny, A. D., Busuttil, R. A., Dollé, M. E., Calder, R. B., Chisholm, G. B., Pollock, B. H., Klein, C. A., Vijg, J. Increased cell-to-cell variation in gene expression in ageing mouse heart. Nature. 441 (7096), 1011-1014 (2006).
- Raj, A., van Oudenaarden, A. Nature, nurture, or chance: stochastic gene expression and its consequences. Cell. 135 (2), 216-226 (2008).
- Elowitz, M., Levine, A., Siggia, E., Swain, P. Stochastic gene expression in a single cell. Science. 297 (5584), 1183-1186 (2002).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados