Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La evaluación de alteraciones hepáticas metabólicas durante la colonización progresiva de la libre de gérmenes de ratón por 1 H espectroscopía de RMN
En este artículo
Resumen
El procedimiento se describe la colonización progresiva de seguir evaluando su impacto sobre el metabolismo hepático de acogida. La colonización se controla de manera no invasiva mediante la evaluación de la excreción urinaria de los metabolitos microbianos co-mediante RMN basados en los perfiles metabólicos, mientras que el metabolismo hepático es evaluado por alto ángulo Resolución Magic Spinning (MAS HR) de perfiles de RMN de la biopsia intactas.
Resumen
Es bien sabido que las bacterias intestinales contribuyen significativamente a la homeostasis de acogida, proporcionando una serie de beneficios tales como la protección inmunológica y la síntesis de la vitamina. También proveen el host con una considerable cantidad de nutrientes, por lo que este ecosistema un órgano metabólico esencial. En el contexto de la creciente evidencia de la relación entre la flora intestinal y el síndrome metabólico, la comprensión de la interacción metabólica entre el huésped y su microbiota intestinal se está convirtiendo en un reto importante de la biología moderna. 1-4
Colonización (también conocido como proceso de normalización) designa a la creación de micro-organismos en un antiguo libre de gérmenes de origen animal. Si bien es un proceso natural que ocurre al nacer, que también se utiliza en adultos libres de gérmenes animales de controlar el ecosistema floral intestinal y más determinar su impacto sobre el metabolismo de acogida. Un procedimiento común para controlar el proceso de colonización es utilizar el método de alimentación forzada con una single o una mezcla de microorganismos. Este método da como resultado una colonización muy rápida y presenta la desventaja de ser muy estresante 5. Por tanto, es útil para minimizar el estrés y para obtener un proceso de colonización más lenta para observar poco a poco el impacto de la creación de bacterias en el metabolismo de acogida.
En este artículo, se describe un procedimiento para evaluar la modificación del metabolismo hepático durante el proceso de colonización gradual mediante una técnica no destructiva del metabolismo de perfiles. Proponemos para controlar la colonización microbiana intestinal mediante la evaluación de la actividad metabólica microbiana intestinal refleja en la excreción urinaria de los metabolitos microbianos co-por un perfil de RMN basado metabólico. Esto permite una apreciación de la estabilidad de la actividad microbiana intestinal más allá del establecimiento estable del ecosistema microbiano intestinal suele evaluarse mediante el control de bacterias fecales por DGGE (electroforesis en gel de gradiente desnaturalizante). 6 Elcolonización se lleva a cabo en un ambiente abierto convencional y se inicia con una arena sucia contaminada con animales convencionales, que servirá como control. Los roedores son animales coprófagos, lo que garantiza una colonización homogénea a lo descrito previamente 7.
Perfiles metabólicos hepáticos se mide directamente a partir de una biopsia de hígado intacto con una H de alta resolución Magic Angle Spinning espectroscopía de RMN. Esta técnica semi-cuantitativa ofrece una forma rápida de evaluar, sin dañar la estructura celular, los principales metabolitos, como los triglicéridos, la glucosa y el glucógeno con el fin de estimar más la compleja interacción entre el proceso de colonización y el metabolismo hepático 70-10. Este método también se puede aplicar a cualquier tejido de la biopsia 11,12.
Protocolo
1. La colonización de animales libres de gérmenes y la recogida de muestras
- Eliminar animales libres de gérmenes de aisladores y de la casa en una sala de cría en jaulas convencionales equipados con filtro delante de los animales convencionales que servirán como controles (Figura 1).
- Mezclar medio de la camada (3 días) tomado de la caja de control convencional con la materia fecal de los animales libres de gérmenes. Siempre mantenga un tercio de la basura convencional sucia cada vez que hay que renovar con el fin de mantener un nivel de bacterias (mantener al menos durante 3 días).
- Recoger la orina en un microtubo de 1,5 ml por el manejo del ratón sobre el tubo y ayudar a la micción por un suave masaje en el intestino. Complemento congelar inmediatamente en nitrógeno líquido. Almacene al menos a -40 ° C hasta su análisis de RMN. Un volumen mínimo de 20 l se requiere para la adquisición de 5 mm de la sonda de RMN, pero se recomienda el uso de 30 l para mejorar la calidad de los perfiles metabólicos.
- Los animales deben be la eutanasia sin el uso de algún tipo de anestesia a fin de evitar resonancias de RMN de confusión debido a un metabolismo hepático de los compuestos anestésicos (por ejemplo, el uso dislocación cervical seguido por la confirmación de la muerte por desangramiento)
2. Recomendación para la recolección de la biopsia hepática
- No use ningún producto que contenga alcohol para evitar la contaminación. Lave las herramientas con solución de agua o solución salina.
- No perfore la vesícula biliar. En caso de fuga de bilis, lavar los tejidos inmediatamente con agua o solución salina.
- Recoger las biopsias de hígado (alrededor de 15-50 mg) en el lóbulo izquierdo, como se muestra en la Figura 2. Para las biopsias reproducible, recoger constantemente en el centro del lóbulo izquierdo evitando las zonas periféricas donde el tejido es más delgada.
- Ajustar las biopsias de congelación en nitrógeno líquido inmediatamente y almacenarlos a -80 ° C hasta el análisis de RMN.
3. 1H NMR adquisición de orina microvolumen
- Prepare el fosfato de sodio 0,2 M tampón en D2O (99,8%), pH 7,4, que contenía 1 mM 3 - (trimetilsilil) de ácido propiónico-d 4 (TSP).
- Mezclar 30 l de orina con 30 l de tampón fosfato de sodio.
- Transferencia de 50 l de solución mixta en 1,7 mm tubo de RMN capilar (Figura 3 (2)) con una jeringa de vidrio de 50 l equipado con una aguja de metal (OD 0.5 mm). Tenga cuidado para evitar las burbujas.
- Ajuste el adaptador capilar (Figura 3) (3) en la parte superior de la muestra de orina que contiene capilares y colocarlo en un microtubo de RMN de 2,5 mm por 5 mm de RMN de la sonda (Figura 3) (1). El uso de esta combinación de tubos como un tubo de RMN regular de 5 mm para la adquisición del espectro.
- Utilice la barra de extracción (Figura 3) (4) para eliminar capilar de 2,5 mm tubo de RMN, atornillando el adaptador capilar con cuidado para sacarlo.
. 4 1 H HR RMN MAS de tejido biopsia del hígado: la preparación de muestras
- MAS componentes del rotor y las herramientasdescritos en la Figura 4.
- Inserte una biopsia (alrededor de 15-50 mg) en el rotor de circonio (Figura 4) (1) y llenar el resto del volumen con puro O D 2 de RMN de bloqueo. Tenga cuidado de no hacer que las burbujas, porque esto altera la calidad de las cuñas posteriores y la calidad de la adquisición de datos.
- Introduzca 50 L espaciador de teflón (Figura 4) (2) utilizando el tornillo cilíndrico (Figura 4) (5). Desenroscar y calibrar usando el medidor de profundidad en el lado corto (Figura 4 (8)). En este paso, es importante prestar una atención específica a la muestra porque parte de ella puede filtrarse a través del orificio del espaciador. Si este es el caso, entonces parte de la biopsia se destruye y el peso de la muestra ya no es confiable. Lo que es necesario volver a empezar desde el principio la preparación de la muestra.
- Coloque el pasador THEAD (Figura 4) (3) y el tornillo suavemente con el destornillador (Figura 4 (6)). Secar el agua residual con un pedazo de tejido.
- Colocar la tapa (figura 4) (4) en elparte superior del rotor y la inserta en el empacador rotor (Figura 4 (6)). Presione firmemente hasta que la tapa está en su lugar. No debe haber ningún espacio entre el rotor y la tapa.
- Marca la mitad de la parte inferior del rotor con un rotulador negro para permitir la detección óptica de la velocidad de rotación.
- Coloque el rotor en el interior del espectrómetro de RMN y comenzar a girar a 5 kHz. Adquirir un espectro de RMN de H con CPMG secuencia de pulsos 13 de acuerdo con las directrices del fabricante.
- El uso de resonancia α glucosa anomérico a 5,22 ppm (doblete) para calibrar los espectros de RMN.
- Para descomprimir el rotor, proceder mediante la eliminación de la tapa con el removedor de la tapa (Figura 4 (9)). Desenroscar y quitar elementos thead pin espaciador de teflón con el tornillo cilíndrico. Lavar exhaustivamente con agua y detergente.
5. Resultados representante
La actividad microbiana del intestino pueden ser monitoreados utilizando un perfil metabólico urinario. Un gran número de microorganismos urinariosco-metabolitos identificados por RMN-1H han sido descritos en la literatura 7,14-17. Estos co-metabolitos microbianos son particularmente útiles para vigilar el proceso de colonización, ya que proporcionan una manera rápida y no invasiva para estimar que el ecosistema de reciente creación es estable. Figura 5A ilustra claramente la aparición de microbios intestinales co-metabolitos sobre el proceso de colonización. Esta cifra muestra un perfil metabólico urinario obtenerse por el procedimiento descrito en el paso 2 para un animal colonizó 20 días usando el procedimiento descrito en el Paso 1. Este animal no excretar sulfato indoxyl y cantidades muy pequeñas de phenylacetylglycine (PAG) y p-cresol sulfato en el estado libre de gérmenes (día 0-azul). Como la colonización avanza, estos tres marcadores del metabolismo de proteínas por la flora intestinal aumentan considerablemente para llegar a un equilibrio en el día 20 (rojo). Esto es particularmente fácil de controlar para un grupo de animales, como se ilustra en la Figura 5B con el PAGresonancia. Este diagrama se obtiene integrando el área bajo la resonancia con fondo gris en la Figura 5 (δ 7.40-7.43), que corresponde a una resonancia específica (triplete) de PAG de un grupo de 7 animales.
1 H de alta resolución angular Magic Spinning (MAS HR) espectroscopía de RMN es una técnica no destructiva que permite la adquisición rápida y reproducible de los perfiles metabólicos de cualquier tipo de biopsia 18. En este protocolo, se utilizó esta técnica de gran alcance para obtener un perfil metabólico hepático de los ratones antes de 2 (azul) y después (rojo) la colonización (Figura 6). Esta figura ilustra bien la información que se puede derivar de un MAS RMN basados en el perfil metabólico. Numerosos aminoácidos, así como los metabolitos derivados del metabolismo energético, como la glucosa, glucógeno, lactato, triglicéridos, (D)-3-hidroxibutirato y nicotinurate puede ser visualizada. Estos perfiles contienen información relevante para el estrés oxidativo (es decir, un ascórbicocid, el glutatión), el metabolismo de nucleótidos (es decir, inosina, uridina) y el metabolismo de metilamina (es decir, la colina, trimetilamina N-óxido). En este ejemplo, es muy claro que la libre de gérmenes del ratón muestra casi ningún glucógeno y cantidades muy bajas de glucosa y triglicéridos, se publicó anteriormente 7.
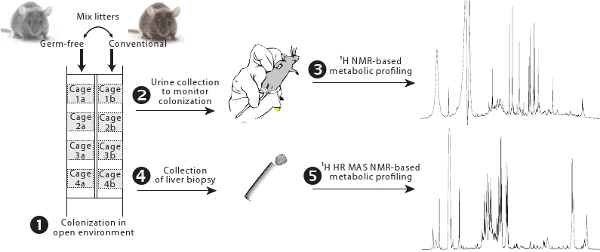
Figura 1. Descripción general del protocolo de la colonización. Animales libres de gérmenes y convencionales están alojados en jaulas equipadas con filtros de lado a lado y sus camadas se intercambian para permitir la colonización progresiva de la flora intestinal convencional (1). La actividad microbiana del intestino se controla mediante un perfil de RMN basado metabólico (2-3). El metabolismo hepático es evaluado por 1 H RMN MAS de recursos humanos basada en el perfil metabólico (4-5).
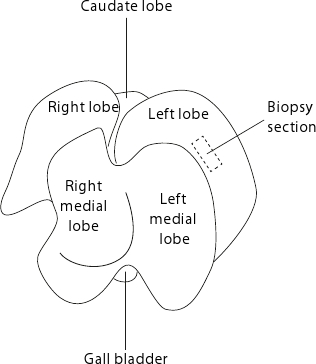
Figura 2. Ratón vivor anatomía. El hígado se muestra como la parte plana del órgano frente a la mesa. Para las biopsias reproducible, se recomienda recoger muestras siempre desde el centro del lóbulo izquierdo según lo indicado por el rectángulo de trazos.

Figura 3 1,7 mm kit RMN capilar para trabajar con microvolúmenes clave:.. 1: 2,5 mm microtubo RMN, 2: 1,7 mm tubo de RMN capilar, 3: adaptador capilar, 4: Extracción de la varilla.

. MAS Figura 4 rotor equipos clave: 1.: Rotor MAS, 2: 50 l espaciador de teflón, 3: Thead pines, 4: gorra, 5: tornillos cilíndricos, 6: destornillador, 7: rotor envasador, 8: medidor de profundidad.
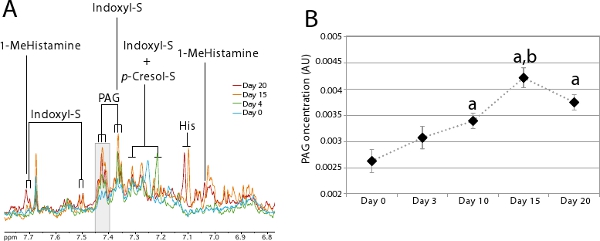
Figura 5. Evolución de los perfiles metabólico urinario durante colonization.
- Zoom en la región aromática del espectro de entre 6.8 a 7.8 ppm, donde co-metabolitos microbianos pueden ser visualizados. 1 H RMN se deriva de una sola persona en el día 0 (azul), 4 (verde), 15 (naranja) y 20 (rojo) post-colonización. La zona gris corresponde a la zona que se ha integrado para hacer el diagrama de la B. Principales: 1-MeHistamine: 1-metilhistamina; indoxil-S: El sulfato de indoxyl;:; Su p-cresol-S Histidina: sulfato de p-cresol, PAG : Phenylacetylglycine.
- Concentración de PAG promedio durante la colonización (n = 7). La t de Student se utilizó la prueba para comparar la diferencia en la concentración de PAG en diferentes puntos de tiempo: a: p
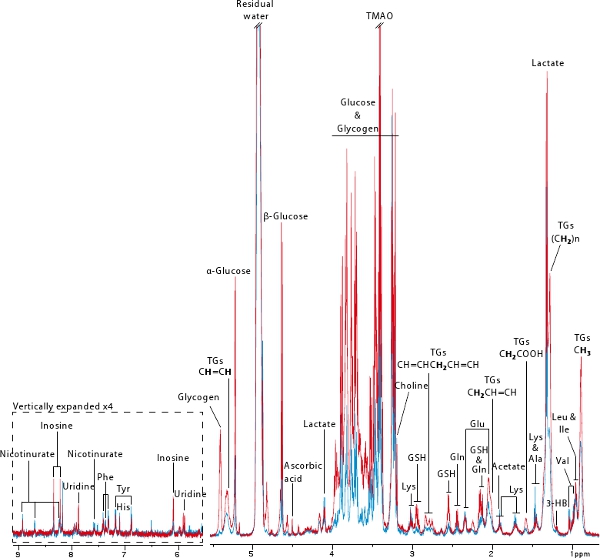
Figura 6. Típicos 600 MHz 1 H HR espectros de RMN MAS de las biopsias hepáticas derivadas de libres de gérmenes (azul) y ex-libre de gérmenes (red) los ratones. Protones en negrita son los responsables de la resonancia de triglicéridos clave:. 3-HB: 3-hidroxibutirato, GSH: glutatión reducido, TG: triglicéridos, OTMA: trimetilamina-N-óxido.
Discusión
En este protocolo, que describe un procedimiento de colonización progresiva en un entorno abierto para seguir investigando el impacto de la microbiota intestinal en el metabolismo hepático evaluado por 1 H RMN de perfiles de recursos humanos del MAS intacta biopsia. Varios métodos de colonización han sido descritos en la literatura. Los métodos más comunes para colonizar los animales con una microbiota definida son una sonda nasogástrica o beber agua contaminada 19,20. Inoculación fecal tam...
Divulgaciones
No tenemos nada que revelar.
Agradecimientos
Todos los espectros de RMN utiliza como ejemplos ilustrativos se derivan de un estudio publicado anteriormente 7, que fue apoyado financieramente por Nestlé.
Materiales
Tabla de reactivos y equipos específicos:
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
|---|---|---|---|
| 2,5 mm microtubo | Nueva Era | NE-H5/2.5-V-Br | |
| 1,7 mm tubo capilar | Sigma-Aldrich | NORS175001 | |
| Adaptador capilar | Nueva Era | NE-325-5/1.7 | |
| La extracción de la barra | Nueva Era | NE-341-5 | |
| HR-MAS rotor BL4 con 50 l esférica de teflón kit de espaciadores | Bruker | HZ07213 | |
| Kit de herramientas para 50 inserciones l | Bruker | B2950 | |
| Avance III 600MHz RMN | Bruker | ||
| 1 H HR MAS RMN sólidos sonda | Bruker | ||
| Óxido de deuterio 99,9% | Sigma-Aldrich | 530867-1L | |
| 3 - (trimetilsilil) propiónico ácido-d 4 (TSP) | Sigma-Aldrich | 269913 |
Referencias
- Cani, P. D., Delzenne, N. M. Gut microflora as a target for energy and metabolic. Curr. Opin. Clin. Nutr. Metab. Care. 10, 729-734 (2007).
- Ley, R. E., Turnbaugh, P. J., Klein, S., Gordon, J. I. Microbial ecology: human gut microbes associated with obesity. Nature. 444, 1022-1023 (2006).
- Raoult, D. Obesity pandemics and the modification of digestive bacterial flora. Eur. J. Clin. Microbiol. Infect. Dis. 27, 631-634 (2008).
- Turnbaugh, P. J., Backhed, F., Fulton, L., Gordon, J. I. Diet-induced obesity is linked to marked but reversible alterations in the mouse distal gut microbiome. Cell. Host. Microbe. 3, 213-223 (2008).
- Balcombe, J. P., Barnard, N. D., Sandusky, C. Laboratory routines cause animal stress. Contemp. Top. Lab. Anim. Sci. 43, 42-51 (2004).
- Muyzer, G., Smalla, K. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology. Antonie van Leeuwenhoek. 73, 127-141 (1998).
- Claus, S. P. Colonization-induced host-gut microbial metabolic interaction. MBio. 2, (2011).
- Waters, N. J. High-resolution magic angle spinning 1H NMR spectroscopy of intact liver and kidney: optimization of sample preparation procedures and biochemical stability of tissue during spectral acquisition. Anal. Biochem. 282, 16-23 (2000).
- Bollard, M. E. High-resolution 1H and 1H-13C magic angle spinning NMR spectroscopy of rat liver. Magnetic resonance in medicine. 44, 201-207 (2000).
- Lindon, J. C., Holmes, E., Nicholson, J. Pattern recognition methods and applications in biomedical magnetic resonance. Progress in Nuclear Magnetic Resonance Spectroscopy. 39, 1-40 (2001).
- Tate, A. R. Distinction between normal and renal cell carcinoma kidney cortical biopsy samples using pattern recognition of (1)H magic angle spinning (MAS) NMR spectra. NMR. Biomed. 13, 64-71 (2000).
- Wang, Y. Topographical variation in metabolic signatures of human gastrointestinal biopsies revealed by high-resolution magic-angle spinning 1H NMR spectroscopy. Journal of Proteome Research. 6, 3944-3951 (2007).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation times. The review of scientific instruments. 29, 688-691 (1958).
- Nicholson, J. K., Holmes, E., Wilson, I. D. Gut microorganisms, mammalian metabolism and personalized health care. Nat. Rev. Microbiol. 3, 431-438 (2005).
- Martin, F. P. Effects of probiotic Lactobacillus paracasei treatment on the host gut tissue metabolic profiles probed via magic-angle-spinning NMR spectroscopy. Journal of Proteome Research. 6, 1471-1481 (2007).
- Swann, J. R. Variation in Antibiotic-Induced Microbial Recolonization Impacts on the Host Metabolic Phenotypes of Rats. J. Proteome. Res. , (2011).
- Jacobs, D. M., Gaudier, E., van Duynhoven, J., Vaughan, E. E. Non-digestible food ingredients, colonic microbiota and the impact on gut health and immunity: a role for metabolomics. Curr. Drug. Metab. 10, 41-54 (2009).
- Beckonert, O. High-resolution magic-angle-spinning NMR spectroscopy for metabolic profiling of intact tissues. Nat. Protoc. 5, 1019-1032 (2010).
- Hooper, L. V., Sansonetti, P., Zychlinsky, A. . Methods in microbiology. 31, 559-589 (2002).
- Rahija, R. J., Fox, J. G. Ch. 7. The mouse in biomedical research. , 217-234 (2007).
- Goodwin, B. L., Ruthven, C. R., Sandler, M. Gut flora and the origin of some urinary aromatic phenolic compounds. Biochemical Pharmacology. 47, 2294-2297 (1994).
- Koopman, J. P. 'Normalization' of germfree mice after direct and indirect contact with mice having a 'normal' intestinal microflora. Lab Anim. 20, 286-290 (1986).
- Nishikata, N., Shikata, N., Kimura, Y., Noguchi, Y. Dietary lipid-dependent regulation of de novo lipogenesis and lipid partitioning by ketogenic essential amino acids in mice. Nutrition and Diabetes. 1, 1-12 (2011).
- Spagou, K. A GC-MS metabolic profiling study of plasma samples from mice on low- and high-fat diets. J. Chromatogr. B. Analyt. Technol. Biomed. Life. Sci. 879, 1467-1475 (2011).
- Sanchez-Patan, F., Monagas, M., Moreno-Arribas, M. V., Bartolome, B. Determination of microbial phenolic acids in human faeces by UPLC-ESI-TQ MS. J. Agric. Food. Chem. 59, 2241-2247 (2011).
- Roux, A., Lison, D., Junot, C., Heilier, J. F. Applications of liquid chromatography coupled to mass spectrometry-based metabolomics in clinical chemistry and toxicology: A review. Clin. Biochem. 44, 119-135 (2011).
- Ryan, D., Robards, K., Prenzler, P. D., Kendall, M. Recent and potential developments in the analysis of urine: a review. Anal. Chim. Acta. 684, 8-20 (2011).
- Nagayama, K., Wuthrich, K., Bachmann, P., Ernst, R. R. Two-dimensional J-resolved 1H n.m.r. spectroscopy for studies of biological macromolecules. Biochem. Biophys. Res. Commun. 78, 99-105 (1977).
- Aue, W. P., Bartholdi, E., Ernst, R. R. Two-dimensional spectroscopy. Application to nuclear magnetic resonance. J. Chem. Phys. 64, 2229-2246 (1975).
- Bodenhausen, G., Ruben, D. J. Natural abundance 15N NMR by enhanced heteronuclear spectroscopy. Chemical. Physics. Letters. 69, 185-189 (1980).
- Fan, T. W. -. M. Metabolite profiling by one- and two-dimensional NMR analysis of complex mixtures. Progress in nuclear magnetic resonance spectroscopy. 28, 161-219 (1996).
- Fan, T., Lane, A. Structure-based profiling of metabolites and isotopomers by NMR. Progress in Nuclear Magnetic Resonance Spectroscopy. 52, 48-48 (2008).
- Fonville, J. M. The evolution of partial least squares models and related chemometric approaches in metabonomics and metabolic phenotyping. Journal of Chemometrics. 24, 636-649 (2010).
- Merrifield, C. A. A metabolic system-wide characterisation of the pig: a model for human physiology. Mol. Biosyst. , (2011).
- Tugnoli, V. Molecular characterization of human gastric mucosa by HR-MAS magnetic resonance spectroscopy. International Journal of Molecular Medicine. 14, 1065-1071 (2004).
- Sitter, B. Comparison of HR MAS MR spectroscopic profiles of breast cancer tissue with clinical parameters. NMR Biomed. 19, 30-40 (2006).
- Beckonert, O. Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma, serum and tissue extracts. Nat. Protoc. 2, 2692-2703 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados