È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Valutare alterazioni metaboliche epatiche durante la colonizzazione progressiva di Germe senza mouse da 1 H spettroscopia NMR
In questo articolo
Riepilogo
Una procedura di progressiva colonizzazione è descritto per valutare ulteriormente il suo impatto sul metabolismo epatico ospitante. La colonizzazione è monitorato non invasivo, valutando l'escrezione urinaria di co-metaboliti microbici da NMR basati su profili metabolici, mentre il metabolismo epatico viene valutata ad alta risoluzione Angle Spinning Magic (MAS HR) profilazione NMR di intatto biopsia.
Abstract
E 'noto che i batteri intestinali contribuiscono significativamente alla omeostasi host, fornendo una serie di benefici come la protezione immunitaria e la sintesi della vitamina. Essi hanno inoltre fornire l'host con una notevole quantità di sostanze nutritive, facendo di questo ecosistema un organo metabolico essenziale. Nel contesto di una crescente evidenza del legame tra la flora intestinale e la sindrome metabolica, comprendere l'interazione metabolica tra l'host e la sua flora intestinale sta diventando una sfida importante della biologia moderna. 1-4
Colonizzazione (noto anche come processo di normalizzazione), designa la creazione di micro-organismi in un antico animale privo di germi. Mentre è un processo naturale che si verificano al momento della nascita, è utilizzato anche in adulti privo di germi animali per controllare l'ecosistema intestinale floreali e ulteriormente determinare il suo impatto sul metabolismo host. Una procedura comune per controllare il processo di colonizzazione è quello di utilizzare il metodo gavage con un singlE o una miscela di microrganismi. Questo metodo comporta una colonizzazione molto veloce e presenta lo svantaggio di essere estremamente stressante 5. E 'quindi utile per ridurre al minimo lo stress e per ottenere un processo di colonizzazione lento ad osservare gradualmente l'impatto di stabilimento sul metabolismo batterico ospite.
In questo manoscritto, si descrive una procedura per valutare la modifica del metabolismo epatico durante un processo di colonizzazione graduale utilizzando una tecnica non distruttiva profili metabolici. Noi proponiamo di monitorare la colonizzazione microbica intestinale, mediante la valutazione dell'attività microbica intestinale metabolica riflette l'escrezione urinaria di metaboliti microbici co-profilatura di 1 H NMR basati metabolica. Questo consente un apprezzamento della stabilità dell'attività microbica intestinale al di là della sede stabile l'ecosistema microbico intestinale solitamente valutata attraverso il monitoraggio dei batteri fecali da DGGE (gel elettroforesi denaturante gradiente). 6 Ilcolonizzazione si svolge in un ambiente convenzionale aperta ed è iniziata da una cucciolata sporca sporca da animali convenzionali, che servirà come controlli. Roditori sono animali coprofagi, questo garantisce una colonizzazione omogenea come descritto in precedenza 7.
Profiling metabolico epatico viene misurato direttamente da una biopsia del fegato intatto con 1 H ad alta risoluzione Magic Angle Spinning spettroscopia NMR. Questo semi-quantitativa tecnica offre un modo rapido per valutare, senza danneggiare la struttura delle cellule, i metaboliti importanti come trigliceridi, glucosio e glicogeno al fine di ulteriormente valutare la complessa interazione tra il processo di colonizzazione e il metabolismo epatico 7-10. Questo metodo può essere applicato a qualsiasi tessuto bioptico 11,12.
Protocollo
1. Colonizzazione di germi senza animali e la raccolta dei campioni
- Rimuovere sterile animali da isolatori e la casa in una stanza di allevamento in gabbie tradizionali dotati di filtro davanti alla animali convenzionali, che servirà come controlli (Figura 1).
- Mescolare mezzo della cucciolata (3 giorni) preso dalla gabbia di controllo convenzionale con la lettiera del germe senza animali. Tenere sempre 1 / 3 della lettiera sporca convenzionale ogni volta che è necessario rinnovarlo al fine di mantenere un livello di batteri (tenerlo almeno per 3 giorni).
- Raccogliere l'urina in un microtubo 1,5 ml gestendo il mouse sopra il tubo e aiutare minzione da massaggiare delicatamente l'intestino. Snap-congelare immediatamente in azoto liquido. Memorizzare almeno a -40 ° C fino al momento dell'analisi NMR. Un volume minimo di 20 l è necessario per l'acquisizione con una sonda NMR 5 millimetri, ma si consiglia di utilizzare 30 microlitri per migliorare la qualità dei profili metabolici.
- Gli animali devono be l'eutanasia, senza uso di anestetico per evitare risonanze NMR confusione a causa del metabolismo epatico di composti anestetici (per esempio, utilizzare dislocazione cervicale seguito da conferma della morte per dissanguamento)
2. Raccomandazione per la raccolta della biopsia epatica
- Non utilizzare alcun prodotto contenente alcol per evitare la contaminazione. Lavare gli attrezzi con acqua o soluzione salina.
- Non perforare cistifellea. In caso di perdita di bile, lavare tessuto immediatamente con acqua o soluzione salina.
- Raccogliere biopsie epatiche (circa 15-50 mg) dal lobo sinistro come mostrato nella Figura 2. Per le biopsie riproducibile, raccogliere costantemente al centro del lobo sinistro evitando le zone periferiche dove il tessuto è più sottile.
- Snap biopsie congelare in azoto liquido immediatamente e conservarli a -80 ° C fino al momento dell'analisi NMR.
3. Acquisizione 1 H NMR di urina microvolumi
- Preparare una soluzione di sodio fosfato 0,2 M tampone in D2O (99,8%), pH 7,4 contenente 1 mM 3 - (trimetilsilil) propionico-d 4 (TSP).
- Mescolare 30 ml di urina con 30 ml di tampone fosfato di sodio.
- Trasferire 50 ml di soluzione mista in tubo di 1,7 millimetri NMR capillare (Figura 3 (2)) usando una siringa da 50 microlitri di vetro dotato di un ago di metallo (diametro 0,5 mm). Attenzione ad evitare le bolle.
- Montare l'adattatore capillare (Figura 3 (3)) sulla parte superiore del campione di urina contenente capillare e mettetelo in un microtubo NMR 2,5 mm per 5 millimetri sonda NMR (Figura 3 (1)). Utilizzare questa combinazione di tubi come un normale tubo NMR 5 mm per l'acquisizione spettrale.
- Utilizzare l'asta di estrazione (Figura 3 (4)) per rimuovere capillare da 2,5 mm Tubo NMR avvitando l'adattatore capillare delicatamente per estrarlo.
. 4 1 H NMR HR MAS di tessuto biopsia epatica: la preparazione del campione
- MAS di componenti di rotori e gli strumenti sonodescritto nella Figura 4.
- Inserire biopsia (circa 15-50 mg) in zirconio rotore (Figura 4 (1)) e riempire il resto del volume con puro D 2 O per NMR di blocco. Fare attenzione a non fare le bolle, perché questo altererebbe la qualità della spessoramento successive e la qualità di acquisizione dei dati.
- Inserire 50 microlitri distanziatore in teflon (figura 4 (2)), utilizzando la vite cilindrica (figura 4 (5)). Svitare e calibrare usando il misuratore di profondità sul lato corto (figura 4 (8)). A questo punto, è importante prestare una specifica attenzione al campione perché una parte di esso può fuoriuscire attraverso il foro distanziatore. Se questo è il caso, allora parte della biopsia viene distrutta e il peso del campione non è più affidabile. E 'quindi necessario ricominciare dall'inizio la preparazione del campione.
- Posizionare il perno thead (Figura 4 (3)) e avvitare delicatamente con il cacciavite (Figura 4 (6)). Asciugare l'acqua residua con un pezzo di tessuto.
- Posizionare il tappo (figura 4 (4)) alsuperiore del rotore e di inserirlo nel packer rotore (Figura 4 (6)). Premere con decisione fino a quando il tappo è a posto. Non ci dovrebbe essere alcuno spazio lasciato tra il rotore e il suo tappo.
- Segna la metà del fondo del rotore con una penna nera marker per consentire il rilevamento ottico velocità di rotazione.
- Posizionare il rotore all'interno dello spettrometro NMR e iniziano a girare a 5 kHz. Acquisire 1 H NMR utilizzando CPMG sequenza di impulsi 13 secondo le linee guida del produttore.
- Utilizzare la risonanza glucosio anomerico α a 5,22 ppm (doppietta) per calibrare gli spettri NMR.
- Per decomprimere il rotore, procedere togliendo il tappo utilizzando il dispositivo di rimozione tappo (figura 4 (9)). Svitare thead pin e rimuovere distanziatore in Teflon con la vite cilindrica. Lavare accuratamente con acqua e detersivo.
5. Rappresentante Risultati
Attività microbica intestinale può essere monitorato tramite il profiling metabolico urinario. Un gran numero di microrganismi urinarico-metaboliti identificabili da 1 H NMR sono stati descritti in letteratura 7,14-17. Questi co-metaboliti microbici sono particolarmente utili per monitorare il processo di colonizzazione in quanto forniscono un modo rapido e non invasivo per valutare quando l'ecosistema di nuova costituzione è stabile. 5A figura illustra chiaramente l'aspetto del microbica intestinale co-metaboliti sul processo di colonizzazione. Questa figura mostra un profilo metabolico urinario ottenute seguendo la procedura descritta al punto 2 per un animale colonizzata 20 giorni utilizzando procedura descritta al punto 1. Questo animale non espellere qualsiasi solfato indossile e quantità molto piccola di phenylacetylglycine (PAG) e p-cresolo solfato presso il germe senza stato (giorno 0-blu). Come colonizzazione avanza, questi 3 marcatori del metabolismo delle proteine dalla flora intestinale aumenta notevolmente per raggiungere un equilibrio al giorno 20 (rosso). Ciò è particolarmente facile da monitorare per un gruppo di animali, come illustrato in Figura 5B con il PAGrisonanza. Questo diagramma è stato ottenuto integrando l'area sotto le risonanze evidenziate in grigio nella Figura 5A (δ 7,40-7,43), corrispondente ad una risonanza specifica (tripletta) di PAG per un gruppo di 7 animali.
1 H ad alta risoluzione Magia Angle Spinning (MAS HR) spettroscopia NMR è una tecnica non distruttiva che permette acquisizioni rapido e riproducibile di profili metabolici di ogni tipo di biopsia 18. In questo protocollo, abbiamo usato questa tecnica potente per ottenere un profilo metabolico epatico di 2 topi prima (blu) e dopo (rosso) colonizzazione (Figura 6). Questa figura illustra bene le informazioni che possono essere derivati da un MAS NMR basati su profilo metabolico. Numerosi aminoacidi e metaboliti derivati da metabolismo energetico come il glucosio, il glicogeno, lattato, trigliceridi, (D)-3-idrossibutirrato e nicotinurate possono essere visualizzati. Questi profili contengono anche informazioni utili per lo stress ossidativo (cioè un ascorbicocid, glutatione), il metabolismo dei nucleotidi (cioè inosina, uridina) e il metabolismo metilammina (cioè colina, Trimetilammina-N-ossido). In questo esempio, è molto chiaro che la priva di germi del mouse viene visualizzata quasi glicogeno e gli importi molto bassi di glucosio e trigliceridi, come è stato precedentemente pubblicato 7.
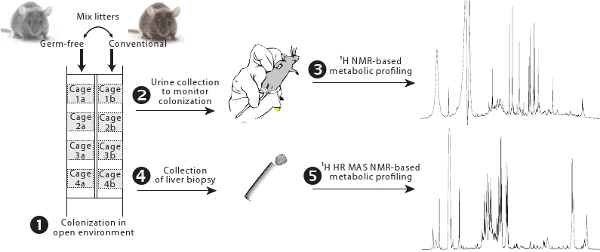
Figura 1. Panoramica del protocollo di colonizzazione. Animali germ-free e convenzionali sono alloggiati in gabbie dotate di filtri fianco a fianco e le loro cucciolate vengono scambiati per consentire progressiva colonizzazione dalla flora intestinale convenzionale (1). Attività microbica intestinale è controllata mediante 1 H NMR del profilo metabolico-based (2-3). Il metabolismo epatico è valutata da 1 H HR MAS NMR basati su profili metabolici (4-5).
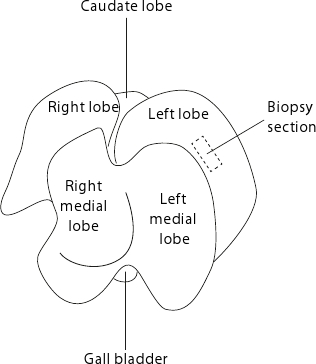
Figura 2. Topo vivor anatomia. Il fegato viene visualizzato come il lato piatto dell'organo di fronte al tavolo. Per le biopsie riproducibile, si consiglia di raccogliere campioni da sempre al centro del lobo sinistro, come indicato dal rettangolo tratteggiato.

Figura 3 1.7 mm Kit NMR capillare di lavorare con microvolumi chiave:.. 1: 2,5 microtubo NMR mm, 2: 1,7 mm Tubo NMR capillare, 3: adattatore capillare, 4: asta di estrazione.

. Figura 4 MAS attrezzature rotore chiave: 1.: Rotore MAS, 2: 50 microlitri distanziatore in Teflon, 3: THEAD pin, 4: cappuccio, 5: vite cilindrica, 6: cacciavite, 7: rotore packer, 8: profondimetro.
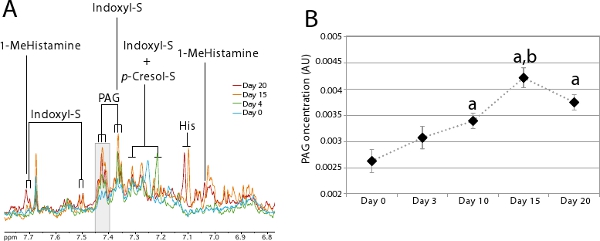
Figura 5. Evoluzione dei profili metabolici urinari durante colonization.
- Zoom sulla regione aromatica degli spettri tra 6,8-7,8 ppm in cui microbica co-metaboliti possono essere visualizzati. Spettri 1 H NMR sono stati ricavati da un singolo individuo, al giorno 0 (blu), 4 (verde), 15 (arancione) e 20 (rosso) post-colonizzazione. La zona grigia corrisponde alla zona che è stata integrata per rendere il diagramma in B. chiave: 1-MeHistamine: 1-methylhistamine; indossile-S: solfato indossile;:; suo p-cresolo, S Istidina: solfato di p-cresolo; PAG : Phenylacetylglycine.
- PAG concentrazione media durante la colonizzazione (n = 7). Student t-test è stato utilizzato per confrontare la differenza di concentrazione PAG a vari time-punti: a: p
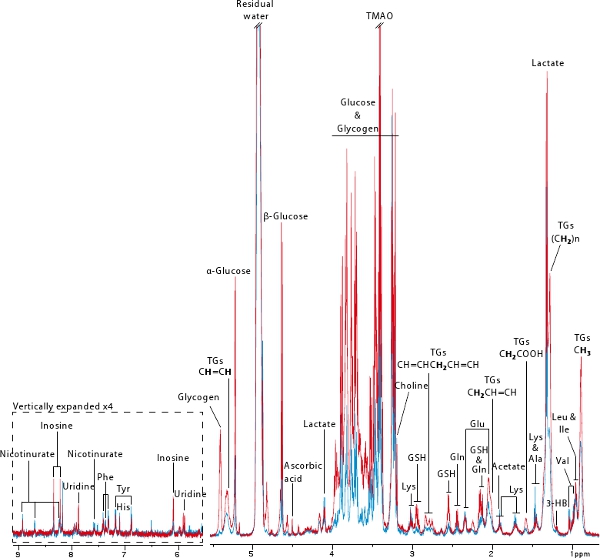
Figura 6. Tipiche 600 MHz 1 H HR spettri NMR MAS di biopsie epatiche derivate da privo di germi (blu) ed ex-priva di germi (rid) i topi. Protoni in grassetto sono responsabili per la risonanza trigliceridi chiave:. 3-MP: 3-idrossibutirrato, GSH: glutatione ridotto, TG: trigliceridi, TMAO: Trimetilammina-N-ossido.
Discussione
In questo protocollo, abbiamo descritto una procedura di progressiva colonizzazione in un ambiente aperto di approfondire l'impatto della flora intestinale sul metabolismo epatico valutate di 1 profiling H NMR HR MAS di intatto biopsia. Vari metodi di colonizzazione sono state descritte in letteratura. I metodi più comuni per colonizzare gli animali con un microbiota definiti sono sonda gastrica o l'acqua potabile contaminata 19,20. Inoculazione fecale può essere utilizzato anche come des...
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Tutti gli spettri NMR utilizzati come esempi illustrativi sono derivati da uno studio precedentemente pubblicato con il 7 che è stato sostenuto finanziariamente dalla Nestlé.
Materiali
Tabella di reagenti e attrezzature specifiche:
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti |
|---|---|---|---|
| 2,5 millimetri microprovette | New Era | NE-H5/2.5-V-Br | |
| 1,7 millimetri tubo capillare | Sigma-Aldrich | NORS175001 | |
| Adattatore capillare | New Era | NE-325-5/1.7 | |
| Estrazione asta | New Era | NE-341-5 | |
| HR-MAS BL4 rotore con 50 microlitri sferica Teflon spacer kit | Bruker | HZ07213 | |
| Kit di strumenti per 50 inserti microlitri | Bruker | B2950 | |
| Advance III 600MHz NMR | Bruker | ||
| 1 H HR MAS NMR solido sonda | Bruker | ||
| Ossido di deuterio 99,9% | Sigma-Aldrich | 530867-1L | |
| 3 - (trimetilsilil) propionico acido-d 4 (TSP) | Sigma-Aldrich | 269913 |
Riferimenti
- Cani, P. D., Delzenne, N. M. Gut microflora as a target for energy and metabolic. Curr. Opin. Clin. Nutr. Metab. Care. 10, 729-734 (2007).
- Ley, R. E., Turnbaugh, P. J., Klein, S., Gordon, J. I. Microbial ecology: human gut microbes associated with obesity. Nature. 444, 1022-1023 (2006).
- Raoult, D. Obesity pandemics and the modification of digestive bacterial flora. Eur. J. Clin. Microbiol. Infect. Dis. 27, 631-634 (2008).
- Turnbaugh, P. J., Backhed, F., Fulton, L., Gordon, J. I. Diet-induced obesity is linked to marked but reversible alterations in the mouse distal gut microbiome. Cell. Host. Microbe. 3, 213-223 (2008).
- Balcombe, J. P., Barnard, N. D., Sandusky, C. Laboratory routines cause animal stress. Contemp. Top. Lab. Anim. Sci. 43, 42-51 (2004).
- Muyzer, G., Smalla, K. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology. Antonie van Leeuwenhoek. 73, 127-141 (1998).
- Claus, S. P. Colonization-induced host-gut microbial metabolic interaction. MBio. 2, (2011).
- Waters, N. J. High-resolution magic angle spinning 1H NMR spectroscopy of intact liver and kidney: optimization of sample preparation procedures and biochemical stability of tissue during spectral acquisition. Anal. Biochem. 282, 16-23 (2000).
- Bollard, M. E. High-resolution 1H and 1H-13C magic angle spinning NMR spectroscopy of rat liver. Magnetic resonance in medicine. 44, 201-207 (2000).
- Lindon, J. C., Holmes, E., Nicholson, J. Pattern recognition methods and applications in biomedical magnetic resonance. Progress in Nuclear Magnetic Resonance Spectroscopy. 39, 1-40 (2001).
- Tate, A. R. Distinction between normal and renal cell carcinoma kidney cortical biopsy samples using pattern recognition of (1)H magic angle spinning (MAS) NMR spectra. NMR. Biomed. 13, 64-71 (2000).
- Wang, Y. Topographical variation in metabolic signatures of human gastrointestinal biopsies revealed by high-resolution magic-angle spinning 1H NMR spectroscopy. Journal of Proteome Research. 6, 3944-3951 (2007).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation times. The review of scientific instruments. 29, 688-691 (1958).
- Nicholson, J. K., Holmes, E., Wilson, I. D. Gut microorganisms, mammalian metabolism and personalized health care. Nat. Rev. Microbiol. 3, 431-438 (2005).
- Martin, F. P. Effects of probiotic Lactobacillus paracasei treatment on the host gut tissue metabolic profiles probed via magic-angle-spinning NMR spectroscopy. Journal of Proteome Research. 6, 1471-1481 (2007).
- Swann, J. R. Variation in Antibiotic-Induced Microbial Recolonization Impacts on the Host Metabolic Phenotypes of Rats. J. Proteome. Res. , (2011).
- Jacobs, D. M., Gaudier, E., van Duynhoven, J., Vaughan, E. E. Non-digestible food ingredients, colonic microbiota and the impact on gut health and immunity: a role for metabolomics. Curr. Drug. Metab. 10, 41-54 (2009).
- Beckonert, O. High-resolution magic-angle-spinning NMR spectroscopy for metabolic profiling of intact tissues. Nat. Protoc. 5, 1019-1032 (2010).
- Hooper, L. V., Sansonetti, P., Zychlinsky, A. . Methods in microbiology. 31, 559-589 (2002).
- Rahija, R. J., Fox, J. G. Ch. 7. The mouse in biomedical research. , 217-234 (2007).
- Goodwin, B. L., Ruthven, C. R., Sandler, M. Gut flora and the origin of some urinary aromatic phenolic compounds. Biochemical Pharmacology. 47, 2294-2297 (1994).
- Koopman, J. P. 'Normalization' of germfree mice after direct and indirect contact with mice having a 'normal' intestinal microflora. Lab Anim. 20, 286-290 (1986).
- Nishikata, N., Shikata, N., Kimura, Y., Noguchi, Y. Dietary lipid-dependent regulation of de novo lipogenesis and lipid partitioning by ketogenic essential amino acids in mice. Nutrition and Diabetes. 1, 1-12 (2011).
- Spagou, K. A GC-MS metabolic profiling study of plasma samples from mice on low- and high-fat diets. J. Chromatogr. B. Analyt. Technol. Biomed. Life. Sci. 879, 1467-1475 (2011).
- Sanchez-Patan, F., Monagas, M., Moreno-Arribas, M. V., Bartolome, B. Determination of microbial phenolic acids in human faeces by UPLC-ESI-TQ MS. J. Agric. Food. Chem. 59, 2241-2247 (2011).
- Roux, A., Lison, D., Junot, C., Heilier, J. F. Applications of liquid chromatography coupled to mass spectrometry-based metabolomics in clinical chemistry and toxicology: A review. Clin. Biochem. 44, 119-135 (2011).
- Ryan, D., Robards, K., Prenzler, P. D., Kendall, M. Recent and potential developments in the analysis of urine: a review. Anal. Chim. Acta. 684, 8-20 (2011).
- Nagayama, K., Wuthrich, K., Bachmann, P., Ernst, R. R. Two-dimensional J-resolved 1H n.m.r. spectroscopy for studies of biological macromolecules. Biochem. Biophys. Res. Commun. 78, 99-105 (1977).
- Aue, W. P., Bartholdi, E., Ernst, R. R. Two-dimensional spectroscopy. Application to nuclear magnetic resonance. J. Chem. Phys. 64, 2229-2246 (1975).
- Bodenhausen, G., Ruben, D. J. Natural abundance 15N NMR by enhanced heteronuclear spectroscopy. Chemical. Physics. Letters. 69, 185-189 (1980).
- Fan, T. W. -. M. Metabolite profiling by one- and two-dimensional NMR analysis of complex mixtures. Progress in nuclear magnetic resonance spectroscopy. 28, 161-219 (1996).
- Fan, T., Lane, A. Structure-based profiling of metabolites and isotopomers by NMR. Progress in Nuclear Magnetic Resonance Spectroscopy. 52, 48-48 (2008).
- Fonville, J. M. The evolution of partial least squares models and related chemometric approaches in metabonomics and metabolic phenotyping. Journal of Chemometrics. 24, 636-649 (2010).
- Merrifield, C. A. A metabolic system-wide characterisation of the pig: a model for human physiology. Mol. Biosyst. , (2011).
- Tugnoli, V. Molecular characterization of human gastric mucosa by HR-MAS magnetic resonance spectroscopy. International Journal of Molecular Medicine. 14, 1065-1071 (2004).
- Sitter, B. Comparison of HR MAS MR spectroscopic profiles of breast cancer tissue with clinical parameters. NMR Biomed. 19, 30-40 (2006).
- Beckonert, O. Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma, serum and tissue extracts. Nat. Protoc. 2, 2692-2703 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon