Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Sola célula de análisis de Bacillus subtilis Las biopelículas mediante microscopía de fluorescencia y citometría de flujo
En este artículo
Resumen
Biopelículas microbianas están constituidas generalmente por distintas subpoblaciones de células especializadas. Sola célula de análisis de estas subpoblaciones requiere el uso de reporteros fluorescentes. A continuación se describe un protocolo para visualizar y controlar varias subpopulationswithin B. subtilis Biopelículas utilizando microscopía de fluorescencia y citometría de flujo.
Resumen
La formación de biopelículas es un atributo general a casi todas las bacterias 1-6. Cuando las bacterias forman biopelículas, las células están encerradas en una matriz extracelular que está mayoritariamente constituida por las proteínas y exopolisacáridos, entre otros 7-10. La comunidad microbiana encerrado dentro de la biopelícula a menudo muestra la diferenciación de las distintas subpoblaciones de células especializadas 11-17. Estas subpoblaciones coexisten y se muestran a menudo la organización espacial y temporal dentro del biofilm 18-21.
La formación de biopelículas en los Bacillus subtilis organismo modelo requiere la diferenciación de las distintas subpoblaciones de células especializadas. Entre ellos, la subpoblación de los productores de matriz, responsables de producir y secretar la matriz extracelular de la biopelícula es esencial para la formación de biopelículas 11,19. Por lo tanto, la diferenciación de los productores de la matriz es una característica de la formación de biopelículas en la B. subtilis.
Hemos utilizado los reporteros fluorescentes para visualizar y cuantificar la subpoblación de los productores de la matriz en las biopelículas de B. subtilis 15,19,22-24. Concretamente, hemos observado que la subpoblación de los productores de matriz diferencia en respuesta a la presencia de auto-producido señal extracelular surfactina 25. Curiosamente, surfactina es producida por una subpoblación de células especializadas diferentes de la subpoblación de los productores de matriz 15.
Hemos expuesto en este informe, el enfoque técnico necesario para visualizar y cuantificar la subpoblación de los productores de la matriz y los productores de surfactina en las biopelículas de B. subtilis. Para hacer esto, los reporteros fluorescentes de los genes necesarios para la producción de matriz y la producción de surfactina se insertan en el cromosoma de B. subtilis. Los reporteros se expresan únicamente en una subpoblación de células especializadas. Entonces, las subpoblaciones puede sercontrolará mediante microscopía de fluorescencia y citometría de flujo (ver Fig. 1).
El hecho de que diferentes subpoblaciones de células especializadas coexisten dentro de las comunidades de bacterias multicelulares nos da una perspectiva diferente sobre la regulación de la expresión génica en procariotas. Este protocolo trata este fenómeno de manera experimental y puede ser fácilmente adaptado a cualquier modelo de otros tratamientos, para dilucidar los mecanismos moleculares que subyacen a la heterogeneidad fenotípica dentro de una comunidad microbiana.
Protocolo
1. Etiquetado de B. subtilis y ensayo de formación de biopelículas
- Amplificar por PCR la región promotora del gen de interés. Se muestra como ejemplo la clonación de P tapa, el promotor de los genes responsables de la producción de TASA proteína de la matriz 26. Clon P tapa en pkm008 vector (creado por el laboratorio Rudner, Escuela Médica de Harvard. Boston, EE.UU.) (Fig. 2).
- Alineado de los plásmidos mediante digestión enzimática (Enzyme recomendada, XhoI).
- Inducir la competencia natural en el B. subtilis cepa 168, siguiendo el protocolo de un solo paso se ha descrito previamente por Harwood y de corte 27.
- Añadir los plásmidos linealizadas en el cultivo de células competentes y seleccionar para resistencia a la espectinomicina, después de dos horas de incubación.
- Las cepas obtenidas ha insertado el constructo en el locus amyE neutra de B. subtilis por recombinación doble (Fig. 4). Para crearuna cepa de doble etiquetado, integrar el segundo reportero en el lugar lacA neutra mediante el uso de la pDR183 plásmido se presenta en la figura 3. Insertar este reportero usando la misma técnica que se ha descrito anteriormente para la inserción de reporteros clonado en pKM008.
- Transferir el reportero de la cepa 168 a NCIB3610 que es capaz de formar biopelículas. Utilice la transducción de fago SPP1 protocolo de 28,29. Crece la tensión de los donantes en medio TY (LB + 10 mM MgSO 4 +10 mM MnSO 4). Mezclar 200 l de la cultura con 100 l de la dilución de stock del fago. Añadir 3 ml de agar blando después de 30 minutos de incubación y permitir que los halos de fagos que surgen a 37 º C.
- Recoger el agar blando. Centrifugar y pasar el sobrenadante cree una jeringa de 0,22 micras filtro. Utilizar el sobrenadante para infectar un cultivo de la cepa receptora crecido en medio TY. Añadir 30 l de 10 ml de cultivo diluido 1:10. Incubar durante 30 min y seleccionar para resistencia a los antibióticos después de 24 h de incubación.
- Senar una colonia y crecer durante la noche en LB a 37 ° C.
- Spot 3 l de cultivo de una noche en el sólido biopelícula inductor medio MSgg 1,5% de agar 30. Permitir que las células crezcan durante 72 horas a 30 ° C (Figura 3). Después de tres días de crecimiento, las biopelículas formadas sobre la superficie del agar MSgg desarrollado una arquitectura morfológica complejo en la superficie del agar.
2. Biofilm dispersión y fijación de la célula
- Retire la forma biofilm de la superficie del agar MSgg utilizando un palillo de dientes o pinzas. La consistencia de la biopelícula debería permitir a la cáscara que fuera desde la superficie del agar en una sola pieza.
- Colocar la biopelícula en 3 ml de tampón PBS y dispersar la biopelícula por el paso repetitivo a través de una pipeta o una aguja. Alternativamente, la dispersión de la biopelícula se puede hacer uso de sonicación suave. Sonicación leve requiere de 12 impulsos con una potencia de 3 y la amplitud de 0,7 segundos.
- Fijar las muestras antes de células único análisis. Resuspensiónd 300 l de la suspensión de células en 1 ml de solución de paraformaldehído al 4% y se incuba a temperatura ambiente durante exactamente siete minutos.
Composición de la solución de paraformaldehído al 4%:
2 g de paraformaldehído
50 ml de tampón PBS
4 l NaOH 10 N
Filtrar la solución a través de un filtro de 0,22 um y alícuota - Lavar las células después de la fijación en tampón PBS tres veces y resuspender en 300 L de tampón PBS.
3. Microscopía de fluorescencia
- Vierta 200 l de agarosa al 0,8% sobre un portaobjetos de microscopio y con cuidado se cubre con otra diapositiva. Quitar la corredera superior suavemente después de 2 minutos para obtener una capa de agarosa unida a la corredera de la parte inferior.
- Punto 2 l de células fijadas en la superficie de la capa de agarosa y se cubre con una cubierta de vidrio microscopio.
- Colocar la muestra en el microscopio de fluorescencia. Nosotros usamos un microscopio de fluorescencia Leica DMI6000B equipado con una Leica iIluminat CRT6000iónico del sistema. Los filtros son para YFP Ejemplo: BP500/20, Em: BP535/30 y PPC son Ejemplo: BP426/20, Em: 480/40
- Expone su muestra a una fluorescencia de excitación entre 50-200 ms. Establecer el período de excitación de acuerdo con un control negativo que no muestra ninguna fluorescencia en las condiciones seleccionadas para el experimento.
- Consulte la imagen de fluorescencia para la misma imagen obtenida con campo claro. Combinar las dos imágenes en una sola imagen. Los resultados obtenidos a partir de microscopía de fluorescencia de flujo utilizando una cepa de una sola etiqueta que alberga el reportero P tapa-YFP están representados en la figura 6.
4. La cuantificación de las células individuales Usando citometría de flujo
- Dispersar la muestra de las células fijas que utilizan ultrasonidos leve. Soníquelos la muestra realizando 2 serie de 12 impulsos con una salida de 5 y la amplitud de 0,7 segundos, para dispersar terrones en células individuales sin causar la lisis celular. Confirmar la eficacia de dispersión de células por microscopía óptica.
- Diluir las muestras 1:100 en tampón PBS antes del análisis de citometría de flujo. Usamos un citómetro de flujo BD FACS Canto II. Para YFP fluorescencia, utilice una excitación láser a 488 nm, junto con un filtro de 530/30. Para PPC fluorescencia, utilice la excitación láser a 405 nm, junto con un filtro de 408/40.
- Calibrar la máquina citómetro de flujo con dos controles negativos. Una muestra de tampón PBS sin células en suspensión debe servir como control negativo para el tamaño de las partículas detectada por el citómetro de flujo. Una muestra marcada con fluorescencia ningún reportero debe servir como control negativo para la sensibilidad de fluorescencia del citómetro de flujo.
- Coloque la muestra marcada con el reportero de fluorescencia en el citómetro de flujo. Para cada muestra, analizar por lo menos 50,000 eventos con un caudal de entre 300 y 3.000 eventos por segundo.
- Captura de datos mediante FACS Diva de software (BD Biosciences) y analizarla con FlowJo 8.5.2 del software. Consulte las señales de fluorescencia en el control que muestrans sin fluorescencia.
- Presentar los datos de seguimiento de la señal de fluorescencia de una sola cepa marcado en un gráfico de dos ejes. Trazar la señal de fluorescencia detectada en el eje X y el número de células que expresan los diferentes niveles de fluorescencia en el eje Y. Los resultados obtenidos a partir del análisis de citometría de flujo utilizando una cepa de una sola etiqueta que alberga el reportero P tapa-YFP están representados en la figura 8.
- Presentar los datos de seguimiento de la señal de fluorescencia de una cepa de doble etiquetado en un gráfico de tres ejes. Trace la señal de fluorescencia de cada uno de los canales monitoreados en los ejes X e Y (por ejemplo, las buenas prácticas agrarias se mide en el eje X y de la PPC en el eje Y). Parcela en el eje Z en el número de células que expresan cada reportero y presentarlos como isolíneas contorno que serían perpendicular al plano del papel (Fig. 9 y 10).
4. Los resultados representativos
Cuando B. subtilis crece en una placa de la biofilm inductor MSgg medio, la formación de biopelículas se observa después de tres días de incubación a 30 ° C 30. La biopelícula muestra consistencia fuerte y podía despegarse de la superficie del agar en una sola pieza. Además, la biopelícula se muestra una arquitectura compleja morfológica que es indicativo de las subpoblaciones de células diferenciadas que participan (Fig. 5). Por ejemplo, la producción de la matriz extracelular en los resultados de biopelículas en la formación de arrugas en la superficie de la colonia. Esta característica puede ser correlacionada con la diferenciación de la subpoblación de los productores de la matriz 19. De manera similar, la elevación de las estructuras aéreas en la superficie de la biopelícula es indicativo de la presencia de una subpoblación de células esporulantes, ya que las esporas localizados en el área apical de estas estructuras 30.
Ejemplos de visualización de la diferenciación celular en una sola r-etiquetados y son una cepa de doble etiquetado mediante microscopía de fluorescenciaepresented en la figura 6 y 7, respectivamente. La única cepa marcada alberga el reportero fluorescente P tapa-PPC, que se expresa en la subpoblación de células productoras de matriz. Esta subpoblación es responsable de producir y secretar la matriz extracelular que constituye la biopelícula (fig. 6). La cepa de doble etiquetado alberga el reportero P tapa-PPC 26 y el reportero de la categoría P-YFP srfAA 31. Este segundo reportero permite monitorizar la subpoblación de las células responsables de secretar la surfactina molécula de señalización, lo que desencadena la cascada de señalización a la diferenciación de los productores de la matriz (Fig. 7). Subpoblación de los productores de la matriz son falsas de color azul, mientras que la subpoblación de los productores de surfactina es falso color amarillo.
Análisis por citometría de flujo utilizando una cepa de una sola etiqueta que alberga el reportero de P tapa-YFP se presenta en la figura 8. Cepa control sin tratar no albergarlos genes de proteínas fluorescentes mostró una fluorescencia única población relativamente baja. Las células que albergan P tapa-YFP en no inducir condiciones de biofilm no diferenciaba la subpoblación de los productores de la matriz y la población en su conjunto mostraron fluorescencia relativa baja. En condición de biopelícula inductor, una subpoblación de células con fluorescencia relativa alta ocurrido, observada como un hombro a la derecha de la baja pico de fluorescencia relativa 23.
Los resultados obtenidos a partir del análisis de citometría de flujo utilizando una cepa de doble etiquetado están representados en la figura 9 y 10. Figura 9 de cerca las subpoblaciones de los productores de la matriz y los productores de surfactina con la cepa de doble etiquetado P tapa-PPC, P srfAA-YFP. En cuanto al control de la fluorescencia de fondo se utilizó una cepa no albergar ningún genes de proteínas fluorescentes. A continuación, se detecta cada subpoblación de los productores de la matriz y los productores de surfactina en cada canal de fluorescencia, utilizando la cepa de una sola etiqueta s como controles. La cepa de doble etiquetado P tapa-PPC, P-YFP srfAA mostró dos subpoblaciones de células que expresan altos niveles de los reporteros fluorescentes. Cada población se enmarca, mostrando que no hay solapamiento en la expresión de los periodistas entre las dos subpoblaciones de células especializadas 15. Del mismo modo, el análisis de citometría de flujo utilizando doble etiquetado cepa P SKF-YFP, P tapa-YFP se presenta en la figura 10. El reportero de la SKF gen controla la diferenciación de la subpoblación de caníbales 32, que se ha descrito para diferenciar de forma coordinada con la subpoblación de 24 productores de la matriz. En este caso, la cepa de doble etiquetado mostró una única subpoblación de células fluorescentes que expresan tanto YFP y PPC. Esto indicó que ambas vías de diferenciación de células se activan coordinadamente en la subpoblación mismo.
1.jpg "/>
Figura 1. Esquema general del experimento. El protocolo se divide en tres pasos principales. El primer paso requiere etiquetado la cepa de B. subtilis con la fusión periodista que supervisa la subpoblación de interés. En segundo lugar, crecen cepas etiquetados en condiciones de inducción de biofilm. En tercer lugar, dispersan la biopelícula y llevar a cabo análisis de células individuales de la población con un microscopio de fluorescencia y citometría de flujo.

Figura 2. Representación esquemática de la pKM008 vector de integración. Este vector se integra la fusión reportero de interés en el amyE lugar neutral por doble recombinación. El promotor de interés (P tapa) se clona en el vector usando los sitios de restricción EcoRI y HindIII. Entonces, la expresión del gen PPC está bajo el control del promotor P tapa. El orientatión de los genes en el plásmido se representa como una flecha.
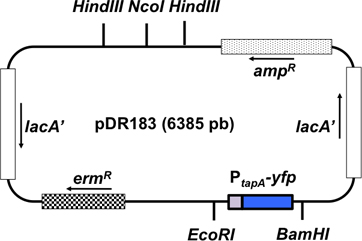
Figura 3. Representación esquemática del vector de integración pDR183. Este vector se integra la fusión reportero de interés en el neutro lacA lugar por recombinación doble. La fusión de interés (P tapa - PPC) se clona en el vector usando los sitios de restricción EcoRI y BamHI. La orientación de los genes en el plásmido se representa como una flecha.

Figura 4. Esquema de la integración de los reporteros en el cromosoma de B. subtilis por recombinación doble. (A) B. subtilis cromosoma tiene dos loci neutrales, amyE y Laca, para integrar fusiones reportero sin afectar el desarrollo de la biopelícula. (B) Proceso de recombinación doble de pKM008. Plásmido linealizado se integra en el genoma de B. subtilis por recombinación doble. La fusión reportero se integra en el lugar geométrico neutro de una manera estable.

Figura 5. La formación de biopelículas de B. subtilis NCIB3610. Proceso de formación bioflm de la cepa B. subtilis NCIB 3610, cuando crecen en el biofilm MSgg medio de inducción durante tres días a 30 ° C. Imágenes secuenciales del desarrollo de la biopelícula se tomaron cada 12 h.

Figura 6. La visualización de la subpoblación de los productores de la matriz bajo el microscopio de fluorescencia. Una muestra de un biofilm de B. subtilis P Tapa - PPC se ha fijado y se examina bajo el microscopio de fluorescencia. 200 ms de la fluorescenciaexcitación evidenció una subpoblación de células que emiten fluorescencia más alto que el resto de las células. Se consideró que esta subpoblación en la subpoblación de células productoras de matriz. Barra de escala es de 3 m.
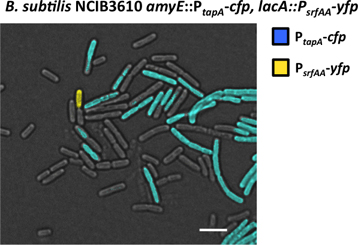
Figura 7. La visualización de la subpoblación de los productores de la matriz y los productores de surfactina bajo el microscopio de fluorescencia. Muestra de un biofilm de la B. subtilis P tapa-PPC, P-YFP srfAA cepa de doble etiquetado se fijó y se examina bajo el microscopio de fluorescencia. Tiempo de exposición de 250 ms evidenciadas dos subpoblaciones de células que emiten fluorescencia más alto que el resto de las células. Una subpoblación expresó YFP y ha sido detectado en exclusiva a través del canal YFP (falso color amarillo). Esta es la subpoblación de productores surfactina. Otra subpoblación expresó PPC y que fue detectado exclusivamente en el chann PPC el. Esta es la subpoblación de los productores de la matriz. Barra de escala es de 3 m.
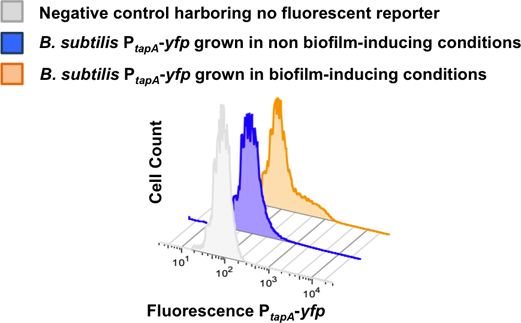
Figura 8. La cuantificación de la subpoblación de los productores de matriz utilizando 2-D citometría de flujo. Dispersed células de una biopelícula de B. subtilis P tapa-YFP fueron controlados mediante citometría de flujo. El citómetro de flujo contado 50.000 eventos y la señal de fluorescencia para cada evento fue monitoreada. Número de células contadas se representa en el eje Y mientras que la intensidad de señal YFP se representa en el eje X. Las células fueron cultivadas en medio LB para obtener no biofilm condiciones de inducción. Las células fueron cultivadas en medio MSgg para obtener inductores de biofilm condiciones. La subpoblación de los productores de la matriz se diferencia sólo en condiciones de inducción de biofilm. Esta cifra se ha adaptado de López et al., PNAS (2009) 106 :280-285.
s/ftp_upload/3796/3796fig9.jpg "/>
Figura 9. La cuantificación de la subpoblación de los productores de la matriz y los productores surfactina en 3-D utilizando citometría de flujo. Señal de fluorescencia de los canales monitorizados se presentan en el eje X (para YFP) y el eje Y (para PPC). El eje Z mide el número de células que expresan cada reportero y se cuantificó como isolíneas contorno perpendiculares al plano del papel. El número de eventos monitoreados en este experimento fue de 50.000 eventos. Panel superior izquierdo presenta un control de la fluorescencia de fondo no albergar los genes de proteínas fluorescentes. Panel superior derecho detecta la subpoblación de los productores de surfactina en el canal de YFP fluorescencia (enmarcado en amarillo) con una sola etiqueta cepa P srfAA-YFP. El panel inferior izquierdo detecta la subpoblación de los productores de la matriz en el canal de la PPC fluorescencia (enmarcada en azul), utilizando una cepa de una sola etiqueta P tapa-PPC. El doble etiquetado cepa P tapa </ Em>-PPC, P srfAA-YFP se supervisará en el panel inferior derecho. Mostraba a dos subpoblaciones que se enmarcan en amarillo y azul. Esta cifra se ha adaptado de López et al., Genes and Development (2009) 23 :1631-1638.
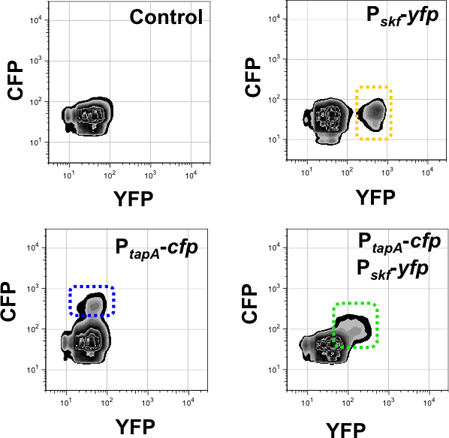
Figura 10. La cuantificación de la subpoblación de los productores de matriz y caníbales en 3-D utilizando citometría de flujo. Señal de fluorescencia de los canales monitorizados se presentan en el eje X (para YFP) y el eje Y (para PPC). El número de eventos monitoreados en este experimento fue 50,000. Panel superior derecho detecta la subpoblación de caníbales en el canal de YFP fluorescencia (enmarcado en amarillo) en una sola cepa marcada con P de SKF-YFP. El panel inferior izquierdo detecta la subpoblación de los productores de la matriz en el canal de la PPC fluorescencia (enmarcada en azul) en una sola cepa marcada con P tapa-PPC. El dOBLE marcado con la cepa P tapa-PPC, P SKF-YFP mostró sólo una subpoblación de células en diagonal a los ejes X e Y (enmarcado en verde). Esta subpoblación se detecta en el canal de YFP y la PPC, ya que expresa las dos periodistas de forma simultánea. López et al, Genes and Development (2009) 23 :1631-1638.
Discusión
El hecho de que las comunidades bacterianas muestran las subpoblaciones de células que expresan el conjunto específico de genes evidencia la complejidad de las comunidades microbianas 33,34. Este protocolo debe ayudar a determinar si la expresión de cualquier gen de interés se limita a una subpoblación particular de células especializadas dentro de la comunidad microbiana. La visualización de estas subpoblaciones requiere el desarrollo de nuevas técnicas, porque los métodos tradicionales para control...
Divulgaciones
No tenemos nada que revelar.
Agradecimientos
Este trabajo está financiado por el Programa de Investigación Investigador Joven, del Centro de Investigación de Enfermedades Infecciosas (Zinf) de la Universidad de Würzburg. Juan C García-Betancur es un compañero de doctorado de la Escuela de Graduados en Ciencias de la Vida (GSL) de la Universidad de Würzburg.
Materiales
| Name | Company | Catalog Number | Comments |
| Técnica | Nombre del reactivo | Empresa | Número de catálogo |
| MSgg composición | 5 mM de fosfato de potasio | Roth | 6878 |
| MOPS 100 mm | Sigma-Aldrich | M1254 | |
| Cloruro de magnesio 2 mM | Roth | 2189.1 | |
| 700μM de cloruro de calcio | Roth | A119.1 | |
| 50 micras de cloruro férrico | Sigma-Aldrich | 157740 | |
| 1um cloruro de cinc | Applichem | A2076 | |
| La tiamina 2μM | Sigma-Aldrich | 74625 | |
| El glicerol 0,5% | Roth | 7533 | |
| Glutacompañero de 0,5% | Sigma-Aldrich | 49621 | |
| El triptófano 50μg/ml | Sigma-Aldrich | T0254 | |
| La fenilalanina 50μg/ml | Sigma-Aldrich | P2126 | |
| La fijación de la célula | Paraformaldehído | Roth | 0335 |
| Nombre del equipo | Empresa | Número de catálogo | |
| Sonicación | Celular Sonicator | Bandelin | D-1000 |
| Microscopía de fluorescencia | Microscopio de fluorescencia | Leica | DMI6000B |
| Nombre del software | Empresa | Número de catálogo | |
| Microscopía de fluorescencia | Asaf | Leica | |
| La citometría de flujo | FCASDiva | BD | |
| La citometría de flujo | FlowJo | Treestar |
Referencias
- Costerton, J. W. Overview of microbial biofilms. J. Ind. Microbiol. 15, 137-140 (1995).
- Davey, M. E., O'Toole, G. A. Microbial biofilms: from ecology to molecular genetics. Microbiol. Mol. Biol. Rev. 64, 847-867 (2000).
- Kolenbrander, P. E. Oral microbial communities: biofilms, interactions, and genetic systems. Annu. Rev. Microbiol. 54, 413-437 (2000).
- O'Toole, G., Kaplan, H. B., Kolter, R. Biofilm formation as microbial development. Annu. Rev. Microbiol. 54, 49-79 (2000).
- Donlan, R. M. Biofilms: microbial life on surfaces. Emerg. Infect. Dis. 8, 881-890 (2002).
- Lopez, D., Vlamakis, H., Kolter, R. Biofilms. Cold Spring Harb. Perspect. Biol. 2, a000398-a000398 (2010).
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- Branda, S. S., Chu, F., Kearns, D. B., Losick, R., Kolter, R. A major protein component of the Bacillus subtilis biofilm matrix. Mol. Microbiol. 59, 1229-1238 (2006).
- Latasa, C., Solano, C., Penades, J. R., Lasa, I. Biofilm-associated proteins. C. R. Biol. 329, 849-857 (2006).
- O'Gara, J. P. ica and beyond: biofilm mechanisms and regulation in Staphylococcus epidermidis and Staphylococcus aureus. FEMS Microbiol Lett. 270, 179-188 (2007).
- Chai, Y., Chu, F., Kolter, R., Losick, R. Bistability and biofilm formation in Bacillus subtilis. Mol. Microbiol. 67, 254-263 (2008).
- Chen, R., Guttenplan, S. B., Blair, K. M., Kearns, D. B. Role of the sigmaD-dependent autolysins in Bacillus subtilis population heterogeneity. J. Bacteriol. 191, 5775-5784 (2009).
- Guttenplan, S. B., Blair, K. M., Kearns, D. B. The EpsE flagellar clutch is bifunctional and synergizes with EPS biosynthesis to promote Bacillus subtilis biofilm formation. PLoS Genet. 6, e1001243-e1001243 (2010).
- Kearns, D. B., Losick, R. Cell population heterogeneity during growth of Bacillus subtilis. Genes Dev. 19, 3083-3094 (2005).
- Lopez, D., Vlamakis, H., Losick, R., Kolter, R. Paracrine signaling in a bacterium. Genes Dev. 23, 1631-1638 (2009).
- Veening, J. W., Smits, W. K., Hamoen, L. W., Jongbloed, J. D., Kuipers, O. P. Visualization of differential gene expression by improved cyan fluorescent protein and yellow fluorescent protein production in Bacillus subtilis. Appl. Environ. Microbiol. 70, 6809-6815 (2004).
- Veening, J. W., Smits, W. K., Hamoen, L. W., Kuipers, O. P. Single cell analysis of gene expression patterns of competence development and initiation of sporulation in Bacillus subtilis grown on chemically defined media. J. Appl. Microbiol. 101, 531-541 (2006).
- Veening, J. W., Kuipers, O. P., Brul, S., Hellingwerf, K. J., Kort, R. Effects of phosphorelay perturbations on architecture, sporulation, and spore resistance in biofilms of Bacillus subtilis. J. Bacteriol. 188, 3099-3109 (2006).
- Vlamakis, H., Aguilar, C., Losick, R., Kolter, R. Control of cell fate by the formation of an architecturally complex bacterial community. Genes Dev. 22, 945-953 (2008).
- Stewart, P. S., Franklin, M. J. Physiological heterogeneity in biofilms. Nat. Rev. Microbiol. 6, 199-210 (2008).
- Veening, J. W., Smits, W. K., Kuipers, O. P. Bistability, epigenetics, and bet-hedging in bacteria. Annu. Rev. Microbiol. 62, 193-210 (2008).
- Aguilar, C., Vlamakis, H., Guzman, A., Losick, R., Kolter, R. KinD is a checkpoint protein linking spore formation to extracellular-matrix production in Bacillus subtilis biofilms. MBio. 1, (2010).
- Lopez, D., Fischbach, M. A., Chu, F., Losick, R., Kolter, R. Structurally diverse natural products that cause potassium leakage trigger multicellularity in Bacillus subtilis. Proc. Natl. Acad. Sci. U.S.A. 106, 280-285 (2009).
- Lopez, D., Vlamakis, H., Losick, R., Kolter, R. Cannibalism enhances biofilm development in Bacillus subtilis. Mol. Microbiol. 74, 609-618 (2009).
- Arima, K., Kakinuma, A., Tamura, G. Surfactin, a crystalline peptidelipid surfactant produced by Bacillus subtilis: isolation, characterization and its inhibition of fibrin clot formation. Biochem. Biophys. Res. Commun. 31, 488-494 (1968).
- Romero, D., Vlamakis, H., Losick, R., Kolter, R. An accessory protein required for anchoring and assembly of amyloid fibres in B. subtilis biofilms. Mol. Microbiol. 80, 1155-1168 (2011).
- Hardwood, C. R., Cutting, S. M. . Molecular Biological Methods for Bacillus. , (1990).
- Novick, R. P. Genetic systems in staphylococci. Methods Enzymol. 204, 587-636 (1991).
- Yasbin, R. E., Young, F. E. Transduction in Bacillus subtilis by bacteriophage SPP1. J. Virol. 14, 1343-1348 (1974).
- Branda, S. S., Gonzalez-Pastor, J. E., Ben-Yehuda, S., Losick, R., Kolter, R. Fruiting body formation by Bacillus subtilis. Proc. Natl. Acad. Sci. U.S.A. 98, 11621-11626 (2001).
- Nakano, M. M. srfA is an operon required for surfactin production, competence development, and efficient sporulation in Bacillus subtilis. J. Bacteriol. 173, 1770-1778 (1991).
- Gonzalez-Pastor, J. E., Hobbs, E. C., Losick, R. Cannibalism by sporulating bacteria. Science. 301, 510-513 (2003).
- Aguilar, C., Vlamakis, H., Losick, R., Kolter, R. Thinking about Bacillus subtilis as a multicellular organism. Curr. Opin. Microbiol. 10, 638-643 (2007).
- Shapiro, J. A. Thinking about bacterial populations as multicellular organisms. Annu. Rev. Microbiol. 52, 81-104 (1998).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados