È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Singola cella Analisi Bacillus subtilis I biofilm usando la microscopia a fluorescenza e citometria a flusso
In questo articolo
Riepilogo
Biofilm microbici sono generalmente costituiti da distinti sottopopolazioni di cellule specializzate. Cella singola analisi di queste sottopopolazioni richiede l'uso di reporter fluorescenti. Qui descriviamo un protocollo di visualizzare e monitorare diversi subpopulationswithin B. subtilis Biofilm mediante microscopia a fluorescenza e citometria a flusso.
Abstract
Formazione di biofilm è un attributo per quasi tutti i batteri 1-6. Quando batteri formano biofilm, le cellule sono racchiusi nella matrice extracellulare che è prevalentemente costituito da proteine e esopolisaccaridi, tra gli altri fattori 7-10. La comunità microbica racchiuso all'interno del biofilm spesso mostra la differenziazione della distinta sottopopolazione di cellule specializzate 11-17. Queste sottopopolazioni coesistono e spesso mostrano organizzazione spaziale e temporale all'interno del biofilm 18-21.
Formazione di biofilm nel modello Bacillus subtilis richiede la differenziazione delle sottopopolazioni di cellule specializzate distinte. Tra questi, la sottopopolazione di produttori matrice, responsabili di produrre e secernere la matrice extracellulare del biofilm è essenziale per la formazione di biofilm 11,19. Pertanto, la differenziazione dei produttori matrice è una caratteristica della formazione di biofilm in B. subtilis.
Abbiamo usato giornalisti fluorescenti per visualizzare e quantificare la sottopopolazione di produttori matrice in biofilm di B. subtilis 15,19,22-24. Concretamente, abbiamo osservato che la sottopopolazione di produttori matrice differenzia in risposta alla presenza di autoprodotto segnale extracellulare surfattina 25. È interessante notare, surfattina è prodotto da una sottopopolazione di cellule specializzate diverse dalla sottopopolazione di produttori matrice 15.
Abbiamo riportato in questa relazione l'approccio tecnico necessario per visualizzare e quantificare la sottopopolazione dei produttori e dei produttori matrice surfattina nei biofilm di B. subtilis. Per fare questo, reporter fluorescenti di geni necessari per la produzione di matrice e di produzione di surfattina sono inseriti nel cromosoma di B. subtilis. Reporter sono espressi solo in una sottopopolazione di cellule specializzate. Quindi, le sottopopolazioni possono esseremonitorata utilizzando la microscopia a fluorescenza e citometria a flusso (Fig. 1).
Il fatto che diverse sottopopolazioni di cellule specializzate coesistono all'interno delle comunità multicellulari di batteri ci dà una prospettiva diversa sulla regolazione dell'espressione genica nei procarioti. Questo protocollo affronta questo fenomeno sperimentalmente e può essere facilmente adattato a qualsiasi modello di altre lavorazioni, per chiarire i meccanismi molecolari alla base eterogeneità fenotipica all'interno di una comunità microbica.
Protocollo
1. Etichettatura B. subtilis e formazione di analisi Biofilm
- Amplificazione PCR dalla regione del promotore del gene di interesse. Si mostra come esempio la clonazione di P tapa, il promotore dei geni responsabili per la produzione di proteina Tasa matrice 26. Clone P tapa in pkm008 vettore (creato dal laboratorio di Rudner, Harvard Medical School. Boston, USA) (Fig. 2).
- Linearizzare i plasmidi per digestione enzimatica (Enzyme consigliato, XhoI).
- Indurre la competenza naturale in B. subtilis ceppo 168 seguendo il one-step protocollo precedentemente descritto da Harwood e Taglio 27.
- Aggiungere i plasmidi linearizzate nella cultura di cellule competenti e selezionate per la resistenza spectinomicina dopo due ore di incubazione.
- I ceppi ottenuti hanno inserito il costrutto nel locus amyE neutro di B. subtilis di ricombinazione doppia (Fig. 4). Per creareun ceppo a doppia etichetta, integrare il secondo reporter nel Laca locus neutro utilizzando il plasmide pDR183 vi presentiamo nella figura 3. Inserire questo reporter utilizzando la stessa tecnica sopra descritta per l'inserimento di reporter clonato in pKM008.
- Trasferire la reporter del ceppo 168 di NCIB3610 che è in grado di formare biofilm. Utilizzare il protocollo di trasduzione del fago SPP1 28,29. Crescere ceppo donatore in TY medio (LB +10 mm MgSO 4 +10 pM MnSO 4). Mescolare 200 microlitri della cultura con 100 microlitri di diluizione magazzino fago. Aggiungere 3 ml di agar morbido dopo 30 minuti di incubazione e permettono aloni fago sorgere a 37 ° C.
- Raccogliere l'agar molle. Centrifugare e passare il surnatante pensato che una siringa da 0,22 micron filtro. Utilizzare questo surnatante per infettare una cultura del ceppo ricevente coltivate in terreno TY. Aggiungere 30 microlitri a 10 ml di 1:10 coltura diluita. Incubare per 30 minuti e selezionare per la resistenza antibiotica dopo 24 h di incubazione.
- Sezionare una colonia e crescere durante la notte il LB a 37 ° C.
- Spot 3 pl della cultura durante la notte su una solida biofilm che induce medie MSgg 1,5% agar 30. Permettere alle cellule di crescere durante 72 ore a 30 ° C (Figura 3). Dopo tre giorni di crescita, biofilm formate sulla superficie di agar MSgg sviluppato un'architettura complessa morfologica nella superficie del agar.
2. Biofilm Dispersione e fissazione cellulare
- Rimuovere il modulo di biofilm sulla superficie del agar MSgg con uno stuzzicadenti o una pinzetta. La consistenza del biofilm dovrebbe consentire di staccatelo dalla superficie di agar in un unico pezzo.
- Posizionare il biofilm in 3 ml di tampone PBS e disperdere il biofilm da ripetitivo passaggio attraverso una pipetta o un ago. In alternativa, la dispersione biofilm può essere fatto utilizzando sonicazione lieve. Sonicazione Mild richiede 12 impulsi con una potenza di 3 e ampiezza di 0,7 secondi.
- Fissare i campioni prima cella singola analisi. Resuspend 300 microlitri della sospensione di cellule in 1 ml di soluzione di paraformaldeide al 4% ed incubare a temperatura ambiente per sette minuti.
Composizione della soluzione di paraformaldeide al 4%:
2 g di paraformaldeide
50 ml di tampone PBS
4 microlitri 10 N NaOH
Filtrare la soluzione attraverso un filtro a 0,22 um e aliquota - Lavare le cellule dopo fissazione in tampone PBS tre volte e risospendere in 300 ul di tampone PBS.
3. Microscopia a fluorescenza
- Versare 200 microlitri di 0.8% agarosio su un vetrino da microscopio e con attenzione coprire con un'altra diapositiva. Rimuovere la slitta superiore dolcemente dopo 2 minuti per ottenere uno strato di agarosio attaccato alla slitta del fondo.
- Spot 2 microlitri di cellule fissate sulla superficie dello strato di agarosio e coprire con un vetro di copertura microscopio.
- Porre il campione nel microscopio a fluorescenza. Usiamo un microscopio a fluorescenza Leica DMI6000B dotato di un CRT6000 Leica iIluminatione sistema. I filtri per YFP sono Ex: BP500/20, Em: BP535/30 e per CFP sono Ex: BP426/20, Em: 480/40
- Esporre il campione un fluorescenza eccitazione tra 50-200 ms. Impostare il periodo di eccitazione in base a un controllo negativo che non mostra fluorescenza nelle condizioni selezionate per l'esperimento.
- Vedere l'immagine di fluorescenza alla stessa immagine ottenuta con campo chiaro. Unire le due immagini in un'unica immagine. I risultati ottenuti dal flusso microscopia a fluorescenza utilizzando un unico ceppo marcato ospitante il reporter sono rappresentati P tapa-YFP in figura 6.
4. La quantificazione delle singole cellule in citometria a flusso
- Disperdere il campione di cellule fissate con sonicazione mite. Sonicare il campione eseguendo 2 serie di impulsi 12 con una potenza di 5 e ampiezza di 0,7 secondi, per disperdere aggregati nelle cellule singole senza causare la lisi cellulare. Verificare l'efficienza di dispersione cellulare mediante microscopia ottica.
- Diluire il campione 1:100 in tampone PBS prima dell'analisi in citometria a flusso. Usiamo un citometro a flusso BD FACS Canto II. Per YFP fluorescenza, utilizzare un laser di eccitazione a 488 nm accoppiato con un 530/30 del filtro. Per CFP fluorescenza, utilizzare l'eccitazione laser a 405 nm accoppiato con un 408/40 filtro.
- Calibrare la macchina citometro a flusso con due controlli negativi. Un campione di tampone PBS privo di cellule in sospensione dovrebbe servire come controllo negativo per la dimensione delle particelle rilevata dal citometro a flusso. Un campione marcato con nessun reporter fluorescenza deve essere servire come controllo negativo per la sensibilità fluorescenza del citometro a flusso.
- Collocare il campione etichettato con il reporter fluorescente nel citometro a flusso. Per ciascun campione, analizzare almeno 50.000 eventi con una portata tra 300 e 3000 eventi al secondo.
- Acquisizione dati utilizzando FACS Diva software (BD Biosciences) e analizzare utilizzando FlowJo 8.5.2 del software. Vedere segnali di fluorescenza al controllo che mostranos no fluorescenza.
- Presentare i dati del monitoraggio del segnale di fluorescenza di un singolo ceppo marcato in uno dei due assi grafica. Tracciare il segnale di fluorescenza rilevato l'asse X e il numero di cellule che esprimono i diversi livelli di fluorescenza sull'asse Y. I risultati ottenuti dall'analisi citometria a flusso utilizzando un unico ceppo marcato ospitante il reporter sono rappresentati P tapa-YFP in figura 8.
- Presentare i dati del monitoraggio del segnale di fluorescenza di un ceppo doppio etichettati in tre assi grafica. Tracciare il segnale di fluorescenza di ciascuno dei canali monitorati in assi X e Y (per esempio, GFP potrebbe essere misurato l'asse X e PCP nel asse Y). Plot in asse Z il numero di cellule che esprimono ciascuna reporter e presentano come isolines contorno che sarebbero perpendicolare al piano del foglio (Fig. 9 e 10).
4. Risultati rappresentativi
Quando B. subtilis cresce su un piatto della biofilm che induce MSgg mezzo, la formazione di biofilm si osserva dopo tre giorni di incubazione a 30 ° C 30. Il biofilm mostra consistenza forte e può essere staccata dalla superficie di agar in un unico pezzo. Inoltre, il biofilm mostra un'architettura complessa morfologica che è indicativo delle sottopopolazioni di cellule aderenti distinti (Fig. 5). Ad esempio, la produzione della matrice extracellulare in biofilm risultato la formazione di rughe sulla superficie della colonia. Questa funzione può essere correlata con la differenziazione della sottopopolazione di produttori matrice 19. Analogamente, l'aumento di strutture aeree sulla superficie del biofilm è indicativa della presenza di una sottopopolazione di cellule sporulanti, poiché le spore localizzato nella zona apicale di queste strutture 30.
Esempi di visualizzazione del differenziamento cellulare in un unico marcato e un ceppo doppio marcato utilizzando microscopia a fluorescenza sono represented in figura 6 e 7, rispettivamente. L'unico ceppo marcato ospita la reporter fluorescente P tapa-CFP, che si esprime nella sottopopolazione di cellule produttrici di matrice. Questa sottopopolazione è responsabile di produrre e secernere la matrice extracellulare che costituisce il biofilm (Fig. 6). Il ceppo doppio etichettato ospita il giornalista P tapa CFP-26 e il giornalista ulteriori P srfAA YFP-31. Questo secondo reporter permette di monitorare la sottopopolazione di cellule responsabili di secernere la surfattina molecola di segnalazione, che innesca la cascata di segnalazione per la differenziazione dei produttori matrice (Fig. 7). Sottopopolazione dei produttori di matrice sono false colorati in blu, mentre la sottopopolazione di produttori surfattina è falso colorata in giallo.
Analisi di citometria a flusso utilizzando un unico ceppo marcato ospitare il reporter P tapa-YFP è presentato in figura 8. Ceppo di controllo non trattato non ospitarei geni delle proteine fluorescenti ha mostrato una singola fluorescenza popolazione relativamente basso. Cellule che ospitano P tapa-YFP non in biofilm che inducono condizioni di non differenziare la sottopopolazione di matrice produttori e l'intera popolazione ha mostrato un basso fluorescenza relativa. In biofilm che inducono condizione, una sottopopolazione di cellule con alta fluorescenza relativa è verificato, osservato come una spalla destra del picco relativa bassa fluorescenza 23.
I risultati ottenuti dall'analisi citometria a flusso utilizzando un ceppo doppio marcato sono rappresentati in figura 9 e 10. Figura 9 monitorato le sottopopolazioni di matrice produttori e dei produttori surfattina utilizzando il doppio etichettato strain P tapa-PCP, P srfAA-YFP. Poiché il controllo di fluorescenza di fondo è stato utilizzato un ceppo non nutrire alcun geni delle proteine fluorescenti. Successivamente, abbiamo rilevato di ciascuna delle sottopopolazioni di produttori della matrice e produttori surfattina in ogni canale di fluorescenza, utilizzando unico ceppo marcato s come controlli. Il doppio etichettato strain P tapa-PCP, P srfAA-YFP ha mostrato due sottopopolazioni di cellule che esprimono alti livelli di i giornalisti fluorescenti. Ogni popolazione è incorniciato, dimostrando che non vi è alcuna sovrapposizione nella espressione dei giornalisti tra le due sottopopolazioni di cellule specializzate 15. Analogamente, l'analisi mediante citometria a flusso doppio marcato ceppo P SKF-YFP, P-tapa YFP è presentato in figura 10. Il reporter per la SKF gene controlla la differenziazione della sottopopolazione di cannibali 32, che è stato descritto per differenziare coordinatamente con la sottopopolazione di produttori di matrice 24. In questo caso, il ceppo doppio marcato mostrato un'unica sottopopolazione di cellule fluorescenti che esprimono sia YFP e PCP. Ciò indica che entrambe le vie della differenziazione delle cellule vengono attivate in modo coordinato sottopopolazione stessa.
1.jpg "/>
Figura 1. Schema generale della Experiment. Il protocollo è diviso in tre fasi principali. La prima fase prevede l'etichettatura il ceppo di B. subtilis con la fusione giornalista che controlla la sottopopolazione di interesse. In secondo luogo, crescere i ceppi identificati in biofilm che inducono condizioni. In terzo luogo, disperdere il biofilm ed effettuare singola cella di analisi della popolazione con microscopio a fluorescenza e citometria a flusso.

Figura 2. Rappresentazione schematica del vettore pKM008 integrazione. Questo vettore integra la fusione giornalista di interesse nel amyE locus neutro dalla ricombinazione doppio. Il promotore di interesse (P tapa) viene clonato nel vettore utilizzando siti di restrizione EcoRI e HindIII. Quindi, l'espressione del gene PCP è sotto il controllo del promotore P tapa. L'Orientationione dei geni nel plasmide è rappresentato come una freccia.
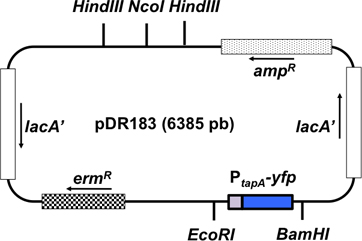
Figura 3. Rappresentazione schematica del vettore di integrazione pDR183. Questo vettore integra la fusione giornalista di interesse nel locus neutro Laca mediante ricombinazione doppio. La fusione di interesse (P tapa - PCP) è clonato nel vettore utilizzando la restrizione siti EcoRI e BamHI. L'orientamento dei geni del plasmide è rappresentato come una freccia.

Figura 4. Schema di integrazione dei reporter nel cromosoma di B. subtilis di ricombinazione doppia. (A) B. cromosoma subtilis ha due loci neutrali, amyE e Laca, per integrare fusioni reporter senza compromettere lo sviluppo del biofilm. (B) di ricombinazione doppio pKM008. Plasmide linearizzato integra nel genoma di B. subtilis di ricombinazione doppia. La fusione giornalista integra nel locus neutro in modo stabile.

Figura 5. Formazione di biofilm di B. subtilis NCIB3610. processo di formazione bioflm del ceppo B. subtilis NCIB 3610 quando cresce il biofilm che induce MSgg medium nel corso di tre giorni a 30 ° C. Immagini sequenziali di sviluppo del biofilm sono stati prelevati ogni 12 ore.

Figura 6. Visualizzazione della sottopopolazione di produttori matrice al microscopio a fluorescenza. Un campione di un biofilm di B. subtilis P tapa - cfp è stato fissato ed esaminato al microscopio a fluorescenza. 200 ms di fluorescenzaeccitazione evidenziato una sottopopolazione di cellule che emettono fluorescenza superiore rispetto al resto delle cellule. Abbiamo considerato questa sottopopolazione come la sottopopolazione di cellule produttrici di matrice. Barra della scala è di 3 micron.
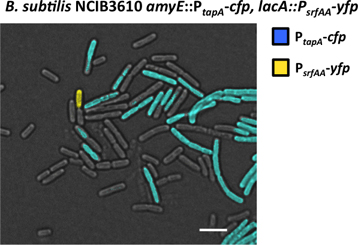
Figura 7. Visualizzazione della sottopopolazione di matrice produttori e produttori surfattina sotto il microscopio a fluorescenza. Campione da un biofilm della B. subtilis P tapa-cfp, P srfAA-YFP ceppo doppio marcato è stato fissato ed esaminato al microscopio a fluorescenza. Tempo di esposizione di 250 ms evidenziato due sottopopolazioni di cellule che emettono fluorescenza superiore rispetto al resto delle cellule. Una sottopopolazione espresso YFP ed è stato rilevato utilizzando esclusivamente il canale YFP (false colorata in giallo). Questa è la sottopopolazione di produttori di surfattina. Un'altra sottopopolazione espresso CFP ed è stato rilevato utilizzando esclusivamente la Chann PCP el. Questa è la sottopopolazione di produttori matrice. Barra della scala è di 3 micron.
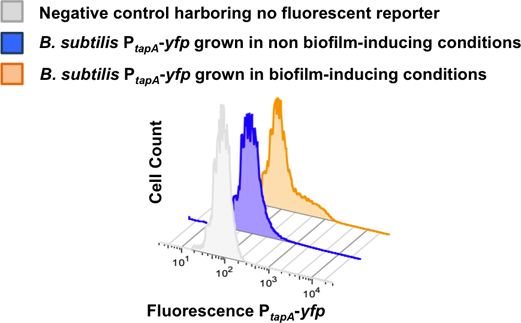
Figura 8. Quantificazione della sottopopolazione di produttori matrice 2-D usando citometria di flusso. Dispersed cellule di un biofilm di B. subtilis P tapa-YFP sono stati monitorati in citometria a flusso. Il citometro a flusso contato 50.000 eventi e il segnale di fluorescenza per ogni evento è stato monitorato. Numero di cellule contate è tracciata in asse Y, mentre l'intensità del segnale YFP viene tracciata sull'asse X. Le cellule sono state coltivate in terreno LB per ottenere non biofilm condizioni che inducono. Le cellule sono state coltivate in mezzo MSgg per ottenere biofilm che inducono condizioni. La sottopopolazione dei produttori matrice differenzia solo nel biofilm che inducono condizioni. Questo dato è stato adattato da López et al., PNAS (2009) 106 :280-285.
s/ftp_upload/3796/3796fig9.jpg "/>
Figura 9. Quantificazione della sottopopolazione di produttori matrice e dei produttori surfattina utilizzando 3-D citometria a flusso. Segnale di fluorescenza dei canali monitorati sono presentati nell'asse X (per YFP) e l'asse Y (per PCP). L'asse Z misura il numero di cellule che esprimono ciascuna reporter e viene quantificata come isolines contorno perpendicolari al piano della carta. Il numero di eventi monitorati in questo esperimento era 50.000 eventi. Sinistra pannello superiore presenta un controllo di fluorescenza di fondo non ospitare geni delle proteine fluorescenti. Diritto pannello superiore rileva la sottopopolazione di produttori surfattina in fluorescenza YFP canale (incorniciata in giallo) utilizzando un unico marcato ceppo P srfAA-YFP. Pannello in basso a sinistra rileva la sottopopolazione di produttori di matrice politica comune della pesca nel canale di fluorescenza (incorniciato in blu) utilizzando un unico ceppo marcato P tapa-CFP. Il doppio etichettato strain tapa P </ Em>-PCP, P srfAA-YFP è monitorata nel pannello in basso a destra. Ha dimostrato due sottopopolazioni che sono incorniciate in giallo e blu. Questo dato è stato adattato da López et al., Geni e lo sviluppo (2009) 23 :1631-1638.
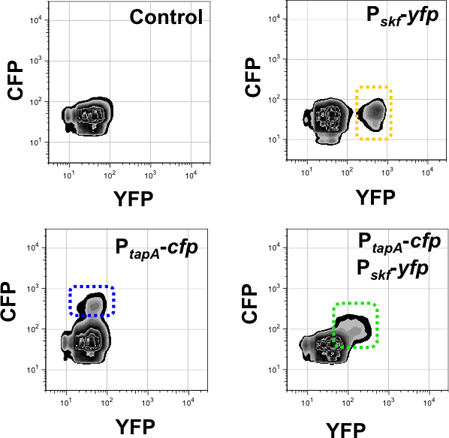
Figura 10. Quantificazione della sottopopolazione di produttori matrice e cannibali utilizzando 3-D citometria a flusso. Segnale di fluorescenza dei canali monitorati sono presentati nell'asse X (per YFP) e l'asse Y (per PCP). Il numero di eventi monitorati in questo esperimento era 50,000. Diritto pannello superiore rileva la sottopopolazione di cannibali in fluorescenza YFP canale (incorniciata in giallo) in un unico marcato ceppo P SKF-YFP. Pannello in basso a sinistra rileva la sottopopolazione di produttori di matrice politica comune della pesca nel canale di fluorescenza (incorniciato in blu) in un unico ceppo marcato P tapa-CFP. Il double marcato ceppo P tapa-PCP, P SKF-YFP ha mostrato solo una sottopopolazione di cellule in diagonale per gli assi X e Y (incorniciato in verde). Questa sottopopolazione viene rilevato nel canale YFP e CFP, perché esprime i due reporter contemporaneamente. López et al. Geni e sviluppo (2009) 23 :1631-1638.
Discussione
Il fatto che le comunità batteriche mostrano sottopopolazioni di cellule che esprimono set specifico di geni evidenzia la complessità delle comunità microbiche 33,34. Questo protocollo dovrebbe aiutare a determinare se l'espressione di un gene di interesse viene limitata a un particolare sottopopolazione di cellule specializzate nella comunità microbica. Visualizzazione di tali sottopopolazioni richiede lo sviluppo di nuove tecniche, perché i metodi tradizionali per monitorare l'espressione genic...
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Questo lavoro è finanziato dal programma Young Investigator Research, dal Centro di ricerca sulle malattie infettive (ZINF) presso l'Università di Würzburg. Juan Garcia-C Betancur è un collega dottorato di ricerca presso la Graduate School of Life Sciences (GSLS) dell'Università di Würzburg.
Materiali
| Name | Company | Catalog Number | Comments |
| Tecnica | Nome del reattivo | Azienda | Numero di catalogo |
| MSgg composizione | fosfato di potassio 5mM | Roth | 6878 |
| 100 mM MOPS | Sigma-Aldrich | M1254 | |
| Cloruro di magnesio 2 mM | Roth | 2.189,1 | |
| Cloruro di calcio 700μM | Roth | A119.1 | |
| 50 micron cloruro ferrico | Sigma-Aldrich | 157740 | |
| Cloruro di zinco 1 pm | Applichem | A2076 | |
| Tiamina 2μM | Sigma-Aldrich | 74625 | |
| Glicerolo 0,5% | Roth | 7533 | |
| Glutacompagno di 0,5% | Sigma-Aldrich | 49621 | |
| Triptofano 50μg/ml | Sigma-Aldrich | T0254 | |
| Fenilalanina 50μg/ml | Sigma-Aldrich | P2126 | |
| Cella di fissaggio | Paraformaldeide | Roth | 0335 |
| Nome dell'apparecchiatura | Azienda | Numero di catalogo | |
| Sonicazione | Cella Sonicator | Bandelin | D-1000 |
| Microscopia a fluorescenza | Microscopio a fluorescenza | Leica | DMI6000B |
| Nome del software | Azienda | Numero di catalogo | |
| Microscopia a fluorescenza | Asaf | Leica | |
| Citometria a flusso | FCASDiva | BD | |
| Citometria a flusso | FlowJo | Treestar |
Riferimenti
- Costerton, J. W. Overview of microbial biofilms. J. Ind. Microbiol. 15, 137-140 (1995).
- Davey, M. E., O'Toole, G. A. Microbial biofilms: from ecology to molecular genetics. Microbiol. Mol. Biol. Rev. 64, 847-867 (2000).
- Kolenbrander, P. E. Oral microbial communities: biofilms, interactions, and genetic systems. Annu. Rev. Microbiol. 54, 413-437 (2000).
- O'Toole, G., Kaplan, H. B., Kolter, R. Biofilm formation as microbial development. Annu. Rev. Microbiol. 54, 49-79 (2000).
- Donlan, R. M. Biofilms: microbial life on surfaces. Emerg. Infect. Dis. 8, 881-890 (2002).
- Lopez, D., Vlamakis, H., Kolter, R. Biofilms. Cold Spring Harb. Perspect. Biol. 2, a000398-a000398 (2010).
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- Branda, S. S., Chu, F., Kearns, D. B., Losick, R., Kolter, R. A major protein component of the Bacillus subtilis biofilm matrix. Mol. Microbiol. 59, 1229-1238 (2006).
- Latasa, C., Solano, C., Penades, J. R., Lasa, I. Biofilm-associated proteins. C. R. Biol. 329, 849-857 (2006).
- O'Gara, J. P. ica and beyond: biofilm mechanisms and regulation in Staphylococcus epidermidis and Staphylococcus aureus. FEMS Microbiol Lett. 270, 179-188 (2007).
- Chai, Y., Chu, F., Kolter, R., Losick, R. Bistability and biofilm formation in Bacillus subtilis. Mol. Microbiol. 67, 254-263 (2008).
- Chen, R., Guttenplan, S. B., Blair, K. M., Kearns, D. B. Role of the sigmaD-dependent autolysins in Bacillus subtilis population heterogeneity. J. Bacteriol. 191, 5775-5784 (2009).
- Guttenplan, S. B., Blair, K. M., Kearns, D. B. The EpsE flagellar clutch is bifunctional and synergizes with EPS biosynthesis to promote Bacillus subtilis biofilm formation. PLoS Genet. 6, e1001243-e1001243 (2010).
- Kearns, D. B., Losick, R. Cell population heterogeneity during growth of Bacillus subtilis. Genes Dev. 19, 3083-3094 (2005).
- Lopez, D., Vlamakis, H., Losick, R., Kolter, R. Paracrine signaling in a bacterium. Genes Dev. 23, 1631-1638 (2009).
- Veening, J. W., Smits, W. K., Hamoen, L. W., Jongbloed, J. D., Kuipers, O. P. Visualization of differential gene expression by improved cyan fluorescent protein and yellow fluorescent protein production in Bacillus subtilis. Appl. Environ. Microbiol. 70, 6809-6815 (2004).
- Veening, J. W., Smits, W. K., Hamoen, L. W., Kuipers, O. P. Single cell analysis of gene expression patterns of competence development and initiation of sporulation in Bacillus subtilis grown on chemically defined media. J. Appl. Microbiol. 101, 531-541 (2006).
- Veening, J. W., Kuipers, O. P., Brul, S., Hellingwerf, K. J., Kort, R. Effects of phosphorelay perturbations on architecture, sporulation, and spore resistance in biofilms of Bacillus subtilis. J. Bacteriol. 188, 3099-3109 (2006).
- Vlamakis, H., Aguilar, C., Losick, R., Kolter, R. Control of cell fate by the formation of an architecturally complex bacterial community. Genes Dev. 22, 945-953 (2008).
- Stewart, P. S., Franklin, M. J. Physiological heterogeneity in biofilms. Nat. Rev. Microbiol. 6, 199-210 (2008).
- Veening, J. W., Smits, W. K., Kuipers, O. P. Bistability, epigenetics, and bet-hedging in bacteria. Annu. Rev. Microbiol. 62, 193-210 (2008).
- Aguilar, C., Vlamakis, H., Guzman, A., Losick, R., Kolter, R. KinD is a checkpoint protein linking spore formation to extracellular-matrix production in Bacillus subtilis biofilms. MBio. 1, (2010).
- Lopez, D., Fischbach, M. A., Chu, F., Losick, R., Kolter, R. Structurally diverse natural products that cause potassium leakage trigger multicellularity in Bacillus subtilis. Proc. Natl. Acad. Sci. U.S.A. 106, 280-285 (2009).
- Lopez, D., Vlamakis, H., Losick, R., Kolter, R. Cannibalism enhances biofilm development in Bacillus subtilis. Mol. Microbiol. 74, 609-618 (2009).
- Arima, K., Kakinuma, A., Tamura, G. Surfactin, a crystalline peptidelipid surfactant produced by Bacillus subtilis: isolation, characterization and its inhibition of fibrin clot formation. Biochem. Biophys. Res. Commun. 31, 488-494 (1968).
- Romero, D., Vlamakis, H., Losick, R., Kolter, R. An accessory protein required for anchoring and assembly of amyloid fibres in B. subtilis biofilms. Mol. Microbiol. 80, 1155-1168 (2011).
- Hardwood, C. R., Cutting, S. M. . Molecular Biological Methods for Bacillus. , (1990).
- Novick, R. P. Genetic systems in staphylococci. Methods Enzymol. 204, 587-636 (1991).
- Yasbin, R. E., Young, F. E. Transduction in Bacillus subtilis by bacteriophage SPP1. J. Virol. 14, 1343-1348 (1974).
- Branda, S. S., Gonzalez-Pastor, J. E., Ben-Yehuda, S., Losick, R., Kolter, R. Fruiting body formation by Bacillus subtilis. Proc. Natl. Acad. Sci. U.S.A. 98, 11621-11626 (2001).
- Nakano, M. M. srfA is an operon required for surfactin production, competence development, and efficient sporulation in Bacillus subtilis. J. Bacteriol. 173, 1770-1778 (1991).
- Gonzalez-Pastor, J. E., Hobbs, E. C., Losick, R. Cannibalism by sporulating bacteria. Science. 301, 510-513 (2003).
- Aguilar, C., Vlamakis, H., Losick, R., Kolter, R. Thinking about Bacillus subtilis as a multicellular organism. Curr. Opin. Microbiol. 10, 638-643 (2007).
- Shapiro, J. A. Thinking about bacterial populations as multicellular organisms. Annu. Rev. Microbiol. 52, 81-104 (1998).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon