Method Article
Fluorescente
En este artículo
Resumen
Entre los tres géneros de mosquitos, es decir, Anopheles, Aedes Y Culex, Técnicas de mapeo físico del genoma se crearon sólo para Anopheles, Cuyos miembros poseen cromosomas politénicos legibles. Para los géneros de Aedes Y Culex Sin embargo, la cartografía citogenético sigue siendo un reto debido a la mala calidad de los cromosomas politénicos. Aquí presentamos un protocolo universal para la obtención de preparados de alta calidad de los cromosomas mitóticos y un protocolo FISH optimizado para los tres géneros de mosquitos.
Resumen
La hibridación fluorescente in situ (FISH) es una técnica utilizada habitualmente por muchos laboratorios para determinar la posición cromosómica de sondas de ADN y ARN. Una aplicación importante de este método es el desarrollo de mapas de alta calidad físicos útiles para mejorar los conjuntos de genoma de varios organismos. El patrón natural de bandas de los cromosomas mitóticos politénicos y proporciona orientación para la ordenación y orientación precisas de los supercontigs genómicos. Entre los tres géneros de mosquitos Anopheles, a saber, Aedes, Culex y, una bien establecida cromosoma técnica basada en la cartografía se ha desarrollado sólo para Anopheles, cuyos miembros poseen cromosomas legibles politénicos 1. Como resultado de los esfuerzos de mapeo del genoma, el 88% de la Un. gambiae genoma se ha colocado para cromosoma posiciones precisas 2,3. Dos géneros de mosquitos Aedes y otros, Culex, han mal polytenized b cromosomasomo de la sobrerrepresentación significativa de elementos transponibles en sus genomas 4, 5, 6. Sólo 31 y 9% de los supercontings genómicas se han asignado sin orden o la orientación de los cromosomas de Ae. aegypti 7 y Cx. quinquefasciatus 8, respectivamente. Preparación mitótica cromosoma para estas dos especies previamente había sido limitada a los ganglios cerebro y líneas celulares. Sin embargo, los portaobjetos cromosómicos preparados a partir de los ganglios cerebro de los mosquitos por lo general contienen un bajo número de placas de metafase 9. Además, aunque una técnica de FISH se ha desarrollado para cromosomas mitóticos de una línea celular de Ae. aegypti 10, la acumulación de múltiples reordenamientos cromosómicos en los cromosomas de líneas celulares 11 los hace inútiles para la cartografía del genoma. Aquí se describe una técnica sencilla y robusta para obtener alta calidad preparaciones de cromosomas mitóticos de los discos imaginales (ID) de 4 º estadio las larvas que cuna se utiliza para todos los tres géneros de mosquitos. Un protocolo FISH estándar 12 está optimizado para el uso de clones de BAC de ADN genómico como una sonda en los cromosomas mitóticos de Ae. aegypti y Cx. quinquefasciatus, y para la utilización de un espaciador intergénico (IGS) región de ADN ribosómico (ADNr) como una sonda en una. cromosomas gambiae. Además de la cartografía física, la técnica desarrollada se puede aplicar a la citogenética de la población y la taxonomía cromosoma / sistemática de los mosquitos y otros grupos de insectos.
Protocolo
1. Cromosoma preparación
Las larvas del mosquito se criaron usando un protocolo estándar descrito en Métodos de Investigación Anopheles disponible en la página web del Centro de Investigación de Paludismo y de reactivos de referencia de recursos (MR4) 13. Las temperaturas de la crianza de mosquitos fueron modificados para proporcionar el mayor número de cromosomas en los discos imaginales y la menor mortalidad de las larvas. Las etapas de desarrollo de las larvas de mosquito se determinaron basándose en los tamaños de sus cápsulas de cabeza 13.
- Los huevos eclosionan los mosquitos a 28 º C, y después de 2-3 días, la transferencia de 2 ª o 3 ª instar larvas a 16 ° C para Ae. aegypti y Cx. quinquefasciatus y a 22 º C durante una. gambiae.
- 4 ª lugar larvas de instar en hielo durante varios minutos para la inmovilización.
- Transferir larva a un portaobjetos con una gota de solución hipotónica fría (0,5% en lomediano o citrato 0,075 M de cloruro de potasio), y ponerlo bajo el microscopio estereoscópico.
- Seleccione larva con ID ovalados (Figura 1 B) para la disección posterior.
- Decapita larva, y cortar la cutícula desde el lado ventral del tórax larval con unas tijeras de disección (Figura 2A). Haga el corte adicional en el segmento abdominal segunda o tercera para diseccionar el intestino de la larva. Las direcciones de los cortes se muestran mediante flechas.
- Abra la cutícula y eliminar el intestino y la grasa corporal de la larva. Retire la solución hipotónica de la diapositiva utilizando papel de filtro, y añada una nueva caída de solución hipotónica directamente a la ID (Figura 2B). Mantener la larva en solución hipotónica durante 10 min a RT.
- Eliminar solución hipotónica usando papel de filtro, y aplicar la solución de Carnoy (etanol / ácido acético en proporción 3:1). Después de añadir la solución de fijación, IDS inmediatamente se vuelven blancas y se convierten fácilmente visibles bajo el microscopio (Figura 2C ).
- El uso de agujas de disección, eliminar ID de la larva (Figura 2D), y transferirlos a una caída de 50% de ácido propiónico. Eliminar cualquier otros tejidos, tales como el intestino y la grasa corporal, a partir de la diapositiva. Cubrir con un ID de cubreobjetos de 22x22 siliconizados, y mantener durante 10 min a RT.
- Cubrir el portaobjetos con papel de filtro y aplastar el tejido tocando la goma de borrar de un lápiz en el perímetro de la hoja de cubierta.
- Brevemente analizar la calidad de la diapositiva con el microscopio de contraste de fases a 100x o 200x (Figura 3). Preparaciones> 50 extensiones de cromosomas pueden ser considerados adecuados para los peces.
- Sumergir y mantener la corredera en nitrógeno líquido hasta que deje de burbujear. Retire la hoja de la cubierta de la corredera usando una hoja de afeitar, y transferir el portaobjetos inmediatamente a un contenedor de 70% de etanol frío a -20 ° C. Almacenar a 4 ° C durante al menos 1 h para el mejor resultado deshidratación (si es necesario, los portaobjetos se pueden almacenar a tsu paso de varios minutos a varios días).
- Deshidratar diapositivas en una serie de etanol (70%, 80%, 100%) a 4 º C durante 5 min cada uno, y el aire seco a temperatura ambiente.
- Guarde portaobjetos secos a -20 ° C antes de utilizarlos para los peces.
2. La extracción de fracciones de ADN repetitivas
Realización de FISH de la sonda de ADN clon BAC en los cromosomas de Ae. aegypti y Cx. quinquefasciatus requiere el uso de fracciones de ADN repetitivas no marcados para bloquear la hibridación inespecífica de las repeticiones de ADN en los cromosomas. La reasociación de ADN de una sola hebra-fragmentado en piezas de varios cientos de pares de bases sigue una curva C 0 0 t donde C es la concentración inicial de ADN de cadena simple y t es el tiempo de reasociación. Fracciones de ADN con valores iguales a C0t -4 10 -10 -1 o 10 ° -10 2, se consideran muy y moderadamente repetitiva, respectivamente.
- Extraer 400-500 ug del DNA genómico de mosquitos adultos entera usando sangre Qiagen y Maxikit cultivo celular, y preparar 100-1.000 ng / l solución de ADN en 1,2 x SSC.
- Desnaturalizar ADN mediante la colocación de un tubo de bloqueo de caja fuerte con ADN genómico en un bloque de calentamiento precalentado a 120 ° C durante 2 min. La alta temperatura ayuda a variar el ADN en fragmentos de 200-500 pb.
- Dependiendo de la concentración de ADN, volver a asociar ADN colocando el tubo a 60 ° C durante 15-150 minutos para obtener fracciones de ADN C 0 t C hasta 0 t3 (Tabla 1).
- Colocar el tubo con el ADN en hielo durante 2 min.
- Transferir el ADN a 42 ° C, añadir precalentado 10x tampón de nucleasa S1 y la nucleasa S1 para una concentración final de 100 U por 1 mg de ADN, y se incuba durante 1 hr.
- Precipitado de ADN mediante la adición de 0,1 volúmenes de acetato sódico 3 M y 1 volumen de isopropanol a temperatura ambiente.
- Centrifugar a 14.000 rpm durante 20 min a 4 ° C.
- Lavar ADN en 70% de etanol, y se centrifuga de nuevo a 14.000 rpm durante 10 mina 4 ° C.
- Secar al aire y disolver precipitado de ADN en tampón TE.
- Medir la concentración de ADN, y visualizar por electroforesis en gel. Por lo general, la cantidad final de fracciones de ADN repetitivas representa 35-50% de la cantidad original de ADN.
3. Sonda de DNA Labeling
Dos protocolos diferentes se usaron para el etiquetado BAC clon de ADN de la sonda y la sonda rDNA IGS.
3,1 etiquetado BAC clon usando nick-translation
- Extraer el ADN BAC clon de la biblioteca BAC usando el kit Qiagen construir grandes.
- Preparar la mezcla de reacción para el etiquetado de la mella en hielo con un volumen final de 50 l: 1 g BAC clon de ADN aislado, 0,05 mM de cada uno de la etiqueta de dATP, dCTP, y dGTP y 0,015 mM de dTTP; 1 l de Cy3-dUTP (u otro fluorocromo); 0,05 mg / ml de BSA, 5 l de 10x tampón de traslación de mella, 20 U de ADN-polimerasa I, y 0,0012 U de DNasa.
- Incubar a 15 ° C durante 20.5 hr.
- Detener la reacción mediante la adición de 1 l de 0,5 M EDTA.
- Tienda sonda a -20 ° C en un lugar oscuro.
IGS 3,2 rDNA etiquetado usando PCR
- Preparar la mezcla de reacción sobre hielo con un volumen final de 50 l: 200 ng de ADN genómico; 0,05 mM de cada uno de la etiqueta de dATP, dCTP, y dGTP; 0,015 mM de dTTP; 1 l de Cy3-dUTP (u otro fluorocromo); 5 l de 10x tampón PCR; 50 pmol de adelante; ONU (GTGTGCCCCTTCCTCGATGT) e inversa; GA (CTGGTTTGGTCGGCACGTTT) cebadores para la amplificación IGS, y 10 U de Taq polimerasa de ADN 14.
- Realizar reacción de PCR utilizando los parámetros estándar de PCR para la amplificación IGS: 95 ° C / 5 min x 1 ciclo; (95 ° C / 30 seg, 50 ° C / 30 seg, 72 ° C / 30 segundos) x 30 ciclos; 72 ° C / 5 min x 1 ciclo; 4 ° C y mantener 14.
- Tienda sonda a -20 ° C en un lugar oscuro.
4. La hibridación fluorescente in situ
Este protocolo FISH incluye dos variantes: la primera para el uso de ADN BAC clon como sonda en los cromosomas mitóticos de Ae. aegypti y Cx. quinquefasciatus y el segundo para el uso de IGS ADNr en los cromosomas mitóticos de An. gambiae. Si se utiliza sondas de ADN BAC clon, saltar tratamiento con RNasa pasos 4,3, 4,4, y simultánea de diapositivas / sonda etapa de desnaturalización 4,19. Si se utiliza la sonda rDNA IGS, preparar la mezcla de hibridación sin C 0 t fracciones de ADN, y pase de diapositivas independiente / sonda desnaturalización medidas 4.10, 4.11, 4.16, y 4.17.
- Incubar los portaobjetos en 2x SSC durante 30 minutos a 37 ° C.
- Deshidratar diapositivas en una serie de 70%, 80%, y 100% de etanol durante 5 min cada uno a temperatura ambiente, y secar al aire. Si se realiza FISH con BAC clon de ADN, vaya directamente al paso 4,5.
- Incubar preparación de cromosomas en 0,1 mg / ml de solución de RNasa bajo Parafilm durante 30 min a 37 ° C.
- Lavar dos veces en 2x SSC durante 5 min cada uno a 37 ° C.
- Ponga diapositivas ina frasco con 0,01% de pepsina y 0,037% de solución de HCl, y se incuba durante 5 min a 37 ° C.
- Lavar los portaobjetos en 1x PBS durante 5 min a RT.
- Fijar preparación de cromosomas en un frasco con formalina 1% en PBS 1x preparado a partir de 10% formalina tamponada neutra durante 10 minutos a RT.
- Lavar los portaobjetos en 1x PBS durante 5 min a RT.
- Deshidratar diapositivas en una serie de 70%, 80%, y 100% de etanol durante 5 min cada uno a temperatura ambiente, y preparaciones de aire seco a 37 ° C. Si se realiza FISH con IGS, vaya directamente al paso 4,12
- Diapositivas desnaturalizar en un tarro con formamida 70% precalentado durante 2 min a 72 ° C.
- Deshidratar diapositivas en una serie de frío (-20 ° C) 70%, 80%, y 100% de etanol durante 5 min cada uno, y de aire seco a 37 ° C.
- Preparar la mezcla de hibridación:. 5 l de ADN sonda marcada desde el paso 3, 10 l de ADN C 0 t de la etapa 2 con una concentración final de 0,5 l ng / l, y 5 de 1 g / l de ADN de esperma de salmón sonicado para FISH con IGS rDNA, prepararhibridación mezcla sin fracciones de ADN C 0 t.
- Precipitado de ADN mediante la adición de 0,1 volúmenes de acetato sódico 3 M y 2 volúmenes de etanol. Almacenar a -20 º C durante 1-3 h.
- Centrifugar a 14.000 rpm a 4 ° C durante 20 min, eliminar el etanol, y el aire seco el precipitado a TA.
- Completamente disolver el precipitado en 10 l de tampón de hibridación:. Formamida al 50%, el 20% de sulfato de dextrano, 2x SSC Si se realiza FISH con IGS, vaya directamente al paso 4,18
- Desnaturalizar la mezcla de hibridación de 7 min a 97 ° C, e inmediatamente se puso en hielo durante 1 min.
- Prehybridize mezcla a 37 º C durante 30 min para evitar la hibridación no específica de ADN repetitivo de los cromosomas.
- Poner 10 l de la mezcla de hibridación en la diapositiva, y cubrir con un cubreobjetos de 22x22. Evitar la formación de burbujas - burbujas de aire deben eliminarse con una leve presión sobre el cubreobjetos Si se realiza FISH con BAC clon de ADN, vaya directamente al paso 4,20 <./ Em>
- Desnaturalizar la sonda y el ADN del cromosoma simultáneamente usando un bloque de calentamiento a 75 ° C durante 5 min.
- Pegamento cubreobjetos en todo el perímetro con cemento de goma.
- Realizar la hibridación durante la noche en una cámara húmeda a 37 ° C.
- Eliminar el cemento de goma y el cubreobjetos de la diapositiva.
- Lave el portaobjetos en solución de 2 min precalentado 1 (0,4 x SSC, 0,3% Nonidet-P40) a 73 ° C.
- Lavar los portaobjetos en la solución de 2 (2x SSC, 0,1% de Nonidet-P40) durante 5 min a RT.
- Contrateñir diapositiva con 0,001 mM YOYO-1 en 1x PBS durante 10 min en una cámara húmeda a temperatura ambiente.
- Montar en una pequeña cantidad de reactivo de Gold Prolong Antifade con un cubreobjetos.
- Analizar preparados bajo un microscopio de fluorescencia usando conjuntos de filtros apropiados en aumento x 1.000 (Figura 4).
5. Los resultados representativos
IDs de insectos se encuentran en cada segmento de la larva. Dependiendo de la posición, they transformarse en diferentes tejidos en el estado adulto del insecto. Los identificadores que se utilizan para la preparación de cromosomas en este protocolo, se desarrollan en las piernas en la etapa adulta del mosquito. Estos ID se encuentra en el lado ventral del tórax larval y son claramente visibles a través de la cutícula en el microscopio (Figura 1). A principios de los años 4 ª etapa estadio larval, IDs tienen una forma redonda (fig. 1A). El mayor número de mitosis, ~ 175 en una ID 9, se acumulan a un después "de forma ovalada" etapa (Figura 1B), que debe ser considerada la etapa óptima para la preparación de los portaobjetos. En este momento, el ID intermedio se divide en dos: una transforma en una pierna y un otro transforma en un ala. Nosotros preferimos utilizar los identificadores grandes de la pierna en la "ovalada" escenario para la preparación de los portaobjetos cromosoma. Figura 1C representa IDs en la última etapa de la 4 ª desarrollo larva instar. En esta etapa, laIDs ya se han desarrollado en las piernas y las alas, y contienen una cantidad significativa de los tejidos diferenciados y un bajo número de mitosis. Esta etapa de desarrollo ID debe evitarse para preparación de cromosomas de diapositivas. También se recomienda la cría de larvas de mosquito a bajas temperaturas:. 16 ° C para Aedes y Culex y 22 ° C para Anopheles Esto ayuda a aumentar la cantidad de mitosis en las identificaciones 9.
La figura 2 ilustra una disección Identificación del tórax de 4 de larva instar. Debido a que la cutícula de un insecto vivo es difícil de analizar, se recomienda utilizar unas tijeras de disección en lugar de las agujas utilizadas para la preparación de larva. El procedimiento más crucial para obtener alta calidad de preparación de cromosomas es el tratamiento de solución hipotónica. Para obtener los mejores resultados, retire el intestino y la grasa corporal desde el tórax larval antes de este tratamiento. Hinchazón de las células ID durante este procedimiento ayuda a difundir el cromosomas en un portaobjetos (Figura 3A). La calidad apropiada de la solución de tratamiento hipotónico se puede reconocer fácilmente por la forma redonda de las células en las preparaciones (Figura 3A, B). Las células con una forma oval indicar tratamiento insuficiente solución hipotónica (Figura 3C). Para ser seleccionado para FISH, preparación de cromosomas debe contener al menos 50 de alta calidad extensiones de cromosomas. Normalmente, ~ 90% de los portaobjetos preparados usando este protocolo tiene suficiente calidad para FISH 9.
Se presentan dos protocolos pesqueros ligeramente diferentes: un protocolo avanzado para FISH utilizando sondas de ADN genómico clon BAC en los cromosomas mitóticos de Aedes y Culex y un protocolo de FISH para ADNr sencillo IGS sonda en los cromosomas mitóticos de Anopheles. Los genomas de Aedes y Culex son muy repetitivos debido a la sobrerrepresentación de los elementos transponibles 7,8. Por lo tanto, la realización de FISH, que utilizes ADN genómico clon BAC como una sonda, requiere la adición de fracciones de ADN repetitivas sin etiqueta a la sonda para bloquear la hibridación inespecífica de las repeticiones de ADN en los cromosomas. Para la extracción de las fracciones de ADN repetitivas, el ADN genómico se desnaturaliza a 120 º C durante 2 min. ADN de ebullición a alta temperatura también ayuda a obtener el ADN en fragmentos de 200-500 pb. ADN se permite volver a asociar después de este tratamiento. Los fragmentos de ADN altamente repetitivas tienden a encontrar su compañero de reasociación más rápido que el ADN con secuencias únicas hace. Como resultado, la reasociación de ADN sigue una curva C 0 x t donde C 0 es la concentración inicial de ADN de cadena simple, y t es el tiempo de reasociación. Fracciones de ADN con C 0 t valores iguales a 10-4 - 10-1 100-102 o se consideran altamente repetitivo y moderada, respectivamente. El tiempo de la reasociación para diferentes fracciones de ADN C 0 t se puede calcular utilizando the fórmula t = 0 t C X × 4,98 / C 0, donde t - tiempo de incubación, C 0 t X - C 0 t fracción (C0t 1 = 1, C 0 t 2 = 2, etc) y C 0 - concentración de ADN inicial en g / l 15 (Tabla 1). Después de la reasociación, el ADN monocatenario se digirió con nucleasa S1. Nosotros preferimos utilizar todas las fracciones de ADN C 0 t a C 0 t 3 juntos en lugar de los de uso común C 0 t 1 fracción de ADN. Estas fracciones C 0 t incluyen algunas de las secuencias de ADN moderadamente repetitivas y juntos representan normalmente un 35-50% de la cantidad original del ADN genómico en Ae. aegypti. La proporción correcta entre la sonda de ADN con y sin etiqueta C 0 t fracción de ADN depende de la componente de ADN repetitivo en particular, cada clon BAC. En promedio, usamos 1:20 sonda C 0 tProporción de ADN para obtener una fracción señales aceptables / Ratio fondo del resultado FISH. La prehibridación de la sonda de ADN con C 0 fracciones de ADN T en un tubo de 30 min antes de la hibridación real de la diapositiva también ayuda a reducir el fondo. Etiquetado, la hibridación en sí, y lavado en este protocolo se llevan a cabo utilizando condiciones estándar 12.
Los resultados de FISH de dos diferentes sondas de ADN marcadas clon BAC en los cromosomas mitóticos de Ae. aegypti y quinquefasciatus Cx. se muestra en las Figuras 4A y B, respectivamente. Las sondas de BAC clon de ADN producir fuertes señales en una sola posición en los cromosomas. Los cromosomas que se muestran en la Figura 1 se tiñen con yoduro de YOYO-1. Este colorante produce los mejores patrones de bandeo de Ae. aegypti cromosomas 9. Alternativamente, otros tintes fluorescentes, tales como DAPI o yoduro de propidio, se puede utilizar para the counterstaining cromosoma. Para photobleaching supresión de las diapositivas, se utiliza Prolongar medio Oro Antifade montaje. Este reactivo tiene una buena capacidad de conservación de la señal y también se puede retirar fácilmente de la corredera mediante lavado en PBS 1x si es necesario utilizar el mismo portaobjetos para hibridaciones varios.
Una versión sencilla del protocolo FISH está diseñado para la hibridación de ADNr IGS sonda en los cromosomas mitóticos de Anopheles. Genes ribosomales en Anopheles se representan como un grupo polimórfico de los genes localizados en 16 cromosomas sexuales. Una sonda de ADN en este protocolo se etiqueta con el estándar de reacción de PCR mediante la adición de la etiqueta fluorescente Cy3 o Cy5 dNTPs. Debido a que el bloqueo de la hibridación no específica de ADN repetitivo en la eucromatina no es necesario, todas las medidas relacionadas con el uso de C 0 t fracciones de ADN se omiten. En cambio, las preparaciones de cromosomas se tratan previamente con RNasa para la prevención de la hibridación de la sonda de ADNr para IGSel nucleolo. Los cromosomas y la sonda de ADN se desnaturalizó por calentamiento simultáneamente la corredera junto con una sonda en un sistema de hibridación a 75 ° C durante 5 min. La hibridación y el lavado en este protocolo también se realizan usando condiciones estándar para FISH 12. El resultado de FISH se demuestra en la Figura 4C: el polimorfismo de la hibridación IGS rDNA entre dos cromosomas X es claramente visible.
C 0 t 3| Concentración de ADN g / l | Reasociación tiempo, min | |
| C 0 t 2 | 0,1 | 100 |
| 0,3 | 33 | |
| 0,5 | 20 | |
| 0,7 | 14 | |
| 0,9 | 11 | |
| 1 | 10 | |
| 0,1 | 150 | |
| 0,3 | 50 | |
| 0,5 | 30 | |
| 0,7 | 21 | |
| 0,9 | 17 | |
| 1 | 15 |
Tabla 1. Concentración de ADN y los tiempos de reasociación para la preparación de C 0 t2 y t3 C 0 fracciones.
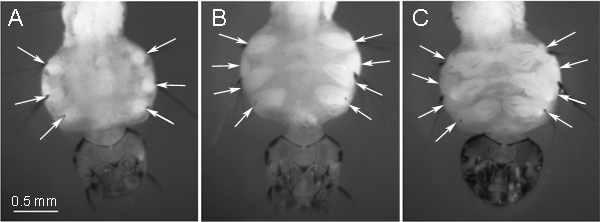
Figura 1 Etapas del desarrollo de ID en 4 ª larva: A) uno de los primeros "forma redonda" nivel de agua; B) un compuesto intermedio "óvalo" etapa - óptimo para la preparación de cromosomas, C) un periodo tardío - apropiado para las preparaciones cromosómicas. . Las posiciones de los ID están indicadas por flechas en el lado ventral del tórax larval.
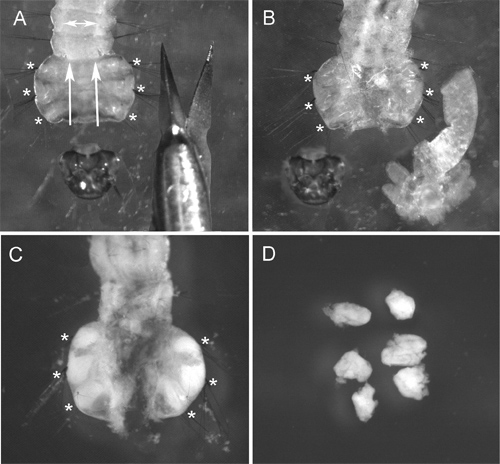
Figura 2 Pasos de disección ID: una larva) decapitada (la dirección de los cortes se indica por las flechas), B) larvas con tripa disecados bajo tratamiento con una solución hipotónica (IDs hinchan y se vuelven casi invisibles); C) larva después de la aplicación solución de Carnoy (. ID de convertirse en blanco y claramente visible), D) disecado ID en solución de Carnoy. Las posiciones de ID en larva se indican con asteriscos.
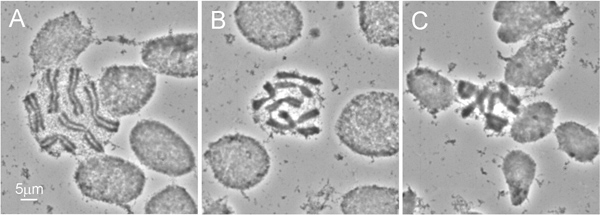
Figura 3 calidades diferentes de las extensiones de cromosomas: A) un margen cromosoma perfecto - forma redonda de las células demuestra tratamiento suficiente de los ID de solución hipotónica, b) un tratamiento hipotónico perfecto - cromosomas son ligeramente undersquashed, c) un cromosoma propagación pobres. - el resultado detratamiento hipotónico insuficiente se indica con forma ovalada de las células.
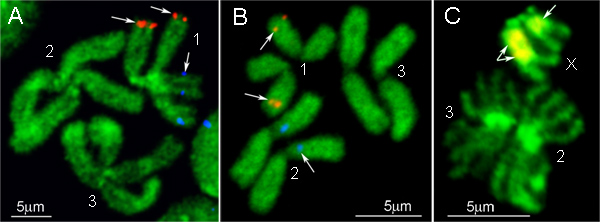
Figura 4. Ejemplos de FISH con clones BAC (A, B) e IGS ADNr (C) en los cromosomas de Ae. aegypti (A), Cx. quinquefasciatus (B), y un. gambiae (C). 1, 2 y 3 - son números de cromosomas; X - cromosoma sexual femenina en una. gambiae.
Discusión
No fluorescentes de hibridación in situ en cromosomas mitóticos de mosquitos se realizó por primera vez en 1990 por A. Kumar y K. Rai 17. En ese estudio, 18S y los genes 28S ribosomal DNA clonado, juntos en un plásmido, se colocaron a los cromosomas de las 20 especies de mosquitos. La sonda de ADN se marcó radiactivamente y se hibridaron a los cromosomas de los ganglios de cerebro. Entre los tres géneros de mosquitos, una técnica de FISH se ha desarrollado sólo para los cromosomas mitóticos a partir de la línea celular de Ae. aegypti 10,18,19 y nunca se ha realizado en los cromosomas mitóticos de mosquitos vivos. Recientemente, hemos desarrollado una técnica sencilla y robusta para obtener alta calidad preparaciones de cromosomas de las IDs de 4 º estadio las larvas 9. Este método permite un elevado número de cromosomas que se obtenga en una diapositiva y puede ser utilizado universalmente para todas las especies de mosquitos. La necesidad de utilizar sta no sólo las larvas, pupa o adultoGES de mosquitos, para la preparación de diapositivas es probablemente la única limitación del método. El método FISH estándar 12 se ha optimizado para el uso de genómica clon BAC y rDNA IGS como sondas para los cromosomas mitóticos de Aedes, Culex, y Anopheles.
Además de estas aplicaciones específicas, los protocolos de FISH descritos aquí también se puede utilizar para otros fines. El protocolo FISH avanzada, que utiliza C 0 fracciones de ADN T para bloquear la hibridación no específica, también se puede aplicar para la hibridación de clones BAC o cualquier otros fragmentos grandes de ADN en las regiones heterocromáticas de Anopheles. Heterochromatic regiones se enriquecen con elementos transponibles y repite otras, y las sondas de estas regiones producen normalmente sólida formación en los 3 cromosomas. Uso no etiquetados C 0 fracciones de ADN T ayudará a reducir la hibridación no específica de la sonda a los cromosomas. La versión simple del FISH protocol puede ser utilizado para cualquier rDNA o sondas de ADN repetitivo en los cromosomas mitóticos de mosquitos y otros insectos. Además, también se puede aplicar para la hibridación de ADN clon BAC en las especies con bajo contenido de ADN repetitivo en euchromatic regiones tales como Anopheles o Drosophila. El protocolo propuesto aquí le ayudará a obtener altamente terminados basados en el cromosoma genoma asambleas para los mosquitos y puede ser ampliamente utilizado para aplicaciones distintas citogenéticas en otros grupos de insectos.
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Damos las gracias a Sergei Demin y Karamysheva Tatyana por su ayuda en la preparación de los cromosomas y FISH en Anopheles. También agradecemos a David Severson por darnos Aedes y Culex ADN genómico clones BAC y Wade Melissa para editar el texto. Este trabajo fue apoyado por dos subvenciones de los Institutos Nacionales de Salud: 1R21 AI88035-01 a Maria V. Sharakhova y 1R21 AI094289-01 a Igor V. Sharakhov.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo / Equipo | Empresa | Número de catálogo | Comentarios |
| MZ6 Leica microscopio estereoscópico | Leica | VA-OM-E194-354 | Un microscopio estereoscópico se pueden utilizar diferentes |
| Olympus CX41 fase microscopio | Olimpo | CX41 | Un microscopio de fase diferente se puede utilizar |
| Olympus BX61 microscopio de fluorescencia | Olimpo | BX61 | Un microscopio fluorescente diferente se puede utilizar |
| Slide ThermoBrite desnaturalización / hibridación Sistema | Abbott Molecular | 30-144110 | Sirve como un bloque de calentamiento y una cámara húmeda |
| Agujas de disección | ScienceTools Artes | 10130-10 | |
| Los porta-agujas | Herramientas Artes Ciencias | 26018-17 | |
| Tijeras de disección | Herramientas Artes Ciencias | 15000-03 | |
| 75x25 doble micro esmerilado se desliza | Corning | 2949-75x25 | |
| 22x22 mm cubreobjetos microscopio | Fisher Scientific | 12-544-10 | |
| Parafilm | Fisher Scientific | 13-374-10 | |
| Adhesivo de goma | Fisher Scientific | 50-949-105 | |
| Ácido acético | Fisher Scientific | A491-212 | |
| Alcohol 200 Proof | Decon Laboratories | 2701 | |
| El ácido propiónico | Sigma-Aldrich | 402907 | |
| Ácido clorhídrico | Fisher Scientific | A144-500 | |
| Sodio dihidrato de citrato | Fisher Scientific | S279-500 | |
| Trihidrato de acetato de sodio | Fisher Scientific | BP334-500 | |
| El cloruro de potasio | Fisher Scientific | BP366-500 | |
| EDTA | Fisher Scientific | S311-500 | |
| Tris base | Fisher Scientific | BP152-1 | |
| 10x PBS | Invitrogen | P5493 | |
| 10% NBF (formalina tamponada neutra) | Sigma-Aldrich | HT501128 | |
| 99% de formamida | Fisher Scientific | BP227500 | |
| El sulfato de dextrano sódico sal | Sigma-Aldrich | D8906 | |
| 20x tampón SSC | Invitrogen | AM9765 | |
| 1 mM de yoduro YOYO-1 (491/509) solución | Invitrogen | Y3601 | |
| Antifade Prolongar reactivo Oro | Invitrogen | P36930 | |
| dATP, dCTP, dGTP, dTTP | Fermentas | R0141, R0151, R0161, R0171 | |
| Cy3-dUTP, Cy5-dUTP | GE Healthcare | PA53022, PA55022 | |
| BSA | Sigma-Aldrich | A3294 | |
| ADN Polimerasa I | Fermentas | EP0041 | |
| DNasa I | Fermentas | EN0521 | |
| Nucleasa S1 | Fermentas | EN0321 | |
| Taq ADN polimerasa | Invitrogen | 18038-042 | |
| RNasa | Sigma-Aldrich | 9001-99-4 | |
| Pepsina | USB | 9001-75-6 | |
| ADN de esperma de salmón | Sigma-Aldrich | D7656 | |
| Nonidet-P40 (NP40) | EE.UU. Biológica | NC9375914 | |
| Qiagen Blood y Cell Culture Maxikit | Qiagen | 13362 | |
| Qiagen Kit construir grandes | Qiagen | 12462 |
Referencias
- Sharakhov, I. V., Sharakhova, M. V., Verrity, J. F., Abbington, L. E. . Chromosome Mapping Research Developments. , (2008).
- Holt, R. A., et al. The genome sequence of the malaria mosquito Anopheles gambiae. Science. 298, 129-149 (2002).
- Sharakhova, M. V., et al. Update of the Anopheles gambiae PEST genome assembly. Genome biology. 8, R5 (2007).
- Campos, J., Andrade, C. F., Recco-Pimentel, S. M. A technique for preparing polytene chromosomes from Aedes aegypti (Diptera, Culicinae). Memorias do Instituto Oswaldo Cruz. 98, 387-390 (2003).
- Campos, J., Andrade, C. F., Recco-Pimentel, S. M. Malpighian tubule polytene chromosomes of Culex quinquefasciatus (Diptera, Culicinae). Memorias do Instituto Oswaldo Cruz. 98, 383-386 (2003).
- McAbee, R. D., Christiansen, J. A., Cornel, A. J. A detailed larval salivary gland polytene chromosome photomap for Culex quinquefasciatus (Diptera: Culicidae) from Johannesburg, South Africa. J. Med. Entomol. 44, 229-237 (2007).
- Nene, V., et al. Genome sequence of Aedes aegypti, a major arbovirus vector. Science. 316, 1718-1723 (2007).
- Arensburger, P., et al. Sequencing of Culex quinquefasciatus establishes a platform for mosquito comparative genomics. Science. 330, 86-88 (2010).
- Sharakhova, M. V., et al. Imaginal discs--a new source of chromosomes for genome mapping of the yellow fever mosquito Aedes aegypti. PLoS neglected tropical diseases. 5, e1335 (2011).
- Brown, S. E., et al. Toward a physical map of Aedes aegypti. Insect Mol. Biol. 4, 161-167 (1995).
- Steiniger, G. E., Mukherjee, A. B. Insect chromosome banding: technique for G- and Q-banding pattern in the mosquito Aedes albopictus. Can. J. Genet. Cytol. 17, 241-244 (1975).
- Garimberti, E., Tosi, S., Bridger, J. M., Volpi, E. V. . Fluorescence in situ hybridization (FISH). , (2010).
- Scott, J. A., Brogdon, W. G., Collins, F. H. Identification of single specimens of the Anopheles gambiae complex by the polymerase chain reaction. The American journal of tropical medicine and hygiene. 49, 520-529 (1993).
- Trifonov, V. A., Vorobyeva, N. N., Rens, W., Leiehr, T. . Fluorescence in situ hybridization (FISH). , (2009).
- Collins, F. H., et al. A ribosomal RNA gene probe differentiates member species of the Anopheles gambiae complex. The American journal of tropical medicine and hygiene. 37, 37-41 (1987).
- Kumar, A., Rai, K. S. Chromosomal localization and copy number of 18S+28S ribosomal RNA genes in evolutionary diverse mosquitoes (Diptera, Culicidae). Hereditas. 113, 277-289 (1990).
- Brown, S. E., Knudson, D. L. FISH landmarks for Aedes aegypti chromosomes. Insect Mol. Biol. 6, 197-202 (1997).
- Brown, S. E., Severson, D. W., Smith, L. A., Knudson, D. L. Integration of the Aedes aegypti mosquito genetic linkage and physical maps. Genetics. 157, 1299-1305 (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados