Method Article
Fluoreszierend
In diesem Artikel
Zusammenfassung
Unter den drei Gattungen Moskito, nämlich Anopheles, Aedes, Und Culex Wurden physikalische Kartierung des Genoms Techniken nur für etablierte Anopheles Besitzen, deren Mitglieder lesbar Polytänchromosomen. Für den Gattungen Aedes Und Culex Bleibt jedoch zytogenetische Zuordnung wegen der schlechten Qualität der Polytänchromosomen Herausforderung. Hier präsentieren wir ein universelles Protokoll für den Erhalt hochwertiger Präparate von mitotischen Chromosomen und eine optimierte FISH-Protokoll für alle drei Gattungen von Mücken.
Zusammenfassung
Fluoreszenz-in-situ-Hybridisierung (FISH) ist eine Technik, die routinemäßig von vielen Labors verwendet werden, um die chromosomale Position von DNA und RNA-Sonden zu bestimmen. Eine wichtige Anwendung dieser Methode ist die Entwicklung von hochwertigen physischen Karten nützlich für die Verbesserung der Genom-Baugruppen für verschiedene Organismen. Die natürliche Bandenmuster polytene und mitotische Chromosomen enthält Leitlinien für die präzise Anordnung und Ausrichtung der genomischen supercontigs. Unter den drei Gattungen Moskito, nämlich Anopheles, Aedes und Culex, ein gut etabliertes Chromosomen-Mapping-Technik ist nur für Anopheles, deren Mitglieder besitzen lesbaren Polytänchromosomen 1 entwickelt worden. Als Ergebnis der Bemühungen Genomkartierung, 88% der Ein. gambiae Genoms hat eine präzise Chromosom Positionen 2,3 gelegt. Zwei weitere mosquito Gattungen Aedes und Culex, haben schlecht Chromosomen b polytenizedeil der signifikante Überrepräsentation von Transposons in ihrem Genom 4, 5, 6. Nur 31 bzw. 9% der genomischen supercontings haben ohne Ordnung und Orientierung an Chromosomen Ae zugewiesen wurde. aegypti 7 und Cx. quinquefasciatus 8 auf. Mitotischen Chromosomen Vorbereitung für diese beiden Arten zuvor auf Gehirn Ganglien und Zelllinien beschränkt. Allerdings Chromosom Dias aus dem Gehirn Ganglien Mücken vorbereitet enthalten in der Regel geringe Zahl von Metaphase Platten 9. Außerdem, obwohl eine FISH-Technik wurde für mitotischen Chromosomen aus einer Zelllinie von Ae entwickelt. aegypti 10, macht die Ansammlung von mehreren chromosomalen Umlagerungen in Zelllinie Chromosomen 11 sie nutzlos für die Genom-Mapping. Hier beschreiben wir eine einfache, robuste Technik zur Gewinnung hochwertiger mitotischen Chromosomen Präparationen aus Imaginalscheiben (IDs) von 4 th Larvenstadium die ceine für alle drei Gattungen von Moskitos verwendet werden. Ein Standard-FISH-Protokoll 12 ist für die Verwendung von BAC-Klone von genomischer DNA als eine Sonde auf mitotischen Chromosomen Ae optimiert. aegypti und Cx. quinquefasciatus, und zur Verwendung einer intergenischen Spacer (IGS) Region der ribosomalen DNA (rDNA) als Sonde auf einem. gambiae Chromosomen. Neben physikalische Kartierung, kann die entwickelte Technik zur Bevölkerung Zytogenetik und Chromosom Taxonomie / Systematik der Mücken und andere Insekten Gruppen angewandt werden.
Protokoll
Ein. Chromosome Vorbereitung
Mückenlarven aufgezogen wurden mit einem Standard-Protokoll in Methods in Anopheles Forschung zur Verfügung beschrieben auf der Website der Malaria Forschung und Reference Reagent Resource Center (MR4) 13. Die Temperaturen von Moskito Aufzucht wurden geändert, um die höchste Anzahl von Chromosomen in Imaginalscheiben und niedrigsten Mortalität der Larven liefern. Die Stufen der Mückenlarven Entwicklung wurden auf der Grundlage der Größe ihres Kopfes Kapseln 13 bestimmt.
- Hatch Mücke Eier bei 28 ° C, und nach 2-3 Tagen, Transfer 2 nd oder 3 rd Larven bis 16 ° C für Ae. aegypti und Cx. quinquefasciatus und auf 22 ° C für eine. gambiae.
- Platz 4 th Larven auf Eis für einige Minuten zur Immobilisierung.
- Übertragen Larve zu einer Folie mit einem Tropfen kalten hypotonen Lösung (0,5%, sodium Citrat oder 0,075 M Kaliumchlorid) und platzieren Sie sie unter dem Stereomikroskop.
- Wählen Larve mit ovalen IDs (Abbildung 1B) zur weiteren Präparation.
- Enthaupten Larve, und schneiden Sie die Nagelhaut von der Bauchseite des larvalen Thorax mit Präparierscheren (Abbildung 2A). Nehmen Sie zusätzliche Schnitt im zweiten oder dritten Abdominalsegment, um den Darm von der Larve zu sezieren. Die Richtungen der Schnitte sind durch Pfeile dargestellt.
- Öffnen Sie die Nagelhaut, und entfernen Sie den Darm und fetten Körper von der Larve. Entfernen Sie die hypotone Lösung aus der Folie mit Filterpapier, und fügen Sie einen frischen Tropfen hypotonen Lösung direkt auf die IDs (Abbildung 2B). Halten Larve in hypotonischer Lösung für 10 min bei RT.
- Entfernen hypotone Lösung mit Filterpapier und gelten Carnoy-Lösung (Ethanol / Essigsäure in Verhältnis 3:1). Nach der Zugabe von Fixierlösung, IDS sofort wieder weiß und werden leicht unter dem Mikroskop sichtbar (Abbildung 2C ).
- Mit Präpariernadeln, entfernen IDs von der Larve (Abbildung 2D), und übertragen Sie sie zu einem Rückgang von 50% Propionsäure. Eventuelle andere Gewebe, wie dem Darm und Fettkörper, von der Folie. Titelbild IDs mit einem unsiliconized 22x22 Deckglas, und halten Sie für 10 min bei RT.
- Bedecken Sie den Objektträger mit Filterpapier und Squash das Gewebe, indem Sie den Radiergummi eines Bleistifts auf dem Umfang des Deckglases.
- Kurz analysieren die Qualität der Folie mit dem Phasenkontrast-Mikroskop mit 100x oder 200x Vergrößerung (Abbildung 3). Zubereitungen mit> 50 Chromosomen Spreads können als geeignet für FISH werden.
- Tauchen Sie und halten Sie die Folie in flüssigem Stickstoff bis er Blasen stoppt. Entfernen des Deckglases von dem Schieber mit einer Rasierklinge, und übertragen die Folie sofort in einen Behälter mit 70% Ethanol bei -20 ° C gekühlt Lagerung bei 4 ° C für mindestens 1 Stunde für die beste Dehydration Ergebnis (wenn nötig, können Dias t gespeichert werdenseinen Schritt von einigen Minuten bis zu mehreren Tagen).
- Dehydratisieren Dias in einer Reihe von Ethanol (70%, 80%, 100%) bei 4 ° C für jeweils 5 min, und bei RT trocknen.
- Trocken lagern Objektträger bei -20 ° C vor Verwendung sie für FISH.
2. Extraktion von Repetitive DNA Fraktionen
Durchführen FISH des BAC-Klons DNA-Sonde auf Chromosomen von Ae. aegypti und Cx. quinquefasciatus erfordert mit unmarkierten repetitiven DNA Fraktionen auf unspezifische Hybridisierung der DNA-Wiederholungen in den Chromosomen blockieren. Die Reassoziation von Einzelstrang-DNA in Stücke von mehreren hundert bp fragmentiert folgt eine C 0 t Kurve wobei C 0 die Anfangskonzentration der Einzelstrang-DNA und t die Zeit Reannealing. Fraktionen mit DNA C0t Werte gleich 10 -4 -10 -1 oder 10 ° -10 2 als hoch und mäßig wiederholende, jeweils betrachtet.
- Auszug 400-500 ug des genomischen DNA aus dem gesamten erwachsenen Mücken mit Qiagen Blood and Cell Culture Maxikit und bereiten 100-1000 ng / ul DNA-Lösung in 1,2 x SSC.
- DNA denaturiert, indem ein sicheres-Schleusenrohr mit genomischer DNA in einen Heizblock vorgewärmt auf 120 ° C für 2 min. Hohe Temperatur hilft, DNA in 200-500 bp Fragmente reichen.
- In Abhängigkeit von der DNA-Konzentration, reassoziieren DNA, indem das Röhrchen bei 60 ° C für 15-150 min bis C 0 t DNA Fraktionen zu erhalten bis C 0 t3 (Tabelle 1).
- Das Röhrchen mit DNA auf Eis für 2 min.
- Übertragung der DNA auf 42 ° C, Hinzufügen vorgewärmten 10x S1 Nuklease-Puffer und S1 Nuklease in einer Endkonzentration von 100 U pro 1 mg DNA und für 1 Stunde inkubiert.
- Präzipitat-DNA durch Zugabe von 0,1 Vol. 3 M Natriumacetat und 1 Volumen Isopropanol bei RT.
- Zentrifugieren bei 14.000 UpM für 20 min bei 4 ° C.
- Waschen DNA in 70% Ethanol und zentrifugieren bei 14.000 UpM für 10 minbei 4 ° C.
- Luft trocknen und lösen DNA-Pellet in TE-Puffer.
- Messung der DNA-Konzentration und Visualisieren durch Gelelektrophorese. Normalerweise ist die Anzahl der repetitiven DNA Fraktionen repräsentiert 35-50% der ursprünglichen DNA-Menge.
3. DNA Probe Labeling
Zwei verschiedene Protokolle wurden für die Kennzeichnung BAC-Klon DNA-Sonde und IGS rDNA Sonde verwendet.
3,1 BAC-Klon Kennzeichnung mit Nick-Translation
- Auszug BAC-Klon-DNA aus dem BAC-Bibliothek mit Qiagen Large Construct Kit.
- Planen Reaktionsmischung zur Nick-Translation Kennzeichnung auf Eis mit Endvolumen von 50 ul: 1 ug DNA isoliert BAC-Klon, 0.05 mM jeweils von unmarkiertem dATP, dCTP und dGTP und 0,015 mM dTTP, 1 ul Cy3-dUTP (oder ein anderes Fluorochrom), 0,05 mg / ml BSA, 5 ul 10x Nick-Translations-Puffer, 20 U DNA-Polymerase I und 0,0012 U DNase.
- Inkubieren bei 15 ° C für 20,5 Std.
- Stoppen der Reaktion durch Zugabe von 1 ul 0,5 M EDTA.
- Shop-Sonde bei -20 ° C an einem dunklen Ort.
3,2 IGS rDNA Kennzeichnung mittels PCR
- Planen Reaktionsgemisch auf Eis mit Endvolumen von 50 ul: 200 ng genomische DNA, 0,05 mM jeweils von unmarkiertem dATP, dCTP und dGTP, 0,015 mM dTTP, 1 ul Cy3-dUTP (oder einem anderen Fluorochrom), 5 ul 10x PCR-Puffer, 50 pmol von vorn; UN (GTGTGCCCCTTCCTCGATGT) und umgekehrt; GA (CTGGTTTGGTCGGCACGTTT) Primer zur Amplifikation IGS, und 10 U Taq DNA-Polymerase 14.
- PCR-Reaktion durchzuführen mittels Standard-PCR-Parameter für IGS Amplifikation: 95 ° C / 5 min x 1 Zyklus; (95 ° C / 30 sec, 50 ° C / 30 sec, 72 ° C / 30 sec) x 30 Zyklen; 72 ° C / 5 min x 1 Zyklus; und 4 ° C zu halten 14.
- Shop-Sonde bei -20 ° C an einem dunklen Ort.
4. Fluoreszenz-in-situ-Hybridisierung
Diese FISH Protokoll umfasst zwei Varianten: die erste für die Verwendung BAC-Klon DNA als Sonde auf mitotische Chromosomen Ae. aegypti und Cx. quinquefasciatus und die zweite für die Verwendung IGS rDNA auf mitotische Chromosomen An. gambiae. Bei der Verwendung von BAC-Klon DNA-Sonden, die Schritte überspringen RNase-Behandlung 4,3, 4,4 und parallele Folienpräsentation / Sonde Denaturierungsschritt 4,19. Bei Verwendung IGS rDNA-Sonde, bereiten Hybridisierung Mischung ohne C 0 t DNA Fraktionen, und überspringen separate Folie / Sonde Denaturierung Schritte 4,10, 4,11, 4,16 und 4,17.
- Die Objektträger in 2x SSC für 30 Minuten bei 37 ° C.
- Entwässern Folien in Serie von 70%, 80% und 100% Ethanol für jeweils 5 min bei RT und an der Luft trocknen. Bei Durchführung FISH mit BAC-Klon DNA, gehen Sie direkt auf 4,5 fort.
- Inkubieren Chromosom Zubereitung in 0,1 mg / ml RNase Lösung unter Parafilm für 30 min bei 37 ° C.
- Zweimal waschen in 2x SSC für jeweils 5 min bei 37 ° C.
- Legen Sie Dias ina jar mit 0,01% Pepsin und 0,037% HCl-Lösung und Inkubation für 5 min bei 37 ° C.
- Waschen Folien in 1x PBS für 5 min bei RT.
- Fix Chromosom Zubereitung in ein Gefäß mit 1% Formalin in PBS 1x von 10% hergestellt neutral gepuffertem Formalin für 10 min bei RT.
- Waschen Folien in 1x PBS für 5 min bei RT.
- Entwässern Folien in Serie von 70%, 80% und 100% Ethanol für jeweils 5 min bei RT und an der Luft trocknen Präparate bei 37 ° C. Bei Durchführung FISH mit IGS, gehen Sie direkt zu 4,12 Schritt
- Denaturieren Dias in einem Glas mit vorgewärmtem 70% Formamid für 2 min bei 72 ° C.
- Entwässern Folien Reihe von Kälte (-20 ° C) 70%, 80% und 100% Ethanol für jeweils 5 Minuten, und an der Luft trocknen bei 37 ° C.
- Bereiten Hybridisierungsmischung:. 5 ul markierten Sonde DNA aus Schritt 3, 10 ul C 0 t DNA aus Schritt 2 mit einer Endkonzentration von 0,5 ng / ul und 5 ul 1 ug / ul beschallte Lachssperma-DNA für Fische mit IGS rDNA, bereitenHybridisierung Mischung ohne C 0 t DNA Fraktionen.
- Präzipitat-DNA durch Zugabe von 0,1 Vol. 3 M Natriumacetat und 2 Volumina Ethanol. Halten Sie bei -20 ° C für 1-3 Stunden.
- Zentrifuge bei 14.000 rpm bei 4 ° C für 20 min, das Ethanol, und an der Luft trocknen das Pellet bei RT.
- Gründlich das Pellet in 10 ul Hybridisierungspuffer:. 50% Formamid, 20% Dextransulfat, 2x SSC Wenn Durchführung FISH mit IGS, gehen Sie direkt zu 4,18 Schritt
- Denaturieren Hybridisierungsgemisch für 7 min bei 97 ° C, und sofort auf Eis gestellt für 1 min.
- Prehybridize Mischung bei 37 ° C für 30 min bis unspezifische Hybridisierung der repetitiven DNA in den Chromosomen verhindern.
- Zeigen 10 ul der Hybridisierung Mischung auf der Folie, und Abdeckung mit einem 22x22 Deckglas. Verhindern Blasenbildung - Luftblasen sollten mit leichtem Druck auf das Deckglas entfernt werden, wenn die Durchführung FISH mit BAC-Klon DNA, gehen Sie direkt zu 4,20 Schritt <./ Em>
- Denaturieren der Sonde und chromosomalen DNA simultan mittels Heizblock bei 75 ° C für 5 min.
- Kleber Deckglas um den Umfang mit Gummi-Zement.
- Führen Hybridisierung über Nacht in einer feuchten Kammer bei 37 ° C.
- Entfernen Gummilösung und Deckglas vom Objektträger.
- Waschen Schlitten 2 min in vorgewärmten Lösung 1 (0,4 x SSC, 0,3% Nonidet-P40) bei 73 ° C.
- Waschen Folien in Lösung 2 (2x SSC, 0.1% Nonidet-P40) für 5 min bei RT.
- Gegenfärbung Folie mit 0,001 mM YOYO-1 in 1x PBS für 10 min in der feuchten Kammer bei RT.
- Montieren in einer kleinen Menge an Gold-Prolong antifade Reagens mit einem Deckglas.
- Analysieren Präparate unter einem Fluoreszenzmikroskop mit geeigneten Filter-Sets bei 1.000-facher Vergrößerung (Abbildung 4).
5. Repräsentative Ergebnisse
Insekten-IDs werden in jedem Segment der Larve befindet. Je nach Stellung, they in verschiedene Gewebe verwandeln im erwachsenen Stadium des Insekts. Die IDs, die für die Herstellung Chromosom in diesem Protokoll verwendet werden, in Beine beim erwachsenen Stadium der Mücke entwickeln. Diese IDs sind an der ventralen Seite des Thorax Larvenstadium befindet und deutlich sichtbar sind durch die Kutikula unter dem Mikroskop (Abbildung 1). Am frühen 4. Larvenstadium Larvenstadium haben IDs eine runde Form (Abbildung 1A). Die größte Anzahl der Mitose werden ~ 175 in ein ID 9, zu einem späteren "ovale" Bühne (Abbildung 1B), die als die optimale Bühne für Präparat muss angesammelt. Zu diesem Zeitpunkt teilt das Zwischenstück in zwei ID: eine Transformationen in einen Schenkel und eine andere verwandelt sich in einen Flügel. Wir bevorzugen mit den großen Bein-IDs auf dem "ovalen" Bühne für das Chromosom Präparat. 1C stellt IDs auf dem neuesten Stand der 4 th instar Larve Entwicklung. In diesem Stadium, dasIDs sind bereits in Beinen und Flügeln entwickelt, und enthalten eine signifikante Menge von differenzierten Gewebe und eine niedrige Anzahl von Mitose. Dieses Stadium der ID Entwicklung sollte für das Chromosom Präparat vermieden werden. Wir empfehlen auch die Aufzucht Mückenlarven bei niedrigen Temperaturen:. 16 ° C für Aedes und Culex und 22 ° C für Anopheles Dies hilft, die Menge der Mitose in IDs 9 erhöhen.
Abbildung 2 zeigt ID Dissektion aus dem Thorax von 4 th instar Larve. Da die Kutikula einer Live-Insekt ist schwer zu sezieren, empfehlen wir die Verwendung Präparierscheren statt der Nadeln häufig für Larven der Herstellung verwendet. Das wichtigste Verfahren zur Gewinnung von hochwertigen Chromosom Vorbereitung ist die hypotonische Lösung Behandlung. Für beste Ergebnisse, entfernen wir den Darm und fetten Körper aus dem Larvenstadium Thorax vor dieser Behandlung. Schwellung der ID-Zellen während dieser Prozedur hilft Chromosom verteilts auf einem Schlitten (3A). Die geeignete Qualität der hypotonischen Lösung Behandlung kann leicht durch die runde Form der Zellen in den Zubereitungen (3A, B) erkannt werden. Zellen mit einer ovalen Form zeigen eine unzureichende Behandlung hypotonischen Lösung (3C). Um FISH gewählt werden, Chromosomen Vorbereitung mindestens 50 hochwertige Chromosom Spreads. Normalerweise haben ~ 90% der Folien hergestellt unter Verwendung dieses Protokolls ausreichende Qualität für FISH 9.
Wir präsentieren zwei leicht unterschiedliche FISH-Protokolle: ein erweitertes Protokoll für FISH unter Verwendung genomischer BAC-Klon DNA-Sonde auf mitotischen Chromosomen Aedes und Culex und einer einfachen FISH-Protokoll für IGS rDNA Sonde auf mitotischen Chromosomen Anopheles. Die Genome von Aedes und Culex sind stark repetitiven wegen der Überrepräsentation von Transposons 7,8. So Durchführung Fische, die utilizes genomischen BAC-Klon-DNA als Sonde, erfordert das Hinzufügen unmarkierten repetitiven DNA Fraktionen an die Sonde, um unspezifische Hybridisierung der DNA-Wiederholungen auf Chromosomen blockieren. Für die Extraktion der repetitiven DNA Fraktionen wird genomische DNA bei 120 ° C für 2 Minuten denaturiert. Siedenden DNA bei einer hohen Temperatur hilft auch DNA in Fragmente von 200-500 bp zu erhalten. DNA läßt man nach dieser Behandlung reassoziieren. Die stark repetitiven DNA-Fragmente neigen dazu, ihre Kumpel für Reassoziation schneller als DNA mit einzigartigen Sequenzen nicht zu finden. Als ein Ergebnis folgt die Reassoziation von DNA einen C 0 x t-Kurve, wobei C 0 die Anfangskonzentration der Einzelstrang-DNA, und t die Zeit Reannealing. DNA Fraktionen mit C 0 t Werte gleich 10-4 - 10-1 oder 100-102 sind hoch und moderat repetitive, als jeweils. Der Zeitpunkt der Neuzuordnungs für verschiedene C 0 t DNA Fraktionen können berechnet werden unter Verwendung the Formel t = C 0 t X × 4,98 / C 0, wobei t - Zeit der Inkubation, C 0 t X - C 0 t Fraktion (C0t 1 = 1, C 0 t 2 = 2, etc.) und C 0 - Ausgangs-DNA-Konzentration in ug / ul 15 (Tabelle 1). Nach Reassoziation wird die Einzelstrang-DNA verdaut mit S1 Nuklease. Wir bevorzugen Verwendung aller C 0 t DNA Fraktionen bis zu C 0 t 3 zusammen anstelle des üblicherweise verwendeten C 0 t 1 DNA-Fraktion. Diese C 0 t Fraktionen gehören einige der moderat repetitiven DNA-Sequenzen und zusammen Regel stellen 35-50% der ursprünglichen Menge der genomischen DNA in Ae. aegypti. Die richtige Verhältnis zwischen markierte DNA-Sonde und unmarkierten C 0 t DNA-Fraktion hängt von der repetitiven DNA-Komponente in jedem einzelnen BAC-Klon. Im Durchschnitt verwenden wir 01.20 Sonde C 0 tDNA-Fraktion Anteil für eine akzeptable Signale / Hintergrund-Verhältnis der FISH Ergebnis. Vorhybridisierung der DNA-Sonde mit C 0 t DNA Fraktionen in einem Röhrchen 30 min vor der eigentlichen Hybridisierung auf der Folie trägt auch zur Hintergrund zu reduzieren. Etikettierung Hybridisierung selbst und das Waschen in diesem Protokoll durchgeführt unter Verwendung von Standardbedingungen 12.
Die FISH Ergebnisse von zwei unterschiedlich markierten BAC-Klon DNA-Sonden auf mitotische Chromosomen Ae. aegypti und Cx. quinquefasciatus sind in 4A und B gezeigt. Die BAC-Klon-DNA-Sonden zu erzeugen starke Signale in einer einzigen Position auf den Chromosomen. Chromosomen in 1 gezeigt sind mit YOYO-1 Iodid gegengefärbt. Dieser Farbstoff produziert die besten Bandenmuster auf Ae. aegypti Chromosomen 9. Alternativ können andere fluoreszierende Farbstoffe wie DAPI oder Propidiumjodid, für th genutzt werdene-Chromosom Gegenfärbung. Zur Unterdrückung Ausbleichen der Folien verwenden wir verlängern Gold-antifade Eindeckmedium. Dieses Reagenz hat eine gute Konservierung Signal Fähigkeiten und kann auch leicht von der Folie werden durch Spülen in 1 × PBS, wenn es notwendig, die für mehrere gleiche Schlitten Hybridisierungen verwenden ist entfernt.
Eine einfache Version des FISH-Protokoll ist für die Hybridisierung von IGS rDNA Sonde auf mitotischen Chromosomen Anopheles konzipiert. Ribosomalen Gene in Anopheles als einer polymorphen Cluster von Genen auf Geschlechtschromosomen 16 befindet vertreten. Eine DNA-Sonde in diesem Protokoll markiert ist mittels Standard-PCR Reaktion durch Zugabe von fluoreszenzmarkierten Cy3 oder Cy5 dNTPs. Weil Blockieren unspezifischer Hybridisierung der repetitiven DNA in Euchromatin nicht benötigt wird, werden alle Schritte in Bezug auf den C 0 t DNA Fraktionen weggelassen. Stattdessen werden Chromosom Präparate mit RNase zur Verhinderung der Hybridisierung der IGS rDNA Sonde vorbehandeltdie Nukleolus. Chromosomen und die DNA-Sonde gleichzeitig durch Erwärmen der Folie zusammen mit einer Sonde in einer Hybridisierung System bei 75 ° C für 5 min denaturiert. Hybridisierung und Waschen in diesem Protokoll sind auch unter Verwendung von Standard-Bedingungen für FISH 12. Das Ergebnis der FISH ist in 4C gezeigt: der Polymorphismus des IGS rDNA Hybridisierung zwischen zwei X-Chromosomen ist deutlich sichtbar.
C 0 t 3| DNA-Konzentration ug / ul | Reannealing, min | |
| C 0 t 2 | 0,1 | 100 |
| 0,3 | 33 | |
| 0,5 | 20 | |
| 0,7 | 14 | |
| 0,9 | 11 | |
| 1 | 10 | |
| 0,1 | 150 | |
| 0,3 | 50 | |
| 0,5 | 30 | |
| 0,7 | 21 | |
| 0,9 | 17 | |
| 1 | 15 |
Tabelle 1. DNA-Konzentration und Reannealing Zeiten für die Herstellung von C 0 t2 und C 0 t3 Fraktionen.
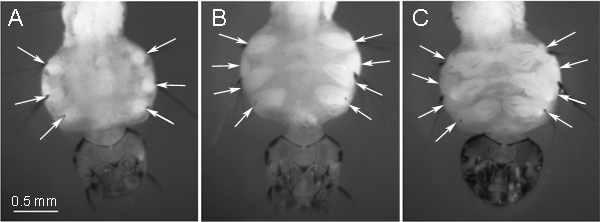
Abbildung 1 Stufen des ID Entwicklung in 4 th instar Larve: A) eine frühe "runde" Bühne; B) ein Zwischenprodukt "oval" stage - optimal für das Chromosom Vorbereitung; C) ein spätes Stadium - ungeeignet für Chromosom Vorbereitungen. . Die Positionen der IDs sind durch Pfeile auf der ventralen Seite des Thorax larvalen angedeutet.
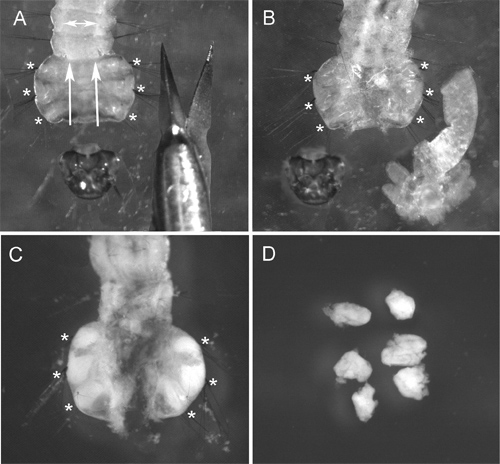
Abbildung 2 Schritte der ID Dissektion: A) enthaupteten Larve (die Richtung der Schnitte sind durch Pfeile angedeutet), B) Larven mit seziert gut unter hypotonen Lösung Behandlung (IDs anschwellen und fast unsichtbar); C) Larve nach Carnoys Lösung (. IDs werden weiß und deutlich sichtbar); D) IDs in Carnoy-Lösung präpariert. Positionen der IDs in Larven sind durch Sternchen gekennzeichnet.
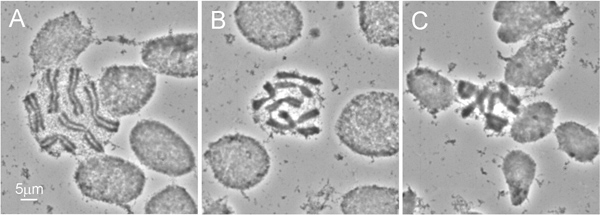
Abbildung 3 unterschiedliche Qualitäten des Chromosoms Spreads: A) eine perfekte Chromosom Spread - runde Form der Zellen zeigt ausreichende Behandlung der IDs in hypotonen Lösung; B) eine perfekte hypotone Behandlung - Chromosomen sind etwas undersquashed; C) eine schlechte Chromosom Verbreitung. - das Ergebnis derunzureichende hypotonische Behandlung wird durch ovale Form der Zellen angegeben.
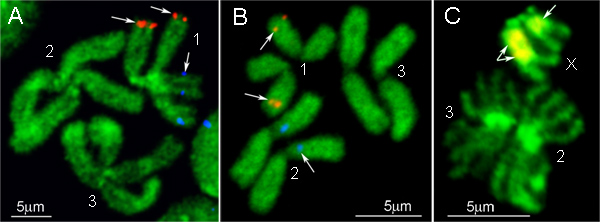
Abbildung 4. Beispiele für FISH mit BAC-Klone (A, B) und IGS rDNA (C) in den Chromosomen Ae. aegypti (A), Cx. quinquefasciatus (B) und ein. gambiae (C). 1, 2 und 3 - Zahlen von Chromosomen, X - weiblichen Geschlechts-Chromosom in eine. gambiae.
Diskussion
Nichtfluoreszierenden in situ Hybridisierung auf mitotischen Chromosomen von Moskitos wurde erstmals 1990 von A. Kumar und K. Rai 17 durchgeführt. In dieser Studie ribosomalen 18S und 28S-DNA-Gene, zusammen in einem Plasmid kloniert wird, wurden auf den Chromosomen 20 Stechmückenarten platziert. Die DNA-Sonde wurde radioaktiv markiert und hybridisiert an die Chromosomen aus Gehirn Ganglien. Zwischen drei Moskito Gattungen wurde eine FISH-Technik nur für mitotischen Chromosomen von der Zelllinie von Ae entwickelt. aegypti 10,18,19 und hat noch nie auf mitotische Chromosomen von Live-Mücken durchgeführt. Kürzlich entwickelten wir eine einfache, robuste Technik für den Erhalt hochwertiger Chromosom Zubereitungen aus IDs von 4 th Larven 9. Diese Methode ermöglicht eine hohe Anzahl von Chromosomen in eine Folie erhalten werden und ist universell für alle Arten von Stechmücken verwendet werden. Die Notwendigkeit der Verwendung von nur Larven, nicht Puppen oder Erwachsener stages von Mücken, zum Präparat ist wahrscheinlich die einzige Einschränkung des Verfahrens. Die Standard-FISH-Methode 12 wurde für die Verwendung von genomischen BAC-Klon und IGS rDNA als Sonden für den mitotischen Chromosomen Aedes, Culex und Anopheles optimiert.
Zusätzlich zu diesen spezifischen Anwendungen können die FISH-Protokolle hier beschriebenen auch für andere Zwecke verwendet werden. Die fortgeschrittene FISH-Protokoll, das C 0 t DNA Fraktionen zur Blockierung unspezifischer Hybridisierung verwendet, können auch für die Hybridisierung von BAC-Klonen oder anderen großen DNA-Fragmente in heterochromatischen Regionen Anopheles aufgebracht werden. Heterochrome Regionen mit Transposons und andere Wiederholungen angereichert und Sonden aus diesen Regionen produzieren normalerweise starken Hintergrund auf den Chromosomen 3. Verwendung unmarkiert C 0 t DNA Fraktionen werden dazu beitragen, unspezifische Hybridisierung der Sonde an die Chromosomen reduzieren. Die einfache Version des FISH protocol kann für jede rDNA oder sich wiederholende DNA-Sonden auf mitotischen Chromosomen von Mücken und anderen Insekten verwendet werden. Darüber hinaus kann es auch für die Hybridisierung von DNA-BAC-Klons in Spezies mit geringem repetitiven DNA-Gehalt in euchromatischen Regionen wie Anopheles oder Drosophila angewendet werden. Die hier vorgeschlagene Protokoll wird dazu beitragen, hoch fertigen Chromosom Genom-basierten Baugruppen für Mücken zu erhalten, und können allgemein für verschiedene zytogenetische Anwendungen in anderen Gruppen von Insekten verwendet werden.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Wir danken Sergei Demin und Tatjana Karamysheva für ihre Hilfe bei Chromosom Vorbereitung und FISH auf Anopheles. Wir danken auch David Severson für die Bereitstellung Aedes und Culex genomische DNA BAC-Klone und Melissa Wade für die Bearbeitung des Textes. 1R21 AI88035-01 bis Maria V. Sharakhova und 1R21 AI094289-01 bis Igor V. Sharakhov: Diese Arbeit wurde durch zwei Zuschüsse aus dem National Institutes of Health unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenz / Equipment | Firma | Katalog-Nummer | Kommentare |
| MZ6 Leica Stereomikroskop | Leica | VA-OM-E194-354 | Eine andere Stereomikroskop benutzt werden |
| Olympus CX41 Phasenkontrastmikroskop | Olymp | CX41 | Eine andere Phase Mikroskop kann verwendet werden |
| Olympus BX61 Fluoreszenzmikroskop | Olymp | BX61 | Eine andere Fluoreszenzmikroskop benutzt werden |
| ThermoBrite Slide Denaturierung / Hybridization System | Abbott Molecular | 30-144110 | Dient als Heizblock und einer feuchten Kammer |
| Präpariernadeln | Feine ScienceTools | 10130-10 | |
| Nadelhalter | Fine Science Tools | 26018-17 | |
| Präparierschere | Fine Science Tools | 15000-03 | |
| 75x25 Doppel frosted micro gleitet | Corning | 2949-75x25 | |
| 22x22 mm Mikroskop Deckgläser | Fisher Scientific | 12-544-10 | |
| Parafilm | Fisher Scientific | 13-374-10 | |
| Rubber Cement | Fisher Scientific | 50-949-105 | |
| Essigsäure | Fisher Scientific | A491-212 | |
| Alcohol 200 Proof | Decon Laboratories | 2701 | |
| Propionsäure | Sigma-ALDRIch | 402907 | |
| Salzsäure | Fisher Scientific | A144-500 | |
| Natriumcitratdihydrat | Fisher Scientific | S279-500 | |
| Natriumacetattrihydrat | Fisher Scientific | BP334-500 | |
| Kaliumchlorid | Fisher Scientific | BP366-500 | |
| EDTA | Fisher Scientific | S311-500 | |
| Tris-Base | Fisher Scientific | BP152-1 | |
| 10x PBS | Invitrogen | P5493 | |
| 10% NBF (neutral gepuffertes Formalin) | Sigma-Aldrich | HT501128 | |
| 99% Formamid | Fisher Scientific | BP227500 | |
| Dextransulfat-Natriumsalz | Sigma-Aldrich | D8906 | |
| 20x SSC-Puffer | Invitrogen | AM9765 | |
| 1 mM YOYO-1-iodid (491/509)-Lösung | Invitrogen | Y3601 | |
| Antifade verlängern Gold-Reagenz | Invitrogen | P36930 | |
| dATP, dCTP, dGTP, dTTP | Fermentas | R0141, R0151, R0161, R0171 | |
| Cy3-dUTP, Cy5-dUTP | GE Healthcare | PA53022, PA55022 | |
| BSA | Sigma-Aldrich | A3294 | |
| DNA-Polymerase I | Fermentas | EP0041 | |
| DNase I | Fermentas | EN0521 | |
| S1 Nuclease | Fermentas | EN0321 | |
| Taq-DNA-Polymerase | Invitrogen | 18038-042 | |
| RNase | Sigma-Aldrich | 9001-99-4 | |
| Pepsin | USB | 9001-75-6 | |
| Lachssperma-DNA | Sigma-Aldrich | D7656 | |
| Nonidet-P40 (NP40) | US Biological | NC9375914 | |
| Qiagen Blood and Cell Culture Maxikit | Qiagen | 13362 | |
| Qiagen Large Construct Kit | Qiagen | 12462 |
Referenzen
- Sharakhov, I. V., Sharakhova, M. V., Verrity, J. F., Abbington, L. E. . Chromosome Mapping Research Developments. , (2008).
- Holt, R. A., et al. The genome sequence of the malaria mosquito Anopheles gambiae. Science. 298, 129-149 (2002).
- Sharakhova, M. V., et al. Update of the Anopheles gambiae PEST genome assembly. Genome biology. 8, R5 (2007).
- Campos, J., Andrade, C. F., Recco-Pimentel, S. M. A technique for preparing polytene chromosomes from Aedes aegypti (Diptera, Culicinae). Memorias do Instituto Oswaldo Cruz. 98, 387-390 (2003).
- Campos, J., Andrade, C. F., Recco-Pimentel, S. M. Malpighian tubule polytene chromosomes of Culex quinquefasciatus (Diptera, Culicinae). Memorias do Instituto Oswaldo Cruz. 98, 383-386 (2003).
- McAbee, R. D., Christiansen, J. A., Cornel, A. J. A detailed larval salivary gland polytene chromosome photomap for Culex quinquefasciatus (Diptera: Culicidae) from Johannesburg, South Africa. J. Med. Entomol. 44, 229-237 (2007).
- Nene, V., et al. Genome sequence of Aedes aegypti, a major arbovirus vector. Science. 316, 1718-1723 (2007).
- Arensburger, P., et al. Sequencing of Culex quinquefasciatus establishes a platform for mosquito comparative genomics. Science. 330, 86-88 (2010).
- Sharakhova, M. V., et al. Imaginal discs--a new source of chromosomes for genome mapping of the yellow fever mosquito Aedes aegypti. PLoS neglected tropical diseases. 5, e1335 (2011).
- Brown, S. E., et al. Toward a physical map of Aedes aegypti. Insect Mol. Biol. 4, 161-167 (1995).
- Steiniger, G. E., Mukherjee, A. B. Insect chromosome banding: technique for G- and Q-banding pattern in the mosquito Aedes albopictus. Can. J. Genet. Cytol. 17, 241-244 (1975).
- Garimberti, E., Tosi, S., Bridger, J. M., Volpi, E. V. . Fluorescence in situ hybridization (FISH). , (2010).
- Scott, J. A., Brogdon, W. G., Collins, F. H. Identification of single specimens of the Anopheles gambiae complex by the polymerase chain reaction. The American journal of tropical medicine and hygiene. 49, 520-529 (1993).
- Trifonov, V. A., Vorobyeva, N. N., Rens, W., Leiehr, T. . Fluorescence in situ hybridization (FISH). , (2009).
- Collins, F. H., et al. A ribosomal RNA gene probe differentiates member species of the Anopheles gambiae complex. The American journal of tropical medicine and hygiene. 37, 37-41 (1987).
- Kumar, A., Rai, K. S. Chromosomal localization and copy number of 18S+28S ribosomal RNA genes in evolutionary diverse mosquitoes (Diptera, Culicidae). Hereditas. 113, 277-289 (1990).
- Brown, S. E., Knudson, D. L. FISH landmarks for Aedes aegypti chromosomes. Insect Mol. Biol. 6, 197-202 (1997).
- Brown, S. E., Severson, D. W., Smith, L. A., Knudson, D. L. Integration of the Aedes aegypti mosquito genetic linkage and physical maps. Genetics. 157, 1299-1305 (2001).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten