Method Article
형광성의
요약
즉 세 모기 장군, 중 아노펠레스, Aedes 및 Culex, 물리적 게놈 매핑 기술은 전용 설립되었다 아노펠레스, 누구의 회원 읽을 수 polytene 염색체를 가지고 있습니다. 의 장군에 대한 Aedes와 Culex 그러나, cytogenetic 매핑 때문에 polytene 염색체의 품질에 도전 남아 있습니다. 여기 고품질의 mitotic 염색체의 준비와 모기의 세 장군을위한 최적화 된 생선 프로토콜을 얻기위한 범용 프로토콜을 제시한다.
초록
현장 하이브리드 화 (물고기)의 형광은 정기적으로 DNA와 RNA 프로브의 염색체 위치를 결정하기 위해 여러 실험실에서 사용하는 기술입니다. 이 방법의 중요한 응용 프로그램은 다양한 생물의 게놈 어셈블리를 개선하기위한 유용한 고품질의 물리적지도의 개발입니다. polytene와 mitotic 염색체의 자연 구간 패턴은 게놈 supercontigs의 정확한 순서와 방향에 대한 지침을 제공합니다. 즉 세 모기 장군, 아노펠레스, Aedes, 그리고 Culex, 중 잘 알려진 염색체 기반의 매핑 기술은 누구의 회원 읽을 수 polytene의 염색체 1을을 가지고 아노펠레스에 대한 개발되었습니다. 게놈 매핑 노력의 88 %의 결과로. gambiae 게놈은 정확한 염색체 위치 2,3에 배치되었습니다. 두 개의 다른 모기 장군, Aedes과 Culex는 제대로 염색체 B를 polytenized 한자신의 genomes 4, 5, 6 transposable 요소의 중요한 overrepresentation의 ecause. 게놈 supercontings 만 31, 9 %는 애의 염색체 명령 또는 방향없이 지정되었습니다. 각각 aegypti 7 CX. quinquefasciatus 8. 이 두 종에 대한 Mitotic의 염색체 준비는 이전에 뇌 신경절과 셀 라인으로 제한 하였다. 그러나, 모기의 뇌 신경절에서 준비 염색체 슬라이드는 일반적으로 metaphase 판 9 낮은 숫자가 포함되어 있습니다. 또한, 물고기 기술이 애의 셀 줄에서 mitotic 염색체을 위해 개발되었습니다 있지만. aegypti 10, 셀 라인 염색체 11에있는 여러 염색체 rearrangements의 축적 게놈지도에 그들을 쓸모합니다. 여기 4 일 instar의 애벌레의 imaginal 디스크 (IDS)에서 높은 품질의 mitotic의 염색체 준비를 얻기위한 간단하고 강력한 기술을 설명하는 C이 모기의 세 장군을 위해 사용될 수 없습니다. 표준 생선 프로토콜 12 애의 mitotic 염색체에 프로브로 게놈 DNA의 BAC 클론의 사용에 최적화되어 있습니다. aegypti 및 CX. quinquefasciatus와의 프로브로 intergenic 스페이서 (IGS) ribosomal DNA의 지역 (rDNA)를 이용하십시오. gambiae 염색체가. 물리적 매핑뿐만 아니라, 개발 된 기술은 인구 cytogenetics와 염색체 분류 / 모기와 다른 곤충 그룹의 systematics에 적용 할 수 있습니다.
프로토콜
1. 염색체 준비
모기 유충은 말라리아 연구 및 참조 시약 리소스 센터의 웹 사이트에서 사용할 수 아노펠레스 연구에 방법에 설명 된 표준 프로토콜을 사용하여 키운 한 (MR4) 13. 모기 양육의 온도는 imaginal 디스크와 애벌레의 낮은 사망률의 염색체 가장 높은 번호를 제공하도록 수정되었습니다. 모기 유충 개발의 단계들은 헤드 캡슐 (13)의 크기에 따라 결정됩니다.
- 28의 해치 모기 계란 ° C, 그리고 애 16 ° C에 2-3일, 이전 2 차 또는 3 instar의 유충 후. aegypti 및 CX. 에 대한 quinquefasciatus, 22 ° C까지. gambiae.
- 고정에 대한 몇 가지 분 얼음에 장소 4 일 instar의 애벌레.
- 추위 hypotonic 솔루션의 한 방울 (0.5 % 때문에 함께 슬라이드에 유충을 전송dium 시트르산 또는 0.075 M의 칼륨 염화물) 및 스테레오 현미경으로 배치합니다.
- 자세한 해부에 타원형 ID를 (그림 1B)와 유충을 선택합니다.
- 유충 목을 벨하고, 해부 가위 (그림 2A)를 사용하여 애벌레의 가슴의 복부 측면에서 큐티클을 잘라. 애벌레에서 직감을 해부하기 위해 제 2 또는 제 3 복부 세그먼트에 추가 몫을합니다. 상처의 방향은 화살표로 표시됩니다.
- 큐티클을 열고 유충의 내장 지방 몸을 제거합니다. 거름 종이를 사용하여 슬라이드에서 hypotonic 솔루션을 제거하고, 직접 ID로 hypotonic 솔루션 (그림 2B)의 신선한 방울을 추가합니다. RT 10 분에 hypotonic 솔루션 유충세요.
- 거름 종이를 사용하여 hypotonic 솔루션을 제거하고, Carnoy의 솔루션 (3시 1분 비율로 에탄올 / 아세트산)를 적용 할 수 있습니다. 정착액 솔루션을 추가 한 후 즉시 (그림 2C 흰색으로 바뀝니다과 현미경으로 쉽게 표시 될 IDS ).
- 해부 바늘을 사용하여 유충 (그림 2D)에서 ID를 제거 50 % 프로피온산 한 방울로 전송할 수 있습니다. 슬라이드에서 같은 내장 지방 몸과 같은 다른 조직을 제거합니다. unsiliconized 22x22 커버 슬립으로 ID를 커버하고, RT에서 10 분 동안 유지.
- 필터 종이 슬라이드, 스쿼시 커버 슬립의 경계에있는 연필의 지우개를 활용하여 조직을 다룹니다.
- 간단히 위상 대비 100x에서 현미경 또는 200x 배율 (그림 3)를 사용하여 슬라이드의 품질을 분석 할 수 있습니다. > 50 염색체 확산과 준비가 물고기에 적합한 것으로 간주 할 수 있습니다.
- 가 버블 링을 멈출 때까지 액체 질소에서 슬라이드를 찍어 누르고 있습니다. 면도날을 사용하여 슬라이드 커버 슬립을 제거하고, -20 ° C.에 냉각 70 % 에탄올의 컨테이너에 즉시 슬라이드를 전송 4 번 스토어 ° C 최적의 탈수 결과 (필요한 경우 최소한 1 시간 동안 슬라이드는 t에 저장할 수 있습니다몇 일 몇 분에서 자신의 단계).
- 5 분마다, 그리고 RT의 공기 건조에 대해 4시 에탄올 일련의 (70 %, 80 %, 100 %) ° C에서 슬라이드를 탈수.
- 생선을 이용하기 전에 -20 ° C에서 건조 슬라이드를 저장합니다.
2. 반복 DNA의 분수의 추출
애의 염색체에있는 BAC 클론의 DNA 프로브의 물고기를 수행하는 중입니다. aegypti 및 CX. quinquefasciatus은 염색체에 DNA 반복의 불특정 하이브리드를 차단하는 라벨이없는 반복적 인 DNA의 분수를 사용이 필요합니다. 수백 BP의 조각으로 조각 단일 스트랜드 DNA의 reassociation는 C 0 단일 좌초 된 DNA의 초기 농도이며, t는 reannealing 시간 C 0t 곡선을 따른다. 10 -4 -10 -1 또는 10 같은 C0t 값 ° -10 2 DNA의 분수는 각각 같은 높은과 적당히 반복 간주됩니다.
- 40 추출0-500 Qiagen의 혈액과 세포 배양 Maxikit를 사용하여 전체 성인 모기의 게놈 DNA의 μg, 그리고 100-1,000 NG / 1.2x SSC에 μl DNA 솔루션을 준비합니다.
- 가열 블록에 게놈 DNA와 안전 잠금 튜브를 삽입하여 변성 DNA는 ° C 2 분에 120 prewarmed. 높은 온도는 200-500 BP 조각으로 DNA를 다양하는 데 도움이됩니다.
- DNA 농도에 따라, C 0 T3 (표 1) C 0t의 DNA 분수를 얻기 위해 15-150 분 동안 60 ° C에서 관을 배치하여 DNA를 다시 연결.
- 2 분을위한 얼음에 DNA와 튜브를 놓습니다.
- 42 DNA를 전송 ° C, DNA의 1 밀리그램 당 100 U의 최종 농도에 preheated 10 배 S1 nuclease 버퍼와 S1 nuclease를 추가하고, 1 시간에 품다.
- 3 M의 아세트산 나트륨과 RT의 이소프로판올 1 볼륨의 0.1 볼륨을 추가하여 석출물 DNA.
- 4 ° C.에 20 분에 14,000 rpm으로 원심 분리기
- 70 % 에탄올에 DNA를 씻어, 10 분 동안 14,000 rpm으로 다시 원심 분리기4 번 ° C.
- 공기 건조 및 TE 버퍼에 DNA 펠릿을 분해.
- DNA 농도를 측정하고, 젤 전기 영동으로 표시합니다. 보통 반복적 인 DNA의 분수의 최종 수량은 원래 DNA 양의 35-50%를 나타냅니다.
3. DNA 프로브 레이블
두 개의 서로 다른 프로토콜 라벨 BAC 클론 DNA 프로브 및 IGS rDNA 프로브에 사용되었다.
대화명 번역을 사용하여 3.1 BAC 클론 라벨
- Qiagen 대형 구조 키트를 사용하여 BAC 라이브러리에서 BAC 클론의 DNA를 추출합니다.
- , Cy3-dUTP 1 μl (또는 다른 1 μg 고립 된 BAC 클론 DNA, 0.05 MM 라벨이없는 dATP, dCTP, 그리고 dGTP 각 및 dTTP의 0.015 MM : 50 μl의 최종 볼륨 얼음에 닉 - 번역 라벨에 대한 반응 혼합물을 준비 fluorochrome), BSA의 0.05 MG / ML, 10 배 닉 번역 버퍼의 5 μl, DNA-중합 효소 20 U I,와 DNase의 0.0012 U.
- 2 15 ° C에서 알을 품다0.5 시간.
- 0.5 M EDTA (에틸렌 다이아 민 테트라 초산) 1 μl를 추가하여 반응을 중지합니다.
- 어두운 장소에서 -20 ° C에서 저장 프로브.
PCR을 사용하여 3.2 IGS rDNA 라벨
- , 0.05 MM 라벨이없는 dATP, dCTP, 그리고 dGTP 각, dTTP의 0.015 MM, Cy3-dUTP (또는 다른 fluorochrome) 1 μl, 5 μl 게놈 DNA 200 NG : 50 μl의 최종 볼륨과 얼음에 반응 혼합물을 준비 10X PCR-버퍼, 앞으로의 50 pmol, UN (GTGTGCCCCTTCCTCGATGT)와 역, IGS 증폭을위한 GA (CTGGTTTGGTCGGCACGTTT) 프리 머 및 DNA 형성 촉매의 DNA 중합 효소 (14)의 10 U.
- (95 ° C / 30 초, 50 ° C / 30 초, 72 ° C / 30 초) X 30주기; 72 ° C - 95 ° C / 5 분 × 1주기 : IGS 증폭을위한 표준 PCR 매개 변수를 사용하여 PCR 반응을 수행 / 5 분 × 1주기,과 4 ° C 14를 개최.
- 어두운 장소에서 -20 ° C에서 저장 프로브.
4. 현장 하이브리드 화에 형광등
이 생선 프로토콜은 두 변형이 포함 애의 mitotic 염색체에 프로브로 BAC 클론의 DNA를 사용하는 첫 번째입니다. aegypti 및 CX. quinquefasciatus와의 mitotic 염색체에 IGS rDNA를 사용하기위한 두 번째. gambiae. BAC 클론의 DNA 프로브를 사용하는 경우, 건너 뛰기 RNase 처리는 4.3, 4.4 단계, 및 동시 슬라이드 / 프로브 변성 단계 4.19. IGS rDNA 프로브를 사용하는 경우, C 0t의 DNA 분수없이 하이브 리다이 제이션 혼합물을 준비하고, 단계 4.10, 4.11, 4.16, 그리고 4.17를 denaturing 별도의 슬라이드 / 프로브를 건너 뛰십시오.
- 37 30 분에 2 배 SSC의 슬라이드를 품다 ° C.
- 5 RT에서 분마다, 에어컨 건조에 70 % 시리즈, 80 %, 100 %의 에탄올에 슬라이드를 탈수. BAC 클론 DNA와 생선을 수행하는 경우, 4.5 단계로 바로 진행합니다.
- 37 ° C에서 30 분에 parafilm 미만의 0.1 MG / ML RNase 솔루션에 염색체 준비를 품다.
- 37 5 분마다 2 배 SSC에 두 번 씻으 ° C.
- 슬라이드 그럼 내가 넣어37 번 0.01 % 펩신과 0.037 % HCL 솔루션, 그리고 5 분에 품다와 NA 항아리 ° C.
- RT에서 5 분에 1 회 PBS로 슬라이드를 씻으십시오.
- RT에서 10 분을위한 포르말린 중립적 인 버퍼 10 % 준비 1X PBS에서 1퍼센트 포르말린과 함께 병 속에 염색체 준비를 수정했습니다.
- RT에서 5 분에 1 회 PBS로 슬라이드를 씻으십시오.
- 37에서 5 RT에서 분마다, 에어컨 건조 준비를 위해 70 % 시리즈, 80 %, 100 %의 에탄올에 슬라이드를 탈수 ° C. IGS와 생선을 수행하는 경우, 4.12 단계로 바로 진행합니다
- 72 ° C.에서 2 분에 prewarmed 70 % 포름 아미드와 병의 변성 슬라이드
- 감기 시리즈 (-20 ° C) 37 70 %, 80 %, 5 분마다, 에어컨 건조를위한 100 % 에탄올 ° C.에 슬라이드를 탈수
- IGS와 생선 3 단계, 1 μg / μl sonicated 연어 정자 DNA의 0.5 / NG μl, 5 μl의 최종 농도 2 단계에서 C 0t의 DNA의 10 μl의 표시 프로브 DNA의 5 μl :. 하이브리드 혼합물을 준비 rDNA는 준비C 0t의 DNA 분수없이 하이브리드 혼합.
- 3 M의 아세트산 나트륨과 에탄올의 2 권의 0.1 볼륨을 추가하여 석출물 DNA. 1-3 시간에 -20 ° C에서 보관.
- 4에 14,000 rpm으로 원심 분리기는 20 분에 ° C, 에탄올을 제거하고 공기 건조 RT의 펠릿.
- 철저하게 하이브 리다이 제이션 버퍼 10 μl의 펠렛을 분해 :. 50 % 포름 아미드, 20 % dextran 황산, 배 SSC는 IGS와 생선을 수행하는 경우, 4.18 단계로 바로 진행합니다
- 7 97시 분 ° C, 즉시에 대한 변성 하이브리드 혼합물은 1 분에 얼음을 넣어.
- 염색체에 반복적 인 DNA의 불특정 하이브리드을 방지하기 위해 30 분에 37 ° C에서 혼합물을 Prehybridize.
- 10 슬라이드의 하이브리드 혼합물의 μl, 그리고 22x22 커버 슬립으로 커버를 놓습니다. 거품 형성을 방지 - 공기 거품은 coverslip에 부드러운 압력을 제거해야 BAC 클론의 DNA와 생선을 수행하는 경우, 4.20 단계로 바로 진행합니다 <./ EM>
- 동시에 5 분에 75에서 가열 블록 ° C를 사용하여 프로브 및 염색체 DNA를 변성.
- 고무 시멘트를 사용하여 둘레 접착제 커버 슬립.
- 37 습한 챔버에 밤새 하이브리드를 수행 ° C.
- 슬라이드에서 고무 시멘트와 coverslip을 제거합니다.
- 73 ° C.에 prewarmed 솔루션 1 (0.4x SSC, 0.3 % Nonidet-P40)에 슬라이드 2 분을 씻으십시오
- RT에서 5 분을위한 솔루션 2 (배 SSC, 0.1 % Nonidet-P40)에 슬라이드를 씻으십시오.
- RT의 습기 챔버에서 10 분 동안 1X PBS에서 0.001 MM 유유-1을 사용하여 슬라이드를 Counterstain.
- 커버 슬립과 골드 antifade 시약을 연장의 작은 금액을 탑재합니다.
- 1000 X 배율 (그림 4)에서 적절한 필터 세트를 사용하여 형광 현미경으로 준비를 분석합니다.
5. 대표 결과
곤충 ID는 유충의 각 세그먼트에 자리 잡고 있습니다. 위치에 따라 일어이은 곤충의 성인용 단계에서 다른 조직으로 변환 할 수 있습니다. 이 프로토콜의 염색체 준비에 사용되는 ID는, 모기의 성인용 단계에서 다리로 개발할 수 있습니다. 이 ID는 애벌레의 가슴의 복부 옆에 위치하고 있으며, 현미경으로 큐티클 (그림 1)을 통해 명확하게 볼 수 있습니다. 초기 4 회 instar 애벌레의 단계에서 ID가 둥근 모양 (그림 1A)를 갖추고 있습니다. 유사 분열의 가장 큰 숫자는 하나 ~ 175 ID 9, 슬라이드 준비에 대한 최적의 무대로 간주해야합니다 뒷부분에있는 "타원형 모양의"단계 (그림 1B)에서 축적되어 있습니다. 현재 중간 ID가 두 개로 분할 : 하나 다리로 변환하고 다른 하나는 날개로 변환합니다. 우리는 염색체 슬라이드 준비에 대해 "타원형 모양의"단계에서 큰 다리 ID를 사용하여 선호합니다. 그림 1C는 4 일 instar의 유충 개발의 최신 단계에서 ID를 나타냅니다. 이 단계에서,ID가 이미 다리와 날개로 발전하고, 차별화 된 조직의 상당한 금액과 유사 분열의 낮은 숫자가 포함되어 있습니다. ID 개발의이 단계 염색체 슬라이드 준비를 위해 피해야한다. 우리는 또한 낮은 온도에서 뒷다리 모기 유충 좋습니다. 16 ° C Aedes과 Culex 22 ° C에 대한 아노펠레스에 대한을이 ID가 9 유사 분열의 양을 증가하는 데 도움이됩니다.
그림 2는 4 일 instar의 유충의 가슴에서 ID의 절개를 보여줍니다. 라이브 곤충의 표피가 해부하기 어려운이기 때문에, 우리는 해부 가위 대신 일반적으로 유충 준비에 사용 된 바늘을 사용하는 것이 좋습니다. 고품질의 염색체 준비를위한 가장 중요한 절차는 hypotonic 솔루션 트리트먼트입니다. 최상의 결과를 얻으려면, 우리는이 치료 전에 애벌레의 가슴에서 내장 지방 몸을 제거합니다. 이 절차를 수행하는 동안 ID 세포의 팽창은 염색체를 확산하는 데 도움이슬라이드 (그림 3A)에 s입니다. hypotonic 솔루션 치료의 적절한 품질은 쉽게 준비에있는 셀의 둥근 모양 (그림 3A, B)에 의해 인식 할 수 있습니다. 타원형 모양으로 세포가 부족 hypotonic 솔루션 치료 (그림 3C)를 나타냅니다. 생선 선택하려면 염색체 준비는 최소 50 고품질의 염색체 확산을 포함해야합니다. 일반적으로,이 프로토콜을 사용하여 준비 슬라이드 ~ 90 % 생선 9 충분한 품질을 가지고 있습니다.
Aedes과 Culex의 mitotic 염색체에 게놈 BAC 클론의 DNA 프로브를 사용하여 생선과 아노펠레스의 mitotic 염색체에 IGS rDNA 프로브에 대한 간단한 생선 프로토콜을위한 고급 프로토콜 : 우리는 두 개의 약간 다른 생선 프로토콜을 제시한다. Aedes과 Culex의 genomes 때문에 transposable 요소 7,8의 overrepresentation의 높은 반복합니다. 따라서, 수행 물고기, UT, 프로브로 게놈 BAC 클론의 DNA를 ilizes 염색체에 DNA 반복의 불특정 하이브리드를 차단하는 프로브에 라벨이없는 반복적 인 DNA의 분수를 추가해야합니다. 반복적 인 DNA의 분수의 추출 들어, 게놈 DNA는 2 분 동안 120 ° C에서 변성되어 있습니다. 높은 온도에서 끓는 DNA는 200-500 BP의 조각에서 DNA를 얻을 수 있습니다. DNA는이 치료 후 다시 연결 할 수 있습니다. 매우 반복적 인 DNA 조각은 빠른 고유 한 시퀀스와 DNA 사람보다 reassociation 위해 짝을 찾는 경향이 있습니다. 그 결과, C 0 단일 좌초 된 DNA의 초기 농도 곳 DNA의 reassociation는 C 0 X t 곡선을 따라, 그리고 t는 reannealing 시간입니다. 10-4와 동일한 C 0t 값과 함께 DNA의 분수 - 10-1 또는 100-102는 각각 높은과 적당히 반복 간주됩니다. 다른 C 0t의 DNA 분수에 대한 reassociation의 시간은 일을 사용하여 계산 될 수있다전자 공식 t = C 0t X × 4.98 / C 0, 어디에서 t - 부화 시간, C 0t X - C 0t 분율 (C0t 1 = 1, C 0t 2 = 2 등) 및 C 0 - μg / μl 15 초기 DNA 농도 (표 1). reassociation 후, 단일 좌초 된 DNA는 S1 nuclease를 사용하여 소화하고 있습니다. 우리는까지 모든 C 0t의 DNA 분수를 사용하여 선호하는 C 0 함께 t 3 대신 일반적으로 사용되는 C 0t 한 DNA 분획. 이 C 0t의 분수는 적당한 반복적 인 DNA 시퀀스의 일부를 포함하고 함께 보통 애의 게놈 DNA의 원래 금액의 35-50%을 나타냅니다. aegypti. 표시 DNA 프로브와 라벨이없는 C 0t의 DNA 분획 사이의 올바른 비율은 각 특정 BAC 클론의 반복적 인 DNA 구성 요소에 따라 달라집니다. 평균적으로, 우리는 C 0t에 1시 20분 프로브를 사용하여허용 신호 / 물고기 결과의 배경 비율을 얻기위한 DNA 분수 비율. 슬라이드의 실제 하이브리드 전에 30 분을위한 관 C 0t의 DNA 분수 DNA 프로브의 Prehybridization 또한 배경을 감소하는 데 도움이됩니다. 라벨, 하이브리드 자체,이 프로토콜에 세탁기는 표준 조건 12를 사용하여 수행됩니다.
애의 mitotic 염색체에 두 개의 다른 표시 BAC 클론의 DNA 프로브의 생선 결과입니다. aegypti 및 CX. quinquefasciatus는 각각,도 4A 및 B에 표시됩니다. BAC 클론의 DNA 프로브는 염색체의 단일 위치에 강력한 신호를 생산하고 있습니다. 그림 1에 표시된 염색체는 유유-1 요오드화물과 counterstained 있습니다. 이 염료는 애에서 제일 구간 패턴을 생산하고 있습니다. aegypti의 염색체 9. 또한, 이러한 DAPI 또는 propidium의 요오드화물과 같은 다른 형광 염료는, 일에 활용 될 수있다전자 염색체 counterstaining. 슬라이드 억제 photobleaching를 들어, 골드 antifade 설치 매체를 연장 사용합니다. 이 시약은 좋은 신호 보존 능력을 갖추고 있으며 또한 쉽게 여러 hybridizations에 대해 동일한 슬라이드를 사용하는 데 필요한 경우 1X PBS로 린스하여 슬라이드에서 제거 할 수 있습니다.
물고기 프로토콜의 간단한 버전은 아노펠레스의 mitotic 염색체에 IGS rDNA 프로브의 하이브리드 위해 설계되었습니다. 아노펠레스의 Ribosomal 유전자는 성 염색체 16에있는 유전자의 다형성 클러스터로 표시됩니다. 이 프로토콜의 DNA 프로브는 휘황 표시 Cy3 또는 Cy5 dNTPs를 추가하여 표준 PCR 반응을 사용하여 라벨이 지정됩니다. euchromatin 반복적 인 DNA의 불특정 하이브리드을 차단하는 것은 필요하지 않기 때문에, C 0t의 DNA 분수를 사용하여 관련된 모든 단계가 생략되어 있습니다. 대신, 염색체 준비가에 IGS rDNA 프로브의 하이브리드을 방지하기위한 RNase와 pretreated 아르nucleolus. 염색체와 DNA 프로브은 5 분 동안 75 ° C에서 하이브리드 시스템의 프로브와 함께 슬라이드를 가열함으로써 동시에 변성되어 있습니다. 이 프로토콜의 하이브리드 및 세척도 물고기 12 표준 조건을 사용하여 수행됩니다. 물고기의 결과는 그림 4C에 증명의 두 X 염색체 사이의 IGS rDNA의 하이브리드의 다형성은 명확 볼 수 있습니다.
C 0t 3| DNA 농도 μg / μl | 시간을 Reannealing, 분 | |
| C 0t 2 | 0.1 | 100 |
| 0.3 | 33 | |
| 0.5 | 20 | |
| 0.7 | 14 | |
| 0.9 | 11 | |
| 1 | 10 | |
| 0.1 | 150 | |
| 0.3 | 50 | |
| 0.5 | 30 | |
| 0.7 | 21 | |
| 0.9 | 17 | |
| 1 | 15 |
표 1. DNA 농도 및 C 0 T2 및 C 0 T3 분수의 준비를위한 reannealing 시간.
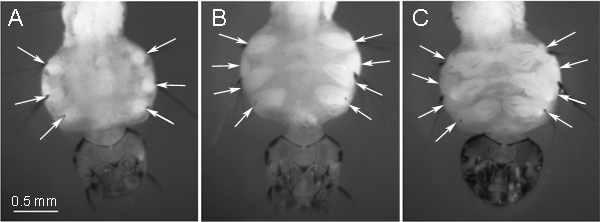
그림 1 4 회 instar의 유충의 ID 개발의 단계 : A) 초기 '둥근 모양'단계, B) 중간 "타원형 모양"단계 - 염색체 준비에 대한 최적, C) 늦은 단계 - 염색체 준비에 대한 부적절한. . ID의 위치는 애벌레의 가슴의 복부 측면에 화살표로 표시됩니다.
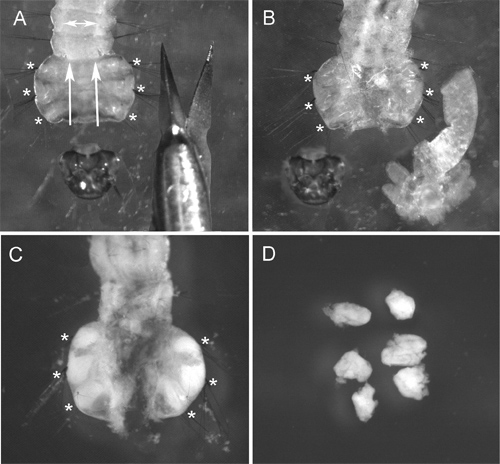
그림 2 ID의 해부의 단계 : A) 참수 유충 (상처의 방향이 화살표로 표시됩니다) hypotonic 고용 화 열처리에 따라 해부 내장 (ID가 팽창하고 거의 눈에 보이지 않는가)와 B) 애벌레, C) 유충 Carnoy의 솔루션 신청 후 (. ID는) 흰색과 명확하게 표시 될, D) Carnoy의 솔루션 ID를 해부 했어요. 유충의 ID의 순위는 별표로 표시됩니다.
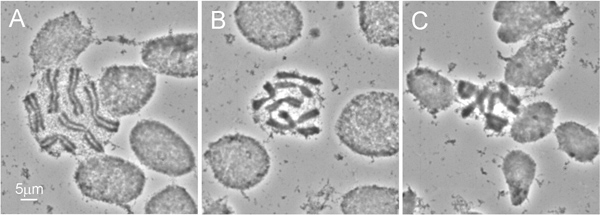
그림 3 염색체 확산의 다른 특성 : A) 완벽한 염색체 확산 - 세포의 둥근 모양은 hypotonic 솔루션 ID의 충분한 치료를 보여, B) 완벽하게 hypotonic 치료 - 염색체는 약간 undersquashed이다; C) 가난한 염색체 확산. - 결과부족 hypotonic 치료는 세포의 타원형 모양으로 표시됩니다.
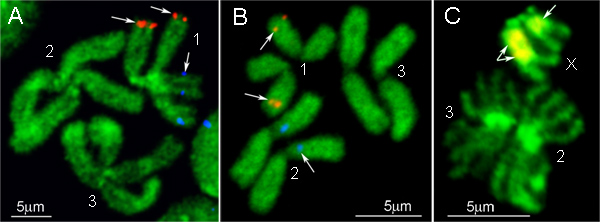
그림 4. BAC 클론 (A, B)와 애의 염색체에 IGS rDNA (C)와 물고기의 예. aegypti (A), CX. quinquefasciatus (B), 그리고. gambiae (C). 1, 2, 3 - 염색체의 숫자, X -에 여성 성 염색체. gambiae.
토론
모기 mitotic 염색체에 원위치 하이브리드 화에 Nonfluorescent는 A. 쿠마르와 K. 라이 17에 의해 1990 년에 처음으로 공연되었다. 이 연구에서, 18S와 28S ribosomal DNA의 유전자는 플라스미드 하나에 함께 복제 된 모기 중 20 종의 염색체에 배치되었습니다. DNA 프로브는 radioactively 표시 및 뇌 신경절에서 염색체에 hybridized되었다. 세 모기 장군 중에서 물고기 기술은 애의 세포 라인에서 mitotic 염색체을 위해 개발되었습니다. aegypti 10,18,19하며 라이브 모기에서 mitotic 염색체에서 수행 된 적이있다. 최근, 우리는 4 일 instar의 애벌레 9의 ID에서 높은 품질의 염색체 준비를 얻기위한 간단하고 강력한 기술을 개발했다. 이 방법은 염색체의 높은 번호가 하나의 슬라이드에서 얻을 수 할 수 있으며, 보편적 모기의 모든 종에 사용될 수 있습니다. 만 애벌레의, pupal 또는 성인되지 역을 사용하는 필요성모기 릿지는 슬라이드 준비에 아마 방법의 한계입니다. 표준 생선 방법 12 Aedes, Culex, 그리고 아노펠레스의 mitotic 염색체에 대한 프로브로 게놈 BAC 클론과 IGS rDNA를 사용에 최적화되었습니다.
이러한 특정 응용 프로그램뿐만 아니라, 여기에 설명 된 생선 프로토콜은 다른 목적을 위해 사용할 수 있습니다. 불특정 하이브리드을 차단을위한 C 0t의 DNA 분수를 활용하여 고급 생선 프로토콜은 또한 BAC 클론 또는 아노펠레스의 heterochromatic 지역의 다른 큰 DNA 조각의 하이브리드에 적용 할 수 있습니다. Heterochromatic 지역은 transposable 요소와 다른 반복과 풍부한,이 지역에서 프로브는 일반적으로 염색체 3 강력한 배경을 생산하고 있습니다. 라벨이없는 C 0t의 DNA 분수를 사용하면 염색체에 프로브의 불특정 하이브리드을 줄이기 위해 도움이 될 것입니다. 물고기 PR의 간단한 버전otocol 어떤 rDNA 또는 모기와 다른 곤충의 mitotic 염색체에 반복적 인 DNA 프로브에 사용할 수 있습니다. 또한, 그것은 또한 아노펠레스 또는 Drosophila 등의 euchromatic 지역에서 낮은 반복적 인 DNA 콘텐츠가 포함 된 종의 BAC 클론의 DNA의 하이브리드에 적용 할 수 있습니다. 여기에 제안 된 프로토콜은 모기에 대한 높은 완성 된 염색체 기반의 게놈 어셈블리를 얻을하는 데 도움이되며, 널리 곤충의 다른 그룹에서 다양한 cytogenetic 응용 프로그램에 사용할 수 있습니다.
공개
관심 없음 충돌이 선언 없습니다.
감사의 말
우리는 아노펠레스의 염색체 준비와 물고기의 도움을 세르게이 Demin와 타티아나 Karamysheva 감사드립니다. 우리는 또한 텍스트를 편집 우리에게 Aedes과 Culex 게놈 DNA BAC 클론과 멜리사 웨이드를 제공하기 위해 데이비드 Severson 감사드립니다. 1R21 AI88035-01 마리아 V. Sharakhova와 1R21 AI094289-01에 이고르 V. Sharakhov :이 작품은 국립 보건원에서 두 보조금에 의해 지원되었다.
자료
| Name | Company | Catalog Number | Comments |
| 시약 / 장비의 이름 | 회사 | 카탈로그 번호 | 코멘트 |
| MZ6 Leica의 stereomicroscope | Leica | VA-OM-E194-354 | 다른 stereomicroscope를 사용할 수 있습니다 |
| 올림푸스 CX41 위상 현미경 | 하늘 | CX41 | 다른 위상 현미경을 사용할 수 있습니다 |
| 올림푸스 BX61 형광 현미경 | 하늘 | BX61 | 다른 형광 현미경을 사용 할 수 있습니다 |
| ThermoBrite 슬라이드 변성 / 하이브리드 시스템 | 애보트 분자 | 30-144110 | 가열 블록 및 습윤 챔버 역할을 |
| 해부 바늘 | 좋은 ScienceTools | 10130-10 | |
| 니들 홀더 | 정밀 과학 도구 | 26018-17 | |
| 해부 가위 | 정밀 과학 도구 | 15000-03 | |
| 75x25 더블 불투명 마이크로 슬라이드 | 코닝 | 2949-75x25 | |
| 22x22 mm 현미경 coverslips | 피셔 과학 | 12-544-10 | |
| Parafilm | 피셔 과학 | 13-374-10 | |
| 고무 시멘트 | 피셔 과학 | 50-949-105 | |
| 아세트산 | 피셔 과학 | A491-212 | |
| 알코올 200 증명 | Decon 연구소 | 2701 | |
| 프로피온산 | 시그마 Aldrich | 402907 | |
| 염산 | 피셔 과학 | A144-500 | |
| 나트륨 시트르산 이수화 | 피셔 과학 | S279-500 | |
| 나트륨 아세테이트 trihydrate | 피셔 과학 | BP334-500 | |
| 염화칼륨 | 피셔 과학 | BP366-500 | |
| EDTA (에틸렌 다이아 민 테트라 초산) | 피셔 과학 | S311-500 | |
| 트리스베이스 | 피셔 과학 | BP152-1 | |
| 10X PBS | Invitrogen | P5493 | |
| 10% NBF (중성는 포르말린 버퍼) | 시그마 - 알드리치 | HT501128 | |
| 99 % 포름 아미드 | 피셔 Scientific | BP227500 | |
| Dextran 황산 나트륨 소금 | 시그마 - 알드리치 | D8906 | |
| 20x SSC 버퍼 | Invitrogen | AM9765 | |
| 1 ㎜ 유유-1 요오드화물 (509분의 491) 솔루션 | Invitrogen | Y3601 | |
| 골드 시약을 연장 Antifade | Invitrogen | P36930 | |
| dATP, dCTP, dGTP, dTTP | Fermentas | R0141, R0151, R0161, R0171 | |
| Cy3-dUTP, Cy5-dUTP | GE 헬스 케어 | PA53022, PA55022 | |
| BSA | 시그마 - 알드리치 | A3294 | |
| DNA의 중합 효소 I | Fermentas | EP0041 | |
| DNase I | Fermentas | EN0521 | |
| S1 Nuclease | Fermentas | EN0321 | |
| DNA 형성 촉매의 DNA 중합 효소의 | Invitrogen | 18038-042 | |
| RNase | 시그마 - 알드리치 | 9001-99-4 | |
| 펩신 | USB | 9001-75-6 | |
| 연어 정자 DNA | 시그마 - 알드리치 | D7656 | |
| Nonidet-P40 (NP40) | 미국 생물 | NC9375914 | |
| Qiagen의 혈액과 세포 배양 Maxikit | Qiagen | 13,362 | |
| Qiagen 대형 건립 키트 | Qiagen | 12,462 |
참고문헌
- Sharakhov, I. V., Sharakhova, M. V., Verrity, J. F., Abbington, L. E. . Chromosome Mapping Research Developments. , (2008).
- Holt, R. A., et al. The genome sequence of the malaria mosquito Anopheles gambiae. Science. 298, 129-149 (2002).
- Sharakhova, M. V., et al. Update of the Anopheles gambiae PEST genome assembly. Genome biology. 8, R5 (2007).
- Campos, J., Andrade, C. F., Recco-Pimentel, S. M. A technique for preparing polytene chromosomes from Aedes aegypti (Diptera, Culicinae). Memorias do Instituto Oswaldo Cruz. 98, 387-390 (2003).
- Campos, J., Andrade, C. F., Recco-Pimentel, S. M. Malpighian tubule polytene chromosomes of Culex quinquefasciatus (Diptera, Culicinae). Memorias do Instituto Oswaldo Cruz. 98, 383-386 (2003).
- McAbee, R. D., Christiansen, J. A., Cornel, A. J. A detailed larval salivary gland polytene chromosome photomap for Culex quinquefasciatus (Diptera: Culicidae) from Johannesburg, South Africa. J. Med. Entomol. 44, 229-237 (2007).
- Nene, V., et al. Genome sequence of Aedes aegypti, a major arbovirus vector. Science. 316, 1718-1723 (2007).
- Arensburger, P., et al. Sequencing of Culex quinquefasciatus establishes a platform for mosquito comparative genomics. Science. 330, 86-88 (2010).
- Sharakhova, M. V., et al. Imaginal discs--a new source of chromosomes for genome mapping of the yellow fever mosquito Aedes aegypti. PLoS neglected tropical diseases. 5, e1335 (2011).
- Brown, S. E., et al. Toward a physical map of Aedes aegypti. Insect Mol. Biol. 4, 161-167 (1995).
- Steiniger, G. E., Mukherjee, A. B. Insect chromosome banding: technique for G- and Q-banding pattern in the mosquito Aedes albopictus. Can. J. Genet. Cytol. 17, 241-244 (1975).
- Garimberti, E., Tosi, S., Bridger, J. M., Volpi, E. V. . Fluorescence in situ hybridization (FISH). , (2010).
- Scott, J. A., Brogdon, W. G., Collins, F. H. Identification of single specimens of the Anopheles gambiae complex by the polymerase chain reaction. The American journal of tropical medicine and hygiene. 49, 520-529 (1993).
- Trifonov, V. A., Vorobyeva, N. N., Rens, W., Leiehr, T. . Fluorescence in situ hybridization (FISH). , (2009).
- Collins, F. H., et al. A ribosomal RNA gene probe differentiates member species of the Anopheles gambiae complex. The American journal of tropical medicine and hygiene. 37, 37-41 (1987).
- Kumar, A., Rai, K. S. Chromosomal localization and copy number of 18S+28S ribosomal RNA genes in evolutionary diverse mosquitoes (Diptera, Culicidae). Hereditas. 113, 277-289 (1990).
- Brown, S. E., Knudson, D. L. FISH landmarks for Aedes aegypti chromosomes. Insect Mol. Biol. 6, 197-202 (1997).
- Brown, S. E., Severson, D. W., Smith, L. A., Knudson, D. L. Integration of the Aedes aegypti mosquito genetic linkage and physical maps. Genetics. 157, 1299-1305 (2001).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유