Method Article
蛍光灯
要約
3蚊属のうち、すなわちハマダラカ、ヤブカと、イエカ物理的なゲノムマッピング技術が確立されたのみハマダラカ、そのメンバーは、読み取り可能な多糸染色体を持っています。の世代のためにヤブカとイエカしかし、細胞遺伝学的マッピングがあるため多糸染色体の質が悪く依然として厳しい。ここでは、高品質の有糸分裂染色体の準備と蚊のすべての3つの属のために最適化されたFISHプロトコルを取得するための汎用プロトコルを提示します。
要約
in situハイブリダイゼーション(FISH) 蛍光 inは、日常的にDNAとRNAプローブの染色体上の位置を決定するために、多くの研究室で使用されるテクニックです。この方法の一つの重要なアプリケーションは、様々な生物のゲノムアセンブリを改善するのに有用で高品質な物理マップの開発である。多糸と糸分裂染色体の自然なバンドパターンは、ゲノムsupercontigsの正確な順序およびオリエンテーションのためのガイダンスを提供します。すなわち3蚊属、 ハマダラカ 、 ヒトスジシマカ、アカイエカとの間で、十分に確立された染色体ベースのマッピング技術は、そのメンバーだけが読める多糸染色体1を所有ハマダラカ 、ために開発されました。ゲノムマッピングの努力の結果として、全体の88%。ハマダラカのゲノムは、正確な染色体位置2,3に配置されています。他の二つの蚊属、 ヤブカとアカイエカは、不十分な染色体bをpolytenizedたそれらのゲノム4、5、6の転移因子の重要overrepresentationのecause。ゲノムsupercontingsは31、9% は 、AEの染色体への順序や向きをせずに割り当てられています。ネッタイシマカ7とそれぞれネッタイイエカ 8。これら2種の有糸分裂染色体の準備は以前に脳基底核および細胞株に限られていた。しかし、蚊の脳の神経節から調製した染色体のスライドは通常中期プレート9の低い数字が含まれています。また、FISH法は、AEの細胞株から分裂期染色体のために開発されてきたが。ネッタイシマカ 10は 、細胞株の染色体11内に複数の染色体再配列の蓄積は、ゲノムマッピングのためにそれらは無用になります。ここでは、C 4 番目の幼虫の成虫から高品質の分裂期染色体標本を得るためのシンプルで堅牢な技術(ID)を記述する蚊のすべての3つの属にも使用できます。標準FISHプロトコル12は AEの有糸分裂染色体上のプローブとしてゲノムDNAのBACクローンを使用するために最適化されています。ネッタイシマカとネッタイイエカ、オンプローブとしてリボソームDNAの遺伝子間スペーサー(IGS)の領域(遺伝子組換え)を利用する。ハマダラカの染色体は、物理マッピングに加えて、開発された技術は、人口細胞遺伝学、染色体分類/蚊や他の昆虫群の系統に適用することができます。
プロトコル
1。染色体調製
蚊の幼虫は、マラリア研究とレファレンス試薬リソースセンターのWebサイトから入手できハマダラカ研究の方法に記載の標準プロトコルを使用して飼育した (MR4)13。蚊飼育の温度は成虫と幼虫の死亡率が最も低いの染色体の数が最も多くを提供するように変更されました。蚊の幼虫の発達の段階は自分の頭のカプセル13の大きさに基づいて決定した。
- 28でハッチ蚊の卵℃、2〜3日後に、16〜移転第2または第3齢幼虫° ヨンエのC。ネッタイシマカとCx。用ネッタイイエカと22℃〜。ハマダラカ。
- 固定化のための数分間、氷上で4 番目の幼虫を置きます。
- 冷低張液(0.5%程度の低下とスライドに幼虫を移すdiumクエン酸または0.075 Mの塩化カリウム)と、実体顕微鏡下に置きます。
- さらに解剖のために楕円形のIDを持つ幼虫( 図1B)を選択します 。
- 幼虫の首を、そして解剖ハサミ( 図2A)を使って幼虫の胸部の腹側からキューティクルをカット。幼虫から腸を解剖する第二または第三腹節で追加カットを行う。カットの方向が矢印で示されている。
- キューティクルを開き、幼虫から腸や体脂肪を取り除く。ろ紙を用いてスライドから低張液を取り出して、直接IDに低張液( 図2B)の新鮮なドロップを追加。室温で10分間低張液中で幼虫を保管してください。
- ろ紙を用いて低張液を取り出して、カルノア液(3:1の割合でエタノール/酢酸)を適用します。固定液を添加した後、直ちに( 図2C白くおよび顕微鏡下で容易に目に見えるようになるIDが)。
- 解剖針を使用して、幼虫( 図2D)からIDを削除し、50%プロピオン酸の滴に転送します。スライドから、そのような腸や体脂肪などの他の組織を削除します。 unsiliconized 22x22のカバースリップでIDを覆い、室温で10分間保つ。
- カバースリップの周囲に鉛筆の消しゴムをタップすることで、ろ紙、スカッシュ組織スライドをカバーしています。
- 簡単に言うと100倍以上200倍( 図3)位相差顕微鏡を使用して、スライドの品質を分析する。 > 50染色体スプレッドを有する製剤をFISHに適して考えることができます。
- それはバブリングを停止するまで液体窒素でスライドを浸したままにします。カミソリの刃を用いてスライドからカバースリップを外し、-20℃に冷やして70%エタノールを容器にすぐにスライドを転送4℃で保存、必要に応じて最善の結果脱水のために少なくとも1時間、Cは(、スライドトンで保存できます。数日〜数分から彼のステップ)。
- それぞれ5分間、そして室温で乾燥した空気のために4℃のエタノールシリーズ(70%、80%、100%)℃でスライドを脱水する。
- 魚のためにこれを利用する前に-20℃で乾燥したスライドを保管してください。
2。反復DNA画分の抽出
AE からの染色体上のBACクローンDNAプローブのFISHを行う 。ネッタイシマカとCx。ネッタイイエカは、 染色体にDNA反復の非特異的ハイブリダイゼーションをブロックするためにラベルのない反復DNA画分を使用する必要があります。数百bpの断片に断片化一本鎖DNAの再アソシエーションは、C 0は 、一本鎖DNAの初期濃度であり、tはアニーリング時間はC 0トンの曲線に従います。 10 -4〜10 -1 10以上C0t値-10℃2とのDNA画分をそれぞれ、高度かつ適度に繰り返しとみなされます。
- エキス400から500キアゲン血液および細胞培養Maxikitを使用して全体の大人の蚊からのゲノムDNAのμg、および1.2×SSC中で100〜1,000 ng /μlにDNA溶液を準備します。
- 加熱ブロックにゲノムDNAを使って安全なロックチューブを配置することによって、変性DNAは2分間120℃に温めた。高温は200から500塩基対の断片にDNAを範囲するのに役立ちます。
- DNA濃度に応じて、C 0と T3(表1)、C 0トンのDNA画分を得るために15から150分間、60℃で真空管を配置することによって、DNAを再度関連付けます。
- 2分間氷上にDNAとチューブを置きます。
- 42にDNAを導入℃、DNAの1 mg当たり100 Uの最終濃度になるように予熱した10倍S1ヌクレアーゼ緩衝液およびS1ヌクレアーゼを追加し、1時間インキュベートします。
- 3 M酢酸ナトリウムとRTでイソプロパノール1体積の0.1倍量を添加することによりDNAを沈殿させる。
- 4℃で20分間、14,000 rpmで遠心
- 70%エタノールでDNAを洗浄し、10分間、14,000 rpmで遠心操作4℃
- 空気乾燥し、TE緩衝液にDNAペレットを溶解する。
- DNA濃度を測定し、ゲル電気泳動によって可視化する。通常反復DNA画分の最終量は、元のDNA量の35から50パーセントを表します。
3。 DNAプローブのラベリング
二つの異なるプロトコルを標識BACクローンDNAプローブとIGSのrDNAプローブのために使用された。
ニックトランスレーションを使用して3.1 BACクローンのラベリング
- キアゲン大コンストラクトキットを使用してBACライブラリーからBACクローンDNAを抽出します。
- ;にCy3-dUTPで(または別の1μlのBACクローンDNAを、0.05mMの未標識のdATP、dCTP、およびdGTPおよびdTTPの0.015mmのそれぞれを単離した1μgの:50μlの最終体積が氷上でニックトランスレーション標識のために反応液を調製蛍光色素)、BSAの0.05 mg / mlで、10倍のニックトランスレーション緩衝液5μl、DNAポリメラーゼ20UのIとDNaseの0.0012 U。
- 2のために15℃でインキュベートする0.5時間。
- 0.5M EDTAを1μlを加え、反応を停止させます。
- 暗い場所では-20℃で保存するプローブ。
PCRを用いて3.2のrDNA IGSのラベリング
- ; 0.05mMのラベルが付いていないのdATP、dCTP、およびdGTPの各; dTTPの0.015ミリメートル;にCy3-dUTPで(または別の蛍光色素)を1μl、5μlのゲノムDNAを200ngの:50μlの最終体積氷上で反応液を調製する10×PCR緩衝液、前方50pmolの、国連(GTGTGCCCCTTCCTCGATGT)とリバース、IGSの増幅のためのGA(CTGGTTTGGTCGGCACGTTT)プライマー;およびTaq DNAポリメラーゼ14 10Uの。
- (95℃/ 30秒、50℃/ 30秒、72℃/ 30秒)×30サイクル;、72℃〜95℃/ 5分×1サイクル:IGSの増幅のための標準的なPCRパラメータを用いてPCR反応を行う/ 5分×1サイクル;及び4℃で14を開催しています 。
- 暗い場所では-20℃で保存するプローブ。
4。 蛍光 in situハイブリダイゼーション
この魚のプロトコルは、2つのバリエーションが含まれます。ネッタイシマカとCx。ネッタイイエカとの分裂期染色体上のrDNA IGSを使用するための第二。ハマダラカ。 BACクローンDNAプローブを使用している場合は、スキップRNase処理は、4.3、4.4を段階的に説明し、同時にスライド/プローブの変性ステップ4.19。 IGSのrDNAのプローブを使用する場合は、C 0トンのDNA分画せずにハイブリダイゼーション混合物を準備し、手順4.10、4.11、4.16、および4.17を変性別のスライド/プローブをスキップします。
- 37℃で30分のために2×SSC中でスライドをインキュベート
- RTで5分間ずつ、空気乾燥し、70%のシリーズ、80%、100%エタノールでスライドを脱水する。BACクローンDNAを用いてFISHを行っている場合は、4.5の手順に進んでください。
- 37℃で30分間パラフィルムの下に0.1 mg / mlのRNaseの溶液中で染色体の準備をインキュベートする。
- 37℃で5分間ずつ2×SSCで二回洗っ℃、
- 私はスライドを入れてNaの0.01%ペプシンと0.037パーセントHCl溶液で瓶、そして37℃で5分間インキュベート℃、
- 室温で5分間1X PBSでスライドを洗浄します。
- 室温で10分間ホルマリン中性緩衝10パーセントから調製1X PBS中1%ホルマリンと瓶の染色体の準備を修正しました。
- 室温で5分間1X PBSでスライドを洗浄します。
- 37℃で5分間室温でそれぞれ、空気乾燥製剤の場合は70%、80%、および100%エタノールの一連のスライドを脱水℃、IGSで魚を行っている場合は、4.12をステップに直接進み
- 72℃で2分間温めておいた70%ホルムアミドと瓶の変性スライド
- 風邪の一連のスライドを脱水(-20℃)37℃で5分間ずつ、空気乾燥で70%、80%、100%エタノール℃の
- ハイブリダイゼーション混合物の準備をします。ステップ3から標識プローブDNA 5μlの、0.5 ng /μlに、1μg/μLの超音波処理サケ精子DNA5μlの最終濃度と、ステップ2からC 0トンのDNA10μlをIGSと魚のためのrDNAの、準備C 0トンのDNA分画せずにハイブリダイゼーション混合物。
- 3 M酢酸ナトリウムとエタノールの2容積の0.1倍量を添加することによりDNAを沈殿させる。 1から3時間-20℃で保管してください。
- 4℃、14,000 rpmで遠心し、20分間℃、エタノールを除去し、空気乾燥した室温でペレット。
- 徹底的にハイブリダイゼーション緩衝液10μlにペレットを溶解:50%ホルムアミド、20%デキストラン硫酸、2×SSC、IGSで魚を行っている場合は、4.18のステップに直接進む
- 変性、ハイブリダイゼーション97で7分間混合℃、直ちに1分間氷上に置く。
- 染色体への反復DNAの非特異的なハイブリダイゼーションを防止するために30分間37℃で混合物をプレハイブリダイゼーション。
- スライド上にハイブリダイゼーション混合物10μlを置き、22x22のカバースリップで覆う。バブルの形成を防ぐ- 。気泡がカバースリップに穏やかな圧力を用いて除去されるべきであるBACクローンDNAとFISHを行っている場合は、4.20のステップに直接進み</ em>の
- 同時に75℃の加熱ブロックを用いたプローブおよび染色体DNAを変性℃で5分間。
- ゴム糊を使用して周囲に接着剤カバースリップ。
- 37℃湿潤チャンバー℃で一晩ハイブリダイゼーションを行っ
- スライドからゴム糊とカバーガラスを外します。
- 73℃に温めておいた溶液1(0.4x SSC、0.3%ノニデット-P40)にスライド2分間洗浄
- 室温で5分間溶液2(2×SSC、0.1%ノニデット-P40)でスライドを洗浄します。
- RTで湿潤チャンバー中で10分間1X PBSで0.001 mMのYOYO-1を使用したスライドを対比染色する。
- カバースリップとGold退色防止試薬を延長少量のマウントします。
- 1,000×倍率( 図4)で、適切なフィルターセットを用いて蛍光顕微鏡下で準備を分析します。
5。代表的な結果
昆虫IDは幼虫の各セグメントに配置されています。位置に応じて、目EYは、昆虫の成虫の段階で異なる組織に変身。このプロトコルにおける染色体の調製のために使用されるIDは、蚊の成虫期における脚へと発展する。これらのIDは、幼虫の胸部の腹側に位置しており、顕微鏡下でキューティクル( 図1)を介して明確に表示されています。初期の4 番目の齢幼虫の段階では、IDが丸い形状( 図1A)を持っています。有糸分裂の最大の番号は、1〜175でIDを図9に示すように 、スライドの準備のための最適な段階を考慮しなければならない後の"楕円形"の段階( 図1B)に蓄積される。この時、中間のIDが2つに分かれ:片足に変身し、もう一つは翼に変身します。我々は染色体スライドの準備のための"楕円形"の段階で大型レッグIDを使用して好む。 図1Cは、 第 4齢幼虫開発の最新の段階でIDを表します。この段階では、IDはすでに脚と翼に開発され、分化した組織のかなりの量や有糸分裂の低い数字を含んでいます。 IDの開発のこの段階は、染色体スライドの準備のために避けるべきである。 16°ヒトスジシマカとアカイエカのためのCと22° ハマダラカのCこれは、IDが9で有糸分裂の量を増やすのに役立ちます:我々はまた、低温での蚊の幼虫の飼育をお勧めします。
図2は、 第 4齢幼虫の胸部からID解剖を示しています。ライブ昆虫のクチクラが解剖するのは難しいですので、解剖ハサミの代わりに、一般的に幼虫の調製のために使用される針を使用することをお勧めします。質の高い染色体の準備を取得するための最も重要な手順は、低張液処理である。最良の結果を得るために、我々は、この治療の前に幼虫の胸部から腸や体脂肪を取り除く。この手順の中にID細胞の腫脹と、染色体を広めるのに役立ちますスライド上の( 図3A)。低張液処理の適切な品質を簡単に準備中の細胞の円形( 図3A、B)によって認識することができます。楕円形の形状を持つ細胞が不十分な低張液処理( 図3C)を示す。魚のために選択されるためには、染色体の準備は、少なくとも50の高品質染色体スプレッドを含める必要があります。通常、このプロトコルを使用して調製したスライドの約90%は魚の9のために十分な品質を持っています。
ヒトスジシマカとアカイエカの分裂期染色体上のゲノムBACクローンDNAプローブを用いたFISHおよびハマダラカの分裂期染色体上のrDNA IGSのプローブのためのシンプルなFISHプロトコルのための高度なプロトコル:我々は2つのわずかに異なる魚のプロトコルを提示します。 ヒトスジシマカとアカイエカのゲノムが原因転移因子7,8のoverrepresentationの高度に反復されています。したがって、実行FISH、UTプローブとしてゲノムBACクローンDNAをilizes染色体にDNA反復の非特異的ハイブリダイゼーションをブロックするプローブに標識されていない反復DNA画分を追加する必要があります。反復DNA画分の抽出のために、ゲノムDNAは、2分間120℃で変性されています。高い温度で沸騰するDNAは、200から500 bpの断片のDNAを得るのに役立ちます。 DNAは、この治療後に再アソシエートを許可されている。反復性の高いDNA断片は速くユニークな配列を持つDNAの場合よりも、再アソシエーションのために彼らの仲間を見つける傾向がある。その結果、DNAの再アソシエーションは、C 0は 、一本鎖DNAの初期濃度であり、tはアニーリング時間はC 0×tの曲線に従います。 10月4日に等しく、C 0、tの値が DNA画分- 10-1または100から102は、それぞれ高度にそして適度に繰り返しとみなされます。異なるC 0トンの DNA画分の再アソシエーションの時間が目を使って計算することができ電子式T = C 0トン X×4.98 /℃0、ここでt -インキュベーション時間は、C 0トン X - C 0トン分画(C0t 1 = 1、C 0トン = 2など)、C 0 - μg/μLの15の初期DNA濃度(表1)。再関連付けの後は、一本鎖DNAはS1ヌクレアーゼを用いて消化される。我々は、最大一緒にC 0トン 3の代わりに、一般的に使用されるC 0 t 1のDNA画分にすべてのC 0トンの DNA画分を使用して好む。これらのC 0トン画分は適度に反復DNA配列の一部を含むと一緒に通常AEのゲノムDNAの元の量の百分の35から50を表す。ネッタイシマカ 。標識したDNAプローブの間の正しい割合および非標識C 0トンの DNA画分は、それぞれの特定のBACクローンで反復DNA成分に依存する。平均して、我々は、C 0トン 〜1:20のプローブを使用許容信号/ FISHの結果のバックグラウンド比を得るためのDNA画分の割合。スライド上の実際のハイブリダイゼーションの前に30分間、チューブ内のC 0トンの DNA画分を用いたDNAプローブのハイブリダイゼーションは、バックグラウンドを低減するのに役立ちます。ラベリング、ハイブリダイゼーション自体、およびこのプロトコルでは、標準的な洗浄条件12を使用して実行されます。
AEの有糸分裂染色体上の2つの異なる標識BACクローンDNAプローブのFISH結果。ネッタイシマカとネッタイイエカ、それぞれ図4AおよびBに示されている。 BACクローンのDNAプローブは、染色体上の単一の位置に強力な信号を生成します。 図1に示されている染色体はYOYO-1ヨウで対比されています。この色素は 、AE上で最高のバンドパターンを生成します。ネッタイシマカ染色体9。あるいは、このようなDAPIまたはプロピジウムヨージドなどの他の蛍光色素は、目のために利用することができる電子染色体の対比。スライドの抑制退色のために、私たちはゴールド退色マウンティングメディウムを延長使用しています。この試薬は、良好な信号保全能力を持っており、また簡単にいくつかのハイブリダイゼーションのための同じスライドを使用する必要がある場合1X PBSでリンスすることによりスライドから削除することができます。
FISHプロトコルのシンプルバージョンはハマダラカの分裂期染色体上のrDNA IGSのプローブのハイブリダイゼーションのために設計されています。 ハマダラカにおけるリボソーム遺伝子は性染色体16上に位置する遺伝子の多型クラスタとして表されます。このプロトコルにおけるDNAプローブは蛍光標識Cy3またはCy5 dNTPを加えることにより、標準的なPCR反応を用いて標識されています。ユークロマチンにおける反復DNAの非特異的なハイブリダイゼーションをブロッキングが必要とされていないので、C 0トンの DNA画分の使用に関連するすべてのステップが省略されている。その代わりに、染色体の準備はしてIGSのrDNAのプローブのハイブリダイゼーションを防止するためにRNase Aで前処理し核小体。染色体やDNAプローブを5分間75℃でハイブリダイゼーションシステム内のプローブと一緒にスライドを加熱することにより同時に変性されています。このプロトコルのハイブリダイゼーションおよび洗浄はまた、FISH 12のための標準的な条件を使用して実行されます。 FISHの結果は、図4Cに示されている:2つのX染色体との間のrDNA IGSのハイブリダイゼーションの多型がはっきりと見える。
Cは0トン3| DNA濃度μg/μLの | アニーリング時間、分 | |
| C 0トン2 | 0.1 | 100 |
| 0.3 | 33 | |
| 0.5 | 20 | |
| 0.7 | 14 | |
| 0.9 | 11 | |
| 1 | 10 | |
| 0.1 | 150 | |
| 0.3 | 50 | |
| 0.5 | 30 | |
| 0.7 | 21 | |
| 0.9 | 17 | |
| 1 | 15 |
表1 DNA濃度およびC 0、C 0、t2とt3の画分の調製のための再アニーリング回。
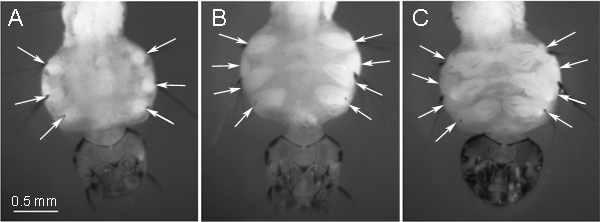
図1 第 4齢幼虫におけるIDの発展段階:)初期の"丸形"の段階、B)中間の"楕円形"ステージ-染色体の準備のための最適な、C)後期-染色体の準備には不適切。 。 IDの位置は幼虫の胸部の腹側の矢印で示されている。
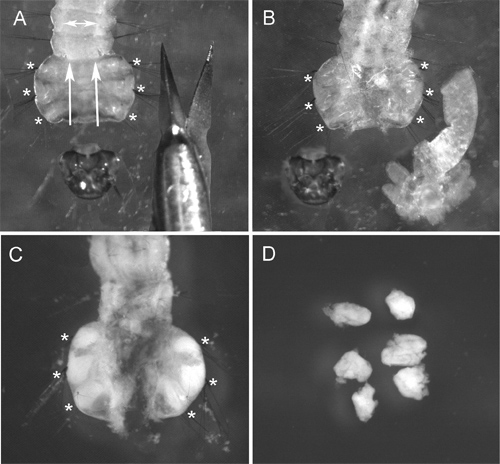
図2の ID清のステップ:)断頭幼虫(カットの方向が矢印で示されている)、低張液処理下で解剖し、腸(IDが膨潤し、ほとんど見えなくなる)とB)の幼虫、C)幼虫カルノア溶液塗布後(。 ID)は白とはっきり見えるようになり、D)カルノア液中のIDを解剖した。幼虫内のIDの位置はアスタリスクで表示されています。
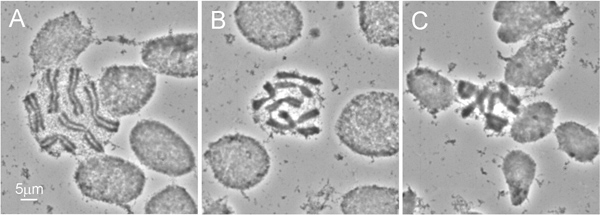
図3染色体スプレッドの異なる性質:)完全染色体の広がり-細胞の丸い形状は低張液中のIDの十分な治療を示し、B)完全な低張処理-染色体が少しundersquashedある; C)は貧しい染色体スプレッド。 - 結果不十分な低張処理は細胞の楕円形で示されます。
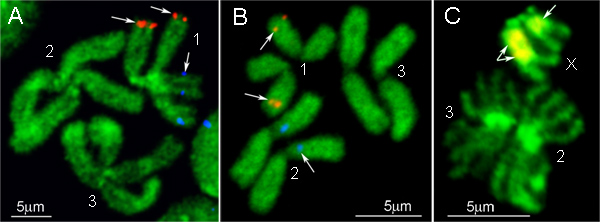
図4:BACクローン(A、B)とAEの染色体におけるrDNAのIGSの(C)とFISHの例。ネッタイシマカ ()、 ネッタイイエカ (B)、および。ハマダラカ (C)である。 1、2、3 -染色体の数であり、X -で女性の性染色体。ハマダラカ。
ディスカッション
蚊の分裂期染色体上の非蛍光in situハイブリダイゼーションは、A.クマーとK.チェンライ17によって1990年に初めて行われた。その研究では、18Sと28SリボソームDNA遺伝子は、プラスミド1で一緒にクローニングされた蚊の20種の染色体に配置した。 DNAプローブを放射性標識し、脳の神経節からの染色体にハイブリダイズさせた。 3蚊属の中では、FISH法は、AEの細胞株から分裂期染色体のために開発されました。ライブ蚊から有糸分裂染色体上ネッタイシマカ 10,18,19および実行されたことがありません。最近、我々は第 4齢幼虫9のIDから、高品質な染色体の準備を取得するための、シンプルで堅牢な技術を開発しました。この方法では、染色体の数が多いが、1スライドに得ることを可能にすると普遍的に蚊のすべての種のために使用することができます。唯一の幼虫、蛹、大人ではないSTAを使用する必要蚊のGESは、スライドの準備のために、おそらく方法の唯一の制限事項です。標準FISH法12は ヒトスジシマカ、アカイエカ、およびハマダラカの分裂期染色体のプローブとしてゲノムBACクローンとIGS rDNAを用いて最適化されています。
これらの特定のアプリケーションに加えて、ここで説明したFISHプロトコルは、他の目的にも使用することができます。非特異的なハイブリダイゼーションを阻止するためのC 0トンのDNA画分を利用して高度なFISHプロトコルも、BACクローンまたはハマダラカのヘテロクロマチン領域における他の大規模なDNA断片のハイブリダイゼーションのために適用することができます。ヘテロクロマチン領域は転移因子や他のリピートを豊かにし、これらの地域からのプローブは通常、染色体3上の強力なバックグラウンドを生産している。ラベルなしのC 0トンのDNA画分を使用すると、染色体へのプローブの非特異的ハイブリダイゼーションを削減するのに役立ちます。 FISHのprのシンプルバージョンotocolは蚊や他の昆虫の分裂期染色体上の任意のrDNAまたは反復的なDNAプローブを使用することができます。さらに、それはまた、 蚊やショウジョウバエなどのユークロマチン領域における低反復DNA含量の種におけるBACクローンDNAのハイブリダイゼーションのために適用することができます。ここで提案されたプロトコルは、蚊の高度に完成した染色体ベースのゲノムアセンブリを取得するのに役立ちますし、広く昆虫の他のグループ内のさまざまな細胞遺伝学的用途に使用することができる。
開示事項
特別な利害関係は宣言されません。
謝辞
我々は、 ハマダラカで染色体の準備と魚と彼らの助けのためにセルゲイデーミンとタチアナKaramyshevaに感謝します。また、テキストを編集するために私達にヒトスジシマカとアカイエカゲノムDNA BACクローンとメリッサウェイドを提供するためのデイビッドSeversonに感謝します。イゴールV. SharakhovにマリアV. SharakhovaとAI094289 1R21-01 1R21 AI88035-01:この作品は、国立衛生研究所からの2の補助金によって支えられている。
資料
| Name | Company | Catalog Number | Comments |
| 試薬/機器の名称 | 会社 | カタログ番号 | 注釈 |
| MZ6ライカの実体顕微鏡 | ライカ | VA-OM-E194-354 | 異なった実体顕微鏡を使用することができます |
| オリンパスCX41位相差顕微鏡 | オリンポス | CX41 | 異なる位相差顕微鏡を使用することができます |
| オリンパスBX61蛍光顕微鏡 | オリンポス | BX61 | 異なる蛍光顕微鏡を使用することができます |
| ThermoBriteスライド変性/ハイブリダイゼーションシステム | アボット分子 | 30-144110 | 加熱ブロック多湿の室となる |
| 解剖針 | ファインScienceTools | 10130から10 | |
| ニードルホルダー | ファイン科学ツール | 26018から17 | |
| 解剖用はさみ | ファイン科学ツール | 15000から03 | |
| 75x25ダブルすりガラスマイクロスライド | コーニング | 2949-75x25 | |
| 22x22のmmの顕微鏡カバースリップ | フィッシャー·サイエンティフィック | 12-544-10 | |
| パラフィルム | フィッシャー·サイエンティフィック | 13-374-10 | |
| ゴムのり | フィッシャー·サイエンティフィック | 50-949-105 | |
| 酢酸 | フィッシャー·サイエンティフィック | A491-212 | |
| アルコール200プルーフ | ディーラボラトリーズ | 2701 | |
| プロピオン酸 | シグマAldr白点病 | 402907 | |
| 塩酸 | フィッシャー·サイエンティフィック | A144-500 | |
| クエン酸ナトリウム二水和物 | フィッシャー·サイエンティフィック | S279-500 | |
| 酢酸ナトリウム三水和物 | フィッシャー·サイエンティフィック | BP334-500 | |
| 塩化カリウム | フィッシャー·サイエンティフィック | BP366-500 | |
| EDTAを | フィッシャー·サイエンティフィック | S311-500 | |
| トリス塩基 | フィッシャー·サイエンティフィック | BP152-1 | |
| 10倍のPBS | インビトロジェン | P5493 | |
| 10パーセントNBF(中性緩衝ホルマリン) | シグマアルドリッチ | HT501128 | |
| 99%ホルムアミド | フィッシャーンスntific | BP227500 | |
| デキストラン硫酸エステルナトリウム塩 | シグマアルドリッチ | D8906 | |
| 20×SSC緩衝液 | インビトロジェン | AM9765 | |
| 1mMのYOYO-1ヨウ(509分の491)ソリューション | インビトロジェン | Y3601 | |
| ゴールド試薬を延長退色 | インビトロジェン | P36930 | |
| は、dATP、dCTP、dGTP、dTTPの | フェルメンタス | R0141、R0151、R0161、R0171 | |
| にCy3-dUTPを、Cy5で-dUTPを | GEヘルスケア | PA53022、PA55022 | |
| BSA | シグマアルドリッチ | A3294 | |
| DNAポリメラーゼI | フェルメンタス | EP0041 | |
| DNase Iを | フェルメンタス | EN0521 | |
| S1ヌクレアーゼ | フェルメンタス | EN0321 | |
| Taq DNAポリメラーゼ | インビトロジェン | 18038-042 | |
| アーゼ | シグマアルドリッチ | 9001-99-4 | |
| ペプシン | USB | 9001-75-6 | |
| サケ精子DNA | シグマアルドリッチ | D7656 | |
| ノニデット-P40(NP40) | 米国の生物 | NC9375914 | |
| キアゲン血液および細胞培養Maxikit | キアゲン | 13362 | |
| キアゲン大コンストラクトキット | キアゲン | 12462 |
参考文献
- Sharakhov, I. V., Sharakhova, M. V., Verrity, J. F., Abbington, L. E. . Chromosome Mapping Research Developments. , (2008).
- Holt, R. A., et al. The genome sequence of the malaria mosquito Anopheles gambiae. Science. 298, 129-149 (2002).
- Sharakhova, M. V., et al. Update of the Anopheles gambiae PEST genome assembly. Genome biology. 8, R5 (2007).
- Campos, J., Andrade, C. F., Recco-Pimentel, S. M. A technique for preparing polytene chromosomes from Aedes aegypti (Diptera, Culicinae). Memorias do Instituto Oswaldo Cruz. 98, 387-390 (2003).
- Campos, J., Andrade, C. F., Recco-Pimentel, S. M. Malpighian tubule polytene chromosomes of Culex quinquefasciatus (Diptera, Culicinae). Memorias do Instituto Oswaldo Cruz. 98, 383-386 (2003).
- McAbee, R. D., Christiansen, J. A., Cornel, A. J. A detailed larval salivary gland polytene chromosome photomap for Culex quinquefasciatus (Diptera: Culicidae) from Johannesburg, South Africa. J. Med. Entomol. 44, 229-237 (2007).
- Nene, V., et al. Genome sequence of Aedes aegypti, a major arbovirus vector. Science. 316, 1718-1723 (2007).
- Arensburger, P., et al. Sequencing of Culex quinquefasciatus establishes a platform for mosquito comparative genomics. Science. 330, 86-88 (2010).
- Sharakhova, M. V., et al. Imaginal discs--a new source of chromosomes for genome mapping of the yellow fever mosquito Aedes aegypti. PLoS neglected tropical diseases. 5, e1335 (2011).
- Brown, S. E., et al. Toward a physical map of Aedes aegypti. Insect Mol. Biol. 4, 161-167 (1995).
- Steiniger, G. E., Mukherjee, A. B. Insect chromosome banding: technique for G- and Q-banding pattern in the mosquito Aedes albopictus. Can. J. Genet. Cytol. 17, 241-244 (1975).
- Garimberti, E., Tosi, S., Bridger, J. M., Volpi, E. V. . Fluorescence in situ hybridization (FISH). , (2010).
- Scott, J. A., Brogdon, W. G., Collins, F. H. Identification of single specimens of the Anopheles gambiae complex by the polymerase chain reaction. The American journal of tropical medicine and hygiene. 49, 520-529 (1993).
- Trifonov, V. A., Vorobyeva, N. N., Rens, W., Leiehr, T. . Fluorescence in situ hybridization (FISH). , (2009).
- Collins, F. H., et al. A ribosomal RNA gene probe differentiates member species of the Anopheles gambiae complex. The American journal of tropical medicine and hygiene. 37, 37-41 (1987).
- Kumar, A., Rai, K. S. Chromosomal localization and copy number of 18S+28S ribosomal RNA genes in evolutionary diverse mosquitoes (Diptera, Culicidae). Hereditas. 113, 277-289 (1990).
- Brown, S. E., Knudson, D. L. FISH landmarks for Aedes aegypti chromosomes. Insect Mol. Biol. 6, 197-202 (1997).
- Brown, S. E., Severson, D. W., Smith, L. A., Knudson, D. L. Integration of the Aedes aegypti mosquito genetic linkage and physical maps. Genetics. 157, 1299-1305 (2001).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved