Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
El uso de LysoTracker para detectar la muerte celular programada en embriones y la diferenciación de células madre embrionarias
En este artículo
Resumen
Se presenta un protocolo simple de visualizar regiones de la muerte celular programada (PCD) en embriones de ratón y diferenciadores madre embrionarias (ES) de cultivos de células utilizando un colorante altamente soluble llamada LysoTracker.
Resumen
La muerte celular programada (PCD) se produce en adultos para mantener la homeostasis del tejido normal y durante el desarrollo embrionario para dar forma a los tejidos y órganos 1,2,6,7. Durante el desarrollo, los productos químicos tóxicos o alteraciones genéticas pueden causar un aumento en la PCD o cambiar los patrones resultantes de PCD en anomalías en el desarrollo y defectos de nacimiento 3-5. Para entender la etiología de estos defectos, el estudio de los embriones puede complementarse con ensayos in vitro que utilizan diferenciar madre embrionarias (ES) de células.
La apoptosis es una forma bien estudiado de PCD, que implica tanto intrínseca y extrínseca de señalización para activar la cascada de enzimas caspasas. Cambios característicos celulares incluyen formación de ampollas en la membrana, contracción nuclear y la fragmentación del ADN. Otras formas de PCD no implican la activación de caspasas y puede ser el resultado final de la autofagia prolongado. Independientemente de la vía de PCD, las células que mueren necesitan ser removidos. En los adultos, las células inmunes performe esta función, mientras que en los embriones, donde el sistema inmune todavía no se ha desarrollado, la eliminación se produce por un mecanismo alternativo. Este mecanismo consiste en células vecinas (llamados "no profesionales" fagocitos) que asumen un papel fagocítico-reconocen la "cómeme" señal en la superficie de la célula que muere y lo engullen 8-10. Después de inmersión, los escombros se lleva a los lisosomas para degradación. Así, independientemente del mecanismo PCD, un aumento de la actividad lisosomal se puede correlacionar con una mayor muerte celular.
Para el estudio de PCD, un ensayo simple de visualizar los lisosomas en los tejidos gruesos y multicapa culturas diferenciadoras pueden ser útiles. LysoTracker colorante es una molécula pequeña altamente soluble que es retenido en ácidos compartimentos subcelulares tales como el lisosoma 11-13. El colorante es absorbido por difusión y por medio de la circulación. Dado que la penetración no es un obstáculo, la visualización de la CPD en los tejidos gruesos y las culturas de varias capas es posible 12,13 14, se limita a muestras pequeñas, secciones histológicas, y cultivos en monocapa debido a que el procedimiento requiere la entrada / permeabilidad de una transferasa terminal.
En contraste con azul de anilina, que se difunde y se disuelve por disolventes, LysoTracker Red DND-99 se puede fijar, brillante y estable. La tinción puede ser visualizada con microscopía confocal fluorescente estándar o en todo el montaje o sección utilizando medio de montaje acuoso o de disolvente 12,13. Aquí se describen los protocolos que utilizan este medio de contraste para ver la CPD en normal y sonic hedgehog embriones de ratones nulos. Además, se demuestra el análisis de PCD en la diferenciación de cultivos de células ES y presentar un método de cuantificación simple. En resumen, la tinción LysoTracker puede ser un gran complemento a otros métodos de detección de PCD.
Protocolo
1. LysoTracker tinción de embriones de ratón
- Generar embriones de ratón mediante la colocación de jóvenes (5-6 semanas de edad) las hembras en jaulas de prisioneros masculinos. Monitorear la mañana siguiente para la aparición de un tapón vaginal que indica que se ha producido el apareamiento. Si la mujer está embarazada, el mediodía de ese día se llama 0,5 dpc (coito días después). El procedimiento que aquí se presenta es ideal para los embriones 13.7 dpc.
- En el día embrionario de interés, la eutanasia a la hembra de acuerdo con los protocolos aprobados y quitar el útero. Retire los embriones de la decidua en una placa de Petri de 10 cm con Hanks BSS (sin rojo fenol). Retire las membranas extra-embrionarias y que por lo menos un enjuague con Hanks BSS para eliminar los tejidos extraños y sangre.
- Preparar embriones LysoTracker solución de tinción (5 mM en LysoTracker Hanks BSS). Un volumen conveniente para 1-2 camadas es de 5 ml. Mezclar suavemente para desembolsar el colorante uniformemente.
- Añadir embriones al Solut LysoTracker tinción de embrionesiones en un tubo de microcentrífuga o vial y se incuba a 37 ° C durante 45 min.
- Enjuague suavemente en Hanks BSS cuatro veces, 5 minutos cada lavado.
- Fijar en 4% paraformaldehido durante la noche.
- Enjuagar una vez en Hanks BSS, 10 min, para eliminar la solución.
- Deshidratar a través de una serie metanol (50%, 75%, 80%, 100%, 5 min cada paso) para eliminar la tinción de fondo. Esto es importante para lograr una buena relación señal a ruido durante la exploración.
- En este punto usted puede almacenar las muestras de embriones indefinidamente en metanol a -20 ° C, protegido de la luz.
Nota: Una muestra de tejido se pueden tomar en cualquier paso para el genotipado de ADN, sin embargo, puede ser más conveniente para tomar una muestra justo antes del almacenamiento de modo que hasta esta etapa todas las muestras pueden ser procesadas por lotes. Se recoge una muestra de tejido de la cola, extremidad, o la cabeza de cada embrión y poner todo en tubos separados. Rehidratar la muestra de tejido y se preparan para genotyping.
2. LysoTracker tinción de diferenciación de las Culturas ES
- Preparar cuerpos embrioides a través del Método de la gota colgante 15 con una densidad inicial de 500 células por 20 l gota.
- Después de 3 días, la transferencia de los cuerpos embrioides al cultivo en suspensión en 10 cm no adherentes placas de Petri (es decir, los que se utilizan para el trabajo bacteriológico). Cultura durante 5 días, el cambio de los medios de comunicación cada 2 días.
- El día 7, trasladar los cuerpos embrioides a gelatinizados portaobjetos de cámara de 8 pocillos (añadir un 0,1% de gelatina a las cámaras, espere 5 minutos, retirar y agregar los medios de comunicación). Traslado 1-2 cuerpos embrioides por cámara.
- Cultura durante 10 días, el cambio de los medios de comunicación todos los días. Tener cuidado para colocar la pipeta en la esquina de la cámara de corredera a fin de no perturbar el cuerpo embrioide unir a la superficie del vidrio. También cuando la adición de medio fresco también colocar la pipeta en la esquina de la cámara y soltar los medios de comunicación lentamente.
- Después de 10 días de cultoUre, para inducir la apoptosis como un control positivo, el tratamiento de algunos de los pocillos con 0,1 mM y 1,0 mM H 2 O 2 (añadir el H 2 O 2 a los medios de comunicación fresca, mezcla, a continuación, añadir a las células) durante 60 min. Aclarar dos veces con D-PBS y cultivo de una noche en medios regulares.
- Aspirar suavemente medios de comunicación existentes y añadir la célula LysoTracker solución de tinción (500 LysoTracker nM (concentración final) en los medios de comunicación, 300 l por cámara).
- Incubar a 37 ° C durante 15 min.
- Aclarar dos veces con D-PBS 2 veces, 5 min cada uno.
- Fijar en 4% de paraformaldehído durante 15 min a TA.
- Aclarar dos veces con D-PBS, 5 min.
3. Viendo embriones LysoTracker manchadas o culturas ES
- Preparar las muestras de células para la microscopía por aspiración de la D-PBS; retirar la cámara del portaobjetos; secar al aire durante aproximadamente 5 min, y luego la adición de un medio acuoso de montaje (Vectashield con DAPI o similar). Coloque una lámina de barrera mientras drying para disminuir blanqueo del fluoróforo. Montar los embriones en un recipiente hondo de cristal-depresión o pequeña placa de Petri en Vectashield o similar.
- Visualizar con un microscopio de disección o compuesto equipado con una rodamina o Texas Red filtro (LysoTracker RED DND-99, de excitación / emisión: 577/590 nm). La coloración suele ser bastante brillante para exposiciones largas no son necesarias.
4. Cuantificación de los resultados mediante técnicas de imagen
- Toma fotos digitales de sus muestras bajo iguales condiciones de exposición en modo manual.
- Abra las imágenes en Adobe Photoshop o un programa de imagen similar. Para determinar la cantidad de manchas LysoTracker, seleccionar el canal rojo y el umbral de la imagen (la imagen se convierte a negro y blanco) de tal manera que la tinción con rojo ahora es representado por píxeles blancos. Seleccione los píxeles blancos y usar la función de histograma para obtener el recuento total.
- Utilice el canal azul para contarel número de núcleos en el campo. Puede ser útil llevar un registro de la cantidad de anotaciones en la imagen.
- Registre los valores y luego calcular el nivel promedio por tinción celular. Repita este análisis con el umbral mismo para todas las imágenes que desea probar.
5. Los resultados representativos
No hay un patrón normal de PCD durante el desarrollo embrionario que puede cambiar cuando un gen del factor de crecimiento, tales como Sonic hedgehog (Shh), se retira 16,17. Desde LysoTracker es un colorante de molécula pequeña que es altamente difusible, el embrión puede ser visualizado para evaluar las áreas de PCD incluso profundamente dentro del mesénquima 12,13 La Figura 1 muestra el patrón de tinción para LysoTracker normales y Shh -. / - Embriones de ratones mutantes en 10.5dpc. En esta etapa, la PCD se ve incrementado para Shh - / - embriones mutantes, en particular en la extremidad y regio somitens. Las porciones del embrión puede ser reflejado en todo el montaje a mayor aumento (1B) para evaluar el patrón general. Partes disecadas también pueden ser seccionados con un micrótomo vibrante (1C) o un criostato (DF) para revelar las regiones de PCD en mayor detalle.
TUNEL análisis se realiza mejor en las secciones delgadas, donde la penetración de la transferasa terminal es menos problemática. Tinción LysoTracker y un ensayo de TUNEL se puede hacer en el mismo tejido (Figura 1D-G). Estos resultados muestran que, aunque no hay una estricta correlación 1:1 entre LysoTracker y tinción de TUNEL, las regiones generales de superposición PCD considerablemente. Con un aumento mayor, es posible ver que puncta tinción positiva con el ensayo de TUNEL, pero no con el colorante LysoTracker, puncta que mancha sólo con el colorante LysoTracker, y puncta que tienen la superposición de tinción.
Cuando una EB se sembraron sobre una superficie recubierta con gelatina, células diferenciadoras se migrate hacia afuera. Después de 10 días de cultivo, las células se han diferenciado en una variedad de tipos de células incluyendo neuronas, células musculares, y cardiomiocitos. Los cultivos que se muestran en la Figura 2 contienen poblaciones mixtas de estos tipos de células, sin embargo se podría adaptar el protocolo para estudiar PCD de un tipo particular de célula de interés. Por ejemplo, el EB podrían ser tratados con ácido retinoico para mejorar la diferenciación de las neuronas 19, y luego el ensayo de LysoTracker podría ser utilizado para evaluar la muerte celular neuronal en condiciones diferentes. En la cultura normal de las células madre embrionarias diferenciadas, hay unas pocas células que se someten a PCD y estos pueden ser detectados con LysoTracker y / o ensayos de TUNEL (Figura 2A). PCD puede ser inducida por un número de diferentes agentes citotóxicos tales como peróxido de hidrógeno (H 2 O 2) que induce daño oxidativo e inicia la ruta apoptótica 20,21. Una dosis baja de H 2 O 2 aumenta tantoLysoTracker y tinción TUNEL en comparación con ningún tratamiento, mientras que se produce una alta dosis de contracción celular y LysoTracker generalizada y la positividad TUNEL. Similar a los resultados en el embrión, es posible ver puncta que la tinción positiva con el ensayo de TUNEL, pero no con el colorante LysoTracker, puncta que se tiñen solamente con el colorante LysoTracker, y puncta que tienen la superposición de tinción. La consecuencia de la aplicación de un agente citotóxico que induce apopotosis se compara con las condiciones de privación de suero que inducen la autofagia (Figura 2D). Las células que se han sometido a un periodo de privación de suero (1 hora) presentan un patrón diferente donde LysoTracker y tinción TUNEL no se correlacionan altamente. Tinción LysoTracker es mucho mayor en comparación con los cultivos sin tratar, sin embargo positividad TUNEL se encuentra a niveles normales.
El empleo de herramientas estándar disponibles en Adobe Photoshop, tinción LysoTracker se puede cuantificar con un método de umbral simpleque permite calcular el nivel de tinción promedio de una población de células. Figura 3A-B muestra cómo se ve una imagen de muestra durante el proceso de cuantificación. A partir de una imagen de referencia con la tinción de LysoTracker en el canal rojo y los núcleos con DAPI-manchado en el canal azul (panel 3A), el canal rojo de esta imagen fue thresholded para convertir la imagen en negro y blanco y los píxeles blancos eran seleccionan entonces y se contaron (Figura 3A '). A nivel de umbral fue elegido que representa el patrón de tinción LysoTracker. Al igual que con cualquier método de cuantificación que utiliza imágenes, es importante evitar la sobreexposición de sus fotos ya que esto podría resultar en un excesivo énfasis en las diferencias. El número de los núcleos en el campo, entonces se puede contar al ver sólo el canal azul (Figura 3B) y el marcado de los núcleos utilizando una herramienta de dibujo tal como un pincel mientras cuenta (Figura 3B '). Entonces se puede expresar the nivel de tinción mediante el cálculo del nivel medio de la tinción por célula (número de píxeles / Número de núcleos). Si se desea utilizar este algoritmo para alta producción, para la detección, la mayoría de los paquetes de detección pueden medir el brillo en un canal en particular y también tienen una función de identificador de objeto que se cuentan los núcleos en un campo, sin embargo para la mayoría de las aplicaciones típicas, el método manual es rápida y simple. Para dar un ejemplo, una región de la muestra en las Figuras 2A-C se cuantificó y confirmar los resultados que el aumento de las concentraciones de H 2 O 2 resultados en un nivel más alto de tinción LysoTracker (Figura 3C).
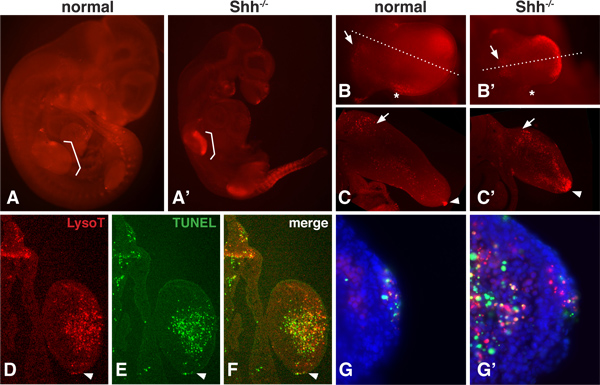
Figura 1. LysoTracker tinción normales y Shh - Embriones - /. A, A 'normales y Shh -. / - Embriones de ratón en 10,5 dpc, se tiñeron con Rojo LysoTracker, montado en Vectashield y se fotografiaron bajoepiflourescence un microscopio de disección. Los paréntesis indican la localización de las extremidades anteriores B, B 'extremidad anterior de normales y Shh -.. / - Embriones fotografiado a mayor aumento. Tenga en cuenta los dominios normales de muerte celular, incluyendo la "zona posterior necrótica '(marcado con un asterisco) y una región proximal en el mesénquima del primordio de la extremidad (marcada con una flecha). Extremidades Shh nulos muestran fuerte tinción en la porción medial y distal . c, c '. secciones medial a través de la yema extremidad anterior de yemas de las extremidades normales y null. Obsérvese la presencia de tinción en el mesénquima proximal (flecha) y ARE (punta de flecha) de la yema de la extremidad normal y la fuerte tinción en el mesénquima dorsal y ventral y distal dentro de la yema del miembro nulo. DF. La misma sección (12 micras, congelados sección) fue fotografiada por LysoTracker y tinción TUNEL (Roche 1.684.795). La sección pasa a través de la porción distal de la mesenc Shh nuloHyme. Los puntos de punta de flecha a la AER. Nótese cómo el patrón general es muy similar con ambos métodos, sin embargo, la tinción no se superponen completamente. G, G '. Con un aumento mayor de la ARE y el mesénquima, uno puede ver tanto la presencia de puntos lagrimales que son LysoTracker o TUNEL positiva sólo así como puntos lagrimales que se solapan las manchas.

Figura 2. LysoTracker tinción en las células ES diferenciadas. Diferenciación de las células se sembraron en portaobjetos de cámara de 8-en ausencia de LIF. Para mostrar cómo LysoTracker y comparar la tinción de TUNEL, la vía de la apoptosis fue inducida con 2 dosis diferentes de H 2 O 2, mientras que la vía de la autofagia se indujo usando 1 hora de privación de suero. Estas imágenes fueron tomadas con un Microscopio Confocal Zeiss LSM5 A-A'':. Cultivos no tratados muestran algunos apoptosis normal como se indica por LysoTRacker y la tinción de TUNEL. El patrón de uso de ambas técnicas en su mayoría de solapamiento (flechas blancas), sin embargo, se puede ver un puncta pocos que son Lysotracker (flechas rojas) positivos sólo B-B'', CC ":. Tratamiento con cantidades crecientes de H 2 O 2 estimula la apoptosis . La dosis inferior muestra más LysoTracker y puncta TUNEL positivo en comparación con las células no tratadas. La dosis más alta induce en LysoTracker aún más y puncta TUNEL positiva. flechas apuntan a las manchas White solapamiento, mientras que las flechas rojas apuntan a puncta que son positivos para LysoTracker sólo. También es posible ver puncta que sólo son positivas para la tinción de TUNEL (flechas verdes) DD ":. privación de suero puede inducir la autofagia, que puede estimular una acumulación de material en los lisosomas y los autophagolysosomes. Tenga en cuenta cuántos puntos lagrimales LysoTracker son visibles en comparación con cultivos no tratados. TUNEL positiva puncta están presentes (la mayoría de ellas se solapan LysoTracker manchaING, flechas blancas), sin embargo, el número no es mucho mayor en comparación con condiciones sin tratamiento. Además, en condiciones de privación de suero, LysoTracker de sólo puncta positivo tienen una alta prevalencia (flechas rojas).
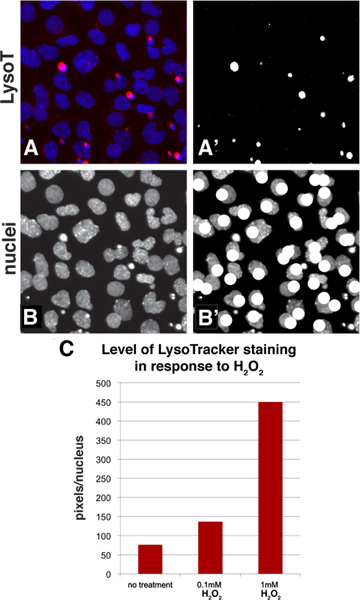
Figura 3. Método de cuantificación para evaluar el nivel de tinción en una población de células usando Adobe Photoshop. A: Una porción de la Figura 2A se ha elegido y la LysoTracker (canal rojo) y tinción con DAPI (núcleos, azul canal) se muestra en este panel 'A:. El canal rojo de la Figura 3A se thresholded para convertirlo en una y negro imagen en blanco. Los píxeles blancos pueden ser seleccionadas y se contaron utilizando la función de histograma B-B ':. El canal azul muestra los núcleos y mientras contando, la imagen puede ser anotado con una función de dibujo tal como un pincel C:. Este es un excuantificación amplio de una muestra a partir de paneles 2A-C. Los datos se representan como píxeles por núcleos. Haga clic aquí para ampliar la cifra .
Discusión
Características importantes de la apoptosis incluyen la detección de daños en el ADN con el ensayo TUNEL, junto con la detección de la actividad caspasa (detectada con mAbs o una molécula de unión tal como zVAD-fmk), la observación de los cambios celulares, y la presentación de la fosfatidilserina (PS ) en la superficie de la membrana (detectado por Anexina V vinculante). Hay algunas desventajas con cada uno de estos ensayos. Por ejemplo, la transferasa terminal utilizado en el ensayo de TUNEL no penetra más al...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Damos las gracias a los miembros del laboratorio Mariani ayuda editando el protocolo. Este trabajo fue financiado por el CIRM Formación Postdoctoral Grant (JLF), un internado CIRM BRIDGES (TZTT), el Robert E. May y R. Wright Foundation (FVM) y la Universidad del Sur de California (FVM).
Materiales
| Name | Company | Catalog Number | Comments | ||||||||||||||
| Nombre del reactivo | Empresa | Número de catálogo | |||||||||||||||
| LysoTracker Red NOM-99 | Invitrogen | # L-7528 | |||||||||||||||
| Hanks BSS | Invitrogen | 14025-076 | |||||||||||||||
| Paraformaldehído | EMD | EM-PX0055-3 | |||||||||||||||
| Vectashield | VECTOR | H-1200 | |||||||||||||||
| DMEM | Cellgro | 10-013 CV- | |||||||||||||||
| No esenciales de aminoácidos | Cellgro | 25 a 025-CI | |||||||||||||||
| Piruvato de sodio | Cellgro | 25 a 000-CI | |||||||||||||||
| FBS | Hyclone | SH30071.02 | |||||||||||||||
| Pen-Strep | Invitrogen | 15140-122 | |||||||||||||||
| b-mercaptoetanol, 50 mM | Invitrogen | 21985-023 | |||||||||||||||
| Labtek-II de la Cámara portaobjetos (8-bien) | Nalge Nunc Internacional | 154534 | |||||||||||||||
| 0,1% de gelatina | Millipore | ES-006-B | |||||||||||||||
| PBS de Dulbecco (D-PBS) | Cellgro | 21-031 CV- | |||||||||||||||
Recetas de Soluciones 4% de paraformaldehído Para 100 ml:
ADVERTENCIA: paraformaldehído en 'volante' forma (pequeños gránulos comprimidos) es menor en polvo y por lo tanto puede ser medido fuera de una campana. Sin embargo, usted todavía debe usar una máscara protectora contra el polvo (N95por lo menos) durante la manipulación. EB Cultura Medios Para 500 ml de medio EB:
|
Referencias
- Baehrecke, E. H. How death shapes life during development. Nat. Rev. Mol. Cell Biol. 3, 779-787 (2002).
- Meier, P., Finch, A., Evan, G. Apoptosis in development. Nature. 407, 796-801 (2000).
- Ikonomidou, C. Ethanol-induced apoptotic neurodegeneration and fetal alcohol syndrome. Science. 287, 1056-1060 (2000).
- Dunty, W. C., Chen, S. Y., Zucker, R. M., Dehart, D. B., Sulik, K. K. Selective vulnerability of embryonic cell populations to ethanol-induced apoptosis: implications for alcohol-related birth defects and neurodevelopmental disorder. Alcohol Clin. Exp. Res. 25, 1523-1535 (2001).
- Price, O. T., Lau, C., Zucker, R. M. Quantitative fluorescence of 5-FU-treated fetal rat limbs using confocal laser scanning microscopy and Lysotracker. Cytometry A. 53, 9-21 (2003).
- Jacobson, M. D., Weil, M., Raff, M. C. Programmed cell death in animal development. Cell. 88, 347-354 (1997).
- Fuchs, Y., Steller, H. Programmed cell death in animal development and disease. Cell. 147, 742-758 (2011).
- Qu, X. Autophagy gene-dependent clearance of apoptotic cells during embryonic development. Cell. 128, 931-946 (2007).
- Grimsley, C., Ravichandran, K. S. Cues for apoptotic cell engulfment: eat-me, don't eat-me and come-get-me signals. Trends Cell Biol. 13, 648-656 (2003).
- Lauber, K., Blumenthal, S. G., Waibel, M., Wesselborg, S. Clearance of apoptotic cells: getting rid of the corpses. Mol. Cell. 14, 277-287 (2004).
- Haller, T., Dietl, P., Deetjen, P., Volkl, H. The lysosomal compartment as intracellular calcium store in MDCK cells: a possible involvement in InsP3-mediated Ca2+ release. Cell Calcium. 19, 157-165 (1996).
- Zucker, R. M., Hunter, S., Rogers, J. M. Confocal laser scanning microscopy of apoptosis in organogenesis-stage mouse embryos. Cytometry. 33, 348-354 (1998).
- Zucker, R. M., Hunter, E. S., Rogers, J. M. Apoptosis and morphology in mouse embryos by confocal laser scanning microscopy. Methods. 18, 473-480 (1999).
- Gavrieli, Y., Sherman, Y., Ben-Sasson, S. A. Identification of programmed cell death in situ via specific labeling of nuclear DNA fragmentation. J. Cell Biol. 119, 493-501 (1992).
- Foty, R. A Simple Hanging Drop Cell Culture Protocol for Generation of 3D Spheroids. J. Vis. Exp. (51), e2720 (2011).
- Chiang, C. Manifestation of the limb prepattern: limb development in the absence of sonic hedgehog function. Dev. Biol. 236, 421-435 (2001).
- Ishibashi, M., McMahon, A. P. A sonic hedgehog-dependent signaling relay regulates growth of diencephalic and mesencephalic primordia in the early mouse embryo. Development. 129, 4807-4819 (2002).
- Schuldiner, M. Induced neuronal differentiation of human embryonic stem cells. Brain Res. 913, 201-205 (2001).
- Bain, G., Kitchens, D., Yao, M., Huettner, J. E., Gottlieb, D. I. Embryonic stem cells express neuronal properties in vitro. Dev. Biol. 168, 342-357 (1995).
- Dumont, A. Hydrogen peroxide-induced apoptosis is CD95-independent, requires the release of mitochondria-derived reactive oxygen species and the activation of NF-kappaB. Oncogene. 18, 747-757 (1999).
- Hampton, M. B., Orrenius, S. Dual regulation of caspase activity by hydrogen peroxide: implications for apoptosis. FEBS Lett. 414, 552-556 (1997).
- Wood, W. Mesenchymal cells engulf and clear apoptotic footplate cells in macrophageless PU.1 null mouse embryos. Development. 127, 5245-5252 (2000).
- Scott, R. C., Juhasz, G., Neufeld, T. P. Direct induction of autophagy by Atg1 inhibits cell growth and induces apoptotic cell death. Curr. Biol. 17, 1-11 (2007).
- Rodriguez-Enriquez, S., Kim, I., Currin, R. T., Lemasters, J. J. Tracker dyes to probe mitochondrial autophagy (mitophagy) in rat hepatocytes. Autophagy. 2, 39-46 (2006).
- Bampton, E. T., Goemans, C. G., Niranjan, D., Mizushima, N., Tolkovsky, A. M. The dynamics of autophagy visualized in live cells: from autophagosome formation to fusion with endo/lysosomes. Autophagy. 1, 23-36 (2005).
- Boya, P., Mellen, M. A., de la Rosa, E. J. How autophagy is related to programmed cell death during the development of the nervous system. Biochem. Soc. Trans. 36, 813-817 (2008).
- Galluzzi, L. Guidelines for the use and interpretation of assays for monitoring cell death in higher eukaryotes. Cell Death Differ. 16, 1093-1107 (2009).
- Klionsky, D. J. Guidelines for the use and interpretation of assays for monitoring autophagy in higher eukaryotes. Autophagy. 4, 151-175 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados