È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
L'uso di LysoTracker per rilevare la morte cellulare programmata in embrioni e differenziazione delle cellule staminali embrionali
In questo articolo
Riepilogo
Vi presentiamo un protocollo semplice per visualizzare le regioni di morte cellulare programmata (PCD) in embrioni di topo e di differenziazione staminali embrionali (ES) colture cellulari utilizzando un colorante chiamato LysoTracker altamente solubile.
Abstract
La morte cellulare programmata (PCD) si verifica negli adulti per mantenere l'omeostasi dei tessuti normali e durante lo sviluppo embrionale di modellare i tessuti e gli organi 1,2,6,7. Durante lo sviluppo, prodotti chimici tossici o alterazioni genetiche possono causare un aumento di PCD o modificare i modelli di PCD con conseguente sviluppo di anomalie e difetti di nascita 3-5. Per comprendere l'eziologia di questi difetti, lo studio di embrioni può essere completata con prove in vitro che utilizzano differenziare staminali embrionali (ES) cellule.
L'apoptosi è un ben studiato forma di PCD che coinvolge sia intrinseca ed estrinseca segnalazione per attivare l'enzima caspasi cascata. Alterazioni cellulari caratteristici sono blebbing membrana, restringimento nucleare, e la frammentazione del DNA. Altre forme di PCD non coinvolgono l'attivazione delle caspasi e possono essere il risultato finale di autofagia prolungata. Indipendentemente dal percorso PCD, cellule morenti devono essere rimossi. Negli adulti, le cellule immunitarie perfOrm questa funzione, mentre in embrioni, in cui il sistema immunitario non ha ancora sviluppato, la rimozione avviene con un meccanismo alternativo. Questo meccanismo comporta cellule vicine (chiamati "non professionali fagociti") assume un ruolo-fagocitica riconoscono il 'mangiami' segnale sulla superficie della cellula morente e inghiottire lo 8-10. Dopo immersione, i detriti viene portato lisosomi per la degradazione. Quindi indipendentemente meccanismo PCD, un aumento dell'attività lisosomiale può essere correlata con la morte cellulare aumentata.
Per studiare PCD, un test semplice per visualizzare lisosomi nei tessuti spessi e multistrato culture differenziazione può essere utile. Colorante LysoTracker è una molecola altamente solubile piccola che è trattenuto in acidi compartimenti subcellulari come il lisosoma 11-13. Il colorante viene ripreso per diffusione e attraverso la circolazione. Dal momento che la penetrazione non è una, la visualizzazione ostacolo di CPS nei tessuti spessi e multistrato culture è possibile 12,13 14, è limitata a piccoli campioni, sezioni istologiche, e culture monostrato perché la procedura richiede l'inserimento / permeabilità di un terminale transferasi.
In contrasto con anilina blu, che si diffonde e si dissolve da solventi, LysoTracker Red ND-99 è risolvibile, luminoso, e stabile. La colorazione può essere visualizzato con un microscopio a fluorescenza o confocale in tutto-mount o sezione utilizzando un mezzo di montaggio acquoso o solvente 12,13. Qui descriviamo i protocolli che utilizzano questo colorante a guardare la CPS in condizioni normali e Sonic hedgehog embrioni di topo null. Inoltre, dimostriamo analisi di PCD in colture di cellule ES differenziare e presentare un metodo semplice di quantificazione. In sintesi, la colorazione LysoTracker può essere un ottimo complemento ad altri metodi di rilevazione PCD.
Protocollo
1. Colorazione LysoTracker di embrioni di topo
- Generazione di embrioni di topo inserendo giovani (5-6 settimana fa) femmine in gabbie prigionieri di sesso maschile. Monitorare il mattino seguente per la comparsa di un tappo vaginale che indica che si è verificato l'accoppiamento. Se la femmina è incinta, a mezzogiorno in quel giorno si chiama 0,5 dpc (giorni dopo il coito). La procedura qui presentata è l'ideale per gli embrioni 7-13 DPC.
- Il giorno embrionale di interesse, l'eutanasia la femmina secondo protocolli approvati e rimuovere l'utero. Rimuovere gli embrioni dalla decidua in una capsula di Petri da 10 cm con Hanks BSS (senza rosso fenolo). Togliere le membrane extra-embrionali e non almeno un risciacquo con Hanks BSS per rimuovere i tessuti estranei e sangue.
- Preparare embrioni Soluzione LysoTracker colorazione (5 mM in LysoTracker Hanks BSS). Un volume utile per 1-2 cucciolate è di 5 ml. Mescolare delicatamente per erogare il colorante in modo uniforme.
- Aggiungi embrioni al Solut colorazione LysoTracker Embryoioni in una provetta per microcentrifuga o flacone e incubare a 37 ° C per 45 min.
- Sciacquare molto delicatamente in Hanks BSS quattro volte, 5 minuti ogni lavaggio.
- Fissare in paraformaldeide al 4% durante la notte.
- Sciacquare una volta in Hanks BSS, 10 min, per rimuovere la correzione.
- Disidratare attraverso una serie di metanolo (50%, 75%, 80%, 100%, 5 min ciascuna fase) per eliminare la colorazione di fondo. Questo è importante per ottenere un buon rapporto segnale-rumore durante l'imaging.
- A questo punto è possibile memorizzare i campioni di embrioni a tempo indeterminato in metanolo a -20 ° C, al riparo dalla luce.
Nota: Un campione di tessuto può essere assunto in qualsiasi fase per la genotipizzazione del DNA, tuttavia, può essere più conveniente prendere un campione prima memoria in modo che fino a questo punto tutti i campioni possono essere elaborazione batch. Raccogliere un campione di tessuto dalla coda, l'incolumità fisica, o la testa da ogni embrione e mettere tutto in tubi separati. Reidratare il campione di tessuto e prepararsi per genotyping.
2. Colorazione LysoTracker di differenziare Culture ES
- Preparare corpi embrionali via il 15 goccia Metodo con una densità iniziale di 500 cellule per 20 microlitri goccia.
- Dopo 3 giorni, trasferire i corpi embrionali di coltura in sospensione in 10 centimetri non aderenti Petri (cioè quelli utilizzati per il lavoro batteriologica). Cultura per 5 giorni, cambiando i media ogni 2 giorni.
- Il giorno 7, trasferire i corpi embrionali per gelatinizzati 8 pozzetti diapositive camera (aggiungere 0,1% di gelatina alle camere, attendere 5 minuti, togliere e aggiungere media). Trasferimento 1-2 corpi embrionali per ogni camera.
- Cultura per 10 giorni, la sostituzione del supporto a giorni alterni. Attenzione a posizionare la pipetta all'angolo della camera di scorrimento in modo da non perturbare il corpo embryoid fissaggio alla superficie di vetro. Anche quando aggiungendo mezzo fresco anche posizionare la pipetta in un angolo della camera e rilasciare il supporto lentamente.
- Dopo 10 giorni di cultoure, per indurre apoptosi, come controllo positivo, trattano alcuni dei pozzetti con 0,1 mM e 1,0 mM di H 2 O 2 (aggiungere l'H 2 O 2 al fresco supporti, mescolare, quindi aggiungere alle cellule) per 60 min. Lavare due volte con D-PBS e della cultura durante la notte in media regolari.
- Aspirare delicatamente media esistenti e aggiungere cellulare LysoTracker soluzione colorante (500 LysoTracker nM (concentrazione finale) in media, 300 ul per ogni camera).
- Incubare a 37 ° C per 15 min.
- Lavare due volte con D-PBS 2 volte, a 5 minuti ciascuno.
- Fissare in paraformaldeide al 4% per 15 minuti a RT.
- Risciacquare due volte con D-PBS, 5 min.
3. Visualizzazione embrioni Stained LysoTracker o Culture ES
- Preparare i campioni di cellule per microscopia aspirando la D-PBS, rimuovere la camera dal vetrino, aria di essiccazione per circa 5 minuti, e quindi aggiungendo un mezzo acquoso di montaggio (Vectashield con DAPI o simile). Inserire un foglio di barriera, mentre drying per diminuire sbiancamento del fluoroforo. Montare gli embrioni in una profonda depressione, piatto di vetro o piccola scatola di Petri in Vectashield o simili.
- Visualizza con un microscopio da dissezione o un composto dotato di un rodamina o Texas Red filtro (DND LysoTracker RED-99, di eccitazione / emissione: 577/590 nm). La colorazione è in genere molto luminoso così lunghe esposizioni di solito non sono necessari.
4. Quantificazione dei risultati utilizzando tecniche di imaging
- Scatta foto digitali dei campioni nelle condizioni di esposizione in modalità manuale.
- Aprire le immagini in Adobe Photoshop o un programma di imaging simile. Per determinare la quantità di colorazione LysoTracker, selezionare il canale rosso e l'immagine di soglia (converte l'immagine in bianco e nero) tale che la colorazione rossa è ora rappresentato da pixel bianchi. Selezionare i pixel bianchi e utilizzare la funzione istogramma per ottenere il conteggio totale.
- Utilizzare il canale blu per contareil numero di nuclei nel campo. Può essere utile per tenere traccia del conteggio annotando l'immagine.
- Registrare i valori e quindi calcolare il livello medio di colorazione per cella. Ripetere questa analisi con l'impostazione stessa soglia per tutte le immagini che si desidera assaggiare.
5. Risultati rappresentativi
Vi è un normale modello di PCD durante lo sviluppo embrionale, che può cambiare quando un gene del fattore di crescita, come Sonic hedgehog (Shh), viene rimosso 16,17. Poiché è un colorante LysoTracker piccola molecola che è altamente diffusibile, l'embrione intero può essere visualizzato per valutare le aree di PCD anche in profondità all'interno del mesenchima 12,13 Figura 1 mostra lo schema di colorazione per LysoTracker normali e Shh -. / - Embrioni di topo mutante a 10.5dpc. In questa fase, PCD è notevolmente aumentata in Shh - / - embrioni mutanti, in particolare l'arto e somite regions. Porzioni di l'embrione può essere ripreso per intero il montaggio a maggiore ingrandimento (1B) per valutare la configurazione generale. Porzioni sezionato può anche essere sezionato con un microtomo vibrante (1C) o un criostato (DF) per rivelare regioni PCD in maggiore dettaglio.
Analisi TUNEL è fatto meglio su sezioni sottili in cui la penetrazione della transferasi terminale è meno problematica. LysoTracker colorazione e un saggio TUNEL può essere fatto nel tessuto stesso (Figura 1D-G). Questi risultati mostrano che, anche se non c'è una stretta correlazione tra LysoTracker 01:01 e colorazione TUNEL, le regioni generali di sovrapposizione PCD notevolmente. Con un ingrandimento superiore, è possibile vedere che puncta colorazione positiva con il saggio TUNEL, ma non con il colorante LysoTracker, puncta che macchia solo con il colorante LysoTracker, e che sono sovrapposti puncta colorazione.
Quando un EB è placcato su una superficie rivestita di gelatina, le cellule si differenziano Migrate verso l'esterno. Dopo 10 giorni di coltura, le cellule si sono differenziate in una varietà di tipi di cellule inclusi neuroni, cellule muscolari, e cardiomiociti. Le culture mostrati in Figura 2 contengono popolazioni miste di questi tipi di cellule, tuttavia si potrebbe migliorare il nostro protocollo per studiare PCD di un particolare tipo cellulare di interesse. Per esempio, la EB potrebbe essere trattata con acido retinoico per migliorare la differenziazione dei neuroni 19, e quindi il saggio LysoTracker potrebbe essere utilizzato per valutare morte neuronale in condizioni diverse. Nella coltura di cellule staminali normali differenziate, ci sono alcune cellule che subiscono PCD e questi possono essere rilevati con LysoTracker e / o saggi TUNEL (Figura 2A). PCD può essere indotta da una serie di diversi agenti citotossici come perossido di idrogeno (H 2 O 2) che induce danno ossidativo e avvia la via apoptotica 20,21. Una bassa dose di H 2 O 2 aumenta siaLysoTracker e colorazione TUNEL rispetto a nessun trattamento, mentre un risultato ad alto dosaggio in restringimento delle cellule e LysoTracker diffusa e positività TUNEL. Simile ai risultati nell'embrione, è possibile vedere che puncta colorazione positiva con il saggio TUNEL, ma non con il colorante LysoTracker, puncta che macchia solo con il colorante LysoTracker, e che sono sovrapposti puncta colorazione. La conseguenza dell'applicazione di un agente citotossico che induce apopotosis viene confrontato condizioni inedia del siero che inducono autofagia (Figura 2D). Le cellule che hanno subito un periodo di inedia del siero (1 ora) presentano un modello diverso in cui LysoTracker e colorazione TUNEL non altamente correlati. LysoTracker colorazione è molto maggiore rispetto alle culture non trattate, tuttavia positività TUNEL è a livelli normali.
L'impiego di strumenti standard disponibili in Adobe Photoshop, colorazione LysoTracker può essere quantificata con un metodo soglia sempliceche permette di calcolare il livello medio di colorazione per una popolazione di cellule. Figura 3A-B mostra come una immagine campione appare durante il processo di quantificazione. Partendo con un'immagine di base con colorazione LysoTracker nel canale rosso e il DAPI-tinto nuclei nel canale blu (pannello 3A), il canale rosso di questa immagine è stata thresholded per convertire l'immagine in bianco e nero ed i pixel bianchi erano poi selezionati e contati (Figura 3A '). Un livello di soglia è stato scelto che rappresenta il pattern di colorazione LysoTracker. Come con qualsiasi metodo di quantificazione che utilizza le immagini, è importante evitare di sovraesporre le foto in quanto ciò potrebbe potenzialmente causare un eccessivo accento sulle differenze. Il numero di nuclei nel campo può essere contato guardando solo il canale blu (Figura 3B) e la marcatura dei nuclei utilizzando un attrezzo di disegno come un pennello, mentre il conteggio (Figura 3B '). Si può quindi esprimere °livello di colorazione e calcolando il livello medio di colorazione per cella (numero di pixel / conteggio nuclei). Se si vuole utilizzare questo algoritmo per alta through-put screening, la maggior parte dei pacchetti di screening può misurare la luminosità in un particolare canale e hanno anche una funzione di identificatore di oggetto che conterà nuclei in un campo, tuttavia per le applicazioni più tipiche il metodo manuale è veloce e semplice. Per fare un esempio, una regione campione da figure 2A-C è stata quantificata ed i risultati confermano che aumentare le concentrazioni di H 2 O 2 risultati in un più alto livello di colorazione LysoTracker (Figura 3C).
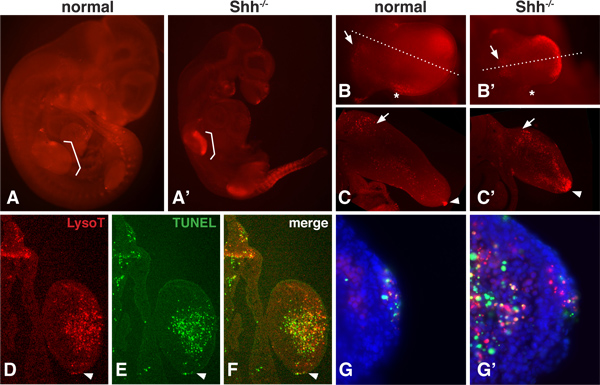
Figura 1. Colorazione LysoTracker in normali e Shh - embrioni - /. A, A 'Normale e Shh -. / - Embrioni di topo a 10,5 dpc, macchiato di rosso LysoTracker, montato in Vectashield e fotografato sottoepiflourescence un microscopio da dissezione. Parentesi indicano la posizione degli arti anteriori B, B 'zampa anteriore di normali e Shh -.. / - Embrioni ripreso a maggiore ingrandimento. Nota: i domini normali di morte cellulare compresa la 'Zona posteriore necrotico' (contrassegnati da un asterisco) e una regione nel mesenchima prossimale della gemma dell'arto (contrassegnati da una freccia). Arti nulli Shh esporre più forte colorazione nella parte mediale e distale . C, C '. sezioni mediali attraverso la gemma delle gemme degli arti arti anteriori normali e null. Si noti la presenza di colorazione nel mesenchima prossimale (freccia) e AER (punta di freccia) della gemma dell'arto normale e il forte colorazione all'interno del mesenchima dorsale e ventrale e distale all'interno della gemma dell'arto nullo. DF. Nella stessa sezione (12 micron, congelati sezione) è stato ripreso per LysoTracker e colorazione TUNEL (Roche 1684795). La sezione passa attraverso la porzione distale del nulla Shh mesenchyme. I punti di punta di freccia alla AER. Si noti come il modello generale è molto simile con entrambi i metodi, tuttavia la colorazione non si sovrappone completamente. G, G '. Un ingrandimento maggiore dell'ARE e mesenchima, si può vedere sia la presenza di puncta che sono LysoTracker o TUNEL-positive Solo così come puncta che sono sovrapposti colorazione.

Figura 2. Colorazione LysoTracker in cellule ES differenziate. Differenziare le cellule sono state piastrate su vetrini 8-camere in assenza di LIF. Per mostrare come LysoTracker e colorazione TUNEL confrontare, il percorso apoptosi è stata indotta con due diverse dosi di H 2 O 2, mentre la via autofagia è stata indotta utilizzando 1 ora di inedia del siero. Queste immagini sono state scattate con un microscopio confocale Zeiss LSM5 A-A'':. Colture non trattate mostrare un po 'normale apoptosi come indicato da LysoTRacker e colorazione TUNEL. Il modello con entrambe le tecniche per lo più sovrapposizioni (frecce bianche), tuttavia, si può vedere un puncta alcuni che sono LysoTracker (frecce rosse) positivi solo B-B'', CC ":. Trattamento con quantità crescenti di H 2 O 2 stimola l'apoptosi . La dose più bassa mostra più LysoTracker e TUNEL puncta positivo rispetto alle cellule non trattate. La dose più alta in LysoTracker induce ancora di più e puncta TUNEL positivi. bianco frecce alle macchie sovrapposte, mentre le frecce rosse scegliere puncta che sono positivi per LysoTracker solo. È anche possibile vedere puncta che sono solo positivo per la colorazione TUNEL (frecce verdi) DD ":. inedia del siero può indurre autofagia, che può stimolare un accumulo di materiale in lisosomi e autophagolysosomes. Nota: il numero di puncta LysoTracker più sono visibili rispetto alle culture non trattati. TUNEL positivi puncta sono presenti (la maggior parte dei quali si sovrappongono LysoTracker macchiazione, frecce bianche), tuttavia, il numero non è notevolmente aumentato rispetto a condizioni non trattati. Inoltre, in condizioni di fame siero, LysoTracker solo puncta positivi sono molto diffusi (frecce rosse).
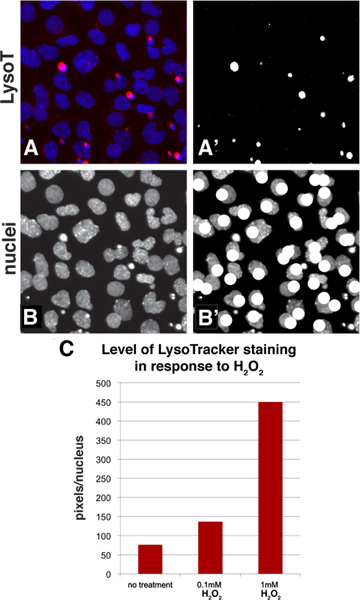
Figura 3. Metodo di quantificazione per valutare il livello di colorazione in una popolazione di cellule utilizzando Adobe Photoshop. A: Una porzione di figura 2A è stato scelto e il LysoTracker (canale rosso) e colorazione DAPI (nuclei, blu canale) è mostrato in questo pannello A ':. Il canale rosso della figura 3A è thresholded per convertirlo in un bianco e immagine in bianco. I pixel bianchi possono essere selezionati e contati utilizzando la funzione istogramma B-B ':. Il canale blu mostra i nuclei e mentre loro conteggio, l'immagine può essere annotati con una funzione di disegno quale un pennello C:. Questo è un exquantificazione ampia di un campione da pannelli 2A-C. I dati sono rappresentati come pixel per nuclei. Clicca qui per ingrandire la figura .
Discussione
Caratteristiche importanti della apoptosi comprendono il rilevamento di danno al DNA con il saggio TUNEL, insieme con la rilevazione dell'attività della caspasi (rilevata con anticorpi monoclonali o una molecola legante come ZVAD-fmk), l'osservazione di cambiamenti cellulari, e la presentazione di fosfatidilserina (PS ) sulla superficie della membrana (rilevata da annessina V vincolante). Ci sono alcuni svantaggi con ciascuno di questi saggi. Per esempio, la transferasi terminale utilizzato nel saggio TUNEL non...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Ringraziamo i membri del laboratorio Mariani aiuto la modifica del protocollo. Questo lavoro è stato finanziato da una sovvenzione di formazione post-dottorato CIRM (JLF), uno stage CIRM BRIDGES (TZTT), E. Robert e maggio R. Wright Foundation (FVM), e la University of Southern California (FVM).
Materiali
| Name | Company | Catalog Number | Comments | ||||||||||||||
| Nome del reagente | Azienda | Numero di catalogo | |||||||||||||||
| LysoTracker Red DND-99 | Invitrogen | # L-7528 | |||||||||||||||
| Hanks BSS | Invitrogen | 14025-076 | |||||||||||||||
| Paraformaldeide | EMD | EM-PX0055-3 | |||||||||||||||
| Vectashield | VECTOR | H-1200 | |||||||||||||||
| DMEM | Cellgro | 10-013-CV | |||||||||||||||
| Aminoacidi non essenziali | Cellgro | 25-025-CI | |||||||||||||||
| Sodio piruvato | Cellgro | 25-000-CI | |||||||||||||||
| FBS | Hyclone | SH30071.02 | |||||||||||||||
| Pen-Strep | Invitrogen | 15140-122 | |||||||||||||||
| b-mercaptoetanolo, 50 mM | Invitrogen | 21985-023 | |||||||||||||||
| Labtek-II Camera diapositive (8 pozzetti) | Nalge Nunc internazionale | 154534 | |||||||||||||||
| 0,1% gelatina | Millipore | ES-006-B | |||||||||||||||
| PBS di Dulbecco (D-PBS) | Cellgro | 21-031-CV | |||||||||||||||
Soluzione Ricette Paraformaldeide al 4% Per 100 ml:
AVVERTENZA: Paraformaldeide in forma 'frill' (compresso piccoli pellet) è meno polverosa e può quindi essere misurata al di fuori di un cappuccio. Tuttavia si deve ancora indossare una maschera protettiva antipolvere (N95almeno) durante la movimentazione. EB Cultura Media Per 500 ml EB Media:
|
Riferimenti
- Baehrecke, E. H. How death shapes life during development. Nat. Rev. Mol. Cell Biol. 3, 779-787 (2002).
- Meier, P., Finch, A., Evan, G. Apoptosis in development. Nature. 407, 796-801 (2000).
- Ikonomidou, C. Ethanol-induced apoptotic neurodegeneration and fetal alcohol syndrome. Science. 287, 1056-1060 (2000).
- Dunty, W. C., Chen, S. Y., Zucker, R. M., Dehart, D. B., Sulik, K. K. Selective vulnerability of embryonic cell populations to ethanol-induced apoptosis: implications for alcohol-related birth defects and neurodevelopmental disorder. Alcohol Clin. Exp. Res. 25, 1523-1535 (2001).
- Price, O. T., Lau, C., Zucker, R. M. Quantitative fluorescence of 5-FU-treated fetal rat limbs using confocal laser scanning microscopy and Lysotracker. Cytometry A. 53, 9-21 (2003).
- Jacobson, M. D., Weil, M., Raff, M. C. Programmed cell death in animal development. Cell. 88, 347-354 (1997).
- Fuchs, Y., Steller, H. Programmed cell death in animal development and disease. Cell. 147, 742-758 (2011).
- Qu, X. Autophagy gene-dependent clearance of apoptotic cells during embryonic development. Cell. 128, 931-946 (2007).
- Grimsley, C., Ravichandran, K. S. Cues for apoptotic cell engulfment: eat-me, don't eat-me and come-get-me signals. Trends Cell Biol. 13, 648-656 (2003).
- Lauber, K., Blumenthal, S. G., Waibel, M., Wesselborg, S. Clearance of apoptotic cells: getting rid of the corpses. Mol. Cell. 14, 277-287 (2004).
- Haller, T., Dietl, P., Deetjen, P., Volkl, H. The lysosomal compartment as intracellular calcium store in MDCK cells: a possible involvement in InsP3-mediated Ca2+ release. Cell Calcium. 19, 157-165 (1996).
- Zucker, R. M., Hunter, S., Rogers, J. M. Confocal laser scanning microscopy of apoptosis in organogenesis-stage mouse embryos. Cytometry. 33, 348-354 (1998).
- Zucker, R. M., Hunter, E. S., Rogers, J. M. Apoptosis and morphology in mouse embryos by confocal laser scanning microscopy. Methods. 18, 473-480 (1999).
- Gavrieli, Y., Sherman, Y., Ben-Sasson, S. A. Identification of programmed cell death in situ via specific labeling of nuclear DNA fragmentation. J. Cell Biol. 119, 493-501 (1992).
- Foty, R. A Simple Hanging Drop Cell Culture Protocol for Generation of 3D Spheroids. J. Vis. Exp. (51), e2720 (2011).
- Chiang, C. Manifestation of the limb prepattern: limb development in the absence of sonic hedgehog function. Dev. Biol. 236, 421-435 (2001).
- Ishibashi, M., McMahon, A. P. A sonic hedgehog-dependent signaling relay regulates growth of diencephalic and mesencephalic primordia in the early mouse embryo. Development. 129, 4807-4819 (2002).
- Schuldiner, M. Induced neuronal differentiation of human embryonic stem cells. Brain Res. 913, 201-205 (2001).
- Bain, G., Kitchens, D., Yao, M., Huettner, J. E., Gottlieb, D. I. Embryonic stem cells express neuronal properties in vitro. Dev. Biol. 168, 342-357 (1995).
- Dumont, A. Hydrogen peroxide-induced apoptosis is CD95-independent, requires the release of mitochondria-derived reactive oxygen species and the activation of NF-kappaB. Oncogene. 18, 747-757 (1999).
- Hampton, M. B., Orrenius, S. Dual regulation of caspase activity by hydrogen peroxide: implications for apoptosis. FEBS Lett. 414, 552-556 (1997).
- Wood, W. Mesenchymal cells engulf and clear apoptotic footplate cells in macrophageless PU.1 null mouse embryos. Development. 127, 5245-5252 (2000).
- Scott, R. C., Juhasz, G., Neufeld, T. P. Direct induction of autophagy by Atg1 inhibits cell growth and induces apoptotic cell death. Curr. Biol. 17, 1-11 (2007).
- Rodriguez-Enriquez, S., Kim, I., Currin, R. T., Lemasters, J. J. Tracker dyes to probe mitochondrial autophagy (mitophagy) in rat hepatocytes. Autophagy. 2, 39-46 (2006).
- Bampton, E. T., Goemans, C. G., Niranjan, D., Mizushima, N., Tolkovsky, A. M. The dynamics of autophagy visualized in live cells: from autophagosome formation to fusion with endo/lysosomes. Autophagy. 1, 23-36 (2005).
- Boya, P., Mellen, M. A., de la Rosa, E. J. How autophagy is related to programmed cell death during the development of the nervous system. Biochem. Soc. Trans. 36, 813-817 (2008).
- Galluzzi, L. Guidelines for the use and interpretation of assays for monitoring cell death in higher eukaryotes. Cell Death Differ. 16, 1093-1107 (2009).
- Klionsky, D. J. Guidelines for the use and interpretation of assays for monitoring autophagy in higher eukaryotes. Autophagy. 4, 151-175 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon