Method Article
Pyrosequencing para la identificación microbiana y caracterización
En este artículo
Resumen
Pirosecuenciación es una técnica versátil que facilita la secuenciación del genoma microbiano que se puede utilizar para identificar las especies bacterianas, discriminar las cepas bacterianas, y detectar las mutaciones genéticas que confieren resistencia a los agentes anti-microbianos. En este vídeo, se demostrará el procedimiento para la generación de amplicón microbiana, amplicón pirosecuenciación, y el análisis de secuencia de ADN.
Resumen
Pirosecuenciación es una técnica versátil que facilita la secuenciación del genoma microbiano que se puede utilizar para identificar las especies bacterianas, discriminar las cepas bacterianas y detectar las mutaciones genéticas que confieren resistencia a los agentes anti-microbianos. Las ventajas de pirosecuenciación para aplicaciones de microbiología incluyen cribado de alto rendimiento rápido y fiable y precisa identificación de los microbios y las mutaciones del genoma microbianas. Pyrosequencing implica la secuenciación de ADN mediante la síntesis de la cadena complementaria de una sola base en un momento, mientras que la determinación del ser específica de nucleótidos incorporados durante la reacción de síntesis. La reacción se produce en inmovilizada plantilla de ADN de cadena sencilla en donde se añaden los cuatro desoxirribonucleótidos (dNTP) secuencialmente y los dNTPs no incorporados se degrada enzimáticamente antes de la adición de la siguiente dNTP a la reacción de síntesis. Detección de la base específica incorporado a la plantilla se controla por la generación de chemilumseñales inescent. El orden de dNTPs que producen las señales quimioluminiscentes determina la secuencia de ADN de la plantilla. La capacidad de secuenciación en tiempo real de la tecnología de pirosecuenciación permite la rápida identificación microbiana en un único ensayo. Además, el instrumento pirosecuenciación, puede analizar la diversidad genética completa de la resistencia a los medicamentos anti-microbiana, incluida la tipificación de SNPs, mutaciones puntuales, inserciones y deleciones, así como la cuantificación de múltiples copias de genes que pueden producirse en un poco de resistencia antimicrobiana patrones.
Introducción
Pirosecuenciación es un método rápido y preciso para la secuencia de ácidos nucleicos que se basa en el principio de "secuenciación por síntesis". "La secuenciación por síntesis" implica el uso de una sola hebra molde de ADN para sintetizar la cadena complementaria de una base a la vez, y la detección de la base incorporada (A, T, G o C) en cada paso por la detección de una señal quimioluminiscente. La reacción pirosecuenciación incluye ADN molde, los dNTP (dATP, dGTP, dTTP, dCTP), ADN polimerasa, ATP sulfurilasa, luciferina, luciferasa, y apirasa. La reacción de síntesis se inició mediante la adición de uno de los cuatro dNTPs y ADN polimerasa a la plantilla de ADN biotinilado de cadena sencilla. ADN polimerasa incorpora el dNTP complementario en la plantilla, con la posterior liberación de pirofosfato (Figura 1). ATP sulfurilasa convierte proporcionalmente el pirofosfato de ATP. ATP actúa como un catalizador para la conversión mediada por la luciferasa de la luciferina a oxiluciferina, la cualgenera luz (Figura 2). La intensidad de la luz es proporcional al número de nucleótidos incorporados y determina si está presente en la cadena molde de forma secuencial (Figura 3) uno o más de dNTP específica (dATP, dTTP, dGTP, dCTP o). Cualquier dNTP no incorporada se degrada por apirasa antes de la adición de la siguiente dNTP para la continuación de la reacción de síntesis. Este "secuenciación por síntesis" reacción se repitió con la adición de cada uno de los cuatro dNTPs hasta que se determine la secuencia de ADN de la plantilla de ADN de cadena única.
Para ver una animación de la reacción pirosecuenciación Haga clic aquí para ver la película .
Protocolo
Introducción: Una sola colonia bacteriana se debe utilizar para inocular un caldo de cultivo apropiado que se incubó durante la noche. Un sedimento bacteriano se procesa a continuación para purificar ADN genómico usando un kit de aislamiento genómico disponibles comercialmente. El ADN genómico purificado debe tener una relación de 260/280 (nm)> 1,8 para asegurar que la muestra está libre de contaminación de proteína. La cantidad de ADN genómico se requiere para la generación de la plantilla de pirosecuenciación es de 10-20 ng.
A. Ampliación de la plantilla
(Manual de PCR PyroMark; www.qiagen.com / manuales 1)
Configuración de la reacción de PCR
Nota: Un cebador debe biotinilado en su extremo 5 ', y se recomienda que sea HPLC purificada para la preparación de la plantilla. Recomendamos el PyroMark Ensayo Diseño de Software 2.0 para PCR y SEprimer diseño cuencia que están optimizados para la secuenciación de corta lectura, sin embargo, Primer-BLAST ( http://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome ) de NCBI también puede ser utilizado para el diseño del cebador.
- Descongelar todas las soluciones requeridas (enumerados en la Tabla 1) y mezclar a fondo cada uno. Configurar la reacción de acuerdo con la Tabla 1. Todo el trabajo se puede realizar a temperatura ambiente.
Nota: La mezcla maestra de PCR contiene MgCl 2 para una concentración final de 1,5 mM, que proporciona resultados satisfactorios en la mayoría de los casos. Si un mayor Mg 2 + de concentración se requiere, hasta 3,5 l de 25 mM de MgCl 2 se puede añadir a una reacción.
- Mezclar bien la mezcla de reacción con la pipeta suavemente hacia arriba y hacia abajo, luego dispensar volúmenes adecuados en tubos de PCR.
- Añadir template ADN a cada tubo de PCR. Recomendamos 10 ng de ADN genómico.
- Programe el termociclador de acuerdo a la Tabla 2. Un mantenimiento final a 4 º C se recomienda para la conveniencia. Si está utilizando un termociclador sin tapa térmica, las reacciones de superposición con 100 l de aceite mineral.
- Coloque los tubos de PCR en el termociclador e iniciar el programa de ciclismo.
B. Vacío Protocolo de estación de trabajo
La inmovilización de los productos de PCR biotinilados
- Hacer una mezcla maestra de acuerdo con la Figura 4.
Nota: Antes de pipeteado, agitar suavemente la botella de perlas de Sepharose recubiertas con estreptavidina para asegurar una suspensión homogénea.
- Dependiendo del volumen de producto de PCR utilizado, dispensar 60-75 mezcla maestra l en cada pocillo de una placa de PCR para dar un volumen total de 80 l por pocillo.
- Añadir 5-20 l producto de PCR biotinilado a cada pocillo.
- Selle los pocillos con cinta caps y agitar la placa de PCR a 1400 rpm durante 5-10 minutos a temperatura ambiente (15-25 ° C) utilizando un agitador orbital.
La desnaturalización de ADN y la adición de cebador de secuenciación
- Diluir los cebadores de secuenciación a 0,3 mM con tampón de reasociación y dispensar 25 l en cada pocillo de una placa de reacción. Coloque la placa en la estación de trabajo.
- Llenar los comederos de estaciones de trabajo de acuerdo con el diagrama de la figura 5.
- Encienda la bomba de vacío, y asegúrese de que el interruptor de la herramienta de vacío está activada (Figura 6). Enjuague la sonda de filtro con agua de alta pureza (Milli-Q 18,2 MW x cm o equivalente) en el canal 5 (Figura 5). Vuelva a llenar la cubeta con agua de alta pureza fresca para su uso en el paso 12.
- Trabajando rápidamente, coloque la placa de PCR con los granos en la estación de trabajo. Asegúrese de que ambas placas están en la misma orientación que cuando se cargaron las muestras.
- Con el interruptor de vacío conectado, reduzca el vacíoherramienta en los pocillos de la placa de PCR de 20 segundos o hasta que todo el volumen ha sido aspirado. Las perlas serán capturadas por la herramienta de vacío (Figura 7).
- Con el vacío todavía en ON, la herramienta de enjuague con etanol al 70% (a través de 1) durante 20 segundos o hasta que todo el volumen ha sido aspirado.
- Con vacío conectado, lave la herramienta con una solución de desnaturalización (canal 2) durante 20 segundos o hasta que el volumen se ha aspirado.
Nota: La solución de desnaturalización contiene hidróxido de sodio, que es un irritante de la piel y los ojos. Siempre use una bata de laboratorio, guantes y gafas de protección.
- Con vacío conectado, lave la herramienta con tampón de lavado (cubeta 3) durante 20 segundos o hasta que el volumen se ha aspirado.
- Con vacío conectado, levante la herramienta de vacío a más de 90 ° en vertical durante 5 segundos, para permitir que todo el líquido se escurra fuera de la herramienta de vacío. Apague la bomba y alinear la herramienta de vacío con la placa de reacción. Mueva la herramienta de vacío en elpozos y agitar suavemente de un lado a otro para liberar las perlas en el cebador de secuenciación que contiene pozos.
- Con el fin de limpiar la herramienta de vacío, agitar las sondas de filtro en agua de alta pureza (canal 4) para 10 seg.
- Encienda la aspiradora y limpiar la herramienta con agua de alta pureza (canal 5) durante 20 segundos o hasta que el volumen se ha aspirado.
- Levante la herramienta de vacío más allá de 90 ° C vertical, durante 5 segundos, luego cambia el vacío OFF y almacenar en la posición de "estacionamiento".
Recocido cebador de secuenciación a hebras de ADN
- Coloque la placa de reacción en un soporte de la placa precalentada.
- Calentar la placa en un bloque de calentamiento a 80 ° C durante 2 min.
- Retire la placa del soporte y el lugar en el mostrador para enfriar a temperatura ambiente durante 5 min.
PyroMark Q24 Operación
Nota: Encienda el instrumento con el interruptor de alimentación situado por encima del cable de alimentación. Estrellatup puede tardar hasta 1 minuto.
- Rellenar un cartucho según las instrucciones proporcionadas en el PyroMark Oro Q24 Reactivos Manual 2. Para determinar los volúmenes necesarios, configurar un archivo de ejecutar en el software del aparato, y en el menú Herramientas, seleccione Información Run Pre
- Si el instrumento ya estaba en marcha, asegurarse de que no se está realizando una carrera y luego abra la tapa. Una señal acústica se emite si la tapa se abre cuando no es seguro hacerlo.
- Abra la puerta del cartucho e inserte el cartucho con la etiqueta hacia arriba (Figura 8). Asegúrese de que el cartucho se ha insertado correctamente (una línea debe ser visible en la parte delantera) y cierre la puerta.
- Abra el marco de la placa de sujeción y la posición de la placa con las muestras en el interior del instrumento.
- Cierre el marco de la placa de sujeción y la tapa del instrumento (Figura 9).
Inicio de una carrera
- Inserte la memoria USB con el archivo de ejecucións creado con el software del instrumento en el puerto USB en la parte frontal del instrumento.
- Seleccione "Ejecutar" en el menú principal y pulse el botón "OK".
- Utilice las flechas de desplazamiento para seleccionar el archivo de ejecución deseado y pulse "Seleccionar".
Nota: El instrumento comenzará a dispensar reactivos cuando se alcanzan toda la presión preestablecida y temperatura (esto puede tardar varios minutos). Durante una ejecución, la pantalla muestra el instrumento de pirograma el pozo seleccionado en tiempo real. Utilice las flechas de desplazamiento para ver el pirograma de otros pozos.
Completar una carrera
- Cuando el instrumento confirma que ha terminado la carrera y el archivo de ejecución se ha guardado en la memoria USB, pulse el botón "Cerrar".
- Retire el dispositivo USB. Si se retira antes de que termine la carrera, inserte la memoria USB. Seleccione "Administración" y luego "Copiar Runs no guardados".
- El archivo de ejecutar ahora se puede abrir y se analizó usando el software del instrumento y se comparaa una biblioteca de microbios conocidos usando el software Identifire.
Resultados
Los resultados de una ejecución típica pirosecuenciación usando el software muestra la ubicación de los cebadores directo e inverso usados para la generación de amplicón, y el cebador de secuenciación utilizado para la reacción pirosecuenciación dentro de la secuencia 16S ribosómica conservado (Figura 10). La secuencia hipervariable entre el cebador permite a los sitios para la identificación de bacterias de un gran número de especies bacterianas utilizando el cebador de secuenciación conservada (5'-TACATGCAAGTCGA). Como un control de calidad interno, la secuencia de ADN horquillado la región variable puede ser comprobado para asegurar que la región de ADN correcta se ha analizado . El software de pirosecuenciación permite la comparación y la alineación de la secuencia generada a una base de datos interna de secuencias ribosómicas bacterianas para la identificación bacteriana. Además, una secuencia puede ser analizada para determinar las mutaciones que confieren resistencia a los antibióticos. Por ejemplo, el análisis de mutaciones en los genes ribosómicos 23S de Helicobpylori rácter muestra dos patrones de mutación (GAA o AGA) que confieren resistencia a los antibióticos (Figura 11). Análisis de múltiples aislados de la misma paciente puede ser ensayada simultáneamente para realizar un seguimiento de la aparición de resistencia a los medicamentos en el tiempo para ayudar en las investigaciones epidemiológicas de brotes de resistencia a fármacos microbianos.
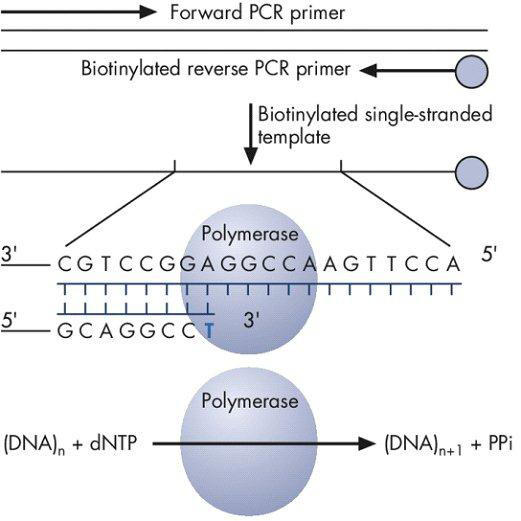
Figura 1. Producto de PCR biotinilado se utiliza como una plantilla para incorporar dNTPs por la ADN polimerasa, lo que lleva a la generación de pirofosfato (PPi).
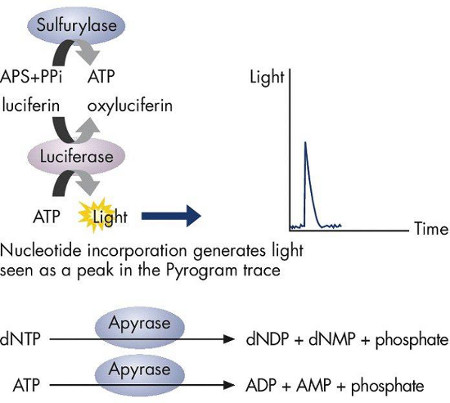
Figura 2. ATP sulfurilasa convierte proporcionalmente pirofosfato de ATP. Actos ATP como un catalizador para la conversión mediada por la luciferasa de la luciferina a oxiluciferina, la cual genera luz que es Proproporcional a la cantidad de ATP. La luz se registra como máximo en la traza pirograma e indica la incorporación de nucleótidos. Los dNTPs no incorporados son degradados por apirasa antes de añadir el siguiente dNTP para la continuación de la síntesis.
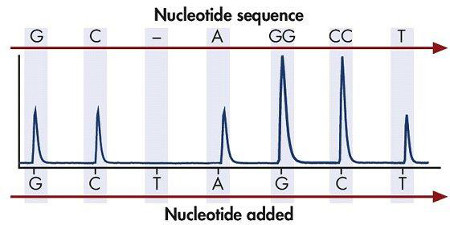
Figura 3. La intensidad de la luz generada indica si se incorpora una o más de dNTP específica (dATP, dTTP, dGTP, dCTP o) sobre la cadena molde secuencialmente.
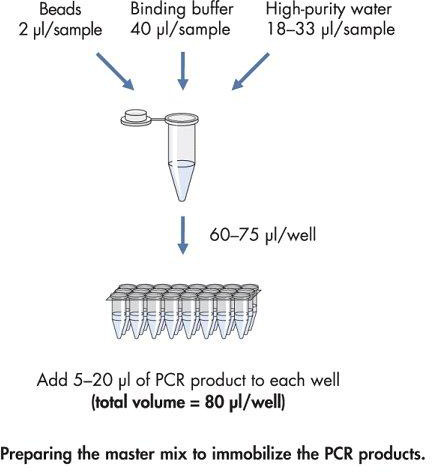
La Figura 4. Diagrama de flujo para la preparación de la mezcla maestra para inmovilizar el producto de PCR biotinilado.
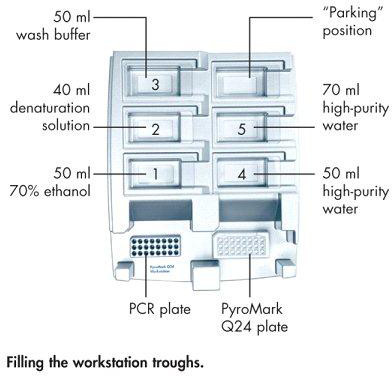
ge = "always"> Figura 5. PyroMark estación de trabajo, con la placa de PCR, placa PyroMark y lugares mínimas.

La Figura 6. Localización del interruptor de vacío posiciones ON y OFF.
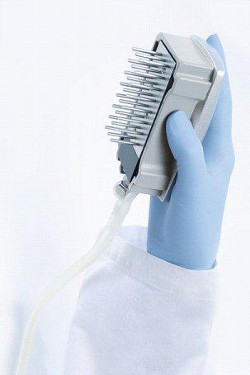
Figura 7. Herramienta de vacío. Manejo adecuado de la herramienta de vacío.

Figura 8. Abrió la puerta del cartucho con el cartucho en su lugar.

La Figura 9. Cartucho insertado correctamente con la puerta cerrada.
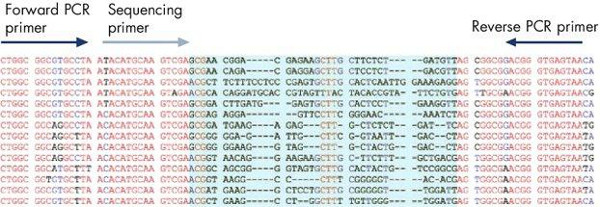
La Figura 10. Pyrosequencing resultados de la identificación de bacterias basados en. Cebadores de la PCR están diseñados para regiones conservadas de la plantilla de ADN, y el cebador de secuenciación se coloca inmediatamente aguas arriba de la identificación de una secuencia de ADN hipervariable bien caracterizado en el amplicón (que se muestra en azul).
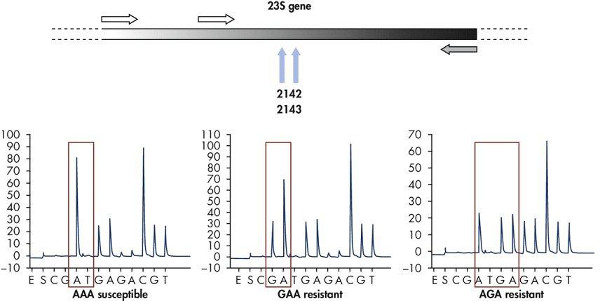
La Figura 11. La detección de la resistencia a los antibióticos en Helicobacter pylori mediante pirosecuenciación. Análisis de mutaciones en los genes 23S que confieren resistencia antibacteriana en Helicobacter pylori contiene GAA o secuencias de AGA. Haz clic aquí para ver más grande la figura .
| Componente | Volumen por reacción | La concentración final |
| Pyro PCR Master Mix, 2x | 12,5 l | 1x |
| Concentrado CoralLoad, 10x | 2,5 l | 1x |
| 25 mM MgCl 2 (opcional) | Variable | ≥ 1,5 mM |
| Q-Solution, 5x (opcional) | 5 l | 1x |
| Primer A / Primer B | Variable / Variable | 0.2 μM/0.2 mM |
| RNasa libre de agua | Variable | - |
| Volumen Total (después de añadir ADN molde) | 25 l |
Tabla 1. Mezcla de reacción de PCR.
| Y nbsp; | Comentarios adicionales | ||
| Etapa de activación inicial de PCR | 15 min | 95 ° C | HotStartTaq ADN polimerasa se activa |
| 3 etapa de ciclo: desnaturalización | 30 seg | 94 ° C | |
| Recocido | 30 seg | 60 ° C 56 ° C | Para el ADN genómico Para bisulfito de ADN convertido |
| Extensión | 30 seg | 72 ° C | |
| Número de ciclos | 45 | ||
| La extensión final | 10 min | 72 ° C |
Tabla 2. Especificaciones del ciclo de PCR.
Discusión
Varios de los nuevos métodos de secuenciación de nueva generación se han comercializado. Tres de los métodos más ampliamente utilizados son la secuenciación de iones de semiconductores, la secuenciación de una sola molécula en tiempo real, y la secuenciación por síntesis (SBS). 3-11 Cada uno de estos métodos depende de secuenciación por síntesis, pero emplean plataformas novedosos para la detección de los nucleótidos incorporados. En la secuenciación de semiconductores de iones, una sola hebra de ADN sirve como molde para la secuenciación de hebra. 12 polimerasa y los nucleótidos se añaden secuencialmente como en pirosecuenciación. Cuando se añade un nucleótido a la cadena de ADN creciente, se libera un ion de hidrógeno. El ion hidrógeno es detectado por un sensor basado en transistor de efecto de campo.
Secuenciación en tiempo real molécula individual depende de la guía de ondas modo cero (ZMW). 13 El ZMW es una pequeña estructura en la que una sola molécula de polimerasa se une a la parte inferior del pozo. Se ilumina de una manera tal que una floremolécula de olor puede ser detectado. Cada nucleótido está etiquetado con una molécula fluorescente. En cuanto se incorpora un nucleótido marcado con fluorescencia, detector identifica el nucleótido. Cuando se añade el siguiente nucleótido, la molécula fluorescente se escinde. 14
La secuenciación por síntesis (SBS) utiliza un método único para amplificar el ADN diana de tal manera que grupos de secuencias únicas se generan. 15 plantillas de cadena sencilla se generan y luego se sintetiza la hebra complementaria. Cada nucleótido está marcado con una molécula fluorescente y después de cada adición de base se registra la fluorescencia de la base añadida.
Divulgaciones
La producción y el libre acceso a este artículo es patrocinado por Qiagen.
Autores Ahmed, Durocher, Jessen y Vardi son empleados de Qiagen Inc., que produce reactivos e instrumentos utilizados en este artículo.
Agradecimientos
Este proyecto se apoyó en parte por la Universidad Johns Hopkins, Oficina del Rector a través de la Iniciativa Científica Gateway y Qiagen Inc.
Materiales
| Name | Company | Catalog Number | Comments |
| PyroMark PCR Master Mix, 2x | Qiagen | 978703 | |
| CoralLoad Concentrate, 10x | Qiagen | 978703, 978705 | |
| Q-Solution, 5x | Qiagen | 978703, 978705 | |
| MgCl2, 25 mM | Qiagen | 978703, 978705 | |
| RNase-Free Water | Qiagen | 129112 | |
| Primers | Qiagen | 978776 or 978777 | Primers should be purchased from an established oligonucleotide manufacturer (i.e. QIAGEN Pyromark Custom Assays, IDT, etc). Lyophilized primers should be dissolved in TE to provide a stock solution of 100 μM. |
| Pyromark Gold Q24 Reagents | Qiagen | 970802 | |
| High-purity water (Milli-Q 18.2 MΩ x cm or equivalent) | |||
| Streptavidin Sepharose High Performance Beads | GE Healthcare | 17-5113-07 | |
| PyroMark Annealing Buffer | Qiagen | 979009 | |
| PyroMark Denaturation Solution | Qiagen | 979007 | |
| PyroMark Wash Buffer | Qiagen | 979008 | |
| PyroMark Q24 | Qiagen | 9001514 | |
| PyroMark Q24 Cartridge | Qiagen | 979202 | |
| PyroMark Q96 HS Tip Holder Box | Qiagen | 979105 | |
| PyroMark Q24 Vacuum Workstation | Qiagen | 9001516 | |
| PyroMark Q24 Plate Holder | Qiagen | 9022273 | |
| Orbital shaker | Fisher | 11-675-297 | |
| Heat block | Fisher | 11-718Q |
Referencias
- . . PyroMark PCR Handbook. , (2009).
- . . PyroMark Q24 User Manual. , (2012).
- Elahi, E., Ronaghi, M. Pyrosequencing: a tool for DNA sequencing analysis. Methods Mol. Bio. 255, 211-219 (2004).
- Fakhrai-Rad, H., Pourmand, N., Ronaghi, M. Pyrosequencing: and accurate detection platform for single nucleotide polymorphisms. Hum. Mutat. 19 (5), 479-485 (2002).
- Langaee, T., Ronaghi, M. Genetic variation analyses by pyrosequencing. Mutat. Res. 573 (1-2), 96-102 (2005).
- Liu, Z., Lozupone, C., Hamady, M., Bushman, F. D., Knight, R. Short pyrosequencing reads suffice for accurate microbial community analysis. Nucleic Acids Res. 35 (18), e120 (2007).
- Novais, R. C., Thorstenson, Y. R. The evolution of Pyrosequencing for microbiology: From genes to genomes. J. Microbiol. Methods. 86 (1), 1-7 (2011).
- J, Metagenomic pyrosequencing and microbial identification. Clin. Chem. 55 (5), 856-866 (2009).
- Quince, C., Lanzen, A., Curtis, T. P., Davenport, R. J., Hall, N., Head, I. M., Read, L. F., Sloan, W. T. Accurate determination of microbial diversity from 454 pyrosequencing data. Nat. Methods. (9), 639-6341 (2009).
- Ronaghi, M., Karamohamed, S., Pettersson, B., Uhlen, M., Nyren, P. Real-time DNA sequencing using detection of pyrophosphate release. Anal. Biochem. 242, 84-89 (1996).
- King, C., Scott-Horton, T. Pyrosequencing: A simple method for accurate genotyping. J. Vis. Exp. (11), e630 (2008).
- Rothberg, J. W., Heinz, , et al. An integrated semi-conductor device that enabling non-optical genome sequencing. Nature. 475, 348-352 (2011).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-Mode Waveguides for Single-Molecule Analysis at high concentrations. Science. 299, 682-686 (2003).
- Eid, J., Fehr, A. Real-Time DNA Sequencing from Single Polymerase Molecules. Science. 323, 133-138 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados