Method Article
Pirosequenciamento de Identificação Microbiana e Caracterização
Neste Artigo
Resumo
Pyrosequencing é uma técnica versátil, que facilita a sequenciação do genoma microbiano que pode ser utilizado para identificar espécies bacterianas, discriminar estirpes bacterianas, e detectar mutações genéticas que conferem resistência a agentes anti-microbianos. Neste vídeo, o procedimento para a geração microbiana amplicon, amplicon pyrosequencing, e análise da seqüência do DNA será demonstrado.
Resumo
Pyrosequencing é uma técnica versátil, que facilita a sequenciação do genoma microbiano que pode ser utilizado para identificar espécies bacterianas, discriminar estirpes bacterianas e detecção de mutações genéticas que conferem resistência a agentes anti-microbianos. As vantagens de pirosequenciamento para aplicações de microbiologia incluem high-throughput screening rápido e confiável e correta identificação de micróbios e mutações do genoma microbianas. Pyrosequencing envolve a sequenciação de ADN, sintetizando a cadeia complementar de uma única base de cada vez, enquanto que a determinação do nucleótido específico ser incorporada durante a reacção de síntese. A reacção ocorre em imobilizada único molde de ADN de cadeia simples onde os quatro desoxirribonucleótidos (dNTP) são adicionados sequencialmente e os dNTPs não incorporados são degradados enzimaticamente, antes da adição do próximo dNTP para a reacção de síntese. Detecção da base específica incorporada no molde é monitorizada por geração de chemilumsinais inescent. A ordem dos dNTPs que produzem os sinais quimioluminescentes determina a sequência de ADN do molde. A capacidade de sequenciação em tempo real de pyrosequencing tecnologia permite a identificação microbiana rápido num único ensaio. Além disso, o instrumento pirosequenciamento, pode analisar a diversidade genética completa de resistência às drogas anti-microbianas, incluindo a tipagem de SNPs, mutações pontuais, inserções e deleções, bem como a quantificação de múltiplas cópias do gene que podem ocorrer em alguns resistência anti-microbiana padrões.
Introdução
Pyrosequencing é um método rápido e preciso de sequências de ácidos nucleicos que se baseia no princípio de "sequenciação por síntese". "Sequenciação por síntese" envolve o uso de um único molde de ADN para sintetizar a cadeia complementar uma cadeia de base de cada vez, e detectando a base incorporada (A, T, G ou C) em cada passo, por detecção de um sinal quimioluminescente. A reacção pirosequenciamento inclui ADN molde, dNTPs (dATP, dGTP, dTTP, dCTP), polimerase de ADN, sulfurilase de ATP, a luciferina, a luciferase e a apirase. A reacção de síntese é iniciada pela adição de um dos quatro dNTPs e DNA polimerase a um único molde de ADN de cadeia simples biotinilado. Polimerase de ADN incorpora o dNTP complementar para o molde, com a subsequente libertação de pirofosfato (Figura 1). Sulfurilase de ATP proporcionalmente converte o pirofosfato de ATP. ATP actua como um catalisador para a conversão mediada pela luciferase de luciferina para oxiluciferina, o qualgera luz (Figura 2). A intensidade da luz é proporcional ao número de nucleótidos incorporados e determina se está presente na cadeia molde sequencialmente (Figura 3) uma ou mais específico do dNTP (dATP, dTTP, dGTP e dCTP). Qualquer dNTP não incorporada é degradada pela apirase, antes da adição do próximo dNTP para a continuação da reacção de síntese. Este "sequenciação por síntese" reacção é repetida com a adição de cada um dos quatro dNTP até a sequência de ADN do molde de ADN de cadeia simples é determinado.
Para ver uma animação da reação pirosequenciamento Clique aqui para ver filme .
Protocolo
Introdução: Uma única colónia bacteriana deve ser usada para inocular um meio de cultura adequado que é incubado durante a noite. Um pelete bacteriana é então processado para purificar ADN genómico usando um kit de isolamento genómico disponíveis comercialmente. O ADN genómico purificado deve ter uma proporção de 260/280 (nm)> 1,8 para assegurar que a amostra está isenta de contaminação de proteína. A quantidade de ADN genómico requerido para a geração do modelo pirosequenciamento é de 10-20 ng.
A. Ampliação do Modelo de
(PyroMark Handbook PCR; www.qiagen.com / manuais 1)
Configurando a reação de PCR
Nota: Uma cartilha deve ser biotinilado na sua extremidade 5 ', e é recomendado que seja HPLC purificada para elaboração do modelo. Recomendamos o PyroMark Ensaio Projeto Software 2.0 para PCR e sedesenho de primers cia que são otimizados para o seqüenciamento de curto leitura, no entanto, Primer-BLAST ( http://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome ) do NCBI também pode ser utilizados para o desenho de primers.
- Descongelar todas as soluções necessárias (listadas na Tabela 1) e misturar completamente cada. Configure a reação de acordo com a Tabela 1. Todo o trabalho pode ser realizado à temperatura ambiente.
Nota: A PCR Master Mix contém MgCl2 para uma concentração final de 1,5 mM, o que proporciona resultados satisfatórios, na maioria dos casos. Se um maior Mg 2 + a concentração é necessária, até 3,5 ul de 25 mM de MgCl 2 pode ser adicionado a uma reacção.
- Misture a mistura de reacção pipetando suavemente para cima e para baixo, então dispensar volumes adequados em tubos de PCR.
- Adicionar template ADN de cada tubo de PCR. Recomendamos 10 ng de DNA genômico.
- Programar o termociclador de acordo com a Tabela 2. Um porão definitivo a 4 ° C, para conveniência. Se estiver usando um termociclador sem tampa aquecida, reações de sobreposição com 100 mL de óleo mineral.
- Coloque os tubos de PCR no termociclador e iniciar o programa de ciclismo.
B. Vacuum Protocolo Workstation
Imobilizar os produtos de PCR biotinilados
- Faça uma mistura mestre de acordo com a Figura 4.
Nota: Antes de pipetagem, agite o frasco de contas Sepharose streptavidin revestidos para garantir uma suspensão homogénea.
- Dependendo do volume de produto de PCR utilizado, dispensar 60-75 ul mistura principal em cada poço de uma placa de PCR para produzir um volume total de 80 uL por poço.
- Adicionar 5-20 ul do produto de PCR biotinilado a cada poço.
- Selar os poços com tira caps e agitar a placa de PCR a 1.400 rpm durante 5-10 min à temperatura ambiente (15-25 ° C), utilizando um agitador orbital.
A desnaturação de ADN e adição de iniciador de sequenciação
- Dilui-se os iniciadores de sequenciação a 0,3 uM com tampão de recozimento e dispensar 25 ul em cada poço de uma placa de reacção. Posicione a placa na estação de trabalho.
- Preencha os bebedouros de estações de trabalho de acordo com o diagrama da Figura 5.
- Ligue a bomba de vácuo, e verifique se a chave na ferramenta de vácuo é ativada (Figura 6). Lave as sondas do filtro com água de elevada pureza (Milli-Q cm x 18,2 MW ou equivalente) na calha 5 (Figura 5). Encher o cocho com água de alta pureza fresca para uso na etapa 12.
- Trabalhando rapidamente, posicionar a placa de PCR com contas na estação de trabalho. Assegure-se que ambas as placas estão na mesma orientação que quando as amostras foram carregadas.
- Com o interruptor de vácuo ON, diminuir o vácuoferramenta para os poços da placa de PCR de 20 segundos ou até que todo o volume foi aspirado. As pérolas serão capturadas pela ferramenta de vácuo (Figura 7).
- Com o vácuo ainda ON, lave a ferramenta com etanol 70% (trough 1) por 20 segundos ou até que todo o volume foi aspirado.
- Com vácuo ON, lave a ferramenta com solução de desnaturação (trough 2) por 20 segundos ou até que todo o volume foi aspirado.
Nota: A solução de desnaturação contém hidróxido de sódio, que é um irritante da pele e dos olhos. Sempre usar um jaleco, luvas e óculos de proteção.
- Com vácuo ON, lave a ferramenta com tampão de lavagem (calha 3) por 20 segundos ou até que todo o volume foi aspirado.
- Com vácuo ON, levantar a ferramenta vácuo para além de 90 ° na vertical por 5 segundos, para permitir que todo o líquido escorrer para fora da ferramenta de vácuo. Desligar a bomba, e alinhar a ferramenta de vácuo com a placa de reação. Baixe a ferramenta de vácuo nopoços e agitar suavemente de um lado para outro para liberar as contas nos poços contendo cartilha seqüenciamento.
- De modo a limpar a ferramenta de vácuo, agitar as sondas de filtro com água de elevada pureza (calha 4) durante 10 seg.
- Desligue o vácuo ON e limpar a ferramenta com água de alta pureza (trough 5) por 20 segundos ou até que todo o volume foi aspirado.
- Levante a ferramenta vácuo para além de 90 ° C vertical, por 5 segundos, em seguida, mudar o vácuo OFF e armazenar na posição "Parking".
Recozimento cartilha sequenciamento de cadeias de DNA
- Colocar a placa de reacção em um suporte da placa pré-aquecida.
- Aquece-se a placa num bloco de aquecimento a 80 ° C durante 2 min.
- Remova a placa do suporte e coloque em cima do balcão para esfriar em temperatura ambiente por 5 min.
Pyromark Q24 Operação
Nota: Ligue o instrumento usando o interruptor localizado acima do cabo de alimentação. Estrelatup pode levar até 1 min.
- Encher um cartucho de acordo com as instruções fornecidas no PyroMark Ouro Q24 Reagentes Manual 2. Para determinar volumes necessários, configurar um arquivo executado no software do aparelho, e no menu Ferramentas, selecione Pré Informações Run
- Se o instrumento já estava, verifique se ele não está realizando uma corrida e, em seguida, abra a tampa. Um sinal de aviso sonoro será emitido se a tampa é aberta quando não é seguro para o fazer.
- Abra a porta do cartucho e insira o cartucho com o rótulo virado para a frente (Figura 8). Certifique-se de que o cartucho está inserido corretamente (a linha deve ser visível na parte da frente) e fechar o portão.
- Abra a moldura da placa de retenção e posicionar a placa com as amostras dentro do instrumento.
- Feche a moldura da placa de retenção ea tampa do aparelho (Figura 9).
Iniciando uma corrida
- Insira o pendrive com o arquivo runs criadas com o software do instrumento na porta USB na parte da frente do aparelho.
- Selecione "Run" no menu principal e pressione "OK".
- Use as setas de rolagem para selecionar o arquivo de execução desejado e pressione "Select".
Nota: O aparelho começará a distribuição de reagentes, quando toda a pressão pré e níveis de temperatura são alcançados (isso pode levar alguns minutos). Durante o funcionamento, o instrumento tela exibe o pyrogram do bem seleccionada em tempo real. Use as setas de rolagem para visualizar o pyrogram de outros poços.
Completar uma corrida
- Quando o instrumento confirma que o prazo terminou eo arquivo de executada, foi salvo no pendrive, clique em "Fechar".
- Retire o stick USB. Se ele foi removido antes do prazo terminar, insira o stick USB. Selecione "Administração" e depois em "Copy é executado não salvos".
- O arquivo de correr agora podem ser abertos e analisados usando o software do aparelho e comparadosde uma biblioteca de micróbios conhecidos utilizando o software Identifire.
Resultados
Os resultados de uma corrida pyrosequencing típica utilizando o programa mostra a localização dos iniciadores para a frente e reverso usados para a geração do fragmento amplificado, e o iniciador de sequenciação utilizada para a reacção dentro do pyrosequencing 16S ribossomal sequência conservada (Figura 10). Hipervariável A sequência entre o primer locais permite a identificação de bactérias de um grande número de espécies bacterianas utilizando o iniciador de sequenciação conservada (5'-TACATGCAAGTCGA). Como um controlo interno de qualidade, a sequência de variação de ADN da região variável pode ser verificado para assegurar que a região de DNA correcta foi analisado . O software pirosequenciamento permite a comparação e alinhamento da sequência gerada a uma base de dados interna de sequências ribossomais bacterianas para identificação bacteriana. Além disso, uma sequência pode ser analisada para determinar as mutações que conferem resistência ao antibiótico. Por exemplo, a análise de mutações nos genes ribossomais 23S do Helicobpylori acter demonstra dois padrões de mutação (ou AGA GAA) que conferem resistência aos antibióticos (Figura 11). Análise dos vários isolados do mesmo paciente pode ser ensaiada simultaneamente para controlar o aparecimento de resistência a drogas ao longo do tempo para auxiliar em investigações epidemiológicas dos surtos microbianas de resistência a drogas.
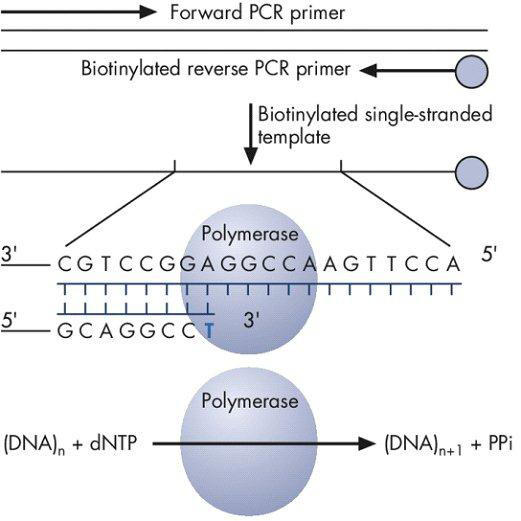
Figura 1. Produto de PCR biotinilado é usado como um modelo para incorporar dNTPs por polimerase do ADN, conduzindo à geração de pirofosfato (PPi).
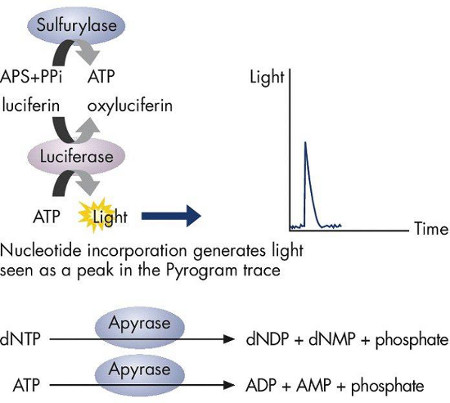
Figura 2. Sulfurilase de ATP proporcionalmente converte pirofosfato de ATP actos ATP. Como um catalisador para a conversão mediada pela luciferase de luciferina para oxiluciferina, o que gera luz que é próproporcional à quantidade de ATP. A luz é registada como o pico do traçado pyrogram e indica a incorporação de nucleótidos. Os dNTPs não incorporados são degradados pela apirase antes do próximo dNTP é adicionado para a continuação da síntese.
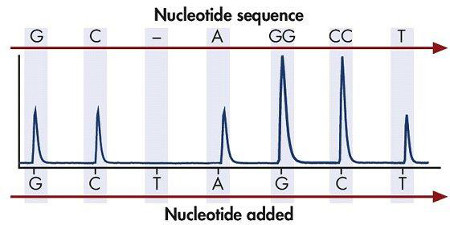
Figura 3. A intensidade de luz gerada indica se foi incorporado um ou mais específico do dNTP (dATP, dTTP, dGTP e dCTP) na cadeia molde sequencialmente.
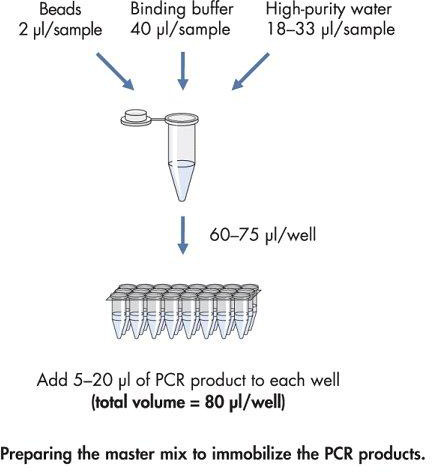
Figura 4. Fluxograma para preparação de mestre mix para imobilizar o produto PCR biotinilado.
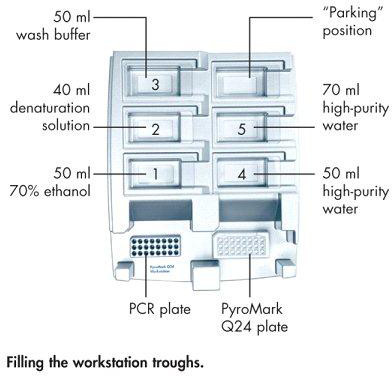
ge = "always"> Figura 5. PyroMark estação de trabalho, com placa de PCR, placa PyroMark e locais da calha.

Figura 6. Localização da chave de vácuo ON e OFF posições.
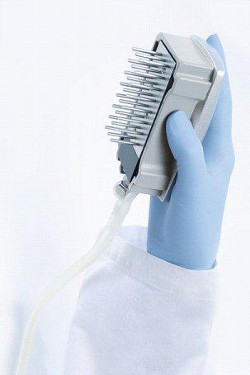
Figura 7. Ferramenta de vácuo. Tratamento adequado da ferramenta de vácuo.

Figura 8. Cartucho portão se abriu com o cartucho no lugar.

Figura 9. Cartucho inserido corretamente com portão fechado.
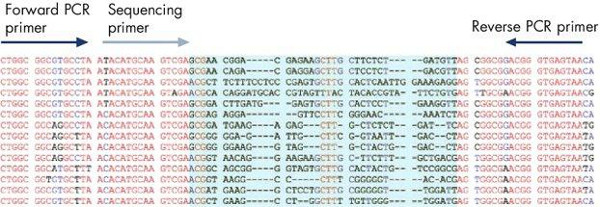
Figura 10. Pyrosequencing bacterianos baseados em resultados de identificação. Iniciadores de PCR foram concebidos para regiões conservadas do molde de ADN, e o iniciador de sequenciação está posicionado imediatamente a montante de uma sequência de ADN de identificação hipervariável bem caracterizados no âmbito do amplicão (mostrado em azul).
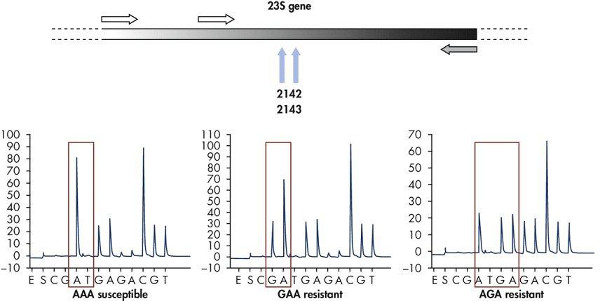
Figura 11. Detecção de resistência aos medicamentos antibióticos em Helicobacter pylori utilizando pyrosequencing. Análise de mutações nos genes 23S que conferem resistência antibacteriana em Helicobacter pylori contendo GAA ou seqüências AGA. Clique aqui para ver a figura maior .
| Componente | Volume por reação | A concentração final |
| Pyro PCR Master Mix, 2x | 12,5 mL | 1x |
| Concentrado CoralLoad, 10x | 2,5 mL | 1x |
| 25 mM de MgCl 2 (opcional) | Variável | ≥ 1,5 mM |
| Q-Solution, 5x (opcional) | 5 ul | 1x |
| Primer A / B Primer | Variável / Variável | ΜM/0.2 0,2 mM |
| Água RNase | Variável | - |
| Volume total (após a adição de ADN-alvo) | 25 ul |
Tabela 1. Mistura de reacção de PCR.
| & Nbsp; | Comentários adicionais | ||
| Passo de activação inicial de PCR | 15 min | 95 ° C | HotStartTaq DNA polimerase é ativado |
| 3 etapa de ciclismo: desnaturação | 30 seg | 94 ° C | |
| Recozimento | 30 seg | 60 ° C 56 ° C | Para obter ADN genómico Para bisulfite DNA convertido |
| Extensão | 30 seg | 72 ° C | |
| Número de ciclos | 45 | ||
| Extensão final | 10 min | 72 ° C |
Tabela 2. Especificações do ciclo de PCR.
Discussão
Vários novos métodos de sequenciamento de próxima geração foram comercializados. Três dos métodos mais utilizados são semicondutores seqüenciamento de íons, única molécula de sequenciamento em tempo real, e seqüenciamento por síntese (SBS). 3-11 Cada um desses métodos depende de seqüenciamento por síntese, mas empregar novas plataformas para a detecção dos nucleotídeos incorporados. No ião semicondutor sequenciação, uma cadeia simples de DNA serve como um molde para a cadeia de sequenciação. Polimerase e 12 nucleótidos são adicionados sequencialmente, tal como no pyrosequencing. Quando um nucleótido é adicionado à crescente cadeia de ADN, um ião de hidrogénio é libertado. O ião de hidrogénio é detectado por um sensor baseado em transistores de efeito de campo.
Única molécula de sequenciação em tempo real depende do modo de guia de ondas de zero (ZMW). ZMW 13 A é uma pequena estrutura em que uma única molécula de polimerase é ligado ao fundo do poço. Ele é iluminado de tal maneira que uma floremolécula de perfume pode ser detectado. Cada nucleótido é marcado com uma molécula fluorescente. Como um nucleótido marcado é incorporado por fluorescência, um detector identifica as sequências de nucleótidos. Quando o próximo nucleótido é adicionada, a molécula fluorescente é clivada 14.
Sequenciação por síntese (SBS) utiliza um método único para amplificar o DNA alvo de tal modo que os conjuntos de sequências únicas são gerados. De 15 modelos de cadeia simples são geradas e, em seguida, a cadeia complementar é sintetizado. Cada nucleótido é marcado com uma molécula fluorescente e depois de cada adição de base da fluorescência de base adicionada é gravado.
Divulgações
Produção e acesso gratuito a este artigo é patrocinado pela Qiagen.
Autores Ahmed, Durocher, Jessen e Vardi são funcionários da Qiagen Inc., que produz reagentes e instrumentos utilizados neste artigo.
Agradecimentos
Este projeto foi o apoio, em parte, pela Johns Hopkins University, Gabinete do Reitor através da Iniciativa Ciência Gateway e Qiagen Inc.
Materiais
| Name | Company | Catalog Number | Comments |
| PyroMark PCR Master Mix, 2x | Qiagen | 978703 | |
| CoralLoad Concentrate, 10x | Qiagen | 978703, 978705 | |
| Q-Solution, 5x | Qiagen | 978703, 978705 | |
| MgCl2, 25 mM | Qiagen | 978703, 978705 | |
| RNase-Free Water | Qiagen | 129112 | |
| Primers | Qiagen | 978776 or 978777 | Primers should be purchased from an established oligonucleotide manufacturer (i.e. QIAGEN Pyromark Custom Assays, IDT, etc). Lyophilized primers should be dissolved in TE to provide a stock solution of 100 μM. |
| Pyromark Gold Q24 Reagents | Qiagen | 970802 | |
| High-purity water (Milli-Q 18.2 MΩ x cm or equivalent) | |||
| Streptavidin Sepharose High Performance Beads | GE Healthcare | 17-5113-07 | |
| PyroMark Annealing Buffer | Qiagen | 979009 | |
| PyroMark Denaturation Solution | Qiagen | 979007 | |
| PyroMark Wash Buffer | Qiagen | 979008 | |
| PyroMark Q24 | Qiagen | 9001514 | |
| PyroMark Q24 Cartridge | Qiagen | 979202 | |
| PyroMark Q96 HS Tip Holder Box | Qiagen | 979105 | |
| PyroMark Q24 Vacuum Workstation | Qiagen | 9001516 | |
| PyroMark Q24 Plate Holder | Qiagen | 9022273 | |
| Orbital shaker | Fisher | 11-675-297 | |
| Heat block | Fisher | 11-718Q |
Referências
- PyroMark PCR Handbook. , [about 27p.]. Available from: http://www.qiagen.com/handbooks (2009).
- PyroMark Q24 User Manual. , [about 27p.]. Available from: http://www.qiagen.com/handbooks (2012).
- Elahi, E., Ronaghi, M. Pyrosequencing: a tool for DNA sequencing analysis. Methods Mol. Bio. 255, 211-219 (2004).
- Fakhrai-Rad, H., Pourmand, N., Ronaghi, M. Pyrosequencing: and accurate detection platform for single nucleotide polymorphisms. Hum. Mutat. 19 (5), 479-485 (2002).
- Langaee, T., Ronaghi, M. Genetic variation analyses by pyrosequencing. Mutat. Res. 573 (1-2), 96-102 (2005).
- Liu, Z., Lozupone, C., Hamady, M., Bushman, F. D., Knight, R. Short pyrosequencing reads suffice for accurate microbial community analysis. Nucleic Acids Res. 35 (18), e120(2007).
- Novais, R. C., Thorstenson, Y. R. The evolution of Pyrosequencing for microbiology: From genes to genomes. J. Microbiol. Methods. 86 (1), 1-7 (2011).
- J, Metagenomic pyrosequencing and microbial identification. Clin. Chem. 55 (5), 856-866 (2009).
- Quince, C., Lanzen, A., Curtis, T. P., Davenport, R. J., Hall, N., Head, I. M., Read, L. F., Sloan, W. T. Accurate determination of microbial diversity from 454 pyrosequencing data. Nat. Methods. (9), 639-6341 (2009).
- Ronaghi, M., Karamohamed, S., Pettersson, B., Uhlen, M., Nyren, P. Real-time DNA sequencing using detection of pyrophosphate release. Anal. Biochem. 242, 84-89 (1996).
- King, C., Scott-Horton, T. Pyrosequencing: A simple method for accurate genotyping. J. Vis. Exp. (11), e630(2008).
- Rothberg, J. W., Heinz,, et al. An integrated semi-conductor device that enabling non-optical genome sequencing. Nature. 475, 348-352 (2011).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-Mode Waveguides for Single-Molecule Analysis at high concentrations. Science. 299, 682-686 (2003).
- Eid, J., Fehr, A. Real-Time DNA Sequencing from Single Polymerase Molecules. Science. 323, 133-138 (2009).
- Sequencing Technology | Sequencing by Synthesis | Illumina [Internet]. , Illumina. Available from: http://www.illumina.com/technology/sequencing_technology.ilmn (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados