Method Article
微生物の同定とキャラクタリゼーションのためのパイロシーケンシング
要約
パイロシーケンシングでは、細菌種を識別する菌株を判別し、抗菌剤に対する耐性を付与する遺伝子の突然変異を検出するために使用することができる微生物のゲノムの配列決定を容易にする汎用性の高い技術である。このビデオでは、微生物のアンプリコンの生成、アンプリコンパイロシーケンシング、およびDNA配列分析のための手順が実証される。
要約
パイロシーケンシングでは、細菌種を識別する菌株を識別および抗微生物剤に対する耐性を付与する遺伝子の突然変異を検出するために使用することができる微生物のゲノムの配列決定を容易にする汎用性の高い技術である。微生物学アプリケーション用パイロシーケンシングの利点は、迅速かつ信頼性の高いハイスループットスクリーニングや微生物や微生物のゲノム変異の正確な識別を含む。パイロは、合成反応中に組み込まれ、特定のヌクレオチドの存在を決定しながら、同時に相補鎖の一塩基を合成することにより、DNAの配列が含まれます。反応には、4つのデオキシリボヌクレオチド(dNTPを)を順次加えて、非法人のdNTPは、合成反応の隣のdNTP添加前に酵素的に分解されて固定化された一本鎖鋳型DNA上で行われます。テンプレートに組み込まれた特定の塩基の検出は、chemilumの発生によって監視されるinescent信号。化学発光シグナルを生成するのdNTP順序は、テンプレートのDNA配列を決定する。パイロシーケンシング技術のリアルタイムシーケンシング機能は、単一のアッセイで迅速な微生物の同定を可能にする。また、パイロシーケンシング器具は、いくつかの抗微生物抵抗で発生する可能性のあるSNPのタイピング、点突然変異、挿入、および削除、ならびに複数の遺伝子コピーの定量化を含む抗微生物薬剤耐性の完全な遺伝的多様性を分析することができパターン。
概要
パイロは "合成による配列決定"の原則に基づいているシーケンス核酸への迅速かつ正確な方法である。 "合成により配列決定は、"一度に一つの基地相補鎖を合成する一本鎖DNAテンプレートを使用し、組み込まれた塩基(A、T、G、C又は)化学発光シグナルの検出によって各段階で検出することを含む。パイロシーケンシング反応は、鋳型DNA、のdNTP(dATPを、dGTPを、dTTPの、dCTPを)、DNAポリメラーゼ、ATPスルフリラーゼ、ルシフェリン、ルシフェラーゼ、およびアピラーゼが含まれています。合成反応は、4つのdNTPおよびビオチン化一本鎖鋳型DNAにDNAポリメラーゼのいずれかを添加することによって開始される。 DNAポリメラーゼは、ピロリン酸の次のリリース( 図1)で、テンプレートの上に補完的なdNTPを組み込まれています。 ATPスルフリラーゼは比例してATPのピロリン酸に変換されます。 ATPは、オキシルシフェリンにルシフェリンルシフェラーゼ媒介変換のための触媒として作用する光( 図2)を生成します。光の強度は、取り込まれたヌクレオチドの数に比例し、一つ以上の特定のdNTPは(のdATP、dTTPを、dGTPを、またはdCTPを)を順次テンプレート鎖( 図3)上に存在するかどうかを判定する。取り込まれていない任意のdNTPは、従来の合成反応の継続のための次のdNTPを添加するアピラーゼによって分解される。一本鎖DNAテンプレートのDNA配列が決定されるまで、この "合成による配列決定"反応は、4つのdNTPを添加して繰り返される。
パイロシーケンシング反応のアニメーションを表示するには、ムービーを表示するには、ここをクリック 。
プロトコル
はじめに:単一の細菌コロニーを一晩インキュベートされ、適切な培養液を接種するために使用されるべきである。細菌ペレットは、その後、市販のゲノム分離キットを用いてゲノムDNAを精製するために処理される。精製されたゲノムDNAは、サンプルは、タンパク質の混入がないことを保証するために280分の260(nm)の比を> 1.8を持っている必要がありますパイロシーケンシングテンプレートの生成に必要なゲノムDNAの量が10〜20 ngのである。
テンプレートのA.増幅
(PyroMark PCRハンドブック; www.qiagen.com /ハンドブック 1)
PCR反応を設定する
注:一方のプライマーは、その5 '末端にビオチン化しなければならず、それはそれは、テンプレート作成用HPLC精製することをお勧めします。我々は、PCRとSEのPyroMarkアッセイデザインソフトウェア2.0を推奨短いリードシーケンシング用に最適化されたquenceプライマーの設計は、しかし、プライマー-BLAST( http://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome NCBIから)もできますプライマー設計のために使用される。
- 必要なすべてのソリューションを( 表1に記載)解凍し、それぞれ徹底的に混ぜる。 表1に従って反応を設定します。すべての作業は室温で行うことができる。
注:PCRマスターミックスは、ほとんどのケースでは満足のいく結果を提供する1.5mmの最終濃度のためのMgCl 2を含んでいます。より高いの Mg 2 +は濃度が必要な場合は、25のMgCl 2の最大3.5μlの反応液に添加することができる。
- 徹底的に軽く上下にピペッティングによって反応ミックスを混ぜて、その後、適切な量をPCRチューブに分注する。
- templatを追加各PCRチューブに電子は、DNA。私たちは、ゲノムDNA 10ngのをお勧めします。
- 表2に従ってサーマルサイクラーをプログラムする。 4の最終ホールド°Cは、利便性をお勧めします。 100μlのミネラルオイルとオーバーレイ反応、加熱蓋なしでサーマルサイクラーを使用している場合。
- サイクラーにPCRチューブをセットし、サイクリングプログラムをスタートする。
B.真空ステーションプロトコル
ビオチン化PCR産物を固定化する
- 図4によるとマスターミックスを作る。
注:ピペッティングの前に、静かに均一な懸濁液を確実にするためにストレプトアビジンコートセファロースビーズのボトルを振る。
- 使用したPCR産物の量に応じて、ウェルあたり80μlの総容積を与えるためにPCRプレートの各ウェルに60〜75μlのマスターミックスを分注する。
- 各ウェルに5-20μlのビオチン化PCR産物を追加します。
- ストリップCAに井戸を封印オービタルシェーカーを用いて室温(15〜25℃)で5〜10分間1,400 rpmでPCRプレートをPSとを攪拌。
DNAの変性とプライマーを配列の追加
- バッファをアニーリングと0.3μMにシーケンシングプライマーを希釈し、反応プレートの各ウェルに25μlずつ分注する。ワークステーション上にプレートを置きます。
- 図5図に従って、ワークステーションの谷を埋める。
- 真空ポンプの電源を入れ、真空ツールのスイッチが( 図6)上に設定されていることを確認してください。トラフ5で高純度水(ミリQ 18.2 MW Xセンチまたは同等品)( 図5)でフィルタープローブをフラッシュします。リフィルステップ12で使用するための新鮮な高純度水でトラフ。
- すぐに働くことは、ワークステーション上のビーズをPCRプレートを配置。両方のプレートはサンプルがロードされたときと同じ向きであることを確認します。
- ON真空スイッチと、真空を下げる20秒またはすべてのボリュームを吸引されるまでPCRプレートのウェル中に工具。ビーズを真空ツール( 図7)によって捕捉される。
- まだON真空で、20秒間、またはすべてのボリュームが吸引されるまで70%エタノール(谷1)を使用してツールをフラッシュします。
- ON真空で、20秒間変性溶液(谷2)ツールをフラッシュまたはすべてのボリュームが吸引されるまで。
注:変性Solutionは目や皮膚刺激性である水酸化ナトリウムが含まれています。常に白衣、手袋、保護用眼鏡を着用してください。
- ON真空で、20秒間洗浄バッファー(トラフ3)でツールをフラッシュまたはすべてのボリュームが吸引されるまで。
- ON真空では、すべての液体が吸引ツールの外に排出することができるように、5秒間90°を超えて垂直に真空ツールを上げる。ポンプの電源をオフにして、反応プレートと真空ツールを揃える。に真空ツールを下げ井戸やそっとでは、配列決定プライマーを含むウェルにビーズを解放するために左右に振る。
- バキューム·ツールをきれいにするために、10秒間高純度水(トラフ4)内のフィルタのプローブを攪拌する。
- ON真空スイッチとすべてのボリュームが吸引されるまで20秒以上のために高純度水(谷5)ツールをフラッシュします。
- 5秒、90°Cを超えて垂直に真空ツールを上げ、その後、真空をOFFに切り替えると "駐車場"の位置に格納します。
DNA鎖に配列決定プライマーをアニーリング
- 予め温めておいたプレートホルダーに反応プレートを置きます。
- 2分間80℃ヒートブロック°Cの上にプレートを加熱する。
- 5分間室温で冷却するようにカウンターの上にホルダーと場所からプレートを取り外します。
Pyromark Q24操作
注:電源コードの上にある電源スイッチを使って楽器をスイッチオン。星TUPは、1分ほどかかる場合があります。
- PyroMarkゴールドQ24試薬ハンドブック2に記載されている指示に従って、カートリッジを埋める。測定器ソフトウェアで実行ファイルを設定し、必要なボリュームを決定し、[ツール]メニューの下に事前実行情報を選択する
- 楽器に既にあった場合、それが実行を行っていないことを確認した後、ふたを開ける。蓋が開いている場合、それはそうすることが安全であるときに可聴警告信号が送られる。
- カートリッジのゲートを開いて、ラベルが( 図8)手前にしてカートリッジを挿入します。カートリッジが正しく(ラインが正面に見えるはずです)が挿入されていることを確認し、ゲートを閉じます。
- 楽器内部のサンプルを用いて、板保持枠と位置プレートを開きます。
- プレート保持枠と機器蓋( 図9)を閉じます。
実行の開始
- 実行ファイルをUSBスティックを挿入sが機器の前面にUSBポートに測定器ソフトウェアで作成された。
- メインメニューの "ファイル名を指定して実行"を選択し、 "OK"を押します。
- 目的の実行ファイルを選択し、 "選択"を押してスクロール矢印を使用します。
注:楽器はすべての設定圧力と温度のレベルに達したとき(これには数分かかる場合があります)試薬の分注を開始します。実行中、計器画面がリアルタイムで選択ウェルのパイログラムを表示します。他の井戸のパイログラムを表示するには、スクロール矢印を使用します。
実行の完了
- 楽器は、実行が終了すると、実行ファイルをUSBスティックに保存されていることを確認すると、 "閉じる"を押します。
- USBスティックを取り外します。実行が終了する前に、それが削除された場合は、USBスティックを挿入します。 "管理"とし、 "コピー保存されていない操業"を選択します。
- 実行ファイルが開かれ、測定器ソフトウェアを用いて分析し、比較することができるIdentifireソフトウェアを使用して、既知の微生物のライブラリ。
結果
典型的なパイロシーケンシングの結果は、ソフトウェアを用いて実行するアンプリコンの生成に使用されるフォワードおよびリバースプライマーの位置、および保存された16Sリボソーム配列( 図10)内のパイロシーケンシング反応に用いたプライマー配列決定を示す。プライマーとの間の可変配列サイトは、保存された配列決定プライマー(5'-TACATGCAAGTCGA)を用いて細菌種の数が多い細菌同定を可能にする。内部品質管理として、DNA配列のブラケットは、可変領域が正しいDNA領域が分析されることを保証するためにチェックすることができる。パイロシーケンシングソフトウェアでは、細菌の同定のための細菌のリボソームの配列の内部データベースに生成されたシーケンスの比較および整列することができます。さらに、配列は、抗生物質耐性を付与する変異を決定するために分析することができる。 Helicobの23Sリボソーム遺伝子の変異の例については、分析アクター·ピロリ菌は、抗生物質耐性を付与する2変異パターン(GAAまたはAGA)( 図11)を示しています。同じ患者からの複数の分離株の分析を同時に微生物の薬剤耐性の発生の疫学的調査を支援するために経時的に薬剤耐性の出現を追跡するためにアッセイすることができる。
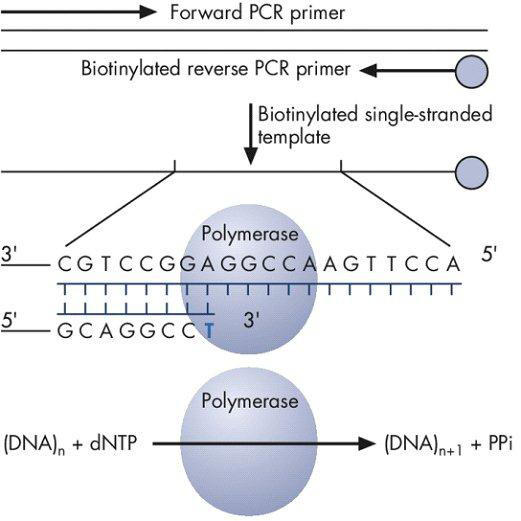
図1。ビオチン化されたPCR産物はピロリン酸(PPiを)の発生につながる、DNAポリメラーゼによってdNTPのを組み込むためのテンプレートとして使用される。
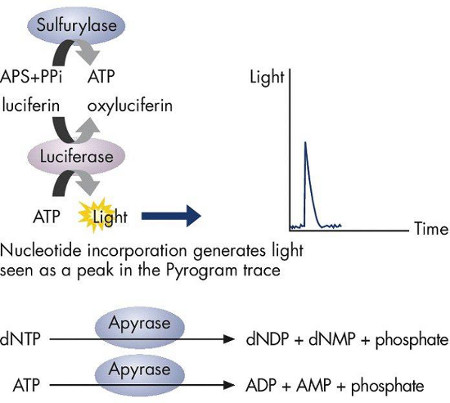
図2。 ATPスルフリラーゼは比例してプロである光を生成するオキシルシフェリンにルシフェリンルシフェラーゼ媒介変換のための触媒としてATP。ATPの行為にピロリン酸に変換ATPの量にportional。光はパイログラムトレース上のピークとして記録され、ヌクレオチドの取り込みを示している。次のdNTP、合成の継続のために追加される前に取り込まれていないのdNTPはアピラーゼによって分解される。
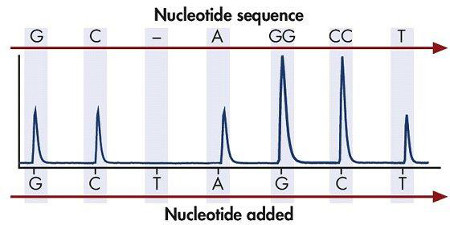
図3。一つ以上の特定のdNTPを(のdATP、dTTPを、dGTPを、またはdCTPを)を順次テンプレート鎖上に組み込まれた場合に発生する光の強度を示している。
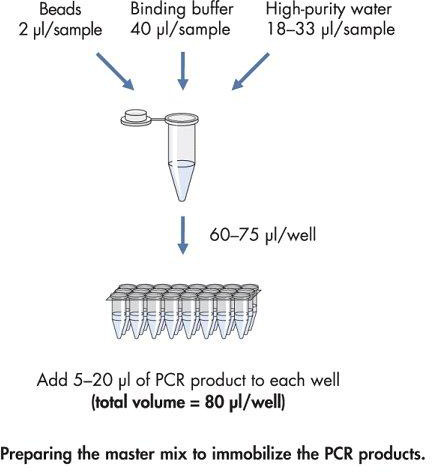
図4。ビオチン化されたPCR産物を固定化するマスターミックスを調製するためのフローチャート。
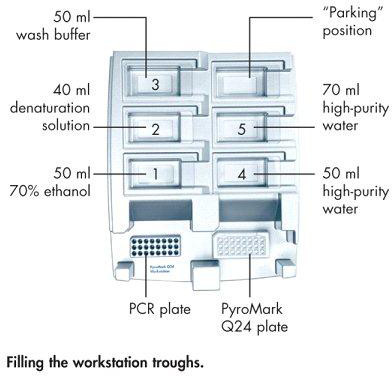
GE = "常に"> 図5。 PCRプレート、PyroMarkプレート、およびトラフの場所とPyroMarkワークステーション。

図6。ポジションONとOFF真空スイッチの位置。
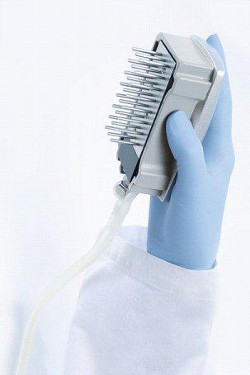
図7。真空ツール。真空ツールの適切な取り扱い。

図8。カートリッジゲートの場所にカートリッジにオープンしました。

図9。ゲートを閉じた状態でカートリッジが正しく挿入。
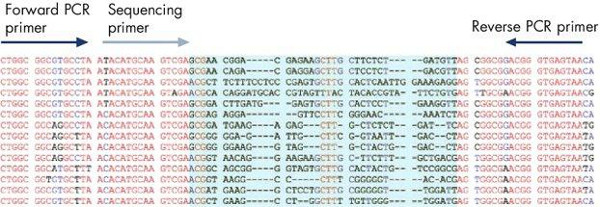
図10。ベース細菌の同定結果をパイロシーケン。PCRプライマーは、DNAテンプレートの保存領域用に設計され、そして配列決定プライマー(青で示されている)アンプリコン内の可変DNA配列を同定十分に特徴付けのすぐ上流に配置されている。
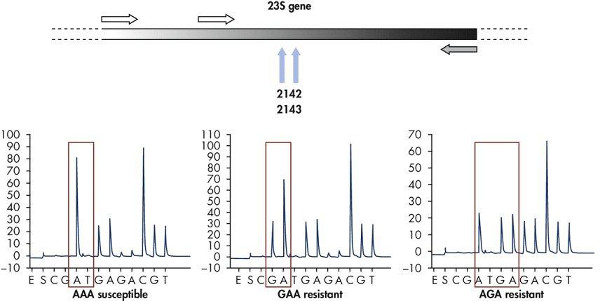
図11。パイロシーケンシングを用いたヘリコバクター·ピロリ菌に抗生物質薬剤耐性の検出。GAAまたはAGA配列を含むヘリコバクター·ピロリに抗菌耐性を付与する23S遺伝子の変異の分析は。 より大きい数字を表示するには、ここをクリックしてください 。
| コンポーネント | 反応あたりの体積 | 最終濃度 |
| パイロPCRマスターミックス、2倍 | 12.5μlの | 1X |
| CoralLoadコンセントレイト、10倍 | 2.5μL | 1X |
| 25のMgCl 2(オプション) | 変数 | ≥1.5 mMの |
| 5X Q-ソリューション(オプション) | 5μlの | 1X |
| プライマー/プライマーB | 可変/可変 | 0.2μM/0.2μM |
| RNaseフリー水 | 変数 | - |
| 総容積(鋳型DNAを添加した後) | 25μlの |
表1。 PCRの反応混合物。
| &NBSPは、 | 追加のコメント | ||
| 最初のPCR活性化ステップ | 15分 | 95°C | HotStartTaq DNAポリメラーゼが活性化される |
| 3ステップサイクリング:変性 | 30秒 | 94°C | |
| アニーリング | 30秒 | 60°C 56°C | ゲノムDNAのために バイサルファイト変換DNAのために |
| 拡張 | 30秒 | 72°C | |
| サイクル数 | 45 | ||
| 最終伸長 | 10分 | 72°C |
表2。 PCRサイクル仕様。
ディスカッション
いくつかの新しい次世代シーケンシング法が実用化されている。最も広く使用されている方法の三人は合成によるイオン半導体シーケンシング、単一分子リアルタイムシーケンシング、およびシーケンシング(SBS)であり、これらのメソッドの3-11各合成による配列決定に依存しますが、取り込まれたヌクレオチドの検出のための新たなプラットフォームを採用しています。イオン半導体シーケンシング、DNAの一本鎖でシーケンストランドのためのテンプレートとして機能します12ポリメラーゼ及びヌクレオチドがパイロシーケンシングのように順次追加されます。ヌクレオチドが成長するDNA鎖に追加されると、水素イオンが放出される。水素イオンは、電界効果トランジスタ基盤のセンサーによって検出される。
単一分子リアルタイムシーケンスは、ゼロモード導波管(ZMW)に依存します13 ZMWが単一ポリメラーゼ分子がウェルの底部に取り付けられている小さな構造です。それは、フロールように点灯している香り分子を検出することができる。各ヌクレオチドは、蛍光分子でタグ付けされています。蛍光標識ヌクレオチドが組み込まれたように、検出器は、ヌクレオチドを識別します。次のヌクレオチドが追加されると、蛍光分子が切断される。14
合成によってシークエンシング(SBS)。15一本鎖テンプレートが生成され、次いで、相補鎖が合成されるユニーク配列のクラスターが生成されるように、標的DNAを増幅するユニークな方法を使用する。各ヌクレオチドは、蛍光分子で標識されており、各基地添加後に添加される塩基の蛍光が記録される。
開示事項
この記事への生産とフリーアクセスはQiagen社が主催しています。
著者アーメド、ドローチャー、ジェッセンとヴァルディは、この記事で使用される試薬および器具を生産キアゲン社の従業員です。
謝辞
このプロジェクトは、ジョンズ·ホプキンス大学、 ゲートウェイ科学イニシアティブ及びキアゲン社を通じて学長のオフィスによって部分的にサポートしていた
資料
| Name | Company | Catalog Number | Comments |
| PyroMark PCR Master Mix, 2x | Qiagen | 978703 | |
| CoralLoad Concentrate, 10x | Qiagen | 978703, 978705 | |
| Q-Solution, 5x | Qiagen | 978703, 978705 | |
| MgCl2, 25 mM | Qiagen | 978703, 978705 | |
| RNase-Free Water | Qiagen | 129112 | |
| Primers | Qiagen | 978776 or 978777 | Primers should be purchased from an established oligonucleotide manufacturer (i.e. QIAGEN Pyromark Custom Assays, IDT, etc). Lyophilized primers should be dissolved in TE to provide a stock solution of 100 μM. |
| Pyromark Gold Q24 Reagents | Qiagen | 970802 | |
| High-purity water (Milli-Q 18.2 MΩ x cm or equivalent) | |||
| Streptavidin Sepharose High Performance Beads | GE Healthcare | 17-5113-07 | |
| PyroMark Annealing Buffer | Qiagen | 979009 | |
| PyroMark Denaturation Solution | Qiagen | 979007 | |
| PyroMark Wash Buffer | Qiagen | 979008 | |
| PyroMark Q24 | Qiagen | 9001514 | |
| PyroMark Q24 Cartridge | Qiagen | 979202 | |
| PyroMark Q96 HS Tip Holder Box | Qiagen | 979105 | |
| PyroMark Q24 Vacuum Workstation | Qiagen | 9001516 | |
| PyroMark Q24 Plate Holder | Qiagen | 9022273 | |
| Orbital shaker | Fisher | 11-675-297 | |
| Heat block | Fisher | 11-718Q |
参考文献
- PyroMark PCR Handbook. , [about 27p.]. Available from: http://www.qiagen.com/handbooks (2009).
- PyroMark Q24 User Manual. , [about 27p.]. Available from: http://www.qiagen.com/handbooks (2012).
- Elahi, E., Ronaghi, M. Pyrosequencing: a tool for DNA sequencing analysis. Methods Mol. Bio. 255, 211-219 (2004).
- Fakhrai-Rad, H., Pourmand, N., Ronaghi, M. Pyrosequencing: and accurate detection platform for single nucleotide polymorphisms. Hum. Mutat. 19 (5), 479-485 (2002).
- Langaee, T., Ronaghi, M. Genetic variation analyses by pyrosequencing. Mutat. Res. 573 (1-2), 96-102 (2005).
- Liu, Z., Lozupone, C., Hamady, M., Bushman, F. D., Knight, R. Short pyrosequencing reads suffice for accurate microbial community analysis. Nucleic Acids Res. 35 (18), e120(2007).
- Novais, R. C., Thorstenson, Y. R. The evolution of Pyrosequencing for microbiology: From genes to genomes. J. Microbiol. Methods. 86 (1), 1-7 (2011).
- J, Metagenomic pyrosequencing and microbial identification. Clin. Chem. 55 (5), 856-866 (2009).
- Quince, C., Lanzen, A., Curtis, T. P., Davenport, R. J., Hall, N., Head, I. M., Read, L. F., Sloan, W. T. Accurate determination of microbial diversity from 454 pyrosequencing data. Nat. Methods. (9), 639-6341 (2009).
- Ronaghi, M., Karamohamed, S., Pettersson, B., Uhlen, M., Nyren, P. Real-time DNA sequencing using detection of pyrophosphate release. Anal. Biochem. 242, 84-89 (1996).
- King, C., Scott-Horton, T. Pyrosequencing: A simple method for accurate genotyping. J. Vis. Exp. (11), e630(2008).
- Rothberg, J. W., Heinz,, et al. An integrated semi-conductor device that enabling non-optical genome sequencing. Nature. 475, 348-352 (2011).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-Mode Waveguides for Single-Molecule Analysis at high concentrations. Science. 299, 682-686 (2003).
- Eid, J., Fehr, A. Real-Time DNA Sequencing from Single Polymerase Molecules. Science. 323, 133-138 (2009).
- Sequencing Technology | Sequencing by Synthesis | Illumina [Internet]. , Illumina. Available from: http://www.illumina.com/technology/sequencing_technology.ilmn (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved