Method Article
Pyrosequencing per identificazione microbica e caratterizzazione
In questo articolo
Riepilogo
Pyrosequencing è una tecnica versatile che facilita microbica sequenziamento del genoma che può essere utilizzato per identificare le specie batteriche, discriminare ceppi batterici, e rilevare mutazioni genetiche che conferiscono resistenza agli agenti antimicrobici. In questo video, verrà dimostrata la procedura per la generazione microbica amplicone, amplicone pyrosequencing, e l'analisi di sequenza del DNA.
Abstract
Pyrosequencing è una tecnica versatile che facilita microbica sequenziamento del genoma che può essere utilizzato per identificare le specie batteriche, discriminare ceppi batterici e rilevare mutazioni genetiche che conferiscono resistenza agli agenti antimicrobici. I vantaggi di pyrosequencing per applicazioni di microbiologia includono rapido e affidabile high-throughput screening e l'identificazione accurata di microbi e mutazioni del genoma microbici. Pyrosequencing comporta sequenziamento del DNA sintetizzando il filamento complementare un'unica base alla volta, mentre la determinazione dell'essere nucleotidica specifica incorporati durante la reazione di sintesi. La reazione avviene in immobilizzata singolo filamento DNA stampo dove vengono aggiunti sequenzialmente le quattro deoxyribonucleotides (dNTP) e le dNTP non incorporati sono degradati enzimaticamente prima dell'aggiunta del successivo dNTP alla reazione di sintesi. Individuazione della base specifica incorporato nel modello viene monitorata da generazione di chemilumsegnali inescent. L'ordine dei dNTP che producono i segnali chemiluminescenti determina la sequenza di DNA del modello. La capacità sequenziamento tempo reale di tecnologia pyrosequencing consente una rapida identificazione microbica in un singolo test. Inoltre, lo strumento pyrosequencing, in grado di analizzare la piena diversità genetica di anti-resistenza ai farmaci antimicrobici, compresi tipizzazione di SNPs, mutazioni puntiformi, inserzioni e delezioni, nonché la quantificazione delle copie multiple del gene che possono verificarsi in una certa resistenza antimicrobica modelli.
Introduzione
Pyrosequencing è un metodo rapido e preciso per gli acidi nucleici di sequenza che si basa sul principio di "sequenziamento per sintesi". "Sequencing per sintesi" comporta l'uso di un singolo filamento stampo di DNA per sintetizzare il filamento complementare una base alla volta, e rilevando la base incorporata (A, T, G, o C) ad ogni passo per rilevamento di un segnale chemiluminescente. La reazione pirosequenziamento comprende DNA stampo, dNTPs (dATP, dGTP, dTTP, dCTP), DNA polimerasi, ATP sulfurilasi, luciferina, luciferasi, e apyrase. La reazione di sintesi è iniziata con l'aggiunta di uno dei quattro dNTP e DNA polimerasi al DNA stampo singolo filamento biotinilato. DNA polimerasi incorpora il dNTP complementare sulla dima, con il successivo rilascio di pirofosfato (Figura 1). ATP sulfurilasi converte proporzionalmente il pirofosfato di ATP. ATP agisce come un catalizzatore per la conversione luciferasi-mediata di luciferina ad oxyluciferin, chegenera luce (Figura 2). L'intensità della luce è proporzionale al numero di nucleotidi incorporati e determina se è presente sul filamento stampo in sequenza (Figura 3) uno o più specifici di dNTP (dATP, dTTP, dGTP, dCTP o). Ogni dNTP non incorporata è degradata dalla apyrase prima dell'aggiunta del successivo dNTP per la continuazione della reazione di sintesi. Questa "sequenziamento mediante sintesi" reazione viene ripetuto con aggiunta di ciascuno dei quattro dNTP finché la sequenza di DNA del template DNA a singolo filamento è determinata.
Per visualizzare un'animazione della reazione pyrosequencing Clicca qui per vedere film .
Protocollo
Introduzione: Una singola colonia batterica dovrebbe essere utilizzata per inoculare un appropriato brodo di coltura che viene incubata per una notte. Un pellet batterico viene quindi elaborato per purificare il DNA genomico usando un kit di isolamento genomico disponibile in commercio. Il DNA genomico purificato deve avere un rapporto di 260/280 (nm)> 1.8 per assicurare che il campione sia esente da contaminazione proteica. La quantità di DNA genomico richiesto per la generazione del modello pirosequenziamento è 10-20 ng.
A. Amplificazione del Template
(PyroMark PCR Handbook; www.qiagen.com / manuali 1)
Impostazione della reazione di PCR
Nota: Una mano di fondo deve essere biotinilato alla sua estremità 5 ', e si raccomanda che sia HPLC purificato per la preparazione del modello. Si consiglia l'PyroMark Assay Software Design 2.0 per PCR e SEprogettazione Primer sequenza che sono ottimizzati per il sequenziamento di breve lettura, tuttavia, Primer-BLAST ( http://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome ) da NCBI può anche essere utilizzato per la progettazione primer.
- Scongelare tutte le soluzioni necessari (elencati nella Tabella 1) e mescolare ogni accuratamente. Impostare la reazione in base alla tabella 1. Tutto il lavoro può essere fatto a temperatura ambiente.
Nota: La PCR Master Mix contiene MgCl 2 per una concentrazione finale di 1,5 mM, che fornisce risultati soddisfacenti nella maggior parte dei casi. Se un maggiore Mg 2 + è richiesta concentrazione, fino a 3,5 ml di 25 mM MgCl2 può essere aggiunto a una reazione.
- Mescolare bene la miscela di reazione pipettando gentilmente su e giù, poi Versare volumi appropriati in provette PCR.
- Aggiungi tEmPLate DNA per ogni provetta PCR. Si consiglia di 10 ng di DNA genomico.
- Programmare il termociclatore secondo la Tabella 2. Una presa finale a 4 ° C è raccomandato per convenienza. Se si utilizza un termociclatore senza coperchio riscaldato, reazioni di sovrapposizione con 100 ml di olio minerale.
- Mettere le provette PCR nel termociclatore e avviare il programma di ciclismo.
B. Vacuum Workstation protocollo
Immobilizzare i prodotti di PCR biotinilato
- Fare un master mix secondo la Figura 4.
Nota: Prima di pipettaggio, agitare delicatamente la bottiglia di streptavidina perline rivestite Sepharose per garantire una sospensione omogenea.
- A seconda del volume del prodotto di PCR utilizzato, dispensare 60-75 microlitri master mix in ciascun pozzetto di una piastra per PCR per ottenere un volume totale di 80 microlitri per pozzetto.
- Aggiungere 5-20 ml di prodotto PCR biotinilato a ciascun pozzetto.
- Sigillare i pozzetti con la striscia di caps e agitare la piastra PCR a 1.400 rpm per 5-10 minuti a temperatura ambiente (15-25 ° C) con un agitatore orbitale.
Denaturazione del DNA e l'aggiunta di sequenziamento Primer
- Diluire i primer di sequenziamento a 0.3 mM tampone con ricottura e dispensare 25 microlitri in ciascun pozzetto di una piastra di reazione. Posizionare la piastra sulla workstation.
- Riempire gli abbeveratoi workstation secondo il diagramma di Figura 5.
- Accendere la pompa del vuoto, e assicurarsi che l'interruttore sullo strumento vuoto è impostato su (Figura 6). Lavare le sonde filtro con acqua ad elevata purezza (Milli-Q 18,2 MW x cm o equivalente) in depressione 5 (Figura 5). Riempire la vasca con acqua ad alta purezza fresco da utilizzare nel passaggio 12.
- Lavorare velocemente, posizionare la piastra di PCR con perline sulla workstation. Assicurarsi che entrambi i piatti sono lo stesso orientamento di quando sono stati caricati i campioni.
- Con il vacuostato ON, abbassare il vuotoutensile nei pozzetti della piastra PCR per 20 secondi o finché tutto volume è stato aspirato. Le perle vengono catturati dallo strumento vuoto (Figura 7).
- Con il vuoto ancora ON, lavare lo strumento con il 70% di etanolo (attraverso 1) per 20 secondi o fino a che tutto il volume è stato aspirato.
- Con vuoto ON, lavare lo strumento con la soluzione di denaturazione (trogolo 2) per 20 secondi o fino a che tutto il volume è stato aspirato.
Nota: La soluzione di denaturazione contiene idrossido di sodio, che è un occhio e irritante per la pelle. Indossare sempre un camice da laboratorio, guanti e occhiali protettivi.
- Con vuoto ON, lavare lo strumento con tampone di lavaggio (depressione 3) per 20 secondi o fino a che tutto il volume è stato aspirato.
- Con vuoto ON, sollevare lo strumento vuoto per oltre 90 ° in verticale per 5 secondi, per consentire a tutti di liquido di defluire fuori dello strumento vuoto. Spegnere la pompa e allineare lo strumento di vuoto con la piastra di reazione. Abbassare l'attrezzo di vuoto nelpozzi e scuotere delicatamente da un lato all'altro per liberare le perline nei pozzetti contenenti Primer di sequenziamento.
- Per pulire lo strumento vuoto, agitare le sonde filtro in acqua ad alta purezza (depressione 4) per 10 sec.
- Spegnere il vuoto e lavare lo strumento con acqua ad alta purezza (depressione 5) per 20 secondi o fino a che tutto il volume è stato aspirato.
- Sollevare lo strumento vuoto per oltre 90 ° C verticale per 5 secondi, quindi passare l'aspirapolvere OFF e memorizzare nella posizione "parcheggio".
Ricottura Primer di sequenziamento di filamenti di DNA
- Posizionare la piastra di reazione in un supporto piatto preriscaldato.
- Riscaldare la piastra su una piastra riscaldante a 80 ° C per 2 min.
- Rimuovere la piastra dal supporto e posto sul bancone a raffreddare a temperatura ambiente per 5 min.
Pyromark Q24 Funzionamento
Nota: Accendere l'apparecchio con l'interruttore di alimentazione che si trova sopra il cavo di alimentazione. Stellatup può richiedere fino a 1 min.
- Riempire una cartuccia seguendo le istruzioni fornite nella PyroMark Oro Q24 Reagenti Manuale 2. Per determinare i volumi necessari, impostare un file di correre nel software dello strumento, e sotto il menu Strumenti selezionare Pre Informazioni Run
- Se lo strumento è già acceso, assicurarsi che non sta eseguendo una corsa e quindi aprire il coperchio. Un segnale acustico viene emesso se il coperchio è aperto, quando non è sicuro di farlo.
- Aprire la porta della cartuccia e inserire la cartuccia con l'etichetta rivolta in avanti (Figura 8). Assicurarsi che la cartuccia sia inserita correttamente (una linea dovrebbe essere visibile nella parte anteriore) e chiudere il cancello.
- Aprire il telaio piastra-holding e posizionare la piastra con i campioni all'interno dello strumento.
- Chiudere il telaio reggipiatti e il coperchio dello strumento (Figura 9).
Avvio di una corsa
- Inserire la chiavetta USB con il file di esecuziones creato con il software dello strumento alla porta USB sulla parte anteriore dello strumento.
- Selezionare "Esegui" nel menu principale e premere "OK".
- Utilizzare le frecce di scorrimento per selezionare il file di esecuzione desiderato e premere "Seleziona".
Nota: Lo strumento inizia la dispensazione reagenti durante tutta la pressione preimpostata e livelli di temperatura raggiunti (l'operazione potrebbe richiedere alcuni minuti). Durante una corsa, lo schermo dello strumento visualizza la pyrogram del ben selezionati in tempo reale. Utilizzare le frecce di scorrimento per visualizzare l'pyrogram di altri pozzi.
Completamento di una corsa
- Quando lo strumento conferma che la corsa è finita e il file di esecuzione è stato salvato sulla chiavetta USB, premere "Chiudi".
- Rimuovere la chiavetta USB. Se è stata rimossa prima che finito la corsa, inserire la chiavetta USB. Selezionare "Amministrazione" e poi "Copy viene eseguito non salvati".
- Eseguire il file può essere aperto e analizzato con il software dello strumento e confrontatoa una libreria di microbi conosciuti utilizzando il software Identifire.
Risultati
I risultati di una tipica corsa pyrosequencing utilizzando il software mostra la posizione dei primer forward e reverse utilizzati per la generazione amplicone, e il primer sequenziamento utilizzato per la reazione pyrosequencing all'interno della sequenza conservata ribosomiale 16S (Figura 10). La sequenza ipervariabile tra il primer siti permette di identificazione batterica di un gran numero di specie batteriche utilizzando il primer di sequenziamento conservata (5'-TACATGCAAGTCGA). Come controllo qualità interno, la sequenza di DNA della regione bracketing variabile può essere controllato per assicurare che la regione di DNA corretta è stata analizzata . Il software pirosequenziamento permette la comparazione e l'allineamento della sequenza generata da un database interno di sequenze ribosomali batteriche di identificazione batterica. Inoltre, una sequenza può essere analizzato per determinare mutazioni che conferiscono resistenza agli antibiotici. Per esempio, l'analisi di mutazioni nei geni 23S ribosomiale Helicobpylori tere dimostra due modelli di mutazione (GAA o AGA) che conferiscono resistenza agli antibiotici (Figura 11). Analisi di isolati multipli dello stesso paziente può essere contemporaneamente analizzati per monitorare la comparsa di resistenza ai farmaci nel tempo per aiutare nelle indagini epidemiologiche microbiche focolai di resistenza ai farmaci.
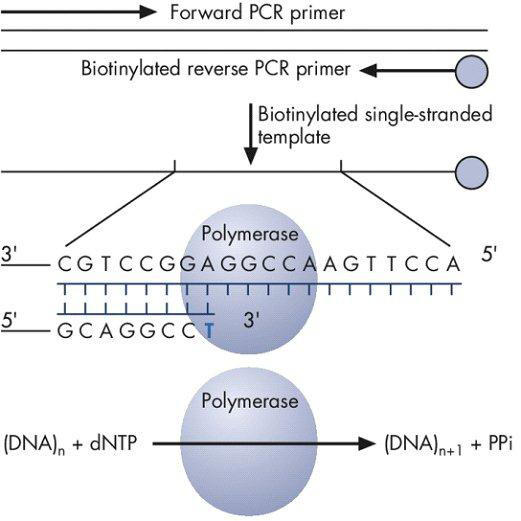
Figura 1. Biotinilato prodotto della PCR è utilizzato come modello per incorporare dNTPs dalla DNA polimerasi, che porta alla generazione di pirofosfato (PPi).
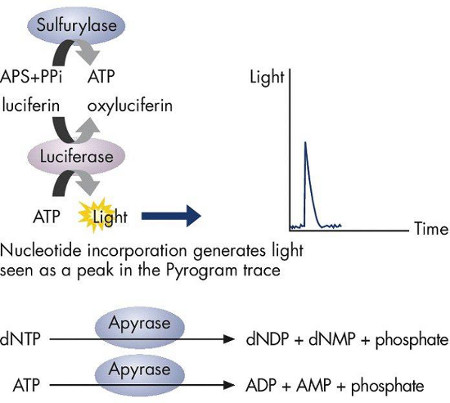
Figura 2. ATP sulfurilasi converte proporzionalmente pirofosfato di ATP. ATP agisce come un catalizzatore per la conversione luciferasi-mediata di luciferina ad oxyluciferin, che genera luce che è proproporzionale alla quantità di ATP. La luce viene registrato come picco sulla traccia pyrogram e indica l'incorporazione di nucleotidi. I dNTPs non incorporati sono degradati da apyrase prima di aggiungere il successivo dNTP per la continuazione della sintesi.
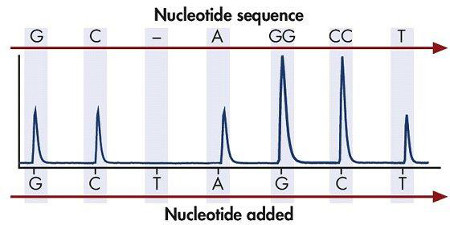
Figura 3. L'intensità della luce generata indica se è stata costituita una o più specifiche di dNTP (dATP, dTTP, dGTP, o dCTP) sul filamento stampo in modo sequenziale.
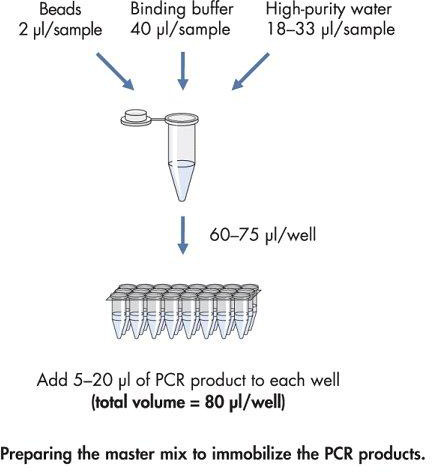
Figura 4. Diagramma di flusso per la preparazione della Master Mix per immobilizzare il prodotto di PCR biotinilato.
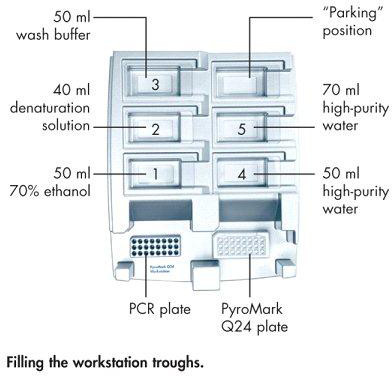
ge = "always"> Figura 5. PyroMark postazione di lavoro, con la piastra PCR, piastra PyroMark, e sedi di valle.

Figura 6. Posizioni dell'interruttore di vuoto posizioni ON e OFF.
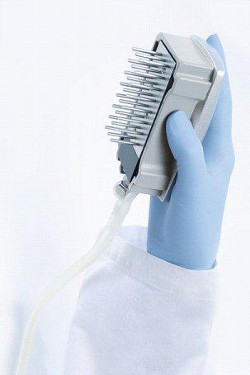
Figura 7. Strumento di vuoto. Corretta gestione dello strumento vuoto.

Figura 8. Cancello cartuccia aperta con cartuccia in posizione.

Figura 9. Cartuccia inserita correttamente con cancello chiuso.
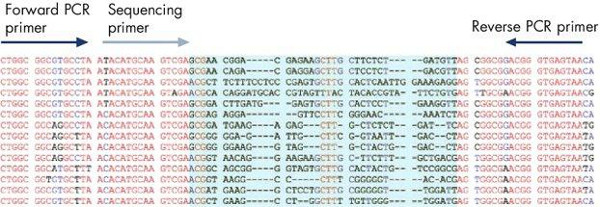
La figura 10. Pirosequenziamento risultati di identificazione batterica based. Primer PCR sono progettati per regioni conservate del DNA stampo, e il primer di sequenziamento è posizionato immediatamente a monte di una sequenza di DNA che identifica ipervariabile ben caratterizzato all'interno amplicone (in blu).
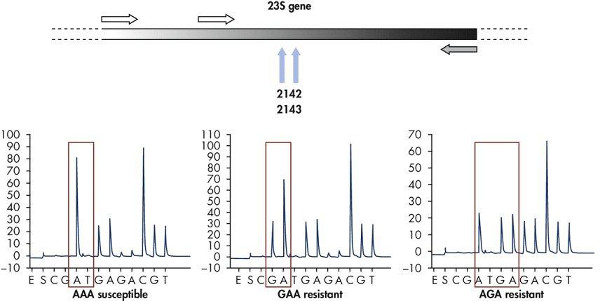
Figura 11. Rilevamento di antibiotico resistenza in Helicobacter pylori mediante pyrosequencing. Analisi di mutazioni nei geni 23S che conferiscono resistenza antibatterica in Helicobacter pylori contenente GAA o sequenze di AGA. Clicca qui per ingrandire la figura .
| Componente | Volume per reazione | Concentrazione finale |
| Pyro PCR Master Mix, 2x | 12,5 ml | 1x |
| CoralLoad Concentrato, 10x | 2,5 microlitri | 1x |
| 25 mM di MgCl2 (opzionale) | Variabile | ≥ 1.5 mM |
| Q-Solution, 5x (opzionale) | 5 microlitri | 1x |
| Primer A / B Primer | Variabile / Variabile | 0.2 μM/0.2 mM |
| Acqua RNase-free | Variabile | - |
| Volume totale (dopo l'aggiunta di DNA stampo) | 25 pl |
Tabella 1. PCR miscela di reazione.
| & Nbsp; | Ulteriori commenti | ||
| Iniziale fase di attivazione PCR | 15 min | 95 ° C | HotStartTaq DNA polimerasi è attivato |
| 3 step ciclismo: denaturazione | 30 sec | 94 ° C | |
| Ricottura | 30 sec | 60 ° C 56 ° C | Per il DNA genomico Per bisolfito DNA convertito |
| Estensione | 30 sec | 72 ° C | |
| Numero di cicli | 45 | ||
| Estensione finale | 10 min | 72 ° C |
Tabella 2. PCR Specifiche ciclabili.
Discussione
Sono stati commercializzati diversi nuovi metodi di sequenziamento di nuova generazione. Tre dei metodi più usati sono ioni semiconduttori sequenziamento, di singola molecola tempo reale sequenziamento, e sequenziamento per sintesi (SBS). 3-11 Ciascuno di questi metodi dipende sequenziamento per sintesi ma impiegano piattaforme innovative per la rilevazione dei nucleotidi incorporati. In ione semiconduttori sequenziamento, un singolo filamento di DNA serve come modello per il filamento sequenziamento. Polimerasi e 12 nucleotidi vengono aggiunti sequenzialmente come nella pyrosequencing. Quando viene aggiunto un nucleotide al filamento di DNA crescente, uno ione idrogeno viene rilasciato. Lo ione idrogeno viene rilevata da un sensore basato transistor ad effetto di campo.
Molecola singola tempo reale sequenziamento dipende dalla modalità di guida d'onda zero (ZMW). 13 La ZMW è una piccola struttura dove una singola molecola polimerasi è attaccata al fondo del pozzo. Viene illuminato in modo tale che un floreprofumo molecola può essere rilevato. Ogni nucleotide è etichettato con una molecola fluorescente. Come un nucleotide fluorescente con tag è incorporato, un rivelatore identifica il nucleotide. Quando è aggiunto il seguente nucleotide, la molecola fluorescente viene scissa. 14
Sequenziamento per sintesi (SBS) utilizza un metodo unico per amplificare il DNA bersaglio tale che i gruppi di sequenze uniche sono generati. 15 singoli modelli stranded sono generati e quindi il filamento complementare è sintetizzato. Ciascun nucleotide è etichettato con una molecola fluorescente e dopo ogni aggiunta di base la fluorescenza la base aggiunta è registrato.
Divulgazioni
Produzione e libero accesso a questo articolo è sponsorizzato da Qiagen.
Autori Ahmed, Durocher, Jessen e Vardi sono dipendenti di Qiagen Inc. che produce reagenti e strumenti utilizzati in questo articolo.
Riconoscimenti
Questo progetto è stato di supporto in parte dalla Johns Hopkins University, Ufficio del prevosto tramite il gateway Science Initiative e Qiagen Inc.
Materiali
| Name | Company | Catalog Number | Comments |
| PyroMark PCR Master Mix, 2x | Qiagen | 978703 | |
| CoralLoad Concentrate, 10x | Qiagen | 978703, 978705 | |
| Q-Solution, 5x | Qiagen | 978703, 978705 | |
| MgCl2, 25 mM | Qiagen | 978703, 978705 | |
| RNase-Free Water | Qiagen | 129112 | |
| Primers | Qiagen | 978776 or 978777 | Primers should be purchased from an established oligonucleotide manufacturer (i.e. QIAGEN Pyromark Custom Assays, IDT, etc). Lyophilized primers should be dissolved in TE to provide a stock solution of 100 μM. |
| Pyromark Gold Q24 Reagents | Qiagen | 970802 | |
| High-purity water (Milli-Q 18.2 MΩ x cm or equivalent) | |||
| Streptavidin Sepharose High Performance Beads | GE Healthcare | 17-5113-07 | |
| PyroMark Annealing Buffer | Qiagen | 979009 | |
| PyroMark Denaturation Solution | Qiagen | 979007 | |
| PyroMark Wash Buffer | Qiagen | 979008 | |
| PyroMark Q24 | Qiagen | 9001514 | |
| PyroMark Q24 Cartridge | Qiagen | 979202 | |
| PyroMark Q96 HS Tip Holder Box | Qiagen | 979105 | |
| PyroMark Q24 Vacuum Workstation | Qiagen | 9001516 | |
| PyroMark Q24 Plate Holder | Qiagen | 9022273 | |
| Orbital shaker | Fisher | 11-675-297 | |
| Heat block | Fisher | 11-718Q |
Riferimenti
- PyroMark PCR Handbook. , [about 27p.]. Available from: http://www.qiagen.com/handbooks (2009).
- PyroMark Q24 User Manual. , [about 27p.]. Available from: http://www.qiagen.com/handbooks (2012).
- Elahi, E., Ronaghi, M. Pyrosequencing: a tool for DNA sequencing analysis. Methods Mol. Bio. 255, 211-219 (2004).
- Fakhrai-Rad, H., Pourmand, N., Ronaghi, M. Pyrosequencing: and accurate detection platform for single nucleotide polymorphisms. Hum. Mutat. 19 (5), 479-485 (2002).
- Langaee, T., Ronaghi, M. Genetic variation analyses by pyrosequencing. Mutat. Res. 573 (1-2), 96-102 (2005).
- Liu, Z., Lozupone, C., Hamady, M., Bushman, F. D., Knight, R. Short pyrosequencing reads suffice for accurate microbial community analysis. Nucleic Acids Res. 35 (18), e120(2007).
- Novais, R. C., Thorstenson, Y. R. The evolution of Pyrosequencing for microbiology: From genes to genomes. J. Microbiol. Methods. 86 (1), 1-7 (2011).
- J, Metagenomic pyrosequencing and microbial identification. Clin. Chem. 55 (5), 856-866 (2009).
- Quince, C., Lanzen, A., Curtis, T. P., Davenport, R. J., Hall, N., Head, I. M., Read, L. F., Sloan, W. T. Accurate determination of microbial diversity from 454 pyrosequencing data. Nat. Methods. (9), 639-6341 (2009).
- Ronaghi, M., Karamohamed, S., Pettersson, B., Uhlen, M., Nyren, P. Real-time DNA sequencing using detection of pyrophosphate release. Anal. Biochem. 242, 84-89 (1996).
- King, C., Scott-Horton, T. Pyrosequencing: A simple method for accurate genotyping. J. Vis. Exp. (11), e630(2008).
- Rothberg, J. W., Heinz,, et al. An integrated semi-conductor device that enabling non-optical genome sequencing. Nature. 475, 348-352 (2011).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-Mode Waveguides for Single-Molecule Analysis at high concentrations. Science. 299, 682-686 (2003).
- Eid, J., Fehr, A. Real-Time DNA Sequencing from Single Polymerase Molecules. Science. 323, 133-138 (2009).
- Sequencing Technology | Sequencing by Synthesis | Illumina [Internet]. , Illumina. Available from: http://www.illumina.com/technology/sequencing_technology.ilmn (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon