Method Article
Pyrosequencing für Mikrobielle Identifizierung und Charakterisierung
In diesem Artikel
Zusammenfassung
Pyrosequencing ist eine vielseitige Technik, die mikrobielle Genom-Sequenzierung, die verwendet werden, um bakterielle Spezies identifizieren, diskriminieren Bakterienstämme und erkennen genetische Mutationen, die eine Resistenz gegen antimikrobielle Mittel werden erleichtert. In diesem Video, wird das Verfahren für die mikrobielle Amplikon Generation Amplikon pyrosequencing und DNA-Sequenzanalyse nachgewiesen werden.
Zusammenfassung
Pyrosequencing ist eine vielseitige Technik, die mikrobielle Genom-Sequenzierung, die verwendet werden, um bakterielle Spezies zu identifizieren, zu unterscheiden und zu erkennen Bakterienstämme genetische Mutationen, die eine Resistenz gegen antimikrobielle Mittel werden erleichtert. Die Vorteile für die Mikrobiologie Pyrosequenzierungs Anwendungen sind schnelle und zuverlässige Hochdurchsatz-Screening und genaue Identifizierung der Mikroorganismen und mikrobielle Genmutationen. Pyrosequencing betrifft die Sequenzierung von DNA durch Synthetisieren des komplementären Strangs einer einzigen Basisstation zu einer Zeit, bei der Ermittlung der spezifischen Nukleotids während der Synthesereaktion eingearbeitet. Die Reaktion erfolgt an immobilisierten einzelsträngigen Matrizen-DNA, wo die vier Desoxyribonukleotide (dNTP) nacheinander zugegeben und nicht-eingebauten dNTPs enzymatisch abgebaut werden vor der Zugabe der nächsten dNTP der Synthesereaktion. Nachweis der spezifischen Base in der Vorlage enthalten ist durch Erzeugung chemilum überwachtinescent Signale. Die Reihenfolge der dNTPs, die die Chemilumineszenz zu erzeugen bestimmt die DNA Sequenz der Matrize. Die Echtzeit-Sequenzierung Fähigkeit Pyrosequenzierungstechnologie ermöglicht eine schnelle Identifizierung von Mikroorganismen in einem einzigen Assay. Darüber hinaus ist die Pyrosequencing Instrument, analysieren die volle genetische Vielfalt der antimikrobiellen Resistenzen, einschließlich Typisierung von SNPs, Punktmutationen, Insertionen, Deletionen und sowie Quantifizierung mehrerer Genkopien, die in irgendeiner Antibiotikaresistenz auftreten können Mustern.
Einleitung
Pyrosequencing ist eine schnelle und genaue Methode zur Sequenzierung von Nukleinsäuren, die auf dem Prinzip der "Sequenzierung durch Synthese" basiert. "Sequenzierung durch Synthese" beinhaltet die Verwendung einer Einzelstrang-DNA-Vorlage, um den komplementären Strang einer Basisstation zu einer Zeit zu synthetisieren, und Erfassen der eingebaute Base (A, T, G oder C) bei jedem Schritt durch die Erfassung eines Chemilumineszenz-Signals. Die Reaktion umfasst Pyrosequenzierungs Matrizen-DNA, dNTPs (dATP, dGTP, dTTP, dCTP), DNA-Polymerase, ATP-Sulfurylase, Luciferin, Luciferase und Apyrase. Die Synthesereaktion wird durch die Zugabe von einer der vier dNTPs und DNA-Polymerase an den biotinylierten einzelsträngigen Matrizen-DNA initiiert. DNA-Polymerase enthält die komplementären dNTP auf die Schablone, mit der anschließenden Freisetzung von Pyrophosphat (Abbildung 1). ATP Sulfurylase anteilig wandelt das Pyrophosphat zu ATP. ATP wirkt als Katalysator für die Luciferase vermittelte Umwandlung von Luciferin zu Oxyluciferin, dasLicht erzeugt (Abbildung 2). Die Intensität des Lichts ist proportional zu der Anzahl von Nucleotiden enthalten und bestimmt, ob ein oder mehrere spezifische dNTPs (dATP, dTTP, dGTP, dCTP oder) ist auf dem Template-Strang sequentiell (Abbildung 3). Jede unincorporated dNTP abgebaut Apyrase vor der Zugabe der nächsten dNTP für die Fortsetzung der Synthesereaktion. Dieses "Sequenzierung durch Synthese" Reaktion wird unter Zusatz von jedem der vier dNTPs wiederholt, bis die DNA-Sequenz der einzelsträngigen DNA-Matrize bestimmt.
Um eine Animation der Pyrosequencing Reaktion sehen Klicken Sie hier, um Film anzusehen .
Protokoll
Einleitung: Eine einzelne Bakterienkolonien sollte verwendet werden, um einen geeigneten Kulturmedium, das über Nacht inkubiert impfen werden. Eine bakterielle Pellet wird dann verarbeitet, um genomische DNA unter Verwendung eines kommerziell erhältlichen genomischen Isolierungskit reinigen. Das gereinigte genomische DNA sollte einen 260/280 (nm)-Verhältnis> 1.8, um sicherzustellen, dass die Probe frei von Protein-Kontamination ist. Die Menge an genomischer DNA zur Erzeugung des Pyrosequenzierungs Schablone erforderlich ist 10-20 ng.
A. Amplifikation der Template
(PyroMark PCR Handbook; www.qiagen.com / Handbücher 1)
Einrichten der PCR-Reaktion
Hinweis: Eine Grundierung muss an seinem 5'-Ende biotinyliert werden, und es wird empfohlen, dass es HPLC gereinigt werden für die Template Vorbereitung. Wir empfehlen die PyroMark Assay-Design Software 2.0 für PCR und seSequenz Primer-Design, die für kurz-Lese-Sequenzierung optimiert sind, jedoch Primer-BLAST ( http://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome Dose) von NCBI auch für die Primer-Design verwendet werden.
- Tauen Sie alle erforderlichen Lösungen (aufgeführt in Tabelle 1) und mischen Sie gründlich. Richten Sie die Reaktion gemäß Tabelle 1. Alle Arbeiten können bei Raumtemperatur durchgeführt werden.
Hinweis: Die PCR Master Mix enthält MgCl 2 für eine endgültige Konzentration von 1,5 mM, die zufriedenstellende Ergebnisse liefert in den meisten Fällen. Wenn eine höhere Mg 2 +-Konzentration benötigt wird, kann bis zu 3,5 ul 25 mM MgCl 2 zu einer Reaktion zugegeben werden.
- Mischen Sie die Reaktion Mischung durch vorsichtiges Auf-und Abpipettieren, dann verzichtet werden entsprechende Volumina in PCR-Röhrchen.
- In template DNA zu jedem PCR-Röhrchen. Wir empfehlen 10 ng genomischer DNA.
- Programmieren Sie den Thermocycler gemäß Tabelle 2. Eine abschließende Halten bei 4 ° C wird der Einfachheit halber empfohlen. Wenn Sie einen Thermocycler ohne beheizte Deckel Reaktionen Overlay mit 100 ul Mineralöl.
- Legen Sie die PCR-Röhrchen in den Cycler und starten Sie das Programm.
B. Vacuum Workstation Protocol
Immobilisierung des biotinylierten PCR-Produkte
- Machen Sie einen Master-Mix nach Abbildung 4.
Hinweis: Vor dem Pipettieren, schütteln die Flasche Streptavidinbeschichteten Sepharosekügelchen um eine homogene Suspension zu gewährleisten.
- Abhängig vom Volumen des PCR-Produkts verwendet wird, verzichtet 60-75 ul Master-Mix in jede Vertiefung einer PCR-Platte, um ein Gesamtvolumen von 80 ul pro Vertiefung geben.
- In 5-20 ul biotinylierten PCR-Produkt in jede Vertiefung.
- Verschließen Sie die Vertiefungen mit Streifen ca.ps und rühren die PCR-Platte bei 1.400 rpm für 5-10 min bei Raumtemperatur (15-25 ° C) mit einem Schüttler.
Denaturierung von DNA und Zugabe von Sequenzierungsprimer
- Verdünnen Sie die Sequenzierungsprimern bis 0,3 um mit Annealingpuffer und verzichten 25 ul in jede Vertiefung einer Reaktion Platte. Positionieren Sie die Platte auf der Arbeitsstation.
- Füllen Sie die Workstation Mulden nach der Abbildung 5 Diagramm.
- Schalten Sie die Vakuumpumpe ein, und stellen Sie sicher, dass der Schalter auf der Vakuum-Werkzeug, um auf (Abbildung 6). Spülen Sie die Filter-Sonden mit hochreinem Wasser (Milli-Q 18,2 MW x cm oder gleichwertig) in Wanne 5 (Abbildung 5). Füllen Sie die Wanne mit frischem hochreinem Wasser für die Verwendung in Schritt 12.
- Arbeiten schnell, positionieren Sie den PCR-Platte mit Perlen auf der Arbeitsstation. Stellen Sie sicher, dass die beiden Platten in der gleichen Richtung sind, wenn die Proben geladen.
- Mit dem Vakuum-Schalter ON, senken das VakuumWerkzeug in die Vertiefungen des PCR-Platte für 20 Sekunden oder bis alle Volumen wurde abgesaugt. Die Perlen werden durch das Vakuum-Werkzeug (Abb. 7) erfasst werden.
- Mit dem Vakuum noch auf, spülen Sie das Tool mit 70% Ethanol (Trog 1) für 20 Sekunden oder bis alle Volumen wurde abgesaugt.
- Mit Vakuum-ON, spülen Sie das Werkzeug mit Denaturierungslösung (Wanne 2) für 20 sec oder bis alle Volumen wurde abgesaugt.
Hinweis: Die Denaturierungsreagenz enthält Natriumhydroxid, das eine Auge und Haut reizend ist. Tragen Sie immer einen Kittel, Handschuhe und Schutzbrille tragen.
- Mit Vakuum-ON, spülen Sie das Tool mit Waschpuffer (Trog 3) für 20 sec oder bis alle Volumen wurde abgesaugt.
- Mit Vakuum-ON, heben Sie den Vakuum-Tool, über 90 ° vertikal für 5 Sekunden, damit alle Flüssigkeit abtropfen aus dem Vakuum-Tool. Schalten Sie die Pumpe AUS, und richten Sie die Vakuum-Tool mit der Reaktion Platte. Senken Sie den Vakuum-Werkzeug in derBrunnen und sanft hin und her zu schütteln, um die Kügelchen in die Vertiefungen, die Sequenzierungsprimer freizugeben.
- Um das Vakuum Werkzeug reinigen, schüttelt die Filter Sonden in Reinstwasser (Wanne 4) für 10 Sekunden.
- Schalten Sie das Vakuum ON und spülen Sie das Tool mit hochreinem Wasser (Wanne 5) für 20 sec oder bis alle Volumen angesaugt worden ist.
- Heben Sie das Vakuum Werkzeug, um mehr als 90 ° C für 5 sec vertikal, dann schalten Sie das Vakuum ab und lagern in der "Parking"-Position.
Glühen Sequenzierungsprimer um DNA-Stränge
- Legen Sie die Reaktion Platte in einer vorgewärmten Platte Inhaber.
- Anschließend wird die Platte auf einem Heizblock bei 80 ° C für 2 min.
- Entfernen Sie die Platte aus der Halterung und auf dem Zähler bei Raumtemperatur für 5 min abkühlen.
PyroMark Q24 Betrieb
Hinweis: Schalten Sie das Gerät über den Netzschalter über das Netzkabel entfernt. Sterntup kann bis zu 1 min.
- Füllen Sie eine Patrone gemäß den Anweisungen in der PyroMark Gold-Q24 Reagenzien Handbook 2 vorgesehen. Um Volumina benötigt, bis eine run-Datei in der Gerätesoftware eingestellt zu bestimmen, und unter dem Menü Extras wählen Pre Run Informationen
- Wenn das Gerät bereits auf, sicherzustellen, dass sie nicht auf der Bühne einen Lauf und dann den Deckel öffnen. Ein akustisches Warnsignal wird ausgegeben, wenn der Deckel geöffnet wird, wenn es nicht sicher ist, dies zu tun.
- Öffnen Sie die Patrone Gate und setzen Sie die Patrone mit dem Etikett nach vorne (Abbildung 8). Stellen Sie sicher, dass die Patrone richtig eingelegt ist (eine Linie sichtbar sein soll an der Front) und schließen Sie die Tür.
- Öffnen Sie die Platte-Halterahmen und positionieren Sie die Platte mit Proben im Inneren des Gerätes.
- Schließen Sie die Platte-Halterahmen und das Instrument Deckel (Abbildung 9).
Ab einen Lauf
- Stecken Sie den USB-Stick mit dem run-Dateis erstellt mit der Gerätesoftware in den USB-Port an der Vorderseite des Gerätes.
- Wählen Sie "Ausführen" im Hauptmenü und drücken Sie "OK".
- Verwenden Sie die Scroll-Pfeile, um die gewünschte Datei auszuwählen run und drücken Sie "Select".
Hinweis: Das Gerät beginnt Abgabe Reagenzien, wenn alle vorgegebenen Druck-und Temperaturniveaus erreicht werden (dies kann einige Minuten dauern). Während eines Laufs, zeigt das Gerät den Bildschirm des ausgewählten Pyrogramm auch in Echtzeit. Verwenden Sie die Scroll-Pfeile, um die Pyrogramm anderer Brunnen zu sehen.
Abschließen einer run
- Wenn das Instrument bestätigt, dass der Lauf beendet ist und die run-Datei auf USB-Stick gespeichert wurden, drücken Sie auf "Schließen".
- Entfernen Sie den USB-Stick. Wenn es entfernt wurde, bevor der Lauf beendet ist, stecken Sie den USB-Stick. Wählen Sie "Verwaltung" und dann "Copy gespeicherte Runs".
- Die run-Datei kann nun geöffnet und analysiert werden mit der Gerätesoftware und verglichenzu einer Bibliothek von bekannten Mikroben mit der Identifire Software.
Ergebnisse
Die Ergebnisse eines typischen pyrosequencing Lauf mit der Software zeigt die Lage der Vorwärts-und Rückwärtsprimer Amplikons für Generation verwendet wird, und der Sequenzierungsprimer die pyrosequencing Reaktion innerhalb der 16S ribosomalen Sequenz (10). Die hypervariable Sequenz zwischen dem Primer Verbindungsstellen ermöglicht Identifizierung von Bakterien aus einer Vielzahl von Bakterienarten mit dem konservierten Sequenzierung (5'-TACATGCAAGTCGA). Als interne Qualitätskontrolle, die DNA-Sequenz Klammerung der variable Bereich überprüft werden, um sicherzustellen, dass die korrekte DNA-Bereich analysiert wurde . Die Pyrosequenzierungs Software ermöglicht den Vergleich und Ausrichtung der erzeugten Sequenz in einer internen Datenbank der bakteriellen ribosomalen Sequenzen für Identifizierung von Bakterien. Darüber hinaus kann eine zu analysierende Sequenz von Mutationen, die Antibiotikum-Resistenz verleihen bestimmen. Beispielsweise Analyse von Mutationen in der 23S ribosomalen Gene HelicobActer pylori zeigt zwei Mutationsmuster (GAA oder AGA), die Antibiotika-Resistenz (Abbildung 11). Die Analyse der verschiedenen Isolate vom gleichen Patienten können gleichzeitig getestet werden, um die Entstehung von Resistenzen im Laufe der Zeit in epidemiologischen Untersuchungen von Resistenzen gegen antimikrobielle Arzneimittel Ausbrüche helfen zu verfolgen.
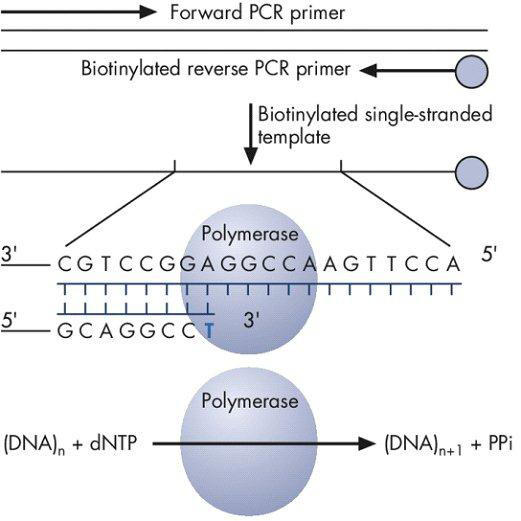
Abbildung 1. Biotinylierte PCR-Produkt wird als Vorlage dNTPs durch DNA-Polymerase enthalten, was zur Erzeugung von Pyrophosphat (PPi) verwendet.
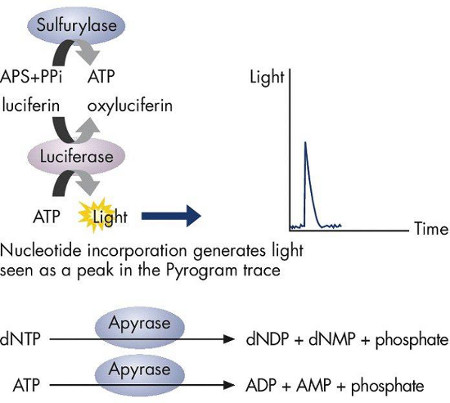
Abbildung 2. ATP Sulfurylase anteilig wandelt Pyrophosphat zu ATP. ATP wirkt als Katalysator für die Luciferase-vermittelte Umwandlung von Luciferin zu Oxyluciferin, das Licht, das Pro ist erzeugtproportional zu der Menge an ATP. Das Licht wird als Höhepunkt am Pyrogramm Trace aufgezeichnet und zeigt Nucleotideinbaus. Nicht-eingebauten dNTPs abgebaut von Apyrase, bevor die nächste dNTP für die Fortsetzung der Synthese zugegeben wird.
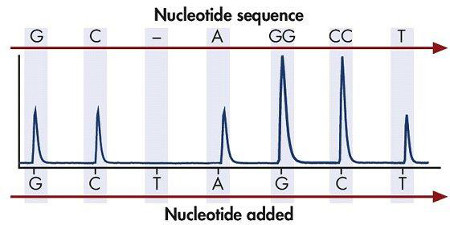
Abbildung 3. Die Intensität des Lichts generiert wird, wenn eine oder mehrere spezifische dNTPs (dATP, dTTP, dGTP, dCTP oder) auf dem Template-Strang eingebaut wurde nacheinander.
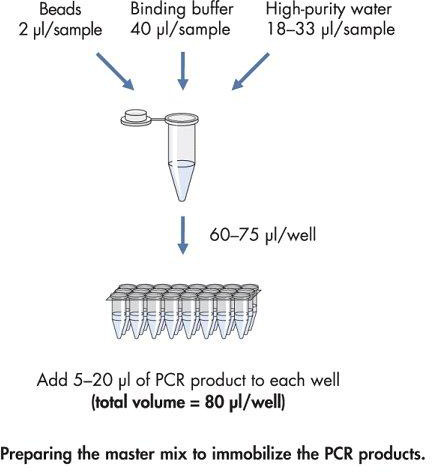
Abbildung 4. Ablaufschema für die Vorbereitung der Master-Mix, um die biotinylierten PCR-Produkt zu fixieren.
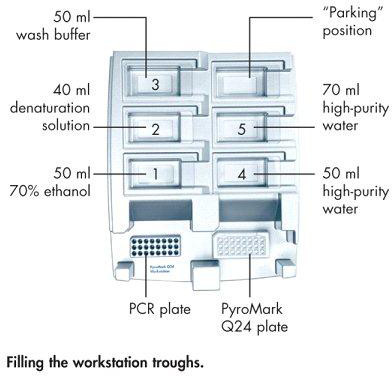
ge = "always"> Abbildung 5. PyroMark Workstation mit PCR-Platte, Platte PyroMark und Trog Standorten.

Abbildung 6. Standorte der Vakuum-Schalter ON und OFF Positionen.
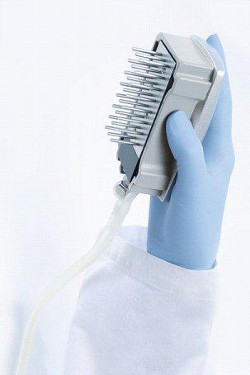
Abbildung 7. Vacuum-Tool. Richtige Handhabung der Vakuum-Tool.

Abbildung 8. Cartridge Tor geöffnet mit Patrone im Ort.

Abbildung 9. Cartridge richtig eingesetzt mit geschlossenem Tor.
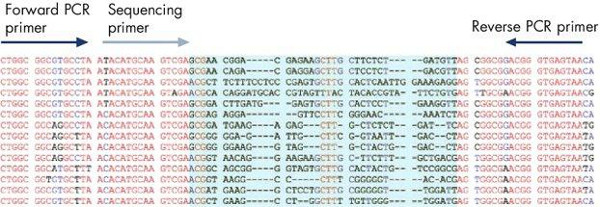
Abbildung 10. Pyrosequencing basierte Identifizierung von Bakterien führt. PCR-Primer für konservierte Regionen der DNA-Matrize ausgebildet ist und der Sequenzierprimer unmittelbar stromaufwärts eines gut charakterisierten Identifizierung hypervariablen DNA-Sequenz innerhalb des Amplikons (blau) angeordnet ist.
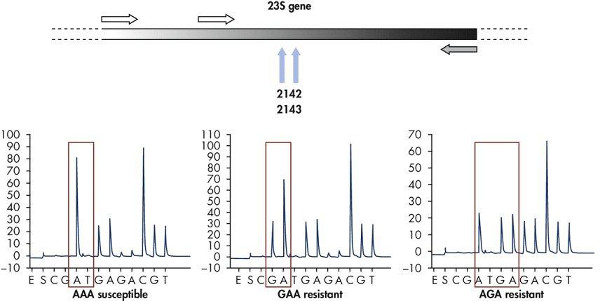
Abbildung 11. Nachweis von Antibiotika-Resistenzen in Helicobacter pylori mit pyrosequencing. Analyse von Mutationen in den Genen, die 23S antibakterielle Resistenz in Helicobacter pylori mit GAA oder AGA-Sequenzen. Klicke hier, um eine größere Abbildung anzuzeigen .
| Komponente | Volumen pro Reaktion | Endkonzentration |
| Pyro PCR Master Mix, 2x | 12,5 ul | 1x |
| CoralLoad Konzentrat, 10x | 2,5 ul | 1x |
| 25 mM MgCl 2 (optional) | Variable | ≥ 1,5 mM |
| Q-Lösung, 5x (optional) | 5 ul | 1x |
| Primer A / B Primer | Variable / Variable | 0,2 μM/0.2 uM |
| RNase-freies Wasser | Variable | - |
| Gesamtvolumen (nach Zugabe von DNA) | 25 ul |
Tabelle 1. PCR-Reaktionsgemisch.
| & Nbsp; | Zusätzliche Kommentare | ||
| Initial PCR Aktivierung Schritt | 15 min | 95 ° C | HotStartTaq DNA Polymerase aktiviert |
| 3 Schritt Radfahren: Denaturierung | 30 sec | 94 ° C | |
| Glühen | 30 sec | 60 ° C 56 ° C | Für genomische DNA Für Bisulfit-konvertierter DNA |
| Erweiterung | 30 sec | 72 ° C | |
| Anzahl der Zyklen | 45 | ||
| Abschließende Verlängerung | 10 min | 72 ° C |
Tabelle 2. PCR-Zyklus Spezifikationen.
Diskussion
Mehrere neue Next Generation Sequencing Methoden wurden kommerzialisiert. Drei der am häufigsten verwendeten Methoden sind Ionen-Halbleiter-Sequenzierung, einzelnes Molekül Echtzeit-Sequenzierung und Sequenzierung durch Synthese (SBS). 3-11 Jede dieser Methoden hängt Sequenzierung durch Synthese, sondern beschäftigen neuartige Plattformen für den Nachweis der eingebauten Nukleotide. In Ion Halbleiter-Sequenzierung, dient ein einzelner DNA-Strang als Vorlage für die Sequenzierung Strang. Polymerase und 12 Nukleotide nacheinander wie in pyrosequencing aufgenommen. Wenn ein Nukleotid auf die wachsende DNA-Strang hinzugefügt wird, wird ein Wasserstoffion freigegeben. Die Wasserstoffionenkonzentration wird durch einen Feldeffekttransistor Basis erfasst.
Einzel-Molekül Echtzeit-Sequenzierung abhängig von der Null-Modus Wellenleiter (ZMW). 13 Die ZMW ist eine kleine Struktur, wo ein einzelnes Molekül der Polymerase Boden des Schachtes befestigt ist. Es wird in einer solchen Weise, dass ein Flore beleuchtetDuft-Molekül erkannt werden können. Jedes Nukleotid mit einem fluoreszierenden Molekül markiert. Als fluoreszierend markierten Nukleotid eingebaut identifiziert ein Detektor die Nukleotidsequenz. Wenn die nächste Nukleotid hinzugefügt wird, wird das fluoreszierende Molekül abgespalten. 14
Sequenzierung durch Synthese (SBS) nutzt eine einzigartige Methode zur Amplifikation der Ziel-DNA, so dass Cluster einzigartige Sequenzen erzeugt werden. 15 Einzel-Matrizen erzeugt werden und dann der komplementäre Strang synthetisiert wird. Jedes Nukleotid mit einem fluoreszierenden Molekül markiert ist und nach jeder Basenzugabe die Fluoreszenz des zugesetzten Base wird aufgezeichnet.
Offenlegungen
Produktion und den freien Zugang zu diesem Artikel von der Firma Qiagen gesponsert.
Autoren Ahmed, Durocher, Jessen und Vardi sind Mitarbeiter von Qiagen Inc., die Reagenzien und Instrumente in diesem Artikel verwendet produziert.
Danksagungen
Dieses Projekt war die Unterstützung teilweise durch Johns Hopkins University, Büro des Provost durch die Gateway-Initiative Wissenschaft im Dialog und Qiagen Inc.
Materialien
| Name | Company | Catalog Number | Comments |
| PyroMark PCR Master Mix, 2x | Qiagen | 978703 | |
| CoralLoad Concentrate, 10x | Qiagen | 978703, 978705 | |
| Q-Solution, 5x | Qiagen | 978703, 978705 | |
| MgCl2, 25 mM | Qiagen | 978703, 978705 | |
| RNase-Free Water | Qiagen | 129112 | |
| Primers | Qiagen | 978776 or 978777 | Primers should be purchased from an established oligonucleotide manufacturer (i.e. QIAGEN Pyromark Custom Assays, IDT, etc). Lyophilized primers should be dissolved in TE to provide a stock solution of 100 μM. |
| Pyromark Gold Q24 Reagents | Qiagen | 970802 | |
| High-purity water (Milli-Q 18.2 MΩ x cm or equivalent) | |||
| Streptavidin Sepharose High Performance Beads | GE Healthcare | 17-5113-07 | |
| PyroMark Annealing Buffer | Qiagen | 979009 | |
| PyroMark Denaturation Solution | Qiagen | 979007 | |
| PyroMark Wash Buffer | Qiagen | 979008 | |
| PyroMark Q24 | Qiagen | 9001514 | |
| PyroMark Q24 Cartridge | Qiagen | 979202 | |
| PyroMark Q96 HS Tip Holder Box | Qiagen | 979105 | |
| PyroMark Q24 Vacuum Workstation | Qiagen | 9001516 | |
| PyroMark Q24 Plate Holder | Qiagen | 9022273 | |
| Orbital shaker | Fisher | 11-675-297 | |
| Heat block | Fisher | 11-718Q |
Referenzen
- PyroMark PCR Handbook. , [about 27p.]. Available from: http://www.qiagen.com/handbooks (2009).
- PyroMark Q24 User Manual. , [about 27p.]. Available from: http://www.qiagen.com/handbooks (2012).
- Elahi, E., Ronaghi, M. Pyrosequencing: a tool for DNA sequencing analysis. Methods Mol. Bio. 255, 211-219 (2004).
- Fakhrai-Rad, H., Pourmand, N., Ronaghi, M. Pyrosequencing: and accurate detection platform for single nucleotide polymorphisms. Hum. Mutat. 19 (5), 479-485 (2002).
- Langaee, T., Ronaghi, M. Genetic variation analyses by pyrosequencing. Mutat. Res. 573 (1-2), 96-102 (2005).
- Liu, Z., Lozupone, C., Hamady, M., Bushman, F. D., Knight, R. Short pyrosequencing reads suffice for accurate microbial community analysis. Nucleic Acids Res. 35 (18), e120(2007).
- Novais, R. C., Thorstenson, Y. R. The evolution of Pyrosequencing for microbiology: From genes to genomes. J. Microbiol. Methods. 86 (1), 1-7 (2011).
- J, Metagenomic pyrosequencing and microbial identification. Clin. Chem. 55 (5), 856-866 (2009).
- Quince, C., Lanzen, A., Curtis, T. P., Davenport, R. J., Hall, N., Head, I. M., Read, L. F., Sloan, W. T. Accurate determination of microbial diversity from 454 pyrosequencing data. Nat. Methods. (9), 639-6341 (2009).
- Ronaghi, M., Karamohamed, S., Pettersson, B., Uhlen, M., Nyren, P. Real-time DNA sequencing using detection of pyrophosphate release. Anal. Biochem. 242, 84-89 (1996).
- King, C., Scott-Horton, T. Pyrosequencing: A simple method for accurate genotyping. J. Vis. Exp. (11), e630(2008).
- Rothberg, J. W., Heinz,, et al. An integrated semi-conductor device that enabling non-optical genome sequencing. Nature. 475, 348-352 (2011).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-Mode Waveguides for Single-Molecule Analysis at high concentrations. Science. 299, 682-686 (2003).
- Eid, J., Fehr, A. Real-Time DNA Sequencing from Single Polymerase Molecules. Science. 323, 133-138 (2009).
- Sequencing Technology | Sequencing by Synthesis | Illumina [Internet]. , Illumina. Available from: http://www.illumina.com/technology/sequencing_technology.ilmn (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten