Method Article
Configuración de un microscopio de luz Hoja simple para
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo se describe la configuración de un microscopio de lámina de luz y su aplicación para la visualización in vivo de C. elegans embriones.
Resumen
Técnicas de imagen fototóxicas rápidos y bajos son pre-requisito para estudiar el desarrollo de los organismos en su totalidad. Microscopía basada Calderería ligera reduce foto-blanqueo y efectos fototóxicas en comparación con la microscopía confocal, al tiempo que proporciona imágenes en 3D con resolución subcelular. Aquí presentamos la configuración de una hoja con base microscopio de luz, que se compone de un microscopio vertical y un pequeño conjunto de elementos opto-mecánicos para la generación de la hoja de la luz. El protocolo se describe cómo crear, alinear el microscopio y caracterizar la lámina de luz. Además, detalla cómo implementar el método para la formación de imágenes en su totalidad de la C. elegans embriones utilizando una cámara simple observación. El método permite la captura de dos colores 3D-películas a intervalos durante las pocas horas de desarrollo. Esto debería facilitar el seguimiento de la forma celular, divisiones celulares y proteínas etiquetadas durante largos períodos de tiempo.
Introducción
La formación y conformación de un organismo implica movimientos celulares, cambios en la forma celular, la división celular y la diferenciación celular. La comprensión de la progresión y coordinación de estos procesos requieren herramientas de imágenes rápidas con capacidad de tres dimensiones y la resolución subcelular. Entre las técnicas in vivo con estas capacidades de imagen en, hoja de luz de iluminación microscopía también llamada selectiva microscopía simple / iluminación avión tiene la ventaja única de la producción de efectos de baja fototóxicas y bajo fotoblanqueo 1,2.
En la iluminación microscopía hoja, la muestra se ilumina desde el lado por una lámina de luz, el cual realiza el seccionamiento óptico. La trayectoria de iluminación y la trayectoria de detección son perpendiculares entre sí y la lámina de luz se alinea para que coincida con el plano focal objeto de la lente del objetivo de detección. La muestra se coloca en la intersección de los dos caminos, unand se puede mover a lo largo del eje de detección para permitir que las imágenes en 3D. Sólo el plano de interés está iluminado y todos los puntos de este plano se detectan al mismo tiempo. Esto aumenta significativamente la velocidad de adquisición y reduce la fotodecoloración, así como fototoxicidad en comparación con la microscopía confocal 3.
Prácticamente, la microscopía de iluminación lámina es fácil de configurar y align: para una imagen bidimensional, no de exploración es necesaria ni para la parte de detección, ni por la parte de iluminación; como una técnica de campo amplio con capacidad de seccionamiento, de formación de imágenes en tres dimensiones se puede conseguir por un solo movimiento del eje de la etapa de la celebración de la muestra.
Aquí presentamos la creación de un microscopio simple lámina de luz y su aplicación en imágenes toto de C. elegans embriones. C. elegans embriones están bien adaptados a vivir en vivo imaging debido a su transparencia, el linaje invariante y estereotipada celular posiciones 4,5. La configuración de lámina de luz desarrollado se basa en un microscopio vertical estándar para la detección. El protocolo presentado a continuación detalla cómo construir y alinear la excitación módulo / path, probar y caracterizar la hoja de la luz y el montaje de la muestra de imágenes en 3D. También proporciona ejemplos de películas a intervalos adquiridos con la instalación en varias cepas que expresan marcadores fluorescentes.
Protocolo
1. Configuración de la Hoja de Ruta de la Luz y la detección
La figura 1 presenta un esquema general de la configuración óptica.
- Colocar y fijar el microscopio y los láseres en la mesa óptica.
- Combinar los láseres de 488 nm, 561 nm y 405 nm con los espejos dicroicos correspondientes. Tras los espejos dicroicos, los láseres tienen que ser alineados conjuntamente con precisión. Tenga en cuenta que la elección de las longitudes de onda de láser, espejos dicroicos, y filtros de luz se determina a partir de los espectros de absorción de los marcadores fluorescentes específicos utilizados en los experimentos.
- Coloque el filtro sintonizable acústico-óptico (AOTF). Utilice la hoja de prueba proporcionado por el proveedor para ajustar la frecuencia y la potencia de la AOTF. Cuando se alinean bien, aproximadamente el 90% de la potencia del láser se transmite a un máximo de sintonización según lo medido por un medidor de potencia. Tenga en cuenta que algunas impresoras láser pueden ser totalmente controlados por software, lo que podría hacer que el AOTF innecesario.
- Colocar y fijarel telescopio. Tenga en cuenta que el telescopio se compone de dos lentes que expanden el haz a una anchura igual o más grande que el diámetro de la abertura posterior de la lente objetivo iluminación. La lente con la longitud focal más pequeño debe ser lo primero.
- Place, alinear y fijar el periscopio llevar hacia arriba el camino óptico más cerca de la abertura posterior de la lente cilíndrica. Tenga en cuenta que los dos espejos de la periscopio están montados de una manera que su eje largo está sentando en la inclinación del espejo con el fin de reflejar el haz completamente expandido.
- Coloque la lente cilíndrica junto a la salida de periscopio. Tenga en cuenta que la lente cilíndrica enfoca el haz en una dirección y deja colimada en la otra dirección, formando de ese modo una lámina de luz.
- Centrar la hoja de luz resultante mediante el uso de la lente del objetivo de la iluminación. Tenga en cuenta que el punto focal de la imagen del objetivo iluminación debe coincidir con el punto focal objeto del objetivo de detección. Proyecte la vigaen una pantalla (por ejemplo, una pared) a una larga distancia. Mueva el objetivo de iluminación a lo largo del eje del haz de iluminación para obtener en la pantalla afilados contornos de la lámina de luz.
- Coloque la línea de cuatro (405/488/561/638) filtro de emisión en el lugar reservado en el microscopio (cubo).
- Coloque la cámara EMCCD en el puerto de salida de microscopio.
- Fijar la primera etapa de traducción manual sobre la platina del microscopio con tornillos en los agujeros perforados en la platina del microscopio. Tenga en cuenta que el borde de la etapa de traducción tiene que estar situado a unos 2 cm de distancia del centro de la platina del microscopio, de manera que la cubeta está centrado debajo de la lente del objetivo de detección.
- Fijar la segunda etapa de traducción manual sobre la primera. Las direcciones de las dos fases tienen que ser perpendicular (X e Y) traducciones.
- Fijar la etapa Z piezoeléctrico en la parte superior de las dos etapas anteriores. Tenga en cuenta que una etapa motorizada puede ser utilizada en lugar de la etapa piezoeléctrico.
- Fijar el holde cubetaR (véase la figura 2) en la parte superior de las etapas. La cubeta tanto, se coloca en la intersección de la trayectoria de iluminación y la trayectoria de detección, que son perpendiculares entre sí.
2. Comprobación de la Hoja de Luz
- Llene una cubeta de vidrio con una solución fluorescente, y colocarlo en el soporte de la muestra en la intersección de los dos caminos ópticos. La hoja de la luz es por lo tanto visible. Tiene que ser horizontal, y simétrica / centrada con respecto a la lente del objetivo de detección.
- Retire la lente cilíndrica y adquirir una imagen para medir el espesor de la lámina de luz. Tenga en cuenta que el espesor de la lámina de luz depende de la apertura numérica (NA) de la iluminación de la lente objetivo (alrededor de 3-4 m para NA = 0,3). Tenga en cuenta que para una lente dada, el espesor de la lámina de luz-se puede aumentar reduciendo el tamaño del haz de entrar en la abertura de vuelta con una hendidura. Tenga en cuenta que las microesferas fluorescentes pueden ser usados para medir el hacharesolución IAL y lateral del microscopio.
- Vuelva a colocar la lente cilíndrica antes de iniciar el experimento.
3. El montaje de C. elegans embriones
Los siguientes pasos se muestran en la Figura 3.
- Utilice una pluma de diamantes-marca para cortar un pedazo de un portaobjetos de vidrio de 10 x 20 x 1 mm.
- Preparar 5% bacto-agar en agua, hervirla y mantenerla en un bloque calefactor a 60 º C.
- Añadir 50 l de poli-L-lisina en un lado de la pieza cortada de diapositivas, deje que se seque.
- Coloque un portaobjetos de microscopio en el banco y arreglarlo.
- Yuxtaponer la pieza cortada de la diapositiva con la corredera fija y añadir una gota de agar derretido en la parte superior. Apriete el agar utilizando otro portaobjetos limpio para obtener una almohadilla fina de agar.
- Deje que la almohadilla de agar se seque antes de retirar el carro superior.
- Corte la almohadilla de agar en el lado de la corredera fija con el fin de dejar un exceso de aproximadamente 3 mm Of agar y remover suavemente la corredera fija.
- Añadir 50 l de poli-L-lisina en el agar y dejar que se seque por completo.
- Poner gusanos grávidas en medio M9 en un vidrio de reloj.
- Cortar los gusanos a nivel de la vulva con un escalpelo para liberar los embriones.
- Bajo un binocular identificar los embriones en la etapa de interés y recoger ellos mediante el uso de una pipeta microcapilar.
- Coloque los embriones en la parte del agar recubiertos con poli-L-lisina que sobresale de la pieza cortada de la corredera. Colocar los embriones justo al lado de la frontera de la diapositiva. No colocar los embriones en la frontera del agar porque no van a ser lo suficientemente estable. Retire con cuidado el líquido de modo que los embriones se pegarán en la capa de poli-L-lisina.
- Alinear rápidamente los embriones para evitar la desecación.
- Colocar el portaobjetos corte en un plato de plástico Petri y agregar suavemente medio M9 para cubrir la diapositiva.
- Revise debajo de un binocular que los huevos todavía están atrapados en el agar.
4. De grabación de C. Desarrollo elegans
- Fijar la preparación anterior (diapositiva corte, agar, más embriones) en el soporte de la muestra y colocarlo en una cubeta. Figura 4 describe el soporte casero y la figura 5 muestra una vista de la cubeta en el contexto de observación.
- Fijar la cubeta en el escenario piezoeléctrico.
- Identificar la posición de los embriones con iluminación de campo brillante.
- Inicie el software de fabricación casera controlar el AOTF, la cámara y el escenario piezoeléctrico. Tenga en cuenta que un software libre, como micromanager, también se puede utilizar.
- Seleccione la línea láser y la potencia (AOTF).
- Ajuste la posición de la hoja de la luz utilizando la etapa 3 direcciones apoyar la lente cilíndrica y la lente de la iluminación. Mover la etapa a lo largo de la dirección lateral (X) para obtener la señal más brillante. Ajuste entonces la posición Z de la hoja de la luz, y la posición longitudinal (Y) para obtener ªe mejor relación señal-ruido. Tenga en cuenta que puede necesitar la posición del foco de lámina de luz que ajustarse de un embrión a otro para corregir las diferencias en longitudes de trayectoria de luz.
- Para adquirir imágenes con lapso de tiempo, seleccione el tiempo de exposición, la ganancia, el tiempo entre dos imágenes, así como el número de imágenes que se deben adquirir, y la potencia del láser. Para la adquisición de color 2, seleccione una segunda línea de láser. Dos imágenes en color se adquieren de forma consecutiva.
- Para adquirir pila az, seleccione también la distancia entre los dos sectores, y el principio y final posiciones del piezoeléctrico.
Resultados
Tres experimentos de grabación de lapso de tiempo en 3D a partir de tres diferentes C. elegans cepas ilustran el tipo de datos que se puede obtener mediante la configuración del microscopio de lámina de luz descrito anteriormente. Comprobamos que en estas condiciones los embriones se desarrollan a una velocidad normal y sobrevivir a la proyección de imagen, de acuerdo con informes anteriores que indican que las imágenes SPIM causa sólo bajos niveles de fototoxicidad en C. elegans embriones 6,7.
La primera cepa expresa una histona fusionado a GFP (zuIs178; stIs10024). De grabación se llevó a cabo durante 2 horas con una pila de 20 rebanadas (distancia entre rebanadas 1 M) tomado cada 37 seg. La figura 6 ilustra un plano de la pila a diferentes puntos de tiempo. Ambos núcleos interfásicos (flecha) y la cromatina mitótico condensada (cabeza de flecha) son claramente visibles.
La segunda cepa expresa una tubulina fusionado a GFP (ruIs57) y una histonafusionado a mCherry (itIs37; stIs10226). De grabación se realizó durante 16 min con una pila de 20 rebanadas (distancia entre rebanadas 1 M) tomado cada 105 seg. La Figura 7 ilustra diferentes planos de la pila y diferentes puntos de tiempo. La película asociado 1 pantallas 3D reconstrucciones en 3 puntos de tiempo sucesivos. Durante la división, el huso mitótico (verde) y los cromosomas condensados (rojo) son claramente visibles lo que permite el seguimiento de las orientaciones de la división celular durante el desarrollo.
La tercera cepa expresa la apolipoproteína VIT-2 fusionado a GFP (pwIs23) y una membrana obligado mCherry (ltIs44). De grabación se realizó durante 25 min con una pila de 10 rebanadas (distancia entre rebanadas 1 M) tomado cada 30 seg. Figura 8 ilustra diferentes planos de la pila y diferentes puntos de tiempo. Los movimientos rápidos de las partículas de lipoproteína de yema (verde) dentro de las células pueden ser fácilmenteseguido.
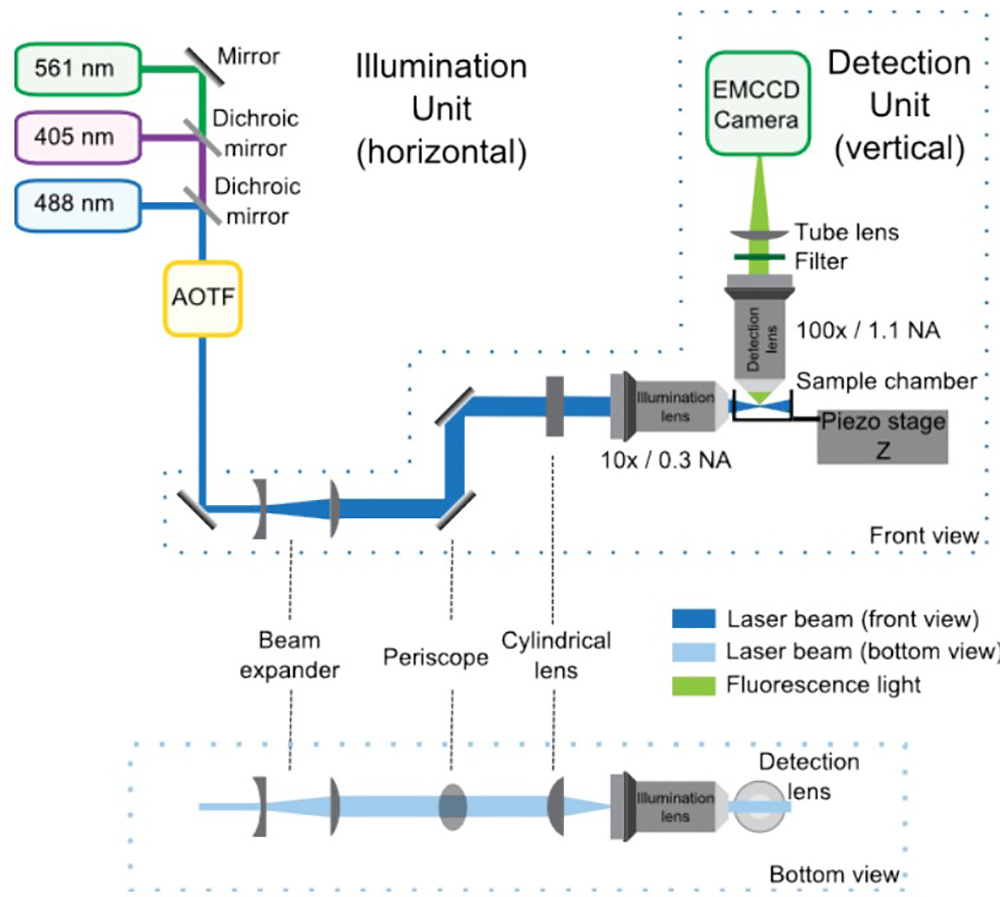
Figura 1: Configuración óptica SPIM compuesto por las unidades de iluminación y detección, delantera y vistas inferiores. En la unidad de iluminación, los láseres combinados por los espejos dicroicos entran en el AOTF, que controla la potencia de cada láser independientemente. Entonces, el telescopio aumenta el tamaño de la viga por 4 veces y el periscopio lo lleva a la altura del microscopio. La lente cilíndrica forma la lámina de luz, que se reorientó por el objetivo de la iluminación. La unidad de detección está integrado en el microscopio vertical y se compone principalmente por la lente de la detección, el filtro, la lente del tubo y la EMCCD. La muestra se coloca en la intersección entre las trayectorias de iluminación y de detección. Una etapa piezoeléctrico permite (Z) desplazamientos verticales de la muestrapara la adquisición 3D.
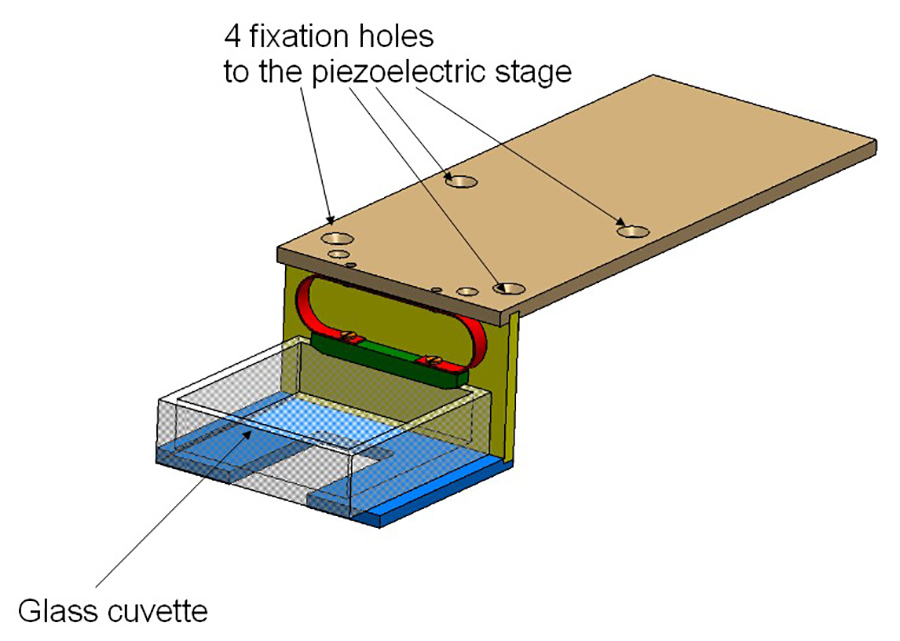
Figura 2:. Portacubetas El titular se compone de 3 placas de aluminio y se atornilla en el escenario piezoeléctrico. La cubeta de vidrio se lleva a cabo por un muelle (en rojo).
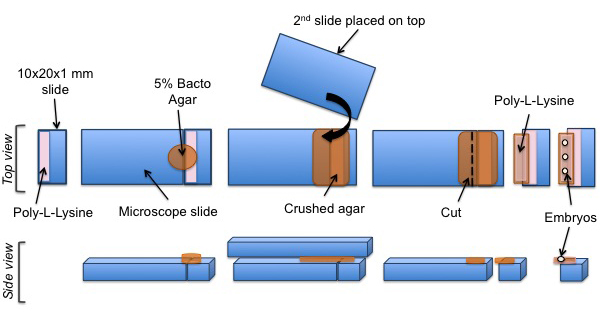
Figura 3: Montaje de protocolo de C. embriones elegans para experimentos SPIM. Una pieza cortada de vidrio está recubierta con poli-L-lisina. Una almohadilla de agar se coloca sobre la superficie recubierta. Poli-L-lisina se añade a continuación, en la almohadilla de agar. Finalmente, C. elegans embriones se alinean en la almohadilla de agar recubiertos con poli-L-lisina.
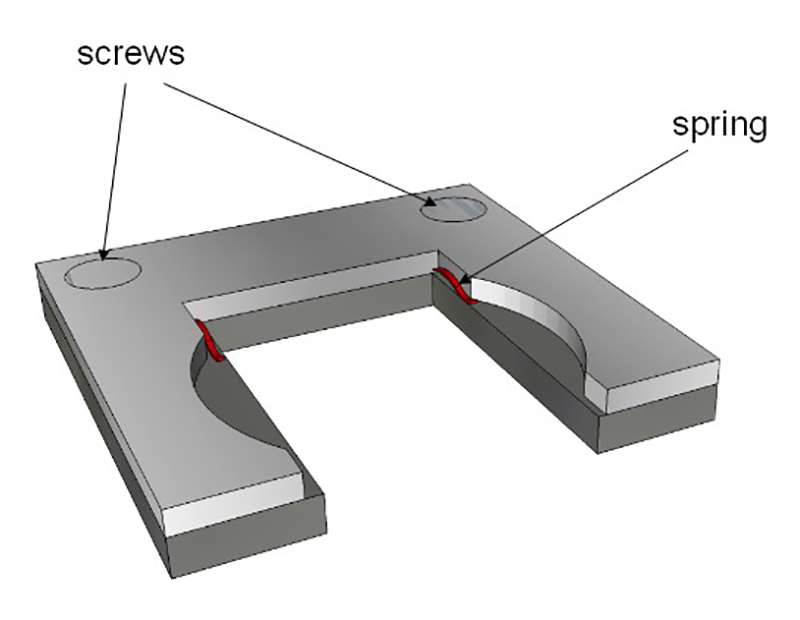
Figura 4: Soporte de la muestra. El soporte se ajusta a las dimensiones interiores de la cubeta de vidrio. Dos resortes (en rojo) mantienen el portaobjetos de vidrio (descrito en la Figura 3). Este soporte se pone entonces en el interior de la cubeta de vidrio.
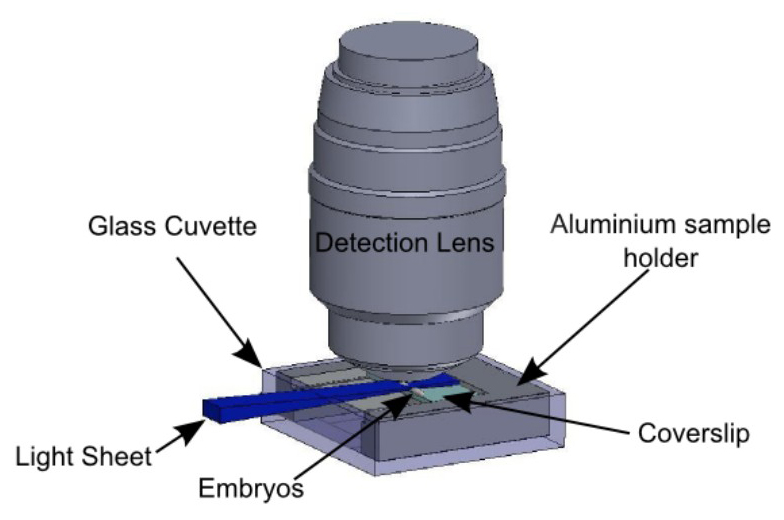
Figura 5:... Soporte de la muestra en el contexto de observación embriones se inmoviliza sobre el portaobjetos de vidrio de acuerdo con el protocolo resumido en la Figura 3 El portaobjetos se soporta por el soporte de la muestra se describe en la Figura 4 el soporte de muestra se inserta en la cubeta de vidrio, en poder del titular describe en la Figura 2. La lámina de luz es horizontal y illuminates los embriones desde el lado. La lente del objetivo de detección es vertical, por encima de la muestra.
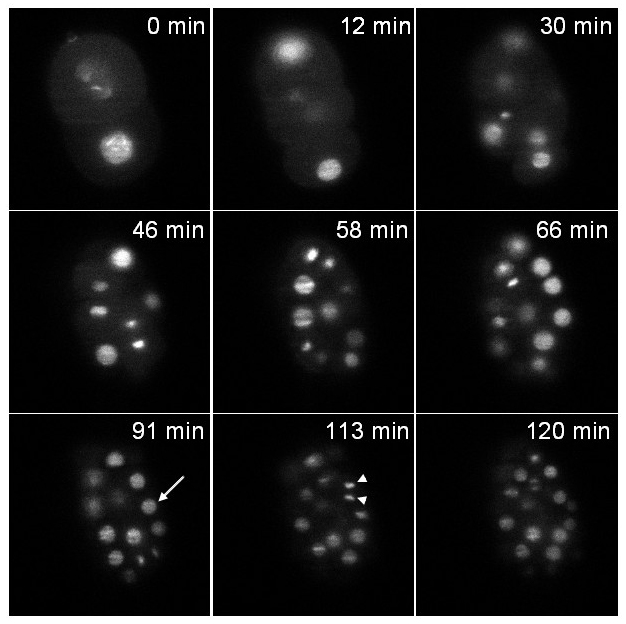
Figura 6: La grabación de un C. elegans embrión expresa una fusión histona :: GFP imágenes planas individuales de un embrión transgénico que expresa una histona :: GFP fusión (zuIs178; stIs10024) a diferentes puntos de tiempo Imágenes extraídas de una grabación de lapso de tiempo 3D:.. pilas hechas de 20 rebanadas ( distancia entre rodajas de 1 M); tiempo de exposición por porción: 30 ms; intervalo de tiempo entre las pilas: 37 seg; tiempo de adquisición en total: 2 hr. Flecha: núcleo interfásicos, punta de flecha: condensa la cromatina mitótico. En t = 0 min el embrión está en la etapa de 2 células y en t = 120 min embrión contiene alrededor de 70 células como se esperaba bajo s de desarrollo normalespis (en cuenta que sólo las células contenidas en un solo plano son visibles en las imágenes y no todas las células del embrión). El poder de la iluminación fue de 30 mW a 488 nm (medida a la salida del objetivo de la iluminación). Por favor, haga clic aquí para ver el vídeo.
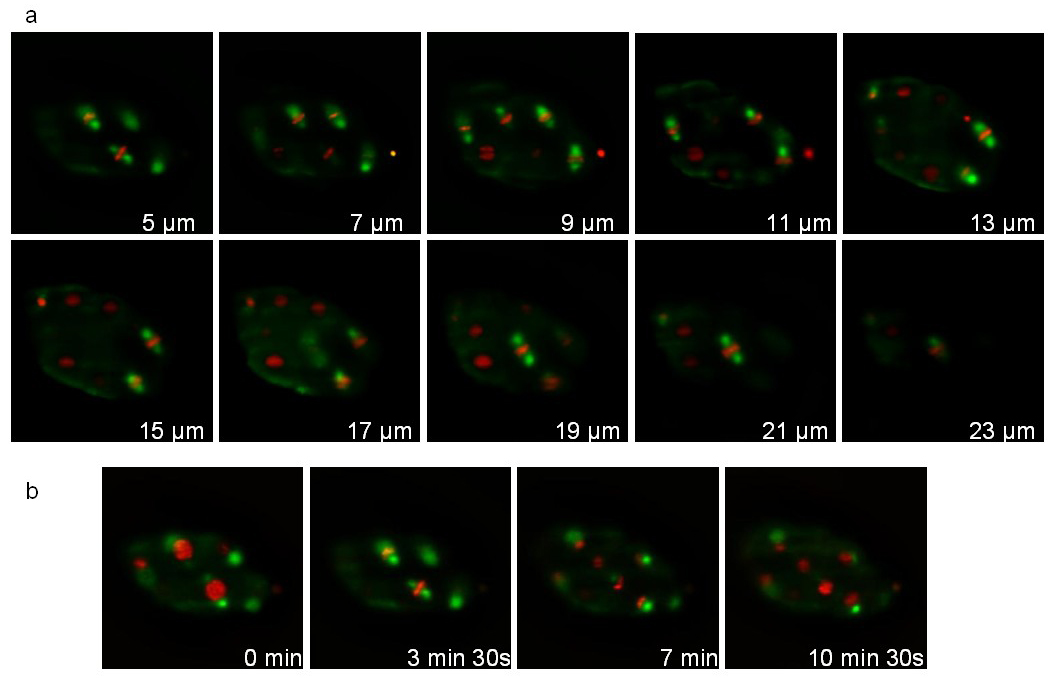
Figura 7: La grabación de un C. elegans embrión expresa una tubulina :: GFP fusión y una histona :: fusión mCherry. Imágenes extraídas de una grabación a intervalos regulares en 3D de una C. elegans embrión expresa una tubulina :: GFP fusión (ruIs57) y una histona :: fusión mCherry (itIs37; stIs10226). Condiciones de grabación: adquisición de una pila de 20 rebanadas (distancia entre rebanadas 1m) cada 105 segundos; tiempo de adquisición total: 16 min; tiempo de exposición: 200 mseg para cada canal. El panel A muestra 10 rebanadas de la misma pila. El panel B muestra la misma rebanada cada 210 seg. El poder de la iluminación fue de 30 mW y 300 mW, para 488 y 561 láseres nm respectivamente (medición en la salida del objetivo de iluminación). Por favor, haga clic aquí para ver el vídeo.
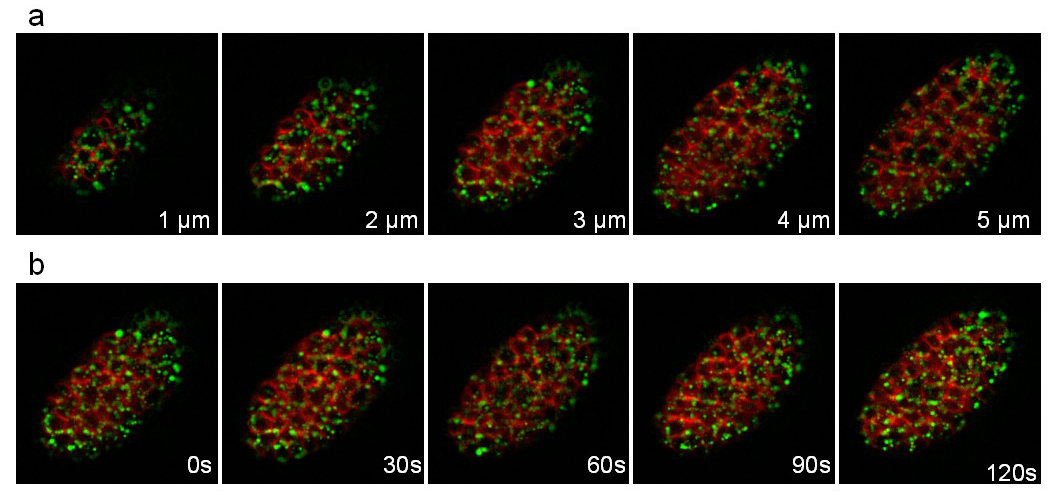
Figura 8: La grabación de un C. elegans embrión expresa una VIT-2 :: GFP fusión y una membrana orientada mCherry. Imágenes extraídas de una grabación a intervalos regulares en 3D de una C. elegans embrión que expresa una fusión VIT-2 :: GFP (PWIs23) y un mCherry membrana orientada (ltIs44). Condiciones de grabación: adquisición de una pila de 10 rebanadas (distancia entre rebanadas 1 mM) cada 30 segundos; tiempo de adquisición total: 25 min; tiempo de exposición por canal: 200 ms. El panel A muestra las 5 primeras rebanadas de la misma pila. El panel B muestra el mismo corte cada 30 seg. Haga clic aquí para ver el vídeo.
Discusión
Este protocolo describe una fácil configuración de la microscopía de lámina de luz para obtener imágenes in vivo de C. elegans embriones. Los elementos ópticos necesarios para crear y alinear la lámina de luz, incluyendo el láser, expansor de haz, colimación y lentes de enfoque, se pueden montar fácilmente en un banco óptico. En combinación con un microscopio vertical para la trayectoria de detección y la cámara, lo que proporciona una solución sencilla para la configuración de un microscopio de lámina de luz.
Esta geometría hace que el entorno de ejemplo simple. El espécimen vivo se coloca en una cubeta de vidrio, que se celebra, colocado y movido por un pequeño conjunto de piezas mecánicas. Como microscopía iluminación general es una técnica de campo ancho, con capacidad de seccionamiento, sólo se requiere un único eje motorizado movimiento para obtener imágenes en 3D. Esto limita el requisito de sincronización a un único elemento de exploración y facilita el desarrollo de software. En comparación con ISPIM 6 nuestra geomet verticalRY permite el uso de alta lente objetivo NA para la detección. En comparación con las configuraciones de SPIM con lentes de objetivo en el plano horizontal el montaje de la muestra y la obtención de imágenes de una serie de embriones son más fáciles. En total, la aplicación de esta técnica es sencilla y se puede lograr con poca experiencia en la óptica. Esta es una alternativa más barata a los microscopios confocal con las ventajas de una mayor velocidad y menor fototoxicidad pero inconveniente de una resolución axial inferior.
También hemos desarrollado una manera fácil y reproducible para montar C. embriones elegans para imágenes in vivo usando este sistema. El procedimiento de montaje es rápido (15 min) y es adecuado para los experimentos de la vida cotidiana. Para C. imágenes elegans, las ventajas de esta configuración simple por lo menos dos pliegues: (i) en imágenes toto es posible sin rotación y por lo tanto la reconstrucción 3D es sencillo; (Ii) el montaje de la muestra es fácil y embriones múltiples se pueden obtener imágenes de forma secuencialen la misma diapositiva. Mostramos ejemplos de películas a intervalos con la resolución temporal suficiente para observar las divisiones celulares y cambios en la forma celular. El poder de la microscopía de lámina de luz para estudiar C. elegans desarrollo también se ha ilustrado recientemente por otros 6, 7, 8. Por lo tanto, esta técnica será muy útil para estudiar la morfogénesis y el patrón de la C. embrión elegans.
¿Puede este sistema también se utiliza para analizar el desarrollo de otros organismos modelo? Es importante reconocer que la aplicación de la iluminación microscopía de hoja para organismo pequeño y transparente, tal como C. elegans es menos estricto que para los organismos más grandes, como los embriones de Drosophila. En las imágenes toto de Drosophila normalmente requiere la rotación y de vistas múltiples reconstrucciones 9, 10. Excitación por dos lados con lentes objetivas de excitación colineales también pueden reducirse los efectos de sombra causado por la attenuation de la luz 11. Sin embargo, la configuración que se presenta aquí está todavía adaptado a la imagen de una cara de los embriones de Drosophila, con una baja de blanqueo y bajo photoxicity. Esta configuración también podría ser útil para imagen pequeña y transparentes embriones como ascidias o pez cebra.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Damos las gracias a todos los miembros de la Lenne y laboratorios de Bertrand, en particular, Olivier Blanc para el desarrollo de software y Jérémie Capoulade para la discusión. También agradecemos a la instalación de imágenes PICsL-IBDM, en particular, Claude Moretti y Brice Detailleur para soporte técnico.
Este trabajo fue apoyado por becas ATIP del CNRS (a p-FL y VB), el Labex INFORM subvención (a P.-FL y VB) y una subvención de Sanofi-Aventis (a VB). Reconocemos la infraestructura France-BioImaging apoyado por la Agence Nationale de la Recherche (ANR-10-INSB-04-01, llamado "Investissements d'Avenir").
Materiales
| Name | Company | Catalog Number | Comments |
| SPIM | |||
| Detection | |||
| upright microscope | ZEISS | Axiophot 1 | |
| 100x W objective (NA 1.1, W.D. 2.5mm) | NIKON | ||
| EMCCD back illuminated | ANDOR | DU-885K-CS0-#VP | |
| Illumination | |||
| LASER 488 nm / 60 mW | TOPTICA | iBeam smart | |
| LASER 405 nm / 120 mW | TOPTICA | iBeam smart | |
| LASER 561 nm / 500 mW | COBOLT | JIVE 500 | for time lapse imaging, 50 or 100mW is enough |
| filter Quad line laser rejectionband ZET 405/488/561/638 | AHF | F57-405 | |
| Dichroic 405 | AHF | F38-405 | |
| Dichroic 488 | SEMROCK | Di01-R488 | |
| AOTF | AA | AOTFnC-400.650-TN | |
| mirror | |||
| lens 50 mm | THORLABS | telescope 5X | |
| lens 200 mm | THORLABS | telescope 5X | |
| periscope | THORLABS | RS99/M | |
| cylindrical lens f=100mm | THORLABS | ACY254-100-A | |
| 10X NA 0,3 | NIKON | ||
| xyz stage | NEWPORT | ||
| Sample | |||
| Bacto Agar | Bectkton Dickinson | 214010 | 5% in water |
| Poly-L-Lysine solution | Sigma | P8920 | |
| Capillaries GC100F-10, 1.0mm oD, 0,58mm ID, 100 mm L | Havard Appartus | 30-0019 | |
| Aspiration tube (mouth pipette) | Dutsher | 55005 | |
| M9 medium: | |||
| KH2PO4 (3g/L) | Any supplier | ||
| Na2HPO4 (6g/L) | Any supplier | ||
| NaCL (5g/L) | Any supplier | ||
| MgSO4 (1 mM final) | Any supplier | ||
| cuvette holder | HOME MADE | made by Brice Detailleur | |
| sample holder | HOME MADE | made by Claude Moretti | |
| x stage | NEWPORT | ||
| coverslip 50x20x1 mm | MARIENFELD | cut then in 10x20x1mm | |
| piezo electric stage with amplifier/controller | PI | P-622.ZCD / E-625.CR | |
| cuvette 40mmx40mmx10mm | HELLMA | 704.002-OG.40 mm- 10mm high | |
| Diamond Point Glass Marking Pencil | VWR | ||
| Software | |||
| C++ (Qt) home made software | developed by Olivier Blanc and Claire Chardès, available on request. Alternative solutions: micromanager or labview | ||
Referencias
- Huisken, J., Swoger, J., Del Bene, F., Wittbrodt, J., Stelzer, E. H. Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305, 1007-1009 (2004).
- Huisken, J., Stainier, D. Y. Selective plane illumination microscopy techniques in developmental biology. Development. 136, 1963-1975 (2009).
- Reynaud, E. G., Krzic, U., Greger, K., Stelzer, E. H. Light sheet-based fluorescence microscopy: more dimensions, more photons, and less photodamage. HFSP J. 2, 266-275 (2008).
- Sulston, J. E., Schierenberg, E., White, J. G., Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Dev Biol. 100, 64-119 (1983).
- Hardin, J. Imaging embryonic morphogenesis in C. elegans. Methods Cell Biol. 106, 377-412 (2011).
- Wu, Y., et al. Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans. Proc Natl Acad Sci U S A. 108, 17708-17713 (2011).
- Giurumescu, C. A., Kang, S., Planchon, T. A., Betzig, E., Bloomekatz, J., Yelon, D., Cosman, P., Chisholm, A. D. Quantitative semi-automated analysis of morphogenesis with single-cell resolution in complex embryos. Development. 139, 4271-4279 (2012).
- Gao, L., Shao, L., Higgins, C. D., Poulton, J. S., Peifer, M., Davidson, M. W., Wu, X. F. Noninvasive Imaging beyond the Diffraction Limit of 3D Dynamics in Thickly Fluorescent Specimens. Cell. 151, 1370-1385 (2012).
- Krzic, U., Gunther, S., Saunders, T. E., Streichan, S. J., Hufnagel, L. Multiview light-sheet microscope for rapid in toto imaging. Nat Methods. 9, 730-733 (2012).
- Tomer, R., Khairy, K., Amat, F., Keller, P. J. Quantitative high-speed imaging of entire developing embryos with simultaneous multiview light-sheet microscopy. Nat Methods. 9, 755-763 (2012).
- Huisken, J., Stainier, D. Y. Even fluorescence excitation by multidirectional selective plane illumination microscopy (mSPIM). Opt Lett. 32, 2608-2610 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados