Method Article
Einrichten eines Simple Light Noten Mikroskop für
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt den Aufbau eines Lichtschnittmikroskop und dessen Umsetzung zur in vivo Bildgebung von C. elegans Embryonen.
Zusammenfassung
Schnelle und phototoxische bildgebenden Verfahren sind Voraussetzung, um die Entwicklung von Organismen in toto zu studieren. Lichtbogen basiert Mikroskopie reduziert Fotobleichen und phototoxische Effekte im Vergleich zu der konfokalen Mikroskopie und bietet 3D-Bilder mit subzellulärer Auflösung. Hier stellen wir den Aufbau einer Lichtschnittmikroskop basiert, die von einem aufrechten Mikroskop und einen kleinen Satz von opto-mechanischen Elementen für die Erzeugung des Lichtblattes zusammengesetzt ist. Das Protokoll beschreibt, wie zu bauen, richten Sie das Mikroskop und prägen das Lichtblatt. Außerdem enthält es, wie die Methode in toto Abbildung C zu implementieren elegans Embryonen mit Hilfe eines einfachen Beobachtungskammer. Das Verfahren ermöglicht die Erfassung von 3D-Zwei-Farben Zeitraffer-Filme über einige Stunden der Entwicklung. Dies sollte die Verfolgung der Zellform, Zellbereiche und markierte Proteine über lange Zeiträume zu erleichtern.
Einleitung
Die Ausbildung und Formgebung des Organismus beinhaltet Zellbewegungen, Änderungen der Zellform, der Zellteilung und Zelldifferenzierung. Das Verständnis der Progression und Koordination dieser Prozesse erfordern schnelle Imaging-Tools mit drei Dimensionen Fähigkeit und subzellulärer Auflösung. Zu den Techniken, mit diesen in-vivo-Imaging-Funktionen, Lichtblatt-Beleuchtung Mikroskopie auch als Einzel / selektiven Plane Illumination Mikroskopie hat den einzigartigen Vorteil der Herstellung von Low-phototoxische Effekte und Low-Photobleaching 1,2.
In Blatt Beleuchtung Mikroskopie wird die Probe von der Seite durch einen Lichtbogen, der die optische Schnitte führt beleuchtet. Der Beleuchtungspfad und der Detektionspfad sind senkrecht zueinander und der Lichtbogen wird ausgerichtet, um mit der Objektbrennebene der Objektivlinse Detektion zusammenfallen. Die Probe wird an dem Schnittpunkt der beiden Pfade angeordnet ist, einnd entlang der Erfassungsachse bewegt werden, um 3D-Bildgebung zu ermöglichen. Nur die interessierende Ebene beleuchtet wird und alle Punkte dieser Fläche werden gleichzeitig erfasst. Dies erhöht die Aufnahmegeschwindigkeit und verringert Photobleichen sowie Phototoxizität im Vergleich zur konfokalen Mikroskopie 3.
Praktisch ist Blatt Beleuchtung Mikroskopie einfach zu installieren und align: für zweidimensionale Bildgebung notwendig weder für den Erfassungsteil, noch für die Beleuchtungsteil ist kein Scannen; als Weitfeldtechnik mit Auftrennung kann die dreidimensionale Bildgebung durch eine einzige Achse der Bewegung des Bühnen Halten der Probe erhalten werden.
Hier präsentieren wir die Einrichtung eines einfachen Lichtmikroskop Blatt und seine Umsetzung in toto Bildgebung von C. elegans Embryonen. C elegans Embryonen sind gut geeignet, um in vivo ima lebenGing aufgrund ihrer Transparenz, invariant Abstammung und stereotype Zelle positioniert 4,5. Das entwickelte Lichtblatt-Setup basiert auf einem Standard aufrechten Mikroskop zur Erkennung basiert. Die nachstehende Protokoll beschreibt, wie zu bauen und richten Sie die Anregung Modul / Pfad, zu testen und zu charakterisieren den Lichtbogen und montieren Sie die Probe für die 3D-Bildgebung. Es bietet auch Beispiele für mit dem Setup auf verschiedenen Stämmen, fluoreszierende Marker erworben Zeitraffer-Filme.
Protokoll
1. Einrichten des Lichtblattes und der Erfassungspfad
Abbildung 1 zeigt ein allgemeines Schema der optischen Aufbau.
- Platzieren und befestigen Sie das Mikroskop und die Laser auf dem optischen Tisch.
- Kombinieren Laser 488 nm, 561 nm und 405 nm mit den entsprechenden dichroitischen Spiegeln. Nach der dichroitischen Spiegel haben die Laser genau miteinander ausgerichtet werden. Man beachte, dass die Auswahl der Laserwellenlängen, dichroitische Spiegel und optische Filter ist aus den Absorptionsspektren der bestimmten Fluoreszenzmarkierungen in den Experimenten verwendet wird, bestimmt.
- Legen Sie die akustisch-optische Filter (AOTF). Verwenden Sie das Testblatt des Lieferanten, die Frequenz und die Macht des AOTF einzustellen. Wenn gut ausgerichtet ist, etwa 90% der Laserleistung bei einer maximalen Abstimmung, wie durch einen Leistungsmesser gemessen übertragen. Beachten Sie, dass einige Laser kann vollständig durch Software gesteuert werden, welche der AOTF unnötig machen könnte.
- Zeigen und fixierendas Teleskop. Beachten Sie, dass das Teleskop aus zwei Linsen, die den Strahl auf eine Breite gleich oder größer als der Durchmesser des hinteren Apertur des Beleuchtungsobjektivlinse zusammen erweitern. Das Objektiv mit der kleinsten Brennweite zuerst kommen sollte.
- Platz, ausrichten und das Sehrohr nach oben bringen den optischen Weg näher an der Rückseite Öffnung der zylindrischen Linse. Beachten Sie, dass die beiden Spiegel des Periskop in einer Weise, dass ihre lange Achse wird mit auf die Neigung des Spiegels, um den vollständig expandierten Strahl zu reflektieren montiert.
- Setzen Sie die Zylinderlinse neben dem Periskop Ausgang. Beachten Sie, dass die Zylinderlinse fokussiert den Strahl in eine Richtung und lässt sie kollimiert in der anderen Richtung, wodurch ein leichtes Blatt bildet.
- Neuausrichtung der resultierende Lichtbogen mit Hilfe der Beleuchtung Objektiv. Beachten Sie, dass der Bildbrennpunkt des Beleuchtungsziel sollte mit dem Gegenstandsbrennpunkt des Erfassungsziel übereinstimmen. Projizieren Sie den Strahlauf einem Bildschirm (beispielsweise einer Mauer) in einem großen Abstand. Bewegen Sie die Beleuchtungsobjektivs entlang der Strahlachse Beleuchtung auf den Bildschirm scharfen Konturen des Lichtblattes erhalten.
- Legen Sie die Quad-Linie (405/488/561/638) Emissionsfilter in dem Ort, an dem Mikroskop (Würfel) reserviert.
- Legen Sie die EMCCD Kamera am Mikroskop Ausgang.
- Befestigen Sie die erste manuelle Übersetzung der Bühne auf dem Mikroskoptisch durch Schrauben in Löchern in den Mikroskoptisch gebohrt. Beachten Sie, dass der Rand der Übersetzungsstufe bei etwa 2 cm von der Mitte des Mikroskoptisches positioniert werden, so daß die Küvette unter der Nachweisobjektivlinse zentriert.
- Befestigen Sie die zweite manuelle Übersetzung Bühne, auf der ersten. Die Richtungen der beiden Stufen müssen senkrecht (X-und Y-Übersetzungen) sein.
- Befestigen Sie den Z-Piezotisch an der Spitze der beiden vorherigen Stufen. Beachten Sie, dass eine motorisierte Stufe kann anstelle des piezoelektrischen Bühne verwendet werden.
- Befestigen Sie die Küvette holder (siehe Abbildung 2) an der Spitze der Stufen. Die Küvette wird somit an der Kreuzung der Beleuchtungspfad und dem Erfassungspfad, die senkrecht zueinander sind, platziert werden.
2. Überprüfung des Lichtblattes
- Füllen einer Glasküvette mit Fluoreszenzlösung, und auf dem Probenhalter an dem Schnittpunkt der beiden optischen Wege. Der Lichtbogen ist somit sichtbar. Es hat horizontal ist, und symmetrisch / zentriert in Bezug auf die Detektionsobjektivlinse.
- Entfernen Sie die Zylinderlinse und erwerben Sie ein Bild, um die Dicke des Lichtblattes messen. Beachten Sie, dass die Dicke der Lichttafel hängt von der numerischen Apertur (NA) der Beleuchtungsobjektivlinse (etwa 3-4 &mgr; m für NA = 0,3). Beachten Sie, dass für eine gegebene Linse kann die Dicke der Licht-Folie durch die Verringerung der Größe des Strahls in die hintere Apertur mit einem Schlitz erhöht werden. Beachten Sie, dass fluoreszierende Mikrosphären können verwendet werden, um die Axt zu messenial und lateralen Auflösung des Mikroskops.
- Setzen Sie die Zylinderlinse vor dem Start des Experiments.
3. Die Montage C. elegans Embryonen
Die folgenden Schritte sind in Fig. 3 dargestellt.
- Verwenden eines diamantMarkierungsStift, um ein Stück einer Glasplatte von 10 x 20 x 1 mm geschnitten.
- Bereiten Sie 5% Bacto-Agar in Wasser kochen und pflegen sie in einem Heizblock bei 60 ° C
- Dann werden 50 &mgr; l Poly-L-Lysin auf einer Seite des Zuschnittes von Folie trocknen lassen.
- Legen Sie ein Mikroskop-Objektträger auf der Bank und zu beheben.
- Juxtapose den Schnitt Stück Folie mit dem festen Rutsche und einen Tropfen geschmolzenen Agar auf der Oberseite. Drücken Sie die Agar mit einem anderen sauberen Objektträger, um eine dünne Agar-Pad erhalten.
- Lassen Sie die Agar-Pad, um vor dem Entfernen der Oberschlitten trocknen.
- Schneiden Sie die Agar-Pad auf der Seite der festen Folie, um einen Überschuss von etwa 3 mm o verlassenf-Agar und entfernen sanft die feste Folie.
- In 50 ul von Poly-L-Lysin auf dem Agar und lassen Sie es vollständig trocknen.
- Setzen trächtige Würmer in M9-Medium in einem Uhrglas.
- Schneiden die Würmer auf der Ebene der Vulva mit einem Skalpell, um die Embryonen zu lösen.
- Unter einem Fernglas erkennen Sie die Embryonen im Stadium von Interesse und sammeln Sie sie mit Hilfe eines Mikrokapillar Pipette.
- Zeigen die Embryonen auf dem Teil des Agars mit Poly-L-Lysin, das von dem Schnittstück des Schiebers ragt beschichtet. Legen Sie die Embryonen nur an der Grenze der Folie. Die Embryonen auf dem Rand des Agar positionieren nicht, weil sie nicht stabil genug. Entfernen Sie vorsichtig die Flüssigkeit, so dass die Embryonen werden auf der Poly-L-Lysin-Schicht-Stick.
- Schnell richten Sie die Embryonen, um ein Austrocknen zu vermeiden.
- Setzen Sie den Schnitt Folie in einer Petrischale aus Kunststoff und sanft hinzufügen M9-Medium, um die Folie zu bedecken.
- Überprüfen unter einem Binokular, dass die Eier noch auf den Agar fest.
4. Aufnahme von C. elegans Entwicklung
- Befestigen Sie die vorherige Vorbereitung (Schnitt Rutsche, Agar-Agar und Embryonen) in den Probenhalter und legen Sie sie in eine Küvette. Abbildung 4 beschreibt die hausgemachten Halter und Abbildung 5 zeigt eine Ansicht der Küvette in Beobachtung Kontext.
- Befestigen Sie die Küvette auf dem piezoelektrischen Bühne.
- Ermitteln Sie die Position der Embryonen mit Hellfeldbeleuchtung.
- Starten Sie das hausgemachte Software-Steuerung der AOTF, die Kamera und den piezoelektrischen Bühne. Beachten Sie, dass eine freie Software, wie z. B. Mikromanager, können ebenfalls verwendet werden.
- Wählen Sie die Laserlinie und Leistung (AOTF).
- Stellen Sie die Position des Lichtblattes mit der Stufe 3 Richtungen Unterstützung der Zylinderlinse und der Beleuchtungslinse. Bewegen der Plattform entlang der seitlichen Richtung (X), um die hellste Signal zu erhalten. Stellen Sie dann die Z-Position des Lichtblattes und die Längsposition (Y), um th erhaltene beste Signal-Rausch-Verhältnis. Beachten Sie, dass die Position des Lichtblattes Fokus muss möglicherweise von einem Embryo zu einem anderen eingestellt werden, um Unterschiede in der Lichtpfadlängen zu korrigieren.
- Zeitraffer-Bilder zu erwerben, wählen Sie die Belichtungszeit, zu gewinnen, Zeit zwischen zwei Bildern, als auch die Anzahl der Bilder, die erworben werden sollen, und die Laserleistung. Für zwei Farberfassung, wählen Sie eine zweite Laserlinie. Zwei Farbbilder werden nacheinander erworben.
- Um az-Stack zu erwerben, wählen Sie auch den Abstand zwischen den zwei Scheiben, und die Anfangs-und Endposition des piezoelektrischen.
Ergebnisse
Drei 3D Zeitrafferaufnahme aus drei verschiedenen Experimenten C. elegans-Stämmen veranschaulichen die Art von Daten, die unter Verwendung des oben beschriebenen Lichtschnittmikroskopaufbau erhalten werden können. Wir überprüften, dass unter diesen Bedingungen die Embryonen entwickeln, mit normaler Geschwindigkeit und überleben die Bildgebung, im Einklang mit früheren Berichten darauf hinweist, dass SPIM Bildgebung verursacht nur geringe Phototoxizität Ebenen in C. elegans Embryonen 6,7.
Der erste Stamm drückt eine Histon-GFP (zuIs178; stIs10024) fusioniert. Aufzeichnung wurde während 2 Stunden unter einem Stapel von Scheiben 20 (Abstand zwischen den Schichten 1 um) genommen jede 37 s durchgeführt. Fig. 6 veranschaulicht eine Ebene des Stapels zu verschiedenen Zeitpunkten. Beide interphasic Kerne (Pfeil) und kondensierte mitotischen Chromatin (Pfeilspitze) sind deutlich sichtbar.
Der zweite Stamm drückt eine Tubulin-GFP (ruIs57) fusioniert und ein Histonzu mCherry (; stIs10226 itIs37) fusioniert. Aufzeichnung wurde während 16 min mit einem Stapel von Scheiben 20 (Abstand zwischen den Schichten 1 um) genommen jede 105 sec durchgeführt. Fig. 7 zeigt verschiedene Ebenen des Stapels und zu verschiedenen Zeitpunkten. Der zugehörige Film 1 zeigt 3D-Rekonstruktionen in 3 aufeinander folgenden Zeitpunkten. Während der Teilung der mitotischen Spindel (grün) und kondensierte Chromosomen (rot) sind deutlich sichtbar so dass die Verfolgung der Zellteilung Orientierungen während der Entwicklung.
Der dritte Stamm drückt die Apolipoprotein VIT-2 bis GFP (pwIs23) fusioniert und eine Membran gebunden mCherry (ltIs44). Aufzeichnung wurde während 25 min mit einem Stapel von Scheiben 10 (Abstand zwischen den Schichten 1 um) genommen alle 30 Sekunden durchgeführt. Fig. 8 zeigt verschiedene Ebenen des Stapels und zu verschiedenen Zeitpunkten. Die schnellen Bewegungen der Eigelb-Lipoprotein-Partikel (grün) innerhalb der Zellen können leichtgefolgt.
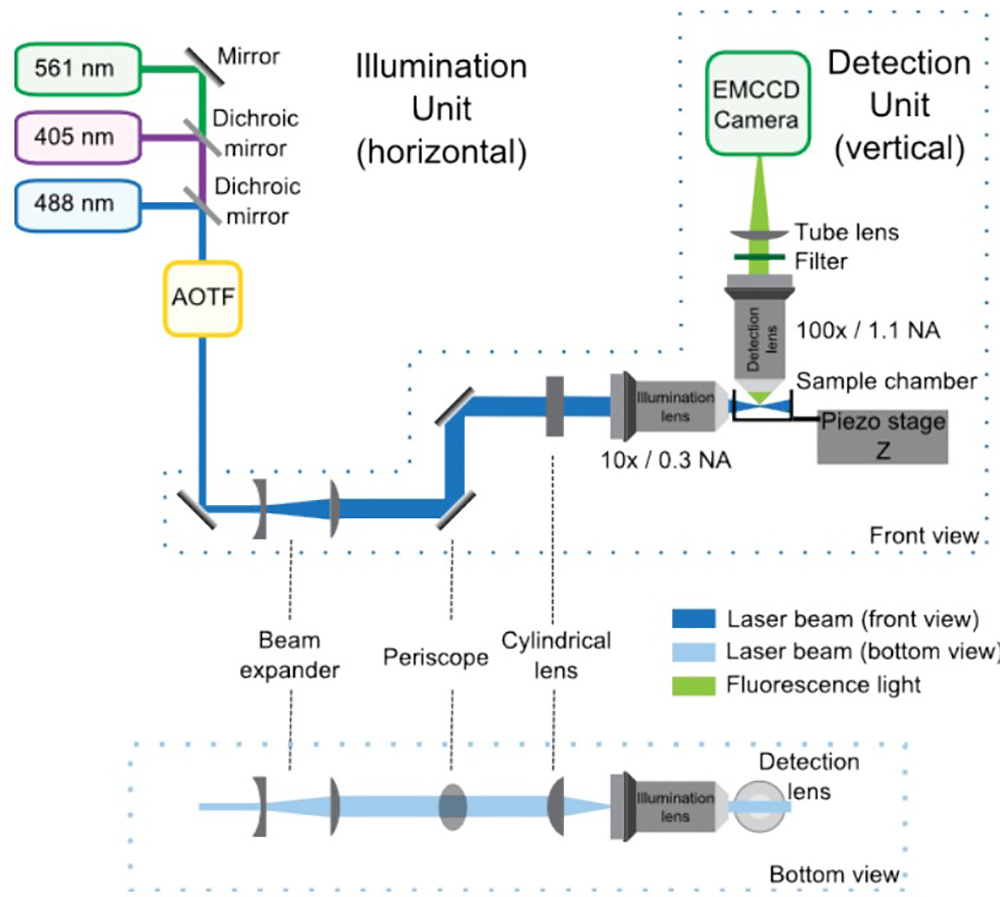
Abbildung 1: SPIM optische Einrichtung der Beleuchtungs-und Detektionseinheiten, Vorder-und Unteransichten zusammen. In der Beleuchtungseinheit, die Laser kombiniert durch die dichroitischen Spiegel in das AOTF, die die Leistung jedes Lasers unabhängig steuert. Dann erhöht das Teleskop die Größe des Strahls durch 4-fach und das Periskop bringt es auf die Höhe des Mikroskops. Die Zylinderlinse bildet den Lichtbogen, die durch die Beleuchtung Ziel neu ausgerichtet wird. Die Erfassungseinheit in der aufrechten Mikroskop integriert und wird hauptsächlich durch die Erfassungslinse, die Filter, die Tubuslinse und EMCCD besteht. Die Probe wird an dem Schnittpunkt zwischen der Beleuchtungs-und Detektionswegen angeordnet. Ein piezoelektrischer Phase ermöglicht vertikale (Z) Verschiebungen der Probefür 3D-Akquisition.
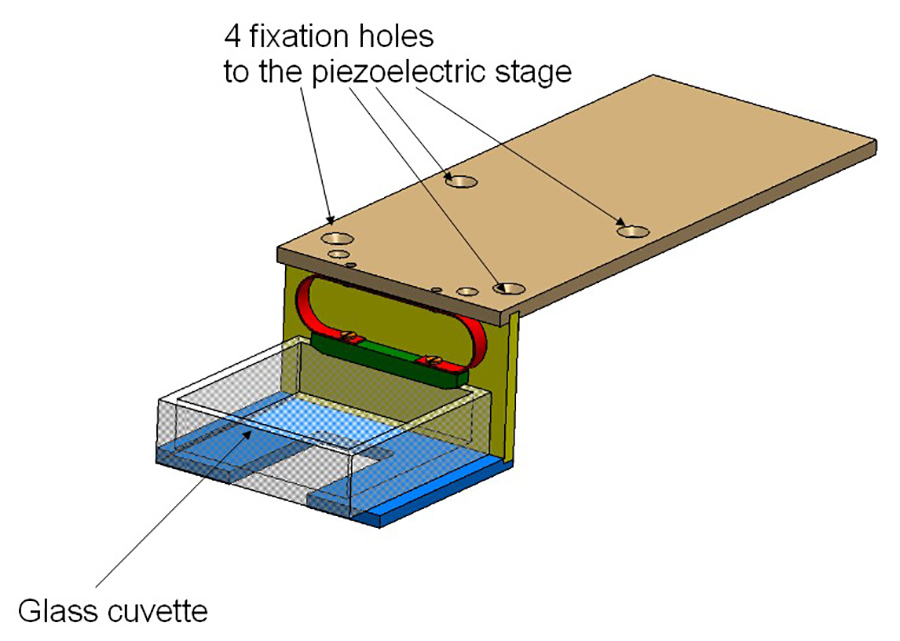
Figur 2:. Küvettenhalter Der Halter besteht aus 3 Aluminium-Platten und auf dem piezoelektrischen Stufe eingeschraubt. Die Glasküvette wird durch eine Feder (in rot) gehalten.
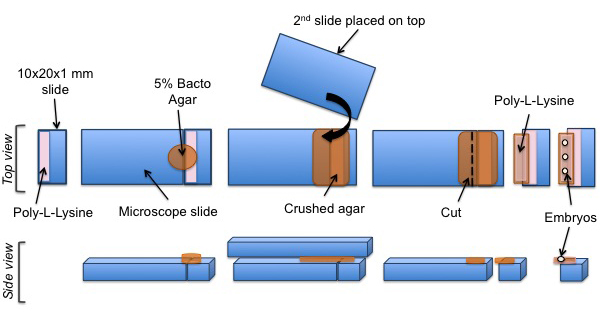
Abbildung 3: Montage der C-Protokoll elegans Embryonen für SPIM Experimenten. Ein Schnittstück aus Glas mit Poly-L-Lysin beschichtet ist. Ein Kissen aus Agar auf der beschichteten Oberfläche positioniert. Poly-L-Lysin wird dann auf die Agar-Pad aufgenommen. Schließlich C. elegans Embryonen werden auf der Poly-L-Lysin beschichteten Agar Kissen ausgerichtet sind.
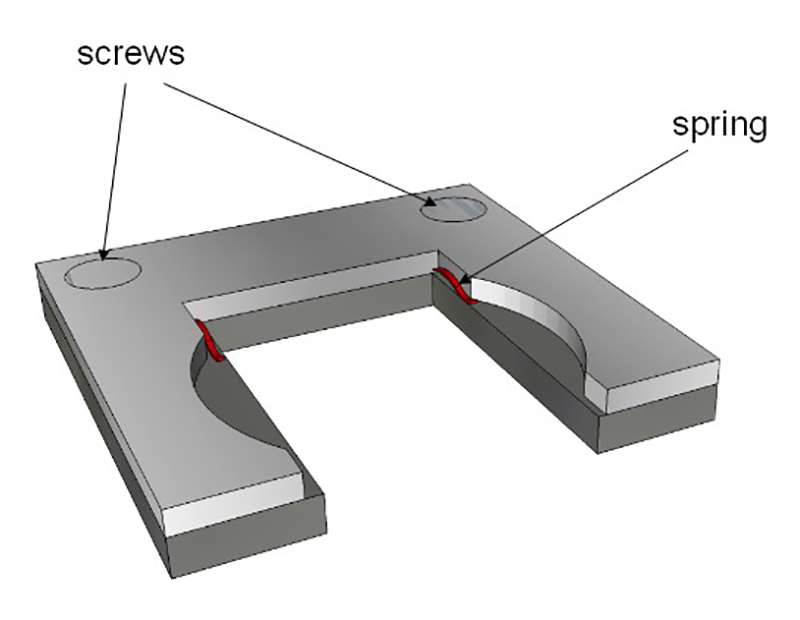
Abbildung 4: Probenhalter. Die Halterung passt in die Innenmaße der Glasküvette. Zwei Federn (in rot) halten den Glasobjektträger (in Abbildung 3 beschrieben). Diese Halterung wird dann im Inneren des Glas Küvette setzen.
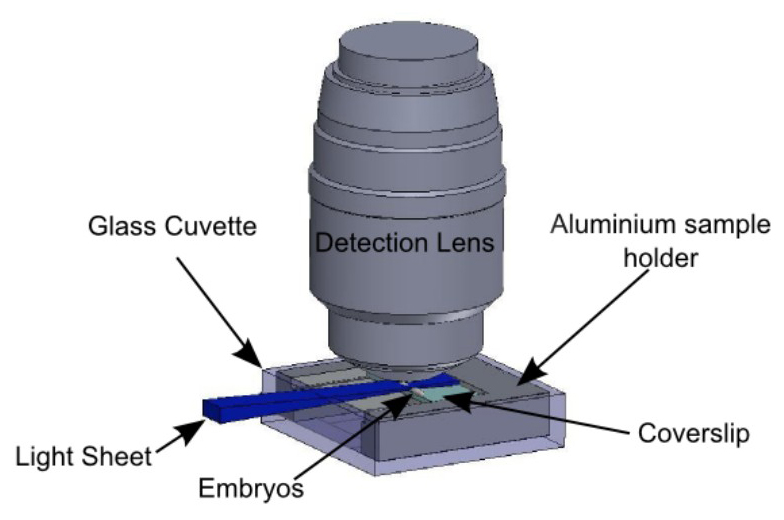
Figur 5:... Probenhalter in der Beobachtungs Zusammenhang Embryonen werden auf dem Objektträger nach dem Protokoll fasste in Fig. 3 immobilisiert wird der Schieber durch die in Fig. 4 beschriebenen Probenhalter gehaltenen Probenhalter in der Glasküvette eingesetzt, durch die in Fig. 2 beschriebene Halter gehalten. Lichtblattes horizontal und illuminates die Embryonen von der Seite. Die Detektionsobjektivlinse vertikal über der Probe.
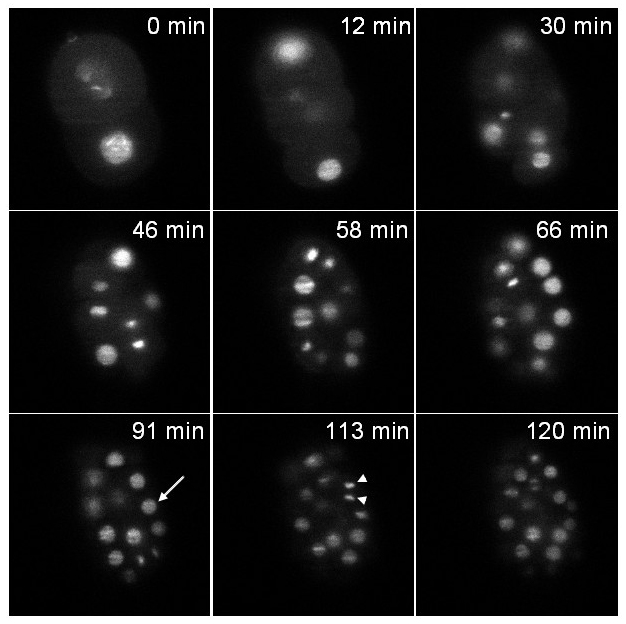
Abbildung 6: Aufnahme eines C. elegans Embryo eine Histon :: GFP-Fusionsebene ausdrückt Einzelbilder eines transgenen Embryos Expression eines Histon :: GFP-Fusions (zuIs178; stIs10024) zu verschiedenen Zeitpunkten aus einem 3D-Bilder Zeitraffer-Aufnahme extrahiert:.. Stapel von 20 Scheiben (gemacht Abstand zwischen den Schichten 1 m); Belichtungszeit pro Stück: 30 ms; Zeitintervall zwischen den Stapeln: 37 sec; Gesamterfassungszeit: 2 Stunden. Pfeil: interphasic Kern, Pfeilspitze: mitotischen Chromatin kondensiert. Bei t = 0 min der Embryo im 2-Zellen-Stadium und bei t = 120 min der Embryo wie unter normalen Entwicklungs s erwartet enthält etwa 70 Zellengepinkelt (beachten Sie, dass nur die in einer einzigen Ebene enthaltenen Zellen sichtbar sind in den Bildern und nicht alle Zellen des Embryos). Die Macht der Beleuchtung war 30 uW bei 488 nm (gemessen am Ausgang des Beleuchtungsziel). Bitte klicken Sie hier, um dieses Video zu sehen.
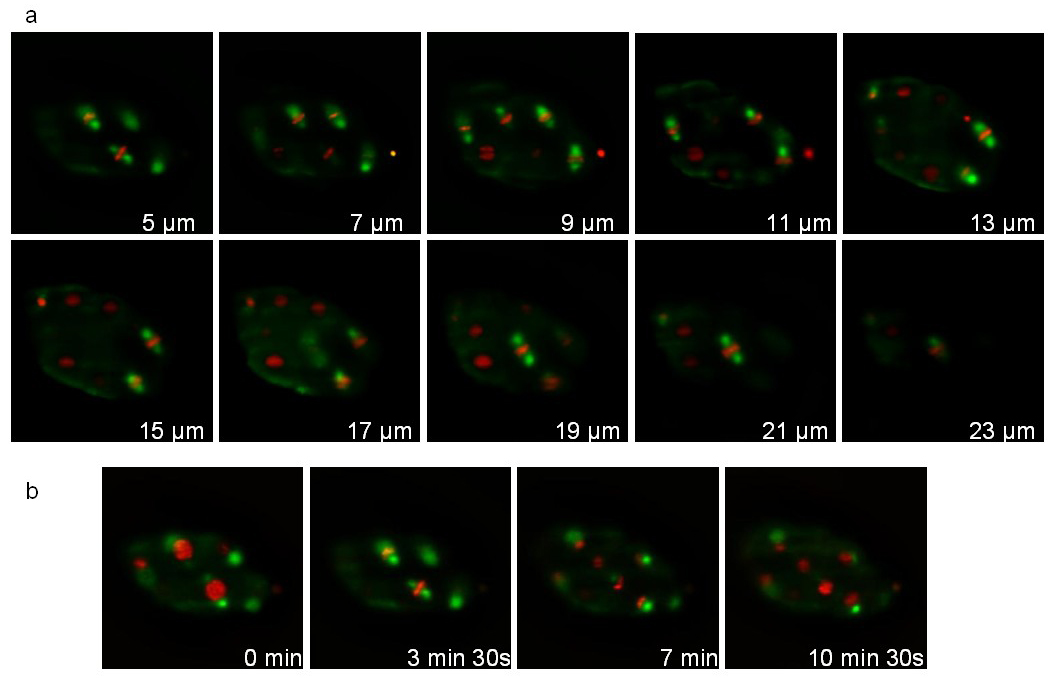
Abbildung 7: Aufnahme eines C. elegans Embryo eine Tubulin :: GFP-Fusions ausdrücken und ein Histon :: mCherry Fusion. Bilder aus einem 3D Zeitrafferaufnahme eines C extrahiert elegans Embryo eine Tubulin :: GFP-Fusions (ruIs57) und einen Histone :: mCherry Fusion (itIs37; stIs10226) ausdrückt. Aufnahmebedingungen: Übernahme von einem Stapel von 20 Scheiben (Abstand zwischen Scheiben einum) alle 105 sec; Gesamtaufnahmezeit: 16 min; Belichtungszeit: 200 ms für jeden Kanal. Feld A zeigt die 10 Scheiben des gleichen Stapels. Panel B zeigt die gleichen Scheibe alle 210 Sekunden. Die Macht der Beleuchtung war 30 uW und 300 uW, für 488 und 561 nm Laser, bzw. (gemessen am Ausgang des Beleuchtungsziel). Bitte klicken Sie hier, um dieses Video zu sehen.
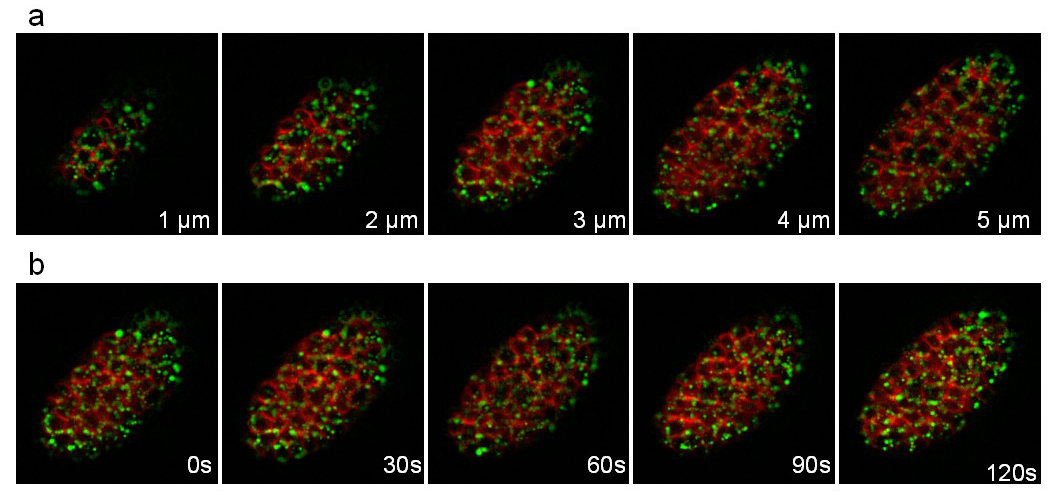
Abbildung 8: Aufnahme eines C. elegans Embryo ein VIT-2 :: GFP-Fusions ausdrücken und ein Membran-bezogene mCherry. Bilder aus einem 3D Zeitrafferaufnahme eines C extrahiert elegans Embryo Ausdruck einer VIT-2 :: GFP-Fusions (PWIs23) und ein Membran-bezogene mCherry (ltIs44). Aufnahmebedingungen: Übernahme von einem Stapel von 10 Scheiben (Abstand zwischen Scheiben 1 m) alle 30 sec; Gesamtaufnahmezeit: 25 min; Belichtungszeit pro Kanal: 200 msec. Tafel A zeigt die ersten 5 Schnitte des gleichen Stapels. Panel B zeigt die gleichen Scheibe jede 30sec. Bitte klicken Sie hier, um dieses Video zu sehen.
Diskussion
Dieses Protokoll beschreibt eine einfache Einstellung der Lichtbogen-Mikroskopie in vivo-Bildgebung von C. elegans Embryonen. Die optischen Elemente, die zum Erstellen und richten Sie den Lichtbogen, darunter Laser, Strahlaufweitung, Bündelung und Fokussierung Linsen, kann leicht auf einer optischen Bank montiert werden. In Kombination mit einem aufrechten Mikroskop für die Detektionspfad und der Kamera bietet dies eine einfache Lösung für das Einrichten eines Lichtschnittmikroskop.
Diese Geometrie macht die Probenumgebung einfach. Die lebende Probe wird in eine Glasküvette, die gehalten wird, angeordnet und durch eine begrenzte Anzahl von mechanischen Teilen verschoben angeordnet ist. Als Blattbeleuchtung Mikroskopie ist ein weites Feld Technik mit Auftrennung wird nur ein einziges motorisierte Achsen-Bewegung für die 3D-Bildgebung erforderlich. Dies begrenzt das Erfordernis der Synchronisation auf einen einzigen Scanelement und erleichtert die Software-Entwicklung. Im Vergleich zu unseren ISPIM 6 aufrecht geory ermöglicht die Verwendung von hohen NA Objektiv für die Erkennung. Im Vergleich zu SPIM Aufbauten mit Objektivlinsen in der horizontalen Ebene die Befestigung der Probe und der Abbildungs einer Reihe von Embryonen zu erleichtern. Insgesamt ist die Implementierung dieser Technik einfach und kann mit wenig Erfahrung in der Optik erreicht werden. Dies ist eine günstige Alternative zum konfokalen Mikroskopen mit den Vorteilen einer höheren Geschwindigkeit und geringere Phototoxizität, aber Nachteil einer geringeren axialen Auflösung.
Wir haben auch eine einfache und reproduzierbare Weise, C. Halterung entwickelt elegans Embryonen für die in vivo-Bildgebung unter Verwendung dieses Systems. Der Montagevorgang ist schnell (15 min) und ist geeignet für den täglichen Experimenten. Für C. elegans-Bildgebung, die Vorteile dieser einfachen Aufbau sind mindestens zwei Falten (i) in toto Abbildungs möglich ist, ohne Drehung und damit die 3D-Rekonstruktion ist einfach; (Ii) Probe Montage ist einfach und mehrere Embryonen nacheinander abgebildet werdenauf derselben Folie. Wir zeigten Beispiele von Zeitraffer-Filmen mit ausreichender zeitlicher Auflösung zu Zellteilungen und Zellformänderungen zu beobachten. Die Macht der Lichtbogen-Mikroskopie zu untersuchen C. elegans Entwicklung wurde auch kürzlich von anderen 6, 7, 8 dargestellt. Daher wird diese Technik sehr nützlich, um die Morphogenese und Strukturieren der Studie C. elegans Embryo.
Kann dieses System auch verwendet werden, um die Entwicklung von anderen Modellorganismen analysieren? Es ist wichtig zu erkennen, dass die Umsetzung der Blattbeleuchtung Mikroskopie für kleine und transparent Organismus wie C. elegans ist weniger streng als für größere Organismen wie Drosophila-Embryonen. In toto Bildgebung von Drosophila erfordert in der Regel Rotation und Multi-View-Rekonstruktionen 9, 10. Anregung von zwei Seiten mit kollineare Anregung Objektive können auch reduzierte die Schatteneffekte durch die Dämpfungs verursachtIon des Lichts 11. Dennoch ist das Setup hier vorge noch Bild einer Seite der Drosophila-Embryonen angepasst, mit geringem Bleichen und niedrigen photoxicity. Diese Einrichtung könnte auch hilfreich, wenn Bilder klein und transparent Embryonen wie Seescheiden oder Zebrafisch sein.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Wir danken allen Mitgliedern der Lenne und Bertrand Labors, insbesondere Olivier Blanc für Software-Entwicklung und Jérémie Capoulade zur Diskussion. Wir danken auch dem PICsL-IBDM Bild Anlage, insbesondere Claude Moretti und Brice Detailleur für technische Unterstützung.
Diese Arbeit wurde durch Zuschüsse aus ATIP CNRS (P.-FL und VB) unterstützt, die Labex INFORM Zuschuss (P.-FL und VB) und einen Zuschuss von Sanofi-Aventis (VB). Wir erkennen France-BioImaging Infrastruktur von der Agence Nationale de la Recherche unterstützt (ANR-10-InSb-04-01, als "Investissements d'Avenir").
Materialien
| Name | Company | Catalog Number | Comments |
| SPIM | |||
| Detection | |||
| upright microscope | ZEISS | Axiophot 1 | |
| 100x W objective (NA 1.1, W.D. 2.5mm) | NIKON | ||
| EMCCD back illuminated | ANDOR | DU-885K-CS0-#VP | |
| Illumination | |||
| LASER 488 nm / 60 mW | TOPTICA | iBeam smart | |
| LASER 405 nm / 120 mW | TOPTICA | iBeam smart | |
| LASER 561 nm / 500 mW | COBOLT | JIVE 500 | for time lapse imaging, 50 or 100mW is enough |
| filter Quad line laser rejectionband ZET 405/488/561/638 | AHF | F57-405 | |
| Dichroic 405 | AHF | F38-405 | |
| Dichroic 488 | SEMROCK | Di01-R488 | |
| AOTF | AA | AOTFnC-400.650-TN | |
| mirror | |||
| lens 50 mm | THORLABS | telescope 5X | |
| lens 200 mm | THORLABS | telescope 5X | |
| periscope | THORLABS | RS99/M | |
| cylindrical lens f=100mm | THORLABS | ACY254-100-A | |
| 10X NA 0,3 | NIKON | ||
| xyz stage | NEWPORT | ||
| Sample | |||
| Bacto Agar | Bectkton Dickinson | 214010 | 5% in water |
| Poly-L-Lysine solution | Sigma | P8920 | |
| Capillaries GC100F-10, 1.0mm oD, 0,58mm ID, 100 mm L | Havard Appartus | 30-0019 | |
| Aspiration tube (mouth pipette) | Dutsher | 55005 | |
| M9 medium: | |||
| KH2PO4 (3g/L) | Any supplier | ||
| Na2HPO4 (6g/L) | Any supplier | ||
| NaCL (5g/L) | Any supplier | ||
| MgSO4 (1 mM final) | Any supplier | ||
| cuvette holder | HOME MADE | made by Brice Detailleur | |
| sample holder | HOME MADE | made by Claude Moretti | |
| x stage | NEWPORT | ||
| coverslip 50x20x1 mm | MARIENFELD | cut then in 10x20x1mm | |
| piezo electric stage with amplifier/controller | PI | P-622.ZCD / E-625.CR | |
| cuvette 40mmx40mmx10mm | HELLMA | 704.002-OG.40 mm- 10mm high | |
| Diamond Point Glass Marking Pencil | VWR | ||
| Software | |||
| C++ (Qt) home made software | developed by Olivier Blanc and Claire Chardès, available on request. Alternative solutions: micromanager or labview | ||
Referenzen
- Huisken, J., Swoger, J., Del Bene, F., Wittbrodt, J., Stelzer, E. H. Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305, 1007-1009 (2004).
- Huisken, J., Stainier, D. Y. Selective plane illumination microscopy techniques in developmental biology. Development. 136, 1963-1975 (2009).
- Reynaud, E. G., Krzic, U., Greger, K., Stelzer, E. H. Light sheet-based fluorescence microscopy: more dimensions, more photons, and less photodamage. HFSP J. 2, 266-275 (2008).
- Sulston, J. E., Schierenberg, E., White, J. G., Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Dev Biol. 100, 64-119 (1983).
- Hardin, J. Imaging embryonic morphogenesis in C. elegans. Methods Cell Biol. 106, 377-412 (2011).
- Wu, Y., et al. Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans. Proc Natl Acad Sci U S A. 108, 17708-17713 (2011).
- Giurumescu, C. A., Kang, S., Planchon, T. A., Betzig, E., Bloomekatz, J., Yelon, D., Cosman, P., Chisholm, A. D. Quantitative semi-automated analysis of morphogenesis with single-cell resolution in complex embryos. Development. 139, 4271-4279 (2012).
- Gao, L., Shao, L., Higgins, C. D., Poulton, J. S., Peifer, M., Davidson, M. W., Wu, X. F. Noninvasive Imaging beyond the Diffraction Limit of 3D Dynamics in Thickly Fluorescent Specimens. Cell. 151, 1370-1385 (2012).
- Krzic, U., Gunther, S., Saunders, T. E., Streichan, S. J., Hufnagel, L. Multiview light-sheet microscope for rapid in toto imaging. Nat Methods. 9, 730-733 (2012).
- Tomer, R., Khairy, K., Amat, F., Keller, P. J. Quantitative high-speed imaging of entire developing embryos with simultaneous multiview light-sheet microscopy. Nat Methods. 9, 755-763 (2012).
- Huisken, J., Stainier, D. Y. Even fluorescence excitation by multidirectional selective plane illumination microscopy (mSPIM). Opt Lett. 32, 2608-2610 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten