Method Article
Identificación de proteínas compañeros de interacción en células de mamífero utilizando SILAC-inmunoprecipitación cuantitativos Proteómica
En este artículo
Resumen
Experimentos de inmunoprecipitación SILAC representan un poderoso medio para descubrir nueva proteína: proteína interacciones. Al permitir la cuantificación relativa precisa de la abundancia de proteínas tanto en el control y las muestras de ensayo, verdaderos interacciones se puede distinguir fácilmente de los contaminantes experimentales, y las interacciones de baja afinidad conservada a través del uso de condiciones de tampón menos estrictos.
Resumen
Proteómica cuantitativa, junto con la purificación de inmunoafinidad, inmunoprecipitación SILAC, representan un poderoso medio para el descubrimiento de la nueva proteína: proteína interacciones. Al permitir la cuantificación relativa precisa de la abundancia de proteínas tanto en el control y las muestras de ensayo, verdaderos interacciones pueden distinguirse fácilmente de los contaminantes experimentales. Interacciones de baja afinidad se pueden preservar mediante el uso de condiciones de tampón menos estrictos y siguen siendo fácilmente identificable. Este protocolo describe el etiquetado de las células de cultivo de tejidos con isótopos estables etiquetados aminoácidos, la transfección y la inmunoprecipitación de una afinidad etiquetado proteína de interés, seguido de la preparación para la presentación a una instalación de espectrometría de masas. Este protocolo a continuación explica cómo analizar e interpretar los datos devueltos por el espectrómetro de masas con el fin de identificar a los socios celulares que interactúan con una proteína de interés. A modo de ejemplo se aplica esta técnica para identificar proteínas de unión a los factores de iniciación de la traducción eucarióticos: eIF4AI y eIF4AII.
Introducción
Un paso esencial en la comprensión de la función de proteínas es la identificación de las proteínas que interactúan pertinentes. Cuando tales proteínas son desconocidos hay una serie de técnicas disponibles, cada uno con sus propias ventajas y desventajas. Estos incluyen el sistema de levadura de dos híbridos, menú desplegable ensayos que utilizan proteínas recombinantes, así como la purificación por afinidad en tándem o TAP-etiquetado 1, 2.
Una adición más reciente a estas técnicas es la combinación de la purificación por afinidad de una proteína de interés a partir de una línea celular de mamífero relevante, seguido de espectrometría de masas cuantitativa utilizando el etiquetado de isótopos estables de aminoácidos en cultivo celular (SILAC) 3. Esto tiene ventajas sobre el enfoque de la levadura de dos híbridos en que la localización celular y modificaciones post-traduccionales no son perturbados, así como las ventajas más tradicional de TAP-de marcado en la que se trata de un enfoque cuantitativo en lugar de cualitativo que permite al usuario REAdily distinguir de forma no específica las proteínas que interactúan y contaminantes, de los factores del huésped que se unen específicamente. Además, como una muestra normalmente se analizó su conjunto, en lugar de las bandas de proteínas como individuales, proteínas de interés no están enmascaradas por la migración de manera similar las proteínas en un gel, ni tampoco suelen necesitar estar presentes en niveles suficientes para ser visibles después de la tinción, lo que lleva a aumento del número de proteínas identificadas con confianza 4.
Para demostrar esta técnica, GFP fusiones del factor de iniciación de la traducción eucariótica estrechamente relacionado eIF4AI y eIF4AII que comparten más de una identidad de aminoácidos del 90% fueron investigados por SILAC-inmunoprecipitación proteómica cuantitativa. EIF4AI humano y II fueron clonados dentro de pEGFP-C1 para formar una proteína de fusión de GFP donde se fusiona con el extremo N-terminal de eIF4A. Para evitar la necesidad de generar líneas celulares estables de transfección transitoria se utilizó para entregar estas construcciones a células 293T de isótopos estables etiquetados.
Las células se marcaron primero durante dos semanas en SILAC medios de cultivo celular, seguido de la transfección del plásmido de ADN que codifica una proteína de interés. Las células se lisaron a continuación, la concentración de proteína normalizaron, y cantidades iguales de afinidad de lisado purificaron en agarosa anti-GFP. Igual cantidad de eluido se combinaron después y se sometieron a análisis LC-MS/MS. Los resultados de este análisis se procesan entonces para identificar proteínas de alta confianza: las interacciones proteína (Figura 1).
Inmunoprecipitación SILAC permite identificar no sólo las interacciones directas, sino también de baja afinidad o interacciones indirectas con complejos de proteínas 4. Usando este sistema, eIF4AI y II inmunoprecipitaciones permitió la identificación reproducible y seguro de la pareja principal de unión eIF4G (isoformas I / II y III) 5, así como las interacciones indirectas con eIF4E, y numerosos componentes del complejo eIF3.
Protocolo
1. Generación y El pase de Líneas Celulares SILAC marcado
Nota: el uso de tripsina-EDTA se debe evitar en todas las etapas de pases y la preparación de las muestras experimentales para el análisis como la tripsina puede contener aminoácidos sin etiqueta, lo que llevaría al etiquetado incompleto de muestras.
- Para preparar una botella de medios SILAC añadir una alícuota de una arginina marcada de manera apropiada-SILAC (84 mg / ml en PBS) y lisina (146 mg / ml en PBS) a una botella de 500 ml de Arg / Lys DMEM libre (que contiene 0,5 ml L-glutamina).
- A continuación, añadir 50 ml de FBS dializado y 5 ml de penicilina / estreptomicina a la botella de 500 ml.
Nota: Las celdas HEK 293T (ATCC) deben mantenerse en medio DMEM que carecen de arginina y lisina y se completará con la luz (R0K0), media (R6K4) o fuerte (R10K8) aminoácidos, suero bovino fetal dializado (10 kDa de corte), y la penicilina / estreptomicina. Las células deben ser mantenidos en los medios de comunicación durante un mínimo de 5 divisiones celulares para asegurar una completaetiquetado. En la mayoría de los casos las células se marcan con facilidad en ≥ 2 semanas. - Para dividir celdas para pases, las células deben ser desalojados de la monocapa golpeando el frasco. Las alternativas incluyen el uso de raspadores de células o mediante el uso de tampón de disociación celular basada en PBS libre de enzimas.
Nota: Para conservar los medios de comunicación, las células deben ser pasados en matraces T25 y el número de células más grandes generan sólo inmediatamente antes de un experimento. - 24 horas antes de la transfección de las células, semillas de 3,5 x 10 6 células SILAC marcado en un único plato de 10 cm 2 para cada condición experimental bajo investigación.
2. La transfección de células marcadas con construcciones pEGFP-fusión
- Retire el papel de las células y reemplazarlo con 9 ml de antibiótico libre SILAC DMEM (Luz, medio o pesado).
Nota: Los antibióticos no deben añadirse a los medios de comunicación ya que pueden interferir con la eficacia de los reactivos de transfección basadas en liposomas. - Prepare una mezcla de 10 g de plásmido apropiado (epEGFP-C1, pEGFP-eIF4A-I, pEGFP-eIF4A-II) en 500 l de antibiótico-libre SILAC DMEM y se mezcla con 500 l de DMEM SILAC libre de antibióticos que contienen 10 l de reactivo de transfección (por ejemplo, lipofectamina 2000). Mezcle la reacción a fondo con la pipeta hacia arriba y abajo varias veces.
- Incubar la mezcla de reacción a temperatura ambiente durante 20 min y a continuación añadir gota a gota a la monocapa de células. Rock the placa suavemente de lado a lado.
- Se incuban las células transfectadas a 37 ° C y 10% de CO 2 durante 24 horas.
Nota: Si la expresión de una proteína de resultados de interés en cualquier toxicidad aparente, entonces puede ser necesario reducir la cantidad de plásmido transfectado y / o la duración del período de expresión posterior.
3. Lisados celulares de cosecha
- Las células de la cosecha del plato en helada PBS utilizando un rascador de células. Recoger las células por centrifugación a 220 xg, 476; C durante 5 min. Lavar las células una 3x más en 10 ml de PBS enfriado en hielo.
- Resuspender el sedimento de células en 200 l de tampón de lisis celular (10 mM de TrisCl / pH 7,5, NaCl 150 mM, EDTA 0,5 mM, 0,5% de NP40) que contiene recién añadido cóctel inhibidor de la proteasa III a una concentración de 1x y cóctel de RNasa (opcional) en 5 l por ml.
Nota: Para las proteínas con ácido nucleico conocida la actividad de unión, puede ser necesario añadir nucleasas al lisado antes de la precipitación. En los casos en donde se añade nucleasa, las muestras se deben incubar en hielo durante 30 min en hielo, con pipeteando cada 10 min. La extracción de ácido nucleico total de una pequeña fracción de la muestra y el análisis por electroforesis en gel de agarosa puede probar la eficacia de la nucleasa. - Centrifugar las muestras a 13.000 xg, 4 ° C durante 10 min retienen el sobrenadante como el lisado celular soluble.
- La concentración del lisado celular debe ser evaluada por ensayo BCA de acuerdo con las instrucciones del fabricante.
- Utilizar una solución tampón de lisis que contenía cóctel inhibidor de la proteasa III para normalizar la concentración de proteína en un volumen final de 500 l.
- Ajustar el volumen a 1 ml con la adición de 500 l de tampón de dilución (10 mM de TrisCl / pH 7,5, NaCl 150 mM, EDTA 0,5 mM) que contenía cóctel inhibidor de la proteasa III a una concentración de 1x final. Una alícuota de 50 l de la muestra debe conservarse como la entrada de la muestra y el lisado se mantuvo en hielo mientras que la preparación perlas anti-GFP (por ejemplo, las buenas prácticas agrarias-trampa).
Nota: Por lo general los rendimientos varían entre 1 a 3,5 mg de proteína en la final de 1 ml de muestra. Mientras que los tampones anteriores son adecuados para muchas proteínas de interés, para otros puede ser necesario modificar los componentes del tampón para asegurar el cebo de proteína se solubiliza y mantener interacciones proteína-proteína. Alteraciones posibles incluyen el agente tampón (fosfato, HEPES), concentración de sal (150-500 mM), la elección del detergente, u otros aditivos.
4. La unión a anti-GCuentas de PF
- Brevemente vórtex con la suspensión de perlas para resuspender las perlas. Con una punta de pipeta de 200 l con el extremo cortado, transferir 25 l de los granos por muestra a un tubo nuevo.
Nota: El usuario debe preparar las cuentas para un solo experimento SILAC como una mezcla maestra para minimizar la variación de muestra a muestra. - Por cada 25 l de suspensión de perlas, añadir 20 volúmenes (1.500 l por 75 l purines) de tampón de dilución, y centrifugar las cuentas a 2.700 xg durante 5 min. A continuación, lavar las perlas de un 2x más en 20 volúmenes de tampón de dilución.
- Añadir 100 l de tampón de dilución por cada 25 l de talón suspensión. Utilizando una punta de 200 l con el extremo cortado, transferir 85 l de esta suspensión se resuspendieron a cada una de las muestras de SILAC marcado de paso 3.6.
- Incubar las muestras con las cuentas de un rotador a 4 º C durante 2 horas.
5. Lavar, elución, y preparación de muestras para análisis MS
- Centrifugar las muestras a 2700 xg, 46; C durante 5 min. 50 l del sobrenadante deben ser retenidos como la muestra no unida, con el resto del sobrenadante descartado.
- Tampón de dilución de 1 ml, debe añadirse a cada tubo para resuspender las perlas y la muestra se centrifugó a 2700 xg, 4 ° C durante 5 min. El sobrenadante se debe desechar. Esto se debe realizar dos veces.
- Eluir la proteína de las perlas por la adición de 50 l de tampón de carga 2x SDS, y calentamiento a 95 ° C durante 10 min. Se precipitan las perlas mediante centrifugación a 2700 xg durante 2 min a 4 ° C
- Conservar el sobrenadante en tubos prelubricados, donde puede ser después se almacenó a -80 ° C hasta que sea necesario. Para el envío a un centro de la espectrometría de masa, mezclar muestras etiquetadas 01:01:01 (por ejemplo, 10 l de cada uno) y presentar la muestra mixta.
Nota: En este punto, las muestras pueden ser probadas por Western blot, tinción de plata o de otros medios de prueba para la interacción con los compañeros de unión conocidos / desconocidos. Un ejemplo se da en la figuraUre 2 que muestra la unión específica de un socio conocido interacción - eIF4G por Western blot, y la aparición de bandas de tinción de plata presentes en las muestras desplegables de una proteína de interés, pero no una muestra de control. - Someter la muestra para el análisis LC-MS/MS.
Nota: una vez satisfecho de que una proteína de la etiqueta de interés se vincula con éxito los compañeros de interacción, volúmenes iguales de cada muestra marcada (ligero, medio y pesado) son combinados y presentados para su análisis LC-MS/MS. Es habitual que presente 30 l total de una muestra de IP para el análisis. Esto implicaría la mezcla 10 l de muestra de luz marcada con 10 l de muestras marcado pesados marcado con medianas y 10 l para dar un total de 30 l.
6 Análisis de Datos I:. Interpretación de los resultados, y la eliminación de Identificaciones de baja confianza
Nota: Una lista de los títulos de las columnas devueltas por el software proteoma Descubridor se da en la Tabla 1 Dif.rentes software (por ejemplo, MSQuant, MaxQuant) volverá a partidas distintas, sin embargo, sólo un subconjunto de ellos son necesarios para el análisis y éstos son comunes a los diferentes paquetes de software. Los datos siempre deben incluir un número de acceso para cada proteína identificada, la relación de la comparación de cada relación de la muestra (luz vs media, ligera vs pesados, medianos vs etc pesado), el número único de péptidos identificados, y algún tipo de tasa de falsos positivos o indicación confianza.
- Antes de ir a través de los datos, en primer lugar, copie los datos en bruto a una nueva hoja de cálculo. A partir de esta hoja de cálculo de eliminar todas las columnas salvo los que den el número de acceso, número único de péptidos, los ratios que comparan muestras, relación de la variabilidad y la descripción de proteínas. Si se realizaron réplicas de los experimentos, estos deben ser combinados en un solo archivo de Excel, con cada experimento que aparece en una ficha independiente.
Nota: los datos de confianza baja incluye proteínas identificadas por un solo péptido único, y los waquí cuantificación no era posible. - Utilice la función 'sort' Sobresale 'para ordenar los datos por el número de péptidos y eliminar las entradas para las proteínas que carecen de más de un péptido. Luego, ordenar por relación y eliminar las proteínas que carecen de relaciones SILAC (proteínas no cuantificados).
- Convertir proporciones SILAC en un registro de 2 valores utilizando la fórmula: '= log (ratio de SILAC, 2)', donde se sustituye «Ratio SILAC 'para el identificador de celda.
Nota: como una relación de SILAC resultados en los datos para las proteínas que muestran una disminución en la abundancia está restringida a valores entre 0 y 1, que es habitual para convertir una relación de SILAC a una relación de SILAC registro 2, ya que esto significa proteínas tanto aumentado o disminuido en una muestra están representados en una escala logarítmica, donde 2 o -2 representa un aumento de 4 veces o disminución de la abundancia, y 3 o -3 un aumento de 9 veces o disminución de la abundancia, respectivamente. A raíz de esta transformación, los datos deben ajustarse a una Gadistribución usia centrado alrededor de una relación de SILAC registro 2 de 0. Un ejemplo de esto se da en la Figura 3. - Crear nuevas columnas en el archivo de Excel y calcular log 2 ratios SILAC para todas las muestras / columnas simuladas. Para la conversión de una relación fingida / muestra a una relación muestra / maqueta, utilice la fórmula: "= 1/ratio"
7 Análisis de Datos II:. Selección de interacciones de alta confianza de estudio adicional
- Abrir Graphpad Prism, seleccione Nuevo> Tabla de datos y gráfico ... Select 'columna' de la lista en el lado izquierdo de la ventana y seleccione el botón ENTER / Importar datos> Introduzca los valores de réplica, apilados en columnas opciones. Presione 'Crear'.
- Seleccione y copie una columna determinada proporción de 2 log de la muestra / Mock SILAC de la hoja de cálculo de Excel en el nuevo archivo de Prism.
- Haga clic en el menú desplegable "Insertar" y seleccione "Nuevo análisis". Conforme al análisis de la columna, seleccione 'Distribución de frecuencias. Mantener las opciones por defecto, haga clic en 'Aceptar'.
Nota: Este paso genera un histograma que ilustra el número de proteínas identificadas en una relación dada. Esto debería formar una distribución gaussiana. - En la carpeta de "Resultados" se habrá generado una nueva sección "Histograma". Seleccione la sección de distribución de frecuencias.
- Haga clic en el menú desplegable "Insertar" y seleccione Nuevo análisis> XY> Regresión no lineal (ajuste de la curva). Haga clic en 'distribución de Gauss' y haga clic en Aceptar.
Nota: Este paso se ajusta a una curva a los datos de distribución de frecuencia generados en la etapa 7.3, los valores que pueden utilizarse posteriormente para calcular los umbrales de significación rendimiento. - En la ventana de resultados que aparece, se dan la media y la desviación estándar. Generar un umbral mediante la adición de 1,96 desviaciones estándar de la media.
Nota: Estos valores representan la media y la desviación estándar de la dist Gaussribution, no el conjunto de datos total. 1.96 desviaciones estándar colocarían un umbral en el límite de confianza del 95% (p ≤ 0,05). 2.58 SD daría el 99%, y el 3,3 SD 99,9%. El umbral debe determinarse individualmente para cada réplica experimento, ya que puede variar.
8 Análisis de Datos III. Fusión de los conjuntos de datos de réplica
Nota: Para tener confianza en la identificación de socios de la interacción de una típica desplegable experimento SILAC idealmente se debe realizar tres veces, con el medio y muestras etiquetadas pesados conmutada por una de las repeticiones de controlar para cualquier efecto de los medios de comunicación de los resultados. Una proteína que se muestra como la interacción en al menos dos de los tres experimentos, con la 'muestra switched'-medios de comunicación ideal que representa uno de estos, es una interacción de alta confianza.
- Volver al archivo de Excel, cree una nueva pestaña llamada 'Combinado'. Columna Etiqueta A 'adhesión' y copiar todos los números de la adhesión de cada uno of los experimentos individuales en esta sola columna.
- Seleccione la pestaña "Datos", y luego la opción "Eliminar Duplicados.
- Crear columnas para las relaciones de SILAC, y la relación de la variabilidad de cada experimento, así como para la columna Nombre / Descripción de la proteína.
- En la ficha Descripción, utilice la fórmula BUSCARV para recoger la descripción de cada número de entrada. Arrastre la fórmula hacia abajo la columna para anotar la descripción de proteínas. La fórmula BUSCARV es: "= BUSCARV (AccessionNo, WheretoLook, ColumnsAccross, False)", donde: AccessionNo es $ aX siendo X el número de fila WheretoLook es la 'pestaña Experiment Name' $ A $ 2:.! $ Y $ Z donde Y y Z son la pieza inferior derecha de los datos en la hoja de cálculo que se hace referencia. ColumnsAccross es el número de columnas a través del número de la adhesiónbre en la columna A un valor deseado miente.
Nota: Por ejemplo, si el número de acceso se encuentra en la columna A, y los datos de interés se encuentra en la columna E. El valor aquí sería 5 (columna A cuenta como 1). Como los números de acceso se agruparon en el paso b, es más probable que vlookup no obtendrá todos los nombres requeridos a partir de un experimento. Cuando regresa N / A, modifique la fórmula para obtener la descripción de cada experimento, a su vez hasta que se adquieran todas las descripciones. - Para resaltar las proteínas que interactúan en la combinación de datos, seleccione cada columna relación individual y haga clic en la ficha Inicio, seguido de reglas células Formato condicional> Resaltar> Más reglas.
- Seleccione el estilo 'Classic'. Formato únicas células que contienen 'Valor de la celda', 'Mayor que o igual a ". En el cuadro, escriba el valor de la desviación estándar de 1,96, y haga clic en 'Aceptar'.
- Evaluar la variabilidad ratio para cada interacción positiva. Si restando la var%ESPONSABILIDAD caería un 'hit' por debajo del valor umbral, este debe ser tratado con precaución.
Nota: Esto representa la consistencia de las relaciones individuales de cada péptido que componen relación SILAC 'unas proteínas son, y se da como un porcentaje. Cuando se toma la variabilidad relación en cuenta y una proteína da una relación de SILAC por encima del umbral, esta proteína representa un éxito. Si la variabilidad relación da un rango que está por debajo del umbral, el 'hit' puede representar un contaminante. - Repita esto para cada una de las columnas de resultados y compararlos a través de experimentos. Proteínas destacadas identificadas en dos o más experimentos representan una interacción de alta confianza.
Nota: En función de la base de datos utilizada para asignar los números de acceso de proteínas, en diferentes experimentos una proteína puede haber sido identificada en una diferente, o incluso múltiples números de acceso. Es importante verificar esto para asegurarse de que las proteínas no se incluyeron, accidentalmente.
Resultados
En un experimento típico de pulldown SILAC, la gran mayoría de las proteínas identificadas (> 90%) representan los contaminantes, así como proteínas de unión no específica a la matriz de afinidad y esto se ilustra en la figura 2B, aun cuando los protocolos de lavado eliminan la mayoría de los contaminantes citoplasmáticos tales como GAPDH (Figura 2A). Sin embargo, el agrupamiento de la unión no específica de proteínas en una distribución normal permite que las proteínas que se unen específicamente a una proteína de interés para ser distinguidos como estos tienen mayores proporciones de muestra / simulacros de que el fondo. Mientras que los contaminantes deben agruparse teóricamente en torno a una relación de SILAC registro 2 de 0, esto no es necesariamente el caso, un ejemplo de esto se da en la Figura 3. Las posibles razones para esto incluyen el etiquetado de SILAC imperfecta de las células, la carga de volúmenes o concentraciones de lisado desiguales en las perlas anti-GFP, la pérdida accidental de los granos durante la purificación o la mezcla desigualción de las muestras al final del procedimiento de purificación 3. Sin embargo, suponiendo que los datos se analizaron sobre la base de una desviación estándar de umbral de la media de los contaminantes normalmente distribuidos, cambios menores en el centrado de los datos no deben afectar a la calidad de los resultados.
Al comparar las diferencias en las interacciones de proteína entre dos proteínas relacionadas, puede producirse una situación similar en el que una proteína de interés se produce a niveles más altos dentro de las células de un segundo (ya sea debido a la variación en la eficiencia de transfección, o una propiedad intrínseca de la proteína o ARNm) . Algunos variación en la expresión (por ejemplo, la Figura 2A) pueden ser corregidos por el análisis de la relación de SILAC para estas dos muestras. En este ejemplo, estos serían los ejemplos de buenas prácticas agrarias-eIF4AI y GFP-eIF4AII. Mediante el análisis de la relación 4AI/4AII SILAC como se discutió en la sección 7, es posible identificar las proteínas cuya unión varía significativamente entre las isoformas.
En la figura 4, una representación de las proteínas de unión a eIF4A-identificados en uno de los experimentos llevados a cabo réplicas se muestra que ilustra la cobertura del complejo de factor de iniciación de este protocolo logra. Las proporciones más altas se observaron típicamente con eIF4G, que se une directamente a eIF4A, con relaciones más bajas para eIF4E, que se une a eIF4G en un sitio lejos del sitio de unión eIF4A-. Se observaron índices más bajos para los miembros del complejo eIF3. Sin embargo, esto ilustra claramente que el experimento identificado satisfactoriamente parejas de unión tanto directos como indirectos de eIF4AI y II. Como se podría esperar a partir de su alta identidad de secuencia 6, las interacciones proteína: proteína aparecieron muy conservada entre las dos isoformas en este sistema experimental 7, 8. Una selección de algunas de las proteínas que interactúan se dan en la Tabla 2, que ilustra el formato de datos.| Encabezado de columna | Descripción |
| Adhesión | Muestra el número de acceso para la secuencia |
| Cobertura | Proporción de la secuencia de la proteína cubierto por los péptidos identificados |
| ♯ PSM | Péptido partido espectral |
| ♯ péptidos | Número total de péptidos únicos identificados para una proteína |
| ♯ AA | La longitud de una proteína en aminoácidos |
| MW (Da) | El peso molecular de una proteína en Daltons. Excluye modificaciones |
| calc. PI | El punto isoeléctrico teórico de una proteína |
| Puntuación | La puntuación total de una proteína (que representa la suma de la Indivlas puntuaciones de péptidos iDual). La puntuación exacta requerida para la significación varía entre los experimentos. Una instalación de MS suele aplicar un corte falso descubrimiento tasa del 5%. |
| Secuencia | La secuencia de aminoácidos que constituyen la proteína |
| Proporción | La intensidad relativa de los péptidos en una muestra marcada con nombre, en comparación con una segunda muestra marcada |
| Count Ratio | El número de relaciones de péptidos que se utilizaron para calcular la relación de una proteína dada |
| Variabilidad Ratio (%) | La variabilidad de los ratios de péptidos individuales que se utilizan para calcular una relación de proteína dada |
| Descripción | El nombre de la proteína |
Tabla 1. Encabezados de columna estándar de un informe proteoma de Discoverer.Mientras que la información útil se puede obtener de todas estas columnas, los críticos de este análisis se muestran en negrita.
| Adhesión | Péptidos | 4AI/Mock | 4AII/Mock | Nombre | Análisis SILAC |
| A8K7F6 | 21 | 100 | 1 | eIF4AI | Proteína 'Bait' |
| Q14240 | 22 | 0.01 | 90.855 | eIF4AII | |
| G5E9S1 | 25 | 47.575 | 30.53 | eIF4GI | Interactuando proteínas |
| Q59GJ0 | 5 | 11.778 | 10.619 | eIF4GII60; | |
| P06730 | 3 | 7.22 | 7.57 | eIF4E | |
| Q5T6W5 | 4 | 0.685 | 0.646 | hnRNPK | Contaminantes no específicamente de unión |
| P62805 | 1 | 0.531 | 0.498 | La histona H4 | |
| H6VRG2 | 18 | - | - | Queratina-1 | Los contaminantes ambientales |
| P35527 | 11 | 0.01 | 0.01 | Queratina-1 citoesqueleto 9 |
Tabla 2. Datos típicos de un experimento de inmunoprecipitación SILAC. Dando datos de ejemplo para una proteína de interés / cebo (altas péptidos, alto cociente), proteínas que interactúan con una proteína de interés (péptidos de alta / baja, alta relación), no específicamente de unión proteínas (alto-bajo Pept /ides, la relación cae por debajo de corte - en este experimento 0.96), y los contaminantes ambientales (a menudo péptidos altos, relación negativa / por debajo del umbral).

Figura 1. Plan experimental. En primer lugar las células se cultivan en medios que carecen de arginina y lisina y sustituidos con isótopos estables marcado con arginina y lisina durante 2 semanas (1). (2) Las células se sembraron en 10 cm 2 platos y transfectadas transitoriamente con plásmidos que codifican GFP (Mock) o proteínas de fusión de GFP (muestras). (3) Las células se lisan y las proteínas GFP o de fusión GFP se inmunoprecipitaron a partir de lisados celulares. (4) Las muestras se combinan en una proporción de 1:1 y se sometieron a análisis de LC-MS/MS. Luego se analiza los datos para eliminar bajo ident proteína confianzacaciones y para seleccionar un nivel de enriquecimiento de proteína correspondiente a las proteínas que interactúan genuinos.
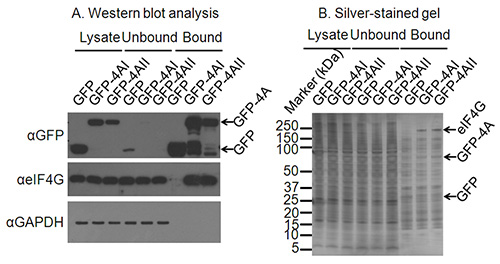
Figura 2. Confirmación de las condiciones de inmunoprecipitación adecuados. A) Análisis de transferencia de Western de lisados de células, así como las fracciones no unidas y unidas de la inmunoprecipitación confirma la expresión y la inmunoprecipitación de la proteína de interés. Un western blot contra GAPDH confirma el agotamiento de proteínas no interaccionan y un western blot, además, una interacción socio conocido de eIF4A confirma la inmunoprecipitación con éxito de las proteínas de unión a la proteína de interés. B) Cuando no se sabe que interactúan los socios de una proteína de interés, un gel teñido con plata puede confirmar inmunoprecipitación de las proteínas que interactúan. En esta plata-mancha bandas de gel ed para GFP y GFP-eIF4AI/II son claras, y una banda que migraba a la talla correcta para eIF4G está presente sólo en los carriles GFP-4AI/II-bound y no en el carril de control GFP.

Figura 3. Los resultados representativos. Histograma que muestra la distribución de las proporciones de proteínas a partir de una repetición de (A) GFP-eIF4AI o (B)-GFP eIF4AII desplegable. El punto de corte 1,96 desviación estándar se marca con una línea discontinua. Proteínas que interactúan caen fuera de los contaminantes normalmente distribuidas-son evidentes a partir de ~ 0,25 y 1 en (A), y ~ 0,3-1,5 en (B).
les/ftp_upload/51656/51656fig4.jpg "/>
Figura 4. Identificación de los miembros del complejo eIF desde una sola réplica de un experimento SILAC IP. Proteínas en verde fueron la proteína de interés utilizada para el pulldown Interactuando proteínas están sombreadas de rojo a blanco, según la relación de log 2 SILAC en el IP SILAC con rojo siendo la proteína más abundante en el análisis, y siendo el blanco el corte 1,96 SD. Proteínas sombreadas en gris no se identificaron en este análisis.
Discusión
La estrategia desplegable SILAC descrito aquí representa un medio muy sensibles y de gran alcance de la detección de la proteína novedosa: las interacciones de proteínas, y además permite la discriminación rápida y simple de los patrones alterados de unión entre las muestras estrechamente relacionados de interés. En este ejemplo se utilizó esta técnica para investigar la proteína: proteína interacciones de las proteínas eIF4AI y eIF4AII 6. Para el conocimiento del autor, este es el primer estudio en la literatura explotar la utilidad de la proteómica SILAC para investigar el interactome celular de estas dos isoformas de eIF4A.
El enfoque descrito anteriormente utiliza un GFP-etiqueta y anti-GFP bolas 9, 10 y, por tanto, las modificaciones pueden ser necesarias para que este enfoque que se utilizará para una proteína específica de interés, por ejemplo, si la etiqueta se coloca en el extremo N-o C-terminal de una proteína. Siempre que sea posible, transferencias Western o ensayos funcionales deberíanllevar a cabo para detectar la unión de una pareja de interacción proteína conocida. En caso de una proteína no tolerar la fusión con una etiqueta de las buenas prácticas agrarias, otra de marcado o estrategias desplegables se han aplicado a jalones SILAC utilizando tanto otras etiquetas (FLAG 11, biotina 12, PEIF (datos propios, sin publicar)), o mediante el uso de anticuerpos primarios contra una proteína de interés en la que siRNA desmontables de la proteína diana ofrece una muestra de control 13. Tales experimentos se han descrito en otra parte en la literatura, pero en breve, paso 1 y pasos 5.4-8 se aplica como anteriormente, con los pasos 2-5.3 modificados según sea apropiado para el sistema de expresión / desplegable de selección utilizando las entradas iguales de proteína como se describe en pasos 2.4 a 2.5. Dado que el grado de cuantificación permiten proteínas de unión no específica a ser retirados en el nivel de análisis, se recomienda omitir la pre-incubación con cuentas de control, o lavados elevadas de sal con el fin de preservar las proteínas de baja afinidad: interacciones de proteínas con una proteína de interés . Una ma nucleasay pueden incluir u omitir de este protocolo de acuerdo con las características específicas de una experiencia particular. Por ejemplo: como las proteínas utilizadas en este método son helicasas de ARN, RNasa cóctel se incluyó en el protocolo para eliminar interacciones indirectas mediadas a través de la ARN (paso 3.2). En algunos casos, sin embargo, podría haber un beneficio de llevar a cabo experimentos paralelos con y sin nucleasa para identificar las interacciones dependientes de ácidos nucleicos.
Dentro de este protocolo, se recomienda el cambio de muestras 'pesados' en replicar experimentos "medio" y para controlar la variación introducida por las diferencias en los medios de comunicación SILAC o el crecimiento celular. Un control alternativo incluye la modificación secuencial de los tres ("light", "medio" y "pesado") los medios de comunicación en la réplica de experimentos. Mientras que este enfoque es potencialmente más estrictas, sí aumenta la complejidad del análisis, como en al menos una repetición, una proteína de interés Wenfermo se produce en las células "light" etiquetados y por lo que es necesario distinguir entre las proteínas identificadas consistentemente en las muestras de "light" (contaminantes ambientales, tales como las queratinas), y los que sólo se enriquecen en las muestras "light" cuando se une a una proteína de interés.
Aunque el uso de datos de cuantificación permite la discriminación de-específica de las interacciones no específicas mediante el uso de un umbral, inevitablemente algunas interacciones genuinas puede ser desechada. El enfoque anterior es un método sencillo y rápido para la identificación de proteínas: proteína interacciones que pueden ser fácilmente intentaron por investigadores sin experiencia previa con la espectrometría de masas, o el análisis de grandes conjuntos de datos de proteínas: proteína interacción. Para la mayoría de usos esto es más que suficiente para la identificación de nuevas proteínas de interés. Otras modificaciones a este enfoque para ayudar a reducir esta pérdida de datos se describen en la literatura y en otros lugares incluyen el uso de un PRbiblioteca frecuencia Otein donde conoce proteínas contaminantes para un conjunto específico de parámetros experimentales (línea celular, matriz de grano, las condiciones de tampón) puede ser excluido 10, 14. Sin embargo, dependiendo de los parámetros experimentales particulares puede ser necesario para realizar un número de experimentos de control para generar un proteoma de talón y por lo tanto esto puede aumentar tanto el gasto y la complejidad del experimento. Para más información sobre esta técnica se encuentra disponible en el sitio web www.peptracker.co.uk 14.
También debe tenerse en cuenta que el protocolo descrito anteriormente consiste en mezclar muestras marcadas de manera diferente en el final del proceso de inmunoprecipitación (denominado una mezcla después de la purificación - MAPA SILAC experimento). Esto se hace en forma de proteínas: proteína interacciones se producen en un equilibrio determinado 15. Debe tenerse en cuenta que otros grupos han combinado este enfoque SILAC MAPA se describe en este protocolo con la incubación de las muestrasantes de la desplegable (purificación después de la mezcla - PAM SILAC) para diferentes periodos de tiempo (20 min a 2 horas se han utilizado en la literatura) 15, 16. Sobre la base de la rapidez con gotas de una relación de proteína a 01:01, es posible investigar cualitativamente afinidades de unión y para definir las proteínas como las proteínas que interactúan estables o dinámicos 15.
En resumen, jalones SILAC representan un medio muy poderoso para identificar las proteínas que interactúan con una determinada proteína de interés, en un ambiente fisiológicamente relevante. La técnica se puede adaptar muy fácilmente a un número de diferentes estrategias de purificación, lo que permite su aplicación a cualquier proteína dada de interés. La cuantificación de los resultados simplifica enormemente la identificación de interacciones auténticos, y permite la relajación de las condiciones de tampón estrictas utilizadas para eliminar los aglutinantes no específicos, y por lo tanto preserva las interacciones de baja afinidad. Como hasta tres muestras se pueden comparar en lo anteriorestrategia, la técnica tiene ventajas claras en comparación de las diferencias en la proteína de unión entre las diferentes isoformas de proteínas, las proteínas mutantes, o el efecto de los inhibidores farmacológicos. A medida que se analizaron cortes de gel enteras en lugar de las bandas individuales que mancha por Coomassie, el número de proteínas identificadas en el alto grado de confianza suelen ser mayores que las señaladas en el GST / TAP-menú desplegable estándar y experimentador sesgo en la selección de proteínas de interés se quita. Por tanto, la técnica se compara muy favorablemente con otras técnicas utilizadas comúnmente utilizados en la identificación de nuevas proteínas-las interacciones (levadura de 2 híbridos, GST / His o TAP Pulldowns).
Divulgaciones
Los autores declaran que no tienen intereses financieros en competencia.
Agradecimientos
Este trabajo fue apoyado por becas de la Wellcome Trust y BBSRC a IG. IG es un Wellcome Senior Fellow.
Materiales
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium (DMEM) | Dundee Cell Products | LM010 | DMEM lacking Arginine and Lysine. (and containing L-glutamine) |
| Dialysed FBS (10 kDa cutoff) 500 ml | Dundee Cell Products | DS1003 | |
| Arginine (R0) 25 g | Sigma-Aldrich | A8094 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R6) 0.5 g | Cambridge Isotope Labs | CLM-2265 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R10) 0.5 g | Cambridge Isotope Labs | CNLM-539 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Lysine (K0) 25 g | Sigma-Aldrich | L8662 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K4) 0.5 g | Cambridge Isotope Labs | DLM-2640 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K8) 0.5 g | Cambridge Isotope Labs | CNLM-291 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Penicillin/streptomycin, Liquid. 100 ml | Gibco | 15140-122 | |
| Lipofectamine 2000 transfection reagent | Invitrogen | 11668-027 | |
| GFP-trap Agarose (500 μl resin) | Chromotek | gta-20 | |
| 5x SDS Sample loading buffer | Fisher Scientific | PN39000 | The use of purchased rather than homemade sample buffer is recommended to minimize keratin contamination. |
| Protease inhibitor cocktail set III | Calbiochem | 539134 | |
| 1.7 ml prelubricated tubes | Costar | 3207 | |
| BCA protein assay kit (1 L) | Pierce | 23225 | |
| Tris (Trizma) | Sigma-Aldrich | T1503 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E6758 | |
| Sodium chloride | Sigma-Aldrich | S3014 | |
| Rnase cocktail | Ambion | AM2286 | |
| NP-40 alternative | Millipore | 492016 |
Referencias
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem Soc Trans. 38, 875-878 (2010).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), (2012).
- Trinkle-Mulcahy, L. Resolving protein interactions and complexes by affinity purification followed by label-based quantitative mass spectrometry. Proteomics. 12, 1623-1638 (2012).
- Emmott, E., et al. The cellular interactome of the coronavirus infectious bronchitis virus nucleocapsid protein and functional implications for virus biology. J Virol. 87, 9486-9500 (2013).
- Li, W., Belsham, G. J., Proud, C. G. Eukaryotic initiation factors 4A (eIF4A) and 4G (eIF4G) mutually interact in a 1:1 ratio in vivo. J Biol Chem. 276, 29111-29115 (2001).
- Nielsen, P. J., Trachsel, H. The mouse protein synthesis initiation factor 4A gene family includes two related functional genes which are differentially expressed. EMBO J. 7, 2097-2105 (1988).
- Galicia-Vazquez, G., Cencic, R., Robert, F., Agenor, A. Q., Pelletier, J. A cellular response linking eIF4AI activity to eIF4AII transcription. RNA. 18, 1373-1384 (2012).
- Zakowicz, H., et al. Mutational analysis of the DEAD-box RNA helicase eIF4AII characterizes its interaction with transformation suppressor Pdcd4 and eIF4GI. RNA. 11, 261-274 (2005).
- Rothbauer, U., et al. A versatile nanotrap for biochemical and functional studies with fluorescent fusion proteins. Mol Cell Proteomics. 7, 282-289 (2008).
- Trinkle-Mulcahy, L., et al. Identifying specific protein interaction partners using quantitative mass spectrometry and bead proteomes. J Cell Biol. 183, 223-239 (2008).
- Dobreva, I., Fielding, A., Foster, L. J., Dedhar, S. Mapping the integrin-linked kinase interactome using SILAC. J Proteome Res. 7, 1740-1749 (2008).
- Mittler, G., Butter, F., Mann, M. A SILAC-based DNA protein interaction screen that identifies candidate binding proteins to functional DNA elements. Genome research. 19, 284-293 (2009).
- Selbach, M., Mann, M. Protein interaction screening by quantitative immunoprecipitation combined with knockdown QUICK. Nat Methods. 3, 981-983 (2006).
- Boulon, S., et al. Establishment of a protein frequency library and its application in the reliable identification of specific protein interaction partners. Mol Cell Proteomics. 9, 861-879 (2010).
- Wang, X., Huang, L. Identifying dynamic interactors of protein complexes by quantitative mass spectrometry. Mol Cell Proteomics. 7, 46-57 (2008).
- Fang, L., et al. Characterization of the human COP9 signalosome complex using affinity purification and mass spectrometry. J Proteome Res. 7, 4914-4925 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados