Method Article
SILAC - 면역 정량 프로테오믹스를 사용하여 포유 동물 세포에있는 단백질 상호 작용 파트너의 식별
요약
단백질 상호 작용 : SILAC의 면역 실험은 새로운 단백질을 발견하기위한 강력한 수단을 나타냅니다. 있도록하여 제어 및 시험 샘플, 진정한 상호 작용 모두에서 단백질 풍부 정확한 상대적 정량화 간단 실험 오염물 구별 될 수 있고, 선호도가 낮은 상호 작용은 덜 엄격한 버퍼 조건의 사용을 통해 유지.
초록
단백질 상호 작용 : 면역 친화 정제, SILAC의 면역과 결합 정량 프로테오믹스는 새로운 단백질의 발견을위한 강력한 수단을 나타냅니다. 제어 및 시료 모두에서 단백질 풍요의 정확한 상대 정량을 허용함으로써, 진정한 상호 작용은 쉽게 실험 오염 물질을 구별 할 수있다. 저 친화력 상호 작용은 덜 엄격한 조건 버퍼의 사용을 통해 유지하고 쉽게 식별 유지 될 수있다. 이 프로토콜은 질량 분석 시설에 제출을위한 준비를 한 다음 그 단백질을 태그 친 화성의 안정 동위 원소 표지 된 아미노산, 형질 전환 및 면역과 조직 배양 세포의 라벨에 대해 설명합니다. 이 프로토콜은 또한 단백질과 상호 작용하는 세포의 파트너를 식별하기 위해 질량 분석기에서 리턴 데이터를 분석하고 해석하는 방법을 논의한다. 예로서이 기술의 iDEN인가eIF4AI 및 eIF4AII : 진핵 세포 번역 개시 인자에 결합하는 단백질을 tify.
서문
단백질 기능을 이해하는 데 필수적인 단계는 관련 상호 작용하는 단백질의 식별입니다. 단백질이 알 수없는 장소에서 사용 가능한 다양한 기술, 자신의 장점과 단점 각각이 있습니다. 이들은 효모 두 하이브리드 시스템, 재조합 단백질을 사용하여 풀다운 분석뿐만 아니라 탠덤 선호도 정화 또는 TAP-태그 1, 2 (가) 있습니다.
이러한 기술에 대한보다 최근의 첨가 (SILAC) 3 세포 배양에서 아미노산의 안정 동위 원소 표지를 사용하여 정량적 질량 분석 하였다 관련된 포유 동물 세포주에서 그 단백질의 친화도 정제의 조합이다. 이는 해당 셀의 현지화 및 번역 후 변형의 효모 두 하이브리드 접근 방식을 통해 장점을 통해 장점뿐만 아니라, 교란되지 않은이 기존의 TAP-태그가 정말이에요에 사용자를 허용하는 양적보다는 질적 접근 방식 인 것을디일 특이 적으로 결합 호스트 요인, 단백질과 오염 물질을 상호 작용이 아닌 구체적으로 구분합니다. 샘플은 일반적으로 오히려 개별 단백질 밴드보다, 전체 분석으로 또한, 그 단백질은 마찬가지로 겔에서 단백질을 마이그레이션 없으며 일반적으로 염색 한 후 볼 수 있도록 충분한 수준에 존재해야합니까, 선도에 의해 마스크되지 않습니다 자신있게 확인 된 단백질 4의 증가 숫자.
이 기술을 설명하기 위해 밀접하게 관련 진핵 번역 개시 인자의 GFP 융합은 eIF4AI 및 eIF4AII 90 %의 동일한 아미노산 서열을 통해 그 점유율은 SILAC - 면역 정량 프로테오믹스에 의해 조사 하였다. 인간 eIF4AI 및 II는 GFP가 eIF4A의 N-말단에 융합되는 융합 단백질을 형성하는 pEGFP-C1에 클로닝 하였다. 일시적 형질 감염은 안정적인 동위 원소 표지 된 293T 세포에 이러한 구조물을 제공하는 데 사용 된 안정한 세포주를 생성하기위한 필요성을 방지한다.
셀은 제 관심 단백질을 코딩하는 플라스미드 DNA의 형질이어서 SILAC 세포 배양 배지에서 2 주 표지 하였다. 세포는 그 후, 용해 단백질 농도는 정규화 및 용해질 친 화성 동등한 양 안티 GFP 아가상에서 정제 하였다. 용출액을 동량이어서 합하고 LC-MS/MS 분석을 위해 제출되었다. 이 분석의 결과는 다음 높은 신뢰성 단백질을 식별하기 위해 처리된다 : 단백질 상호 작용 (도 1).
SILAC의 면역 직접 상호 작용뿐만 아니라 선호도가 낮은 또는 단백질 복합체 4 간접적 인 상호 작용뿐만 아니라 식별 할 수 있습니다. 이 시스템을 사용하여, eIF4AI 및 II immunoprecipitations는 기본 바인딩 파트너 eIF4G (이소 I / II 및 III) 5의 재현과 신분을 확인뿐만 아니라 eIF4E에 간접 상호 작용, 그리고 eIF3 단지의 다양한 구성 요소를 허용했다.
프로토콜
1. 생성 및 SILAC 표지 세포 라인과 Passaging
참고 : 트립신이 샘플의 불완전 라벨로 이어질 것이 레이블이없는 아미노산을 포함 할 수 있으므로 트립신-EDTA의 사용은 분석을위한 실험 샘플의 계대 및 준비의 모든 단계에서 피해야한다.
- SILAC 미디어의 병을 준비하려면 (포함의 Arg /리스 무료 DMEM의 500 ㎖ 병에 적절하게 SILAC 표지 아르기닌 (84 ㎎ / PBS에 ML)과 라이신 (PBS에서 146 ㎎ / ㎖)의 0.5 ㎖ 분취 량을 추가 L-글루타민).
- 다음으로, 500 ㎖ 병으로 투석 FBS 50 ㎖ 페니실린 / 스트렙토 마이신 5 ㎖를 추가합니다.
참고 : HEK의 293T 세포 (ATCC)가 부족 DMEM 매체에서 유지되어야한다 아르기닌과 라이신 등 (R0K0) 보충, 중간 (R6K4) 또는 무거운 (R10K8) 아미노산, 투석 소 태아 혈청 (10 kDa의 컷오프), 페니실린 / 스트렙토 마이신. 세포는 완전한 보장하기 위해 5 세포 분열의 최소 매체에 유지해야라벨. 대부분의 경우 세포는 쉽게 ≥ 2 주에 표시되어 있습니다. - 계대에 대한 세포를 분리하기 위해, 세포가 플라스크를 타격하여 단일 층에서 탈락한다. 대안 세포 스크레이퍼 또는 효소 무료, PBS 기반의 세포 분리 버퍼를 사용하여 사용을 포함한다.
참고 : 용지를 절약하기 위해 세포는 T25 플라스크에 계대해야 더 큰 세포 수는 직전에 실험을 생성합니다. - 이전에 형질 세포에 24 시간 조사에서 각각의 실험 조건에 대해 하나의 10cm 2 접시에 3.5 × 10 6 SILAC 표지 된 세포를 씨앗.
pEGFP 융합 구조로 표기 세포의 2. 형질
- 세포에서 용지를 제거하고 무 항생제 SILAC의 DMEM (빛, 중간 무거운) 미디어의 10 ML로 교체합니다.
참고 : 그들은 리포좀 기반의 형질 전환 시약의 효율성을 방해 할 수 있기 때문에 항생제가 미디어에 추가 할 수 없습니다. - Prepa적절한 플라스미드의 10 μg의 혼합 다시 (epEGFP-C1, pEGFP-eIF4A-I, pEGFP-eIF4A-II) 무 항생제 SILAC DMEM 500 μL와 항생제없는 SILAC의 DMEM (10)를 포함하는 500 μL와 혼합 형질 전환 시약 ㎕의 (예를 들어, 리포 펙 타민 2000). 그것을 피펫 아래로 여러 번에 의해 철저하게 반응을 섞는다.
- 실온에서 20 분 동안 반응 혼합물을 배양한다 후 적가 세포 단층을 추가. 좌우로 부드럽게 플레이트 록.
- 37 ° C에서 형질 전환 세포를 품어 10 % CO 2 24 시간 동안.
참고 : 명백한 독성 나타낼 결과 단백질의 발현이, 그 후에 형질 감염된 플라스미드의 양 및 / 또는 후속 발현 기간의 지속 시간을 감소시킬 필요가있을 수있는 경우.
3. 수확 세포 용 해물
- 세포 스크레이퍼를 사용하여 얼음 차가운 PBS로 접시에서 수확 세포. 4, 220 XG에서 원심 분리하여 세포를 수집76; 5 분 C. 얼음처럼 차가운 PBS의 10 ML에있는 세포에게 더 배를 씻으십시오.
- 세포 용해 버퍼 200 μL를 5 μL의 1X 농도의 RNase 칵테일 (옵션)에 새롭게 추가 된 프로테아제 억제제 칵테일 III을 포함하는 (10 mM의 TrisCl /의 pH 7.5, 150 mM의 염화나트륨, 0.5 mM의 EDTA, 0.5 % NP40)에서 세포 펠렛을 재현 탁 ML 당.
주 : 결합 활성을 공지 된 핵산과 단백질의 경우, 이전 석출 파쇄물에 클레아 제를 추가 할 필요가있다. 클레아가 추가 될 경우, 샘플은 매 10 분을 피펫 팅하여, 얼음 위에서 30 분 동안 얼음에서 인큐베이션한다. 아가로 오스 겔 전기 영동에 의해 샘플 및 분석의 작은 부분에서 전체 핵산을 추출하면 클레아의 효과를 시험 할 수있다. - 13,000 XG에 원심 분리기 샘플, 10 분 동안 4 ° C는 수용성 세포 용 해물로 뜨는을 유지합니다.
- 세포 용 해물의 농도는 제조자의 지시에 따라 BCA 분석에 의해 평가되어야한다.
- 500 μL의 최종 부피 단백질 농도를 정상화 프로테아제 억제제 칵테일 III을 함유하는 용해 버퍼를 사용한다.
- 희석액 500 μL를 추가 최종 1X 농도에서 단백질 분해 효소 억제제 칵테일 III를 포함 (10 mM의 TrisCl /의 pH 7.5, 150 mM의 염화나트륨, 0.5 mM의 EDTA) 1 ML에 볼륨을 조절합니다. 샘플의 50 μL 나누어지는 샘플 입력으로 유지되어야하고, 용해 액 (예를 들면, GFP-트랩) 항-GFP의 구슬을 준비하는 동안 얼음에 보관.
참고 : 일반적으로 수익률이 최종 1 ㎖ 샘플에서 단백질의 1 ~ 3.5 mg의 사이에 다릅니다. 위의 버퍼가 관심이 많은 단백질에 적합한 반면, 다른 사람들은 미끼 단백질이 용해되어 있는지 확인하고, 단백질 - 단백질 상호 작용을 유지하기 위해 버퍼의 구성 요소를 수정해야 할 수도 있습니다. 가능한 변경은 완충제 (인산염, HEPES), 소금 농도 (150 ~ 500 ㎜), 세제의 선택, 또는 기타 첨가제를 포함한다.
4. 안티-G에 바인딩FP의 구슬
- 간단히 구슬을 재현 탁하는 비드 슬러리를 소용돌이. 끝으로 200 μL 피펫 팁을 사용하여 잘라 신선한 튜브에 샘플 당 구슬의 25 μl를 전송합니다.
참고 : 사용자는 샘플 간 편차를 최소화하기 mastermix 같은 단일 SILAC 실험을 위해 비드를 준비해야한다. - 비드 슬러리의 각 25 μL를 들어, 5 분 2,700 XG에서 구슬을 희석 버퍼의 20 권 (75 μL 슬러리 1,500 μL)를 추가하고, 원심 분리기. 다음으로, 희석액의 20 권의 구슬에게 더 배를 세척한다.
- 25 μl의 구슬 슬러리 당 100 ㎕의 희석 버퍼를 추가합니다. 끝으로 200 ㎕의 팁을 사용하여 절단 단계 3.6 SILAC 표지 된 각각의 샘플이 재현 탁 슬러리의 85 μl를 전송합니다.
- 2 시간 동안 4 ° C에서 회전에 구슬 샘플을 품어.
MS 분석을위한 시료 5. 세척, 용출, 및 준비
- 2,700 XG에 원심 분리기 샘플, 46, 5 분 동안 C. 상층 액 50 ㎕를 폐기 상층 액의 나머지, 언 바운드 샘플로 유지해야한다.
- 1 ㎖ 희석 버퍼는 구슬을 재현 탁 각 튜브에 추가해야합니다 샘플을 5 분 동안 4 ° C, 2,700 XG에서 원심 분리. 상등액을 폐기해야합니다. 이 두 번 수행해야합니다.
- 2X SDS 로딩 버퍼 50 μL를 첨가하여 구슬에서 단백질을 용출하고, 10 분 동안 95 ° C에서 가열. 4 ° C에서 2 분 동안 2,700 XG에서 원심 분리하여 구슬을 펠렛
- 필요한 때까지 다음 -80 ° C에 저장할 수 있습니다 prelubricated 튜브에 뜨는을 유지합니다. 질량 분석 시설에 제출, 표지 샘플을 1시 1분 1초 (예를 들어, 각 10 μL) 혼합과 혼합 된 샘플을 제출합니다.
참고 :이 시점에서 샘플을 웨스턴 블롯, 실버 염색 또는 알려진 / 알려지지 않은 바인딩 파트너들과의 상호 작용을 테스트하는 다른 방법으로 테스트 할 수있다. 예를 그림에 제시되어있다서양 얼룩에 의해 eIF4G, 관심의 단백질에서 풀다운 샘플에 존재하는 실버 염색 밴드의 모양,하지만 컨트롤 샘플 - 알려진 상호 작용 파트너의 특이 적 결합을 보여주는 우레 2. - LC-MS/MS 분석을 위해 샘플을 제출하십시오.
참고 : 관심 태그 단백질이 성공적으로 상호 작용 파트너, 레이블이있는 각 샘플의 동일한 볼륨 (빛, 중간 무거운) 결합 LC-MS/MS 분석을 위해 제출 결합되어 한 번에 만족을. 이 분석을 위해 IP 샘플 30 μL의 총을 제출하는 것이 일반적이다. 이것은 30 μL의 총을주는 매체라는 10 μL 무거운 표지 샘플 10 ㎕의 빛 표지 샘플의 10 μl를 혼합 수반한다.
6 데이터 분석 I :. 결과를 이해하고, 저 신뢰 식별 제거
참고 : 프로테옴 Discoverer는 소프트웨어에 의해 반환 된 열 제목의 목록은 표 1에 제시되어있다 DIF에게.ferent 소프트웨어 (예를 들어, MSQuant, MaxQuant)는, 그러나, 이들 중 일부만을 다른 제목을 반환 분석에 필요한 이러한 다양한 소프트웨어 패키지에 공통된다. 데이터는 항상 비율의 각 샘플 비율을 비교 확인 된 각각의 단백질에 대한 접근 번호 (중간 대 빛을, 무거운 등 대 무거운, 중간 대 등)를 포함해야한다, 확인 된 고유 한 펩타이드의 수, 위양성률 또는 신뢰 표시의 형태.
- 데이터를 이동하기 전에, 먼저 새 스프레드 시트에 원시 데이터를 복사합니다. 이 스프레드 시트에서 접근 번호를 부여 제외한 모든 열을 제거, 독특한 펩타이드의 수, 샘플 비율의 변화, 및 단백질에 대한 설명을 비교하는 비율. 복제 실험이 수행 된 경우, 이러한 각각의 실험은 별도의 탭에 나타나는로, 하나의 엑셀 파일로 결합해야합니다.
참고 : 낮은 신뢰 데이터는 단일 고유 펩티드에 의해 식별 단백질을 포함하고, 그 w여기 정량화 할 수 없었습니다. - 펩티드의 숫자로 데이터를 주문하고 하나 이상의 펩티드가없는 단백질에 대한 항목을 제거하는 탁월한 '정렬'기능을 사용합니다. 그런 비율로 정렬하고 SILAC 비율 (정량화 단백질)이 부족한 단백질을 제거합니다.
- 'SILAC 비율이'셀 식별자를 대체한다 '= 로그 (SILAC 비율, 2)': 수식을 사용하여 로그 2 값으로 SILAC 비율을 변환 할 수 있습니다.
주 : 0과 1 사이의 값으로 제한되지 풍요의 감소를 나타내는 단백질에 대한 데이터 SILAC 비율의 결과로서,이 증가 또는 감소 모두 단백질 수단으로서, 로그이 SILAC 비율 SILAC 비율을 변환하는 통상 샘플은 2 또는 -2 4 배 증가 또는 감소 풍부하고, 3 또는 -3 9 배 증가 또는 각각 풍부 감소를 나타내는 로그 스케일로 표현된다. 이 변환 후, 데이터는 조지아 맞아야0의 로그 2 SILAC 비율을 중심으로 ussian 분포.이의 예를 그림 3에 제시되어있다. - 엑셀 파일에 새 열을 생성하고 모든 샘플 / 모의 열 2 SILAC 비율을 기록 계산합니다. "= 1/ratio"샘플 / 모의 비율을 모의 / 샘플 비율의 변환의 경우, 공식을 사용
7 데이터 분석 II :. 또한 연구에 대한 높은 신뢰의 상호 작용을 선택
- 오픈을 Graphpad 프리즘, 뉴> 데이터 표와 그래프 창의 왼쪽에있는 목록에서 선택 ... '열'을 선택하고 입력 / 데이터 가져 오기를 선택> 열 옵션에 쌓인 복제 값을 입력합니다. 을 눌러 '만들기'.
- 선택하고 새 프리즘 파일에 Excel 스프레드 시트에서 주어진 로그 2 샘플 / 모의 SILAC 비율 열을 복사합니다.
- 드롭 다운 '삽입'메뉴를 클릭하고 '새 해석'을 선택합니다. 열 분석에서 '선택주파수 분배 '. 기본 옵션을 유지하는 것은 '확인'을 클릭합니다.
주의 :이 단계는 소정의 비율로 식별 된 단백질의 수를 나타내는 히스토그램을 생성한다. 이 가우스 분포를 형성한다. - '결과'폴더에 새로운 '히스토그램'섹션이 생성 된 것입니다. 주파수 분포 섹션을 선택합니다.
- 드롭 다운 '삽입'메뉴를 클릭하고 새로운 분석을 선택> XY> 비선형 회귀 (곡선 맞춤). '가우스 분포'를 클릭하고 확인을 클릭합니다.
주 :이 단계는 이후 중요성에 대한 임계 값을 계산하는데 사용될 수있는 값을 산출 단계 7.3에서 생성 도수 분포 곡선 데이터에 적합하다. - 나타나는 결과 창에서 평균과 표준 편차가 주어집니다. 평균치에 표준 편차 1.96을 추가하여 임계 값을 생성한다.
참고 :이 값은 평균과 가우스 DIST의 표준 편차를 나타냅니다ribution 아닌 총 집합. 1.96 표준 편차는 95 % 신뢰 한계 (0.05 ≤ P)에서 임계 값을 배치합니다. 2.58 SD는 99 %, 3.3 SD 99.9 %를 줄 것이다. 이 될 수 있으므로 임계 값은 각각의 복제 실험에 대해 개별적으로 결정되어야한다.
8 데이터 분석 III :. 복제 데이터 집합을 병합
참고 : 일반적인 SILAC 풀다운 실험이 이상적으로 결과에 대한 미디어의 영향에 대해 제어 할 수있는 반복 중 하나에 대한 전환 매체와 무거운 표시된 샘플, 세 번 수행해야합니다 상호 작용 파트너를 식별하는 자신감을. 최적의 이들 중 하나를 나타내는 'switched' 매체 샘플과 세 실험 중 적어도 둘을 상호 작용으로 나타난다 단백질은 높은 신뢰성의 상호 작용이다.
- , Excel 파일 찾기 '결합'이라는 새로운 탭을 만들 수 있습니다. 라벨 열 A '기탁'및 O 각각의 모든 접근 번호를 복사이 하나의 컬럼에 F 각각의 실험.
- '데이터'탭을 클릭 한 다음 '중복 제거'옵션을 선택합니다.
- SILAC 비율, 각 실험에서 비의 변화뿐만 아니라 단백질 이름 / 설명 열의를위한 열을 만듭니다.
- 설명 탭에서 각각의 접근 번호의 설명을 수집하는 VLOOKUP 수식을 사용합니다. 단백질 설명을 채우기 위해 열 아래 공식을 끕니다. VLOOKUP 수식은 "= VLOOKUP (AccessionNo, WheretoLook, ColumnsAccross, 거짓)", 여기서.! AccessionNo는 X가 WheretoLook는 '실험 탭 이름'$ 2 $입니다 행 번호 인에 $ 도끼입니다 : $ Y $ Z 어디 Y 및 Z는 스프레드 시트에 참조되는 데이터의 오른쪽 아래 부분입니다. ColumnsAccross가 기탁 숫자 맞은 열 수 있습니다BER 열에서 원하는 값이있다.
참고 : 기탁 번호가 열이며, 그 데이터가 열 E.에 자리 잡고 있습니다 예를 들어, 여기에 값 (열 1로 계산) 5 것입니다. 접근 번호가 b 단계에서 모으고, 그것은 VLOOKUP 한 실험에서 필요한 모든 이름을 해낼 수있을 가능성이 높습니다. 그것은 N / A를 반환하는 경우, 모든 설명이 획득 될 때까지 차례로 각 실험에서 기술을 취득하기 위해서 공식을 수정합니다. - 결합 된 데이터 집합의 단백질 상호 작용을 강조하기 위해 개별적으로 각 비율 열을 선택하고> 조건부 서식> 셀을 강조 표시 규칙에 따라, 홈 탭에 대한 자세한 규칙을 클릭합니다.
- 선택 스타일 '고전'. '셀 값'가 포함 된 형식으로 만 셀 '보다 크거나 같다'. 상자, 1.96 표준 편차 값을 입력하고,에서 '확인'을 클릭합니다.
- 각각의 긍정적 인 상호 작용에 대한 비율의 변화를 평가합니다. %의 편차를 뺀 경우iability가 임계 값 이하 '충돌'을 (를) 삭제할 것, 이것은 신중하게 취급되어야한다.
참고 :이 함께 단백질 'SILAC 비율을 구성하는 각각의 펩타이드에 대한 개별 비율이 얼마나 일관성 나타내고, 백분율로 지정됩니다. 비 변동이 고려 및 단백질이 임계 값 이상 SILAC 비율을 제공하는 경우,이 단백질은 히트를 나타낸다. 비율의 변동성이 임계 값 이하로 떨어질 범위를 제공하는 경우, '히트'는 오염 물질을 나타낼 수있다. - 결과 각 열에 대해이 과정을 반복 실험을 통해 비교. 두 개 이상의 실험에서 식별 강조 단백질은 높은 신뢰성의 상호 작용을 나타낸다.
주의 : 다른 실험에서 단백질이 다른, 또는 다중 접근 번호에 따라 확인되고 있습니다 단백질 접근 번호를 할당하는 데 사용되는 데이터베이스에 따라. 그것은 단백질이 실수로 생략되지 않도록하려면이 옵션을 선택하는 것이 중요합니다.
결과
전형적인 SILAC 풀다운 실험에서 확인 된 단백질 (> 90 %)의 대부분은 오염 물질뿐만 아니라, 친 화성 기질에 비 - 특이 적으로 결합하는 단백질을 나타내고 세척 프로토콜 세포질 오염 물질의 대부분을 제거 할 때에도 이것은,도 2b에 도시 같은 GAPDH (그림 2A)로. 그러나, 비 구체적으로 정규 분포에 결합 단백질의 클러스터링은 특히이 같은 구별 할 관심의 단백질에 결합 단백질이 배경보다 높은 샘플 / 모의 비율이 있습니다. 오염 물질은 이론적으로 0의 로그 2 SILAC 비율 주위 클러스터해야하는 동안, 이것은 반드시 사실이 아니다, 이것의 예는 그림 3에 나와있다. 이런 경우에 대한 가능성이있는 이유보기에 동일하지 않은 볼륨 또는 용해 액의 농도를로드, 세포의 불완전한 SILAC 라벨을 포함 안티 GFP 구슬, 정제 또는 동일하지 않은 혼합하는 동안 구슬의 실수로 손실정제 과정 (3)의 단부에 시료 주입. 그러나, 가정 데이터가 정규 분포 오염물의 평균으로부터 표준 편차를 임계치에 기초 분석, 데이터 중심의 사소한 변화가 결과의 품질에 영향을주지해야한다.
이 연관성 단백질 간의 단백질 상호 작용의 차이를 비교하면 그 한 단백질 (하나 의한 형질 전환 효율의 편차, 또는 단백질 또는 mRNA를 본질적인 속성) 초보다 세포 내에 높은 수준으로 생성되는 경우, 유사한 상황이 발생할 수있다 . 어떤 표정의 변화 (예를 들면,도 2a)이이 샘플 SILAC 비율을 분석함으로써 보정 될 수있다. 이 예에서 이러한 GFP-eIF4AI과 GFP-eIF4AII 샘플 것입니다. 섹션 7에서 논의 된 바와 같이 4AI/4AII SILAC 비율을 분석함으로써, 그 바인딩 동형간에 상당히 변화 단백질을 식별하는 것이 가능하다.
그림 4에서 클래스 = "jove_content">, 실시 복제 실험 중 하나에서 확인 된 eIF4A 결합 단백질의 표현은이 프로토콜이 달성 개시 인자 복합체의 범위를 나타내는 표시됩니다. 가장 높은 비율은 일반적으로 멀리 eIF4A 바인딩 사이트에서 사이트에 eIF4G에 바인딩의 eIF4E, 더 낮은 비율로, eIF4A에 직접 결합 eIF4G으로 관찰되었다. 낮은 비율은 eIF3 단지의 구성원에 대한 관찰되었다. 그러나, 이것은 분명히 실험이 만족 eIF4AI과 II의 직간접 바인딩 파트너를 식별 것을 보여줍니다. 높은 서열 동일성 6에서 예상 할 수 있듯이 단백질 : 단백질 상호 작용은 대부분이 실험 시스템 7, 8의 두 가지 아형 사이에 보존 나타났다. 상호 작용 단백질의 일부의 선택은 데이터 포맷을 나타낸 표 2에 나타내었다.| 열 머리글 | 기술 |
| 기탁 | 시퀀스의 접근 번호를 표시합니다 |
| 적용 범위 | 식별 된 펩티드에 의해 덮여 단백질 서열의 비율 |
| ♯ PSM | 펩타이드 스펙트럼의 경기 |
| 펩티드를 ♯ | 단백질 식별 고유 한 펩타이드의 총 수 |
| ♯ AA 형 | 아미노산의 단백질의 길이 |
| MW (다) | 달톤의 단백질의 분자량. 수정 제외 |
| CALC. PI | 단백질의 이론적 등전점 |
| 점수 | 개별 선택의 합을 나타내는 단백질 (의 총 점수idual 펩타이드 점수). 중요성에 필요한 정확한 점수는 실험에 따라 다를 것입니다. MS 시설은 일반적으로 5 %의 거짓 발견 율 차단을 적용합니다. |
| 순서 | 단백질을 구성하는 아미노산의 서열 |
| 비 | 두 번째 레이블 샘플에 비해 이름이 표시된 샘플의 펩티드의 상대 강도, |
| 비율 카운트 | 주어진 단백질의 비율을 계산하는 데 사용 된 펩티드 비율의 개수 |
| 비율 변화 (%) | 주어진 단백질의 비율을 계산하는 데 사용되는 개별 펩티드 비율의 변동성 |
| 기술 | 단백질의 이름 |
표 1. 프로테옴 Discoverer는 보고서에서 표준 열 머리글.유용한 정보는이 모든 열에서 얻은 수있는하는 동안,이 분석에 중요한 사람들은 굵게 표시됩니다.
| 기탁 | 펩티드 | 4AI/Mock | 4AII/Mock | 이름 | SILAC 분석 |
| A8K7F6 | 21 | (100) | 1 | eIF4AI | '미끼'단백질 |
| Q14240 | 22 | 0.01 | 90.855 | eIF4AII | |
| G5E9S1 | 25 | 47.575 | 30.53 | eIF4GI | 단백질 상호 작용 |
| Q59GJ0 | 5 | 11.778 | 10.619 | eIF4GII60; | |
| P06730 | 3 | 7.22 | 7.57 | 의 eIF4E | |
| Q5T6W5 | 4 | 0.685 | 0.646 | hnRNPK | 비특이적 결합 오염물 |
| P62805 | 1 | 0.531 | 0.498 | 히스톤 H4 | |
| H6VRG2 | 18 | - | - | 각질-1 | 환경 오염 물질 |
| P35527 | 11 | 0.01 | 0.01 | 각질-1 세포 골격 9 |
표 2. SILAC의 면역 실험에서 일반적인 데이터. 관심 / 미끼 단백질 예를 들어 데이터를주고 (높은 펩티드, 높은 비율),이자 (최고 / 최저 펩티드, 높은 비율)의 단백질과 상호 작용하는 단백질, 비 특이 적으로 결합 단백질 (고 / 저 pept)이 실험 0.96에, 환경 오염 물질 (종종 높은 펩타이드, 임계치 이하의 음의 비율 /) - 아이즈, 비는 컷오프 아래로 떨어질.

그림 1. 실험 계획. 첫째 셀을 2 주 동안 아르기닌과 라이신이 부족 미디어에서 성장 및 안정 동위 원소 표지 아르기닌과 라이신으로 치환된다 (1). (2) 세포는 10cm 2 접시에 씨앗을 품고 및 일시적 (샘플) (모의) 또는 GFP 융합 단백질 GFP를 코딩하는 플라스미드로 형질됩니다. (3) 세포를 용균 및 GFP 또는 GFP 융합 단백질은 세포 용 해물로부터 면역 침전된다. (4) 시료를 1:1의 비율로 결합 LC-MS/MS 분석을 위해 제출됩니다. 데이터는 신뢰도가 낮은 단백질 답하라를 제거하는 분석명시된 내용과 진정한 상호 작용하는 단백질에 해당하는 단백질 농축의 수준을 선택합니다.
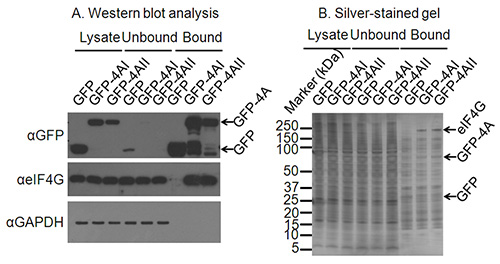
그림 2. 적절한 면역 상태를 확인. A) 세포 용 해물의 웨스턴 블롯 분석뿐만 아니라 면역의 언 바운드 및 바운드 분수는 표현과 관심의 단백질의 면역을 확인한다. GAPDH에 대한 서양 얼룩은 eIF4A의 알려진 상호 작용 파트너가 관심의 단백질의 상호 작용 파트너가 알 수없는 관심. B의 단백질)에 결합 단백질의 성공적인 면역을 확인되지 않은 상호 작용하는 단백질과 다른 서양 얼룩의 고갈을 확인 실버 묻은 겔은 상호 작용하는 단백질의 면역을 확인 할 수 있습니다. 이 실버 얼룩에 GFP 및 GFP-eIF4AI/II 에드 젤 밴드는 깨끗한 지, eIF4G에 대한 올바른 크기로 마이그레이션하는 밴드는 GFP-4AI/II-bound 차선이 아닌 GFP 제어 차선에 존재합니다.

그림 3. 대표 결과. 막대 그래프 (A) GFP-eIF4AI 또는 (B) GFP-eIF4AII 풀다운의 한 반복에서 단백질 비율의 분포를 표시합니다. 1.96 표준 편차 차단은 점선으로 표시되어 있습니다. 일반적으로 분산 된 오염 물질의 범주 밖에있는 상호 작용하는 단백질 (B)에서 0.25 ~ 1 (A)에서, 그리고 ~ 0.3 ~ 1.5에서 명백하다.
les/ftp_upload/51656/51656fig4.jpg "/>
그림 4. SILAC IP 실험의 단일 복제본 EIF 복잡한 회원의 식별. 단백질이 녹색으로 단백질 상호 작용 풀다운 메뉴에 사용되는 관심의 단백질했다가 빨간색 SILAC IP에서 2 SILAC 비율을 기록 흰색있어서, 빨간색에서 음영 분석에서 가장 풍부한 단백질되고, 흰색은 1.96 SD 차단되고. 회색 음영 단백질은이 분석에서 확인되지 않았다.
토론
여기에 설명 된 SILAC 풀다운 전략은 새로운 단백질을 검출하는 매우 민감하고 강력한 수단을 나타냅니다 단백질 상호 작용을하고, 또한 그 밀접하게 관련 샘플 사이의 변경된 바인딩 패턴의 신속하고 간단하게 차별 할 수 있습니다. eIF4AI eIF4AII 단백질 및 (6)의 단백질 상호 작용이 예에서이 기술은 단백질을 조사 하였다. 저자의 지식에,이 eIF4A의 두 가지 아형의 세포 interactome을 조사하기 위해 SILAC의 프로테오믹스의 유틸리티를 악용 문학의 첫 번째 연구이다.
전술 한 바와 같이 방법은 GFP-태그와 항-GFP 비즈 09 10 따라서 변형이 태그가 N-또는 배치되어 있는지 여부를, 예를 들어 관심있는 특정 단백질에 사용되는이 방법을 사용하기 위해 필요할 수있다을 사용 단백질의 C-말단. 가능하다면, 서부 말 또는 기능 분석해야공지 된 단백질의 상호 작용 파트너의 결합을 검출하기 위해 수행 될 수있다. 단백질 GFP 태그, 다른 태그 또는 풀다운 전략과 융합을 용납하지 않겠 다른 태그를 모두 사용하여 SILAC의 풀다운에 적용된 (FLAG 11, 비오틴 12 STREP (자신의 데이터, 게시되지 않은)), 또는 단백질에 대한 일차 항체를 사용하여 관심 대상의 단백질의 siRNA 녹다운는 대조 샘플 (13)을 제공하는 경우. 이러한 실험은 문헌에 밖에 기재되어있다, 그러나 간략히, 단계 1 및 단계는 5.4-8에 설명 동등한 단백질 입력을 사용하여 선택 식 / 풀다운 체제에 따라 변경됨 단계 2-5.3로 상기와 같이 적용 할 것인지 2.4-2.5 단계를 반복합니다. 정량화 단계는 비 특이 적 결합 단백질 분석 수준에서 제거 할 수 있도록, 그것은 낮은 친화력 단백질을 유지하기 위해 제어 구슬, 또는 염분 세척 사전 배양을 생략하는 것이 좋습니다 : 관심의 단백질과 단백질 상호 작용 . 클레아 제 엄마y를 포함하거나 특정 실험의 특성에 따라이 프로토콜에서 생략 될 수있다. 예를 들어이 방법에 사용되는 단백질이 RNA 헬리 한, RNA 분해 효소 칵테일은 RNA (단계 3.2)를 통해 매개되는 간접적 인 상호 작용을 제거하는 프로토콜에 포함되었다. 어떤 경우에는 그러나, 핵산 따라 상호 작용을 확인하는 핵산 분해 효소와 사용하지 병렬 실험을 실행이 혜택을 누릴 수 있습니다.
이 프로토콜 내에서 '매체'와 복제 실험에서 '무거운'샘플의 전환은 SILAC 미디어 또는 세포의 성장의 차이에 의해 도입 된 변화를 제어하는 것이 좋습니다. 다른 컨트롤은 복제 실험에서 세 가지 ( '빛', '중간', 그리고 '무거운') 미디어의 순차적 전환을 포함한다. 이 접근법은 잠재적으로 더 엄격한 반면, 적어도 하나의 복제에 관심 단백질 w 같이 분석의 복잡성을 증가 않는다병이 '라이트'라는 세포에서 생성 될 수 그리고 그것은 지속적으로 '빛'샘플 (예 : 각질 등의 환경 오염 물질), 그리고 단백질에 결합하는 경우에만 '빛'샘플에서 농축 된 것을 식별 단백질을 구별 할 필요가있다 관심.
정량 데이터의 사용을 허용하는 동안 임계 값의 사용을 통해 특정 -에서 비특이적 상호 작용의 차별은 불가피 진짜 상호 작용은 삭제 될 수 있습니다. 위의 방법은 단백질을 식별하는 간단하고 빠른 방법입니다 : 쉽게 더 이전의 질량 분석에 대한 경험, 또는 큰 단백질의 분석과 연구자에 의해 시도 될 수있는 단백질 상호 작용 : 단백질 상호 작용 데이터 세트. 대부분의 용도를 위해이 관심의 새로운 단백질을 식별하기위한 충분한 이상입니다. 이러한 데이터 손실을 줄일 수있는이 방법의 변형은 또 다른 곳 문헌에 기술 및 PR의 사용을 포함한다실험적인 매개 변수 (세포주, 비드 매트릭스, 버퍼 조건)의 특정한 세트에 대해 오염 단백질을 공지 otein 주파수 라이브러리 (10), (14)를 제외 할 수있다. 그러나, 특정 실험 매개 변수에 따라서는 비드 프로테옴을 생성하도록 제어 실험의 수를 실행할 필요가있을 수 있고 따라서 이것은 실험의 비용과 복잡성을 증가시킬 수있다. 이 기술에 대한 자세한 정보는 www.peptracker.co.uk 웹 사이트 14에서 사용할 수 있습니다.
또한 프로토콜 (- MAP의 SILAC 실험을 정제 한 후 믹싱 지칭) 면역 과정의 끝에서 서로 다르게 표지 된 샘플을 혼합 수반 상술 주목해야한다. 이것은 단백질로 이루어집니다 : 단백질 상호 작용은 주어진 평형 (15)에서 발생합니다. 그것은 다른 그룹은 샘플의 배양에이 프로토콜에 설명이 MAP의 SILAC 접근 방식을 결합했다 주목해야한다이전 풀다운 (혼합 한 후 정제 - PAM SILAC)에 시간의 길이가 다른 15, 16 (2 시간 20 분은 문헌에 사용되었다). 단백질 비율을 1:1으로 인하 얼마나 빨리 기반으로, 품질 적 결합 친화도를 조사 및 안정 또는 동적 상호 작용 단백질 15로 단백질을 정의하는 것이 가능하다.
요약하면, SILAC의 풀다운은 생리학 관련 설정에서, 관심의 특정 단백질과 상호 작용하는 단백질을 식별하는 매우 강력한 방법을 나타냅니다. 기술은 매우 쉽게 또한 임의의 주어진 단백질에의 응용을 허용하는 다른 정제 전략의 수에 적응 될 수있다. 결과의 정량화가 크게 진정한 상호 작용의 식별을 단순화하고, 비 특정 바인더를 제거하는 데 엄격한 버퍼 조건의 완화를 허용, 따라서 선호도가 낮은 상호 작용을 유지합니다. 최대 3 개의 시료는 상기에서 비교 될 수있는 바와 같이전략 기술은 다른 단백질 이소 형, 돌연변이 단백질, 또는 약물 억제제의 효과 사이에 결합 단백질의 차이를 비교하는 명확한 장점을 가지고 있습니다. 전체 젤 조각이 아니라 그 쿠마에 의해 얼룩 개별 밴드보다 분석으로, 높은 신뢰성에 확인 된 단백질의 수는 일반적으로 표준 GST / TAP-풀다운에서 확인 된 것보다 더 높은, 그리고 관심의 단백질을 제거한다 선택에 편견을 실험. 이 기술은 따라서 새로운 단백질 상호 작용 (효모 2 - 하이브리드, GST / 그의 또는 TAP 풀다운)의 식별에 사용되는 다른 일반적으로 사용되는 기술로 매우 호의적으로 비교합니다.
공개
저자는 더 경쟁 재정적 이익이 없다는 것을 선언합니다.
감사의 말
이 작품은 IG의 웰컴 트러스트 (Wellcome Trust)와 BBSRC에서 교부금에 의해 지원되었다. IG는 웰컴 선임 연구원입니다.
자료
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium (DMEM) | Dundee Cell Products | LM010 | DMEM lacking Arginine and Lysine. (and containing L-glutamine) |
| Dialysed FBS (10 kDa cutoff) 500 ml | Dundee Cell Products | DS1003 | |
| Arginine (R0) 25 g | Sigma-Aldrich | A8094 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R6) 0.5 g | Cambridge Isotope Labs | CLM-2265 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R10) 0.5 g | Cambridge Isotope Labs | CNLM-539 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Lysine (K0) 25 g | Sigma-Aldrich | L8662 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K4) 0.5 g | Cambridge Isotope Labs | DLM-2640 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K8) 0.5 g | Cambridge Isotope Labs | CNLM-291 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Penicillin/streptomycin, Liquid. 100 ml | Gibco | 15140-122 | |
| Lipofectamine 2000 transfection reagent | Invitrogen | 11668-027 | |
| GFP-trap Agarose (500 μl resin) | Chromotek | gta-20 | |
| 5x SDS Sample loading buffer | Fisher Scientific | PN39000 | The use of purchased rather than homemade sample buffer is recommended to minimize keratin contamination. |
| Protease inhibitor cocktail set III | Calbiochem | 539134 | |
| 1.7 ml prelubricated tubes | Costar | 3207 | |
| BCA protein assay kit (1 L) | Pierce | 23225 | |
| Tris (Trizma) | Sigma-Aldrich | T1503 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E6758 | |
| Sodium chloride | Sigma-Aldrich | S3014 | |
| Rnase cocktail | Ambion | AM2286 | |
| NP-40 alternative | Millipore | 492016 |
참고문헌
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem Soc Trans. 38, 875-878 (2010).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), (2012).
- Trinkle-Mulcahy, L. Resolving protein interactions and complexes by affinity purification followed by label-based quantitative mass spectrometry. Proteomics. 12, 1623-1638 (2012).
- Emmott, E., et al. The cellular interactome of the coronavirus infectious bronchitis virus nucleocapsid protein and functional implications for virus biology. J Virol. 87, 9486-9500 (2013).
- Li, W., Belsham, G. J., Proud, C. G. Eukaryotic initiation factors 4A (eIF4A) and 4G (eIF4G) mutually interact in a 1:1 ratio in vivo. J Biol Chem. 276, 29111-29115 (2001).
- Nielsen, P. J., Trachsel, H. The mouse protein synthesis initiation factor 4A gene family includes two related functional genes which are differentially expressed. EMBO J. 7, 2097-2105 (1988).
- Galicia-Vazquez, G., Cencic, R., Robert, F., Agenor, A. Q., Pelletier, J. A cellular response linking eIF4AI activity to eIF4AII transcription. RNA. 18, 1373-1384 (2012).
- Zakowicz, H., et al. Mutational analysis of the DEAD-box RNA helicase eIF4AII characterizes its interaction with transformation suppressor Pdcd4 and eIF4GI. RNA. 11, 261-274 (2005).
- Rothbauer, U., et al. A versatile nanotrap for biochemical and functional studies with fluorescent fusion proteins. Mol Cell Proteomics. 7, 282-289 (2008).
- Trinkle-Mulcahy, L., et al. Identifying specific protein interaction partners using quantitative mass spectrometry and bead proteomes. J Cell Biol. 183, 223-239 (2008).
- Dobreva, I., Fielding, A., Foster, L. J., Dedhar, S. Mapping the integrin-linked kinase interactome using SILAC. J Proteome Res. 7, 1740-1749 (2008).
- Mittler, G., Butter, F., Mann, M. A SILAC-based DNA protein interaction screen that identifies candidate binding proteins to functional DNA elements. Genome research. 19, 284-293 (2009).
- Selbach, M., Mann, M. Protein interaction screening by quantitative immunoprecipitation combined with knockdown QUICK. Nat Methods. 3, 981-983 (2006).
- Boulon, S., et al. Establishment of a protein frequency library and its application in the reliable identification of specific protein interaction partners. Mol Cell Proteomics. 9, 861-879 (2010).
- Wang, X., Huang, L. Identifying dynamic interactors of protein complexes by quantitative mass spectrometry. Mol Cell Proteomics. 7, 46-57 (2008).
- Fang, L., et al. Characterization of the human COP9 signalosome complex using affinity purification and mass spectrometry. J Proteome Res. 7, 4914-4925 (2008).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유