Method Article
Identificazione delle proteine partner di interazione in cellule di mammifero Uso SILAC-immunoprecipitazione quantitativi Proteomica
In questo articolo
Riepilogo
Esperimenti di immunoprecipitazione SILAC rappresentano un potente mezzo per scoprire nuova proteina: interazioni proteina. Consentendo l'accurata quantificazione relativa abbondanza di proteine sia nel controllo e campioni di prova, veri interazione può essere facilmente distinto da contaminanti sperimentali e interazioni bassa affinità conservata attraverso l'uso di condizioni di buffer meno stringenti.
Abstract
Proteomica quantitativa in combinazione con la purificazione immuno-affinità, SILAC immunoprecipitazione, rappresentano un potente mezzo per la scoperta di nuova proteina: interazioni proteina. Consentendo la precisa quantificazione relativa di proteine abbondanza sia nel controllo e campioni, veri interazioni possono essere facilmente distinti dai contaminanti sperimentali. Interazioni bassa affinità possono essere conservati mediante l'uso di condizioni di buffer meno stringenti e rimangono facilmente identificabili. Questo protocollo discute l'etichettatura di cellule di coltura tissutale con isotopi stabili amminoacidi marcati, trasfezione e immunoprecipitazione di un'affinità targhetta proteina di interesse, seguita dalla preparazione per la presentazione di un impianto di spettrometria di massa. Questo protocollo illustra poi come analizzare e interpretare i dati restituiti dal spettrometro di massa al fine di individuare partner cellulari che interagiscono con una proteina di interesse. Come esempio di questa tecnica è applicata a idenduare proteine di legame dei fattori di inizio della traduzione eucariotiche: eIF4AI e eIF4AII.
Introduzione
Un passo essenziale per comprendere la funzione delle proteine è l'identificazione delle relative proteine che interagiscono. Qualora tali proteine sono noti ci sono una serie di tecniche disponibili, ciascuno con i propri pregi e difetti. Questi includono il sistema del doppio ibrido di lievito, saggi tendina con proteina ricombinante, così come la depurazione tandem affinità o TAP-codifica 1, 2.
Una più recente aggiunta a queste tecniche è la combinazione di purificazione per affinità di una proteina di interesse da una linea cellulare di mammifero rilevante, seguita da spettrometria di massa quantitativa utilizzando marcatura isotopica stabile di amminoacidi in coltura cellulare (SILAC) 3. Questo ha dei vantaggi rispetto al lievito approccio del doppio ibrido in quella localizzazione cellulare e modificazioni post-traduzionali non siano disturbati, così come vantaggi rispetto tradizionale TAP di marcatura che è un approccio quantitativo piuttosto che qualitativo permettendo all'utente di readily distinguere non specificamente proteine interagenti e contaminanti, da fattori dell'ospite che si legano specificamente. Inoltre, come un campione è tipicamente analizzato tutto, piuttosto che bande proteiche come singoli, proteine di interesse non sono mascherati da simile migrazione di proteine su un gel, né hanno in genere bisogno di essere presente a livelli sufficienti per essere visibile dopo colorazione, portando a aumento del numero di proteine identificate con sicurezza 4.
Per dimostrare questa tecnica, fusioni GFP del fattore eucariotico traduzione iniziazione strettamente correlati eIF4AI e eIF4AII che la quota di oltre il 90% di identità aminoacidica sono stati studiati da SILAC-immunoprecipitazione proteomica quantitativa. EIF4AI umana e II sono stati clonati in pEGFP-C1 per formare una proteina di fusione GFP in cui è fusa all'N-terminale di eIF4A. Per evitare la necessità di generare linee cellulari stabili trasfezione transiente è stato usato per fornire questi costrutti di isotopi cellule 293T etichettati stabili.
cellule sono state prima etichettato per due settimane in SILAC RPMI, seguita da trasfezione di DNA plasmidico codificante una proteina di interesse. Le cellule sono state lisate, la concentrazione proteica normalizzati, e la stessa quantità di lisato affinità purificato su agarosio anti-GFP. Uguali quantità di eluato sono stati poi combinati e sottoposti ad analisi LC-MS/MS. I risultati di questa analisi vengono poi elaborati per identificare alta proteina fiducia: proteina (Figura 1).
SILAC immunoprecipitazione consente di individuare non solo le interazioni dirette, ma anche una bassa affinità o interazioni indirette con complessi proteici 4. Utilizzando questo sistema, eIF4AI e II immunoprecipitazioni permesso l'identificazione riproducibile e fiducioso del partner di legame primario eIF4G (isoforme I / II e III) 5, nonché le interazioni indirette con eIF4E, e numerosi componenti del complesso eIF3.
Protocollo
1. Generazione ed Passaging di linee cellulari SILAC marcato
Nota: l'uso di tripsina-EDTA deve essere evitata in tutte le fasi di passaging e preparazione dei campioni sperimentali per l'analisi, come la tripsina può contenere amminoacidi senza etichetta, che porterebbero a etichettatura incompleta dei campioni.
- Per preparare una bottiglia di supporto SILAC aggiungere 0,5 ml aliquota di una arginina opportunamente SILAC-passo (84 mg / ml in PBS) e lisina (146 mg / ml in PBS) per una bottiglia da 500 ml di Arg / Lys DMEM privo (contenente L-glutammina).
- Successivamente, aggiungere 50 ml di dializzato FBS e 5 ml di penicillina / streptomicina per la bottiglia da 500 ml.
Nota: le cellule HEK 293T (ATCC) dovrebbero essere mantenute in DMEM privo di mezzi di arginina e lisina e integrato con la luce (R0K0), medio (R6K4) o pesante (R10K8) aminoacidi, siero fetale bovino dializzato (10 kDa cutoff), e penicillina / di streptomicina. Le cellule devono essere mantenuti in mezzi per un minimo di 5 divisioni cellulari per garantire la completaetichettatura. Nella maggior parte dei casi le cellule sono facilmente etichettati in ≥ 2 settimane. - Per dividere le celle per passaging, le cellule devono essere sloggiati dal monostrato colpendo il pallone. Alternative includono l'uso di raschietti o cellulari utilizzando senza enzima, basato PBS memoria di cella-dissociazione.
Nota: Per risparmiare i media, le cellule dovrebbero essere diversi passaggi in fiasche T25 e il numero di cellule di grandi dimensioni generati solo immediatamente prima di un esperimento. - 24 ore prima cellule trasfettando, semi di 3,5 x 10 6 cellule SILAC marcato in un unico piatto di 10 centimetri 2 per ogni condizione sperimentale sotto inchiesta.
2. Trasfezione di cellule marcate con costrutti pEGFP-fusione
- Rimuovere il supporto dalle cellule e sostituirlo con 9 ml di libera-antibiotico SILAC DMEM (leggero, medio o pesante) media.
Nota: Gli antibiotici non dovrebbero essere aggiunti al terreno in quanto possono interferire con l'efficienza di transfezione reagenti a base di liposomi. - Prepare un mix di 10 microgrammi di plasmide appropriato (epEGFP-C1, pEGFP-eIF4A-I, pEGFP-eIF4A-II) in 500 ml di antibiotico-libera SILAC DMEM e mescolare con 500 ml di antibiotico-libero SILAC DMEM contenente 10 ml di reagente di trasfezione (es. lipofectamina 2000). Mescolare la reazione accuratamente pipettando su e giù parecchie volte.
- Incubare la miscela di reazione a temperatura ambiente per 20 min e poi aggiungere goccia a goccia per il monostrato cellulare. Oscillare delicatamente la piastra da un lato all'altro.
- Incubare le cellule trasfettate a 37 ° C e 10% di CO 2 per 24 ore.
Nota: Se l'espressione di una proteina di risultati interesse in qualsiasi tossicità apparente, allora può essere necessario ridurre la quantità di plasmide trasfettate e / o la durata del periodo di espressione successiva.
3. Raccolta lisati cellulari
- Celle di raccolta del piatto in PBS freddo ghiaccio con un raschietto cellulare. Raccogliere le cellule per centrifugazione a 220 xg, 476; C per 5 min. Lavare le cellule di un ulteriore 3 volte in 10 ml di PBS freddo.
- Risospendere il pellet cellulare in 200 ml di tampone di lisi cellulare (10 mm TrisCl / pH 7.5, 150 mM NaCl, EDTA 0,5 mM, 0,5% NP40) contenente recente aggiunta di inibitori delle proteasi cocktail III a concentrazione 1x e RNasi cocktail (opzionale) a 5 ml per ml.
Nota: Per le proteine con nota acido nucleico attività di legame, può essere necessario aggiungere nucleasi al lisato prima della precipitazione. Nei casi in cui viene aggiunto nucleasi, i campioni devono essere incubati in ghiaccio per 30 minuti sul ghiaccio, con pipettaggio ogni 10 min. Estrazione acido nucleico totale da una piccola frazione del campione e l'analisi mediante elettroforesi su gel di agarosio può testare l'efficacia della nucleasi. - Centrifugare i campioni a 13.000 xg, a 4 ° C per 10 min mantengono il surnatante come il lisato cellulare solubile.
- La concentrazione del lisato cellulare dovrebbe essere valutata mediante saggio BCA secondo le istruzioni del produttore.
- Utilizzare tampone di lisi contenente inibitori delle proteasi cocktail III per normalizzare la concentrazione di proteine in un volume finale di 500 microlitri.
- Regolare il volume di 1 ml con l'aggiunta di 500 ml di tampone di diluizione (10 mM TrisCl / pH 7,5, NaCl 150 mM, EDTA 0,5 mM) contenente inibitori delle proteasi cocktail III a concentrazione finale 1x. Un'aliquota di 50 microlitri del campione deve essere mantenuto come ingresso campione e il lisato tenuta in ghiaccio, mentre la preparazione perle anti-GFP (ad esempio, GFP-trap).
Nota: in genere le rese variano tra 1-3,5 mg di proteine nella finale di 1 ml di campione. Mentre i buffer sopra sono adatti per molte proteine di interesse, per altri può essere necessario modificare i componenti tampone per assicurare la proteina esca viene solubilizzato e mantenere le interazioni proteina-proteina. Possibili alterazioni includono il agente tamponante (Phosphate, HEPES), concentrazione salina (150-500 mM), la scelta del detersivo, o altri additivi.
4. Associazione a Anti-GBeads FP
- Brevemente vortice l'impasto tallone risospendere le sfere. Usando una pipetta 200 microlitri con l'estremità tagliata, trasferire 25 ml di perline per campione in una nuova provetta.
Nota: L'utente deve preparare le perline per un esperimento SILAC singolo come mastermix minimizzare variazione da campione a campione. - Per ogni 25 ml di tallone slurry, aggiungere 20 volumi (1,500 microlitri per 75 microlitri slurry) di tampone di diluizione, e centrifugare le perline a 2.700 xg per 5 min. Successivamente, lavare le perline un'ulteriore 2x in 20 volumi di tampone di diluizione.
- Aggiungere 100 microlitri di buffer di diluizione per 25 microlitri tallone liquami. Utilizzando una punta di 200 microlitri con l'estremità tagliata, trasferire 85 microlitri di questo slurry risospese a ciascuno dei campioni SILAC marcato dal punto 3.6.
- Incubare i campioni con perline su un rotatore a 4 ° C per 2 h.
5. Lavaggio, eluizione, e la preparazione dei campioni per MS Analysis
- Centrifugare i campioni a 2.700 xg, 46; C per 5 min. 50 ml di surnatante siano conservati come il campione non legato, con il resto del supernatante scartato.
- 1 ml di tampone di diluizione deve essere aggiunto a ciascuna provetta per risospendere le sfere e il campione centrifugato a 2700 xg, a 4 ° C per 5 min. Il surnatante deve essere eliminata. Questo dovrebbe essere eseguita due volte.
- Eluire proteine delle perle con l'aggiunta di 50 microlitri di tampone di caricamento SDS 2x, e riscaldamento a 95 ° C per 10 min. Agglomerare le perline per centrifugazione a 2.700 xg per 2 min a 4 ° C
- Conservare il supernatante in tubi prelubrificati, dove può essere poi conservato a -80 ° C fino all'utilizzo. Per l'invio di un impianto di spettrometria di massa, mescolare campioni etichettati 01:01:01 (ad esempio, 10 ml di ciascuno) e inviare il campione misto.
Nota: A questo punto i campioni possono essere analizzati mediante western blot, argento colorazione o altro mezzo per testare l'interazione con i partner di legame conosciuti / sconosciuti. Un esempio è riportato in Figure 2 che mostra il legame specifico di un partner noto interazione - eIF4G mediante western blot, e la comparsa di bande di colorazione argento presenti nei campioni pulldown da una proteina di interesse, ma non un campione di controllo. - Invia campione per l'analisi LC-MS/MS.
Nota: una volta accertato che una proteina tag di interesse è successo vincolante partner di interazione, volumi uguali di ogni campione marcato (leggeri, medi e pesanti) vengono combinati e sottoposti ad analisi LC-MS/MS. Si è soliti presentare 30 microlitri totale di un campione per l'analisi IP. Ciò comporterebbe mescolando 10 ml di campione Light-marcato con 10 ml di campioni pesanti marcato 10 microlitri medie etichettati e per dare un totale di 30 microlitri.
6 Analisi dei dati I:. Comprendere i risultati, e la rimozione di identificazioni Low-fiducia
Nota: Un elenco delle intestazioni di colonna restituiti dal software Proteome Discoverer è riportata nella tabella 1 Dif.software ferenti (ad esempio, MSQuant, MaxQuant) restituirà voci diverse, però, solo un sottoinsieme di questi sono necessari per l'analisi e questi sono comuni ai vari pacchetti software. I dati devono sempre includere un numero di adesione per ogni proteina identificata, il rapporto di confrontando ogni rapporto campione (luce vs medio, chiaro vs pesante, medio vs pesante ecc), il numero di peptidi unici identificati, e una qualche forma di tasso di falsi positivi o indicazione di fiducia.
- Prima di passare attraverso i dati, copiare prima i dati grezzi in un nuovo foglio di calcolo. Da questo foglio di rimuovere tutte le colonne ad eccezione di quelle che dà il numero di accesso, il numero di peptidi unici, rapporti di confronto campioni, rapporto di variabilità, e la descrizione della proteina. Se sono stati eseguiti esperimenti di replica, questi dovrebbero essere combinati in un unico file di Excel, con ogni esperimento che appare su una scheda separata.
Nota: dati a bassa fiducia comprende proteine identificate da un solo peptide unico, e quelli wqui quantificazione non è stato possibile. - Utilizzare la funzione 'ordina' Eccelle 'per ordinare i dati per il numero di peptidi e rimuovere le voci per le proteine che mancano più di un peptide. Poi l'ordinamento rapporto e rimuovere le proteine che mancano rapporti di SILAC (proteine non quantificate).
- Convertire i rapporti SILAC a un registro 2 valori utilizzando la formula: '= log (Rapporto SILAC, 2)', dove 'Rapporto SILAC' è sostituito con l'identificativo della cella.
Nota: come rapporto SILAC comporta alcun dato per le proteine che mostrano una diminuzione in abbondanza limitarsi ai valori tra 0 e 1, è usuale per convertire un rapporto SILAC ad un rapporto SILAC log 2, come questo significa proteine sia aumentato o diminuito in un campione siano rappresentati su scala logaritmica in cui 2 o -2 rappresenta un incremento di 4 volte o diminuzione in abbondanza, e 3 o -3 un aumento di 9 volte o diminuzione in abbondanza rispettivamente. A seguito di questa trasformazione, i dati dovrebbero montare un Gadistribuzione ussian centrato su un rapporto log 2 SILAC di 0. Un esempio di questo è dato in Figura 3. - Creare nuove colonne nel file excel e calcolare log 2 rapporti SILAC per tutti i campioni / colonne finte. Per la conversione di un rapporto finto / campione ad una / rapporto finto campione, utilizzare la formula: "= 1/ratio"
7 Analisi dei dati II:. Selezione Interazioni alta fiducia per approfondire lo studio
- Aperto Graphpad Prism, selezionare Nuovo> tabella di dati e grafico ... Seleziona 'colonna' dalla lista sul lato sinistro della finestra, selezionare il tasto ENTER / Importa dati> Immettere i valori di replica, accatastati in opzioni colonne. Premere 'Crea'.
- Selezionare e copiare un determinato log 2 Sample / Mock SILAC rapporto colonna dal foglio di calcolo Excel nel nuovo file Prism.
- Fare clic sul menu a discesa 'Inserisci' e selezionare 'New analisi'. Sotto analisi colonna selezionare 'Distribuzione di frequenza '. Mantenere le opzioni predefinite fare clic su 'OK'.
Nota: Questo passaggio genera un istogramma che illustra il numero di proteine identificate in un determinato rapporto. Ciò dovrebbe costituire una distribuzione gaussiana. - Nella cartella 'Risultati' sarà stata generata una nuova sezione 'Istogramma'. Selezionare la sezione distribuzione di frequenza.
- Fare clic sul menu a discesa 'Inserisci' e selezionare Nuovo analisi> XY> Regressione non lineare (curva fit). Fare clic su 'distribuzione gaussiana' e fare clic su OK.
Nota: Questo passaggio si inserisce una curva ai dati di distribuzione di frequenza generati nella fase 7.3, ottenendo valori che possono essere successivamente utilizzati per calcolare le soglie di rilevanza. - Nella finestra dei risultati che appare, la media e la deviazione standard sono dati. Generare una soglia aggiungendo 1.96 deviazioni standard alla media.
Nota: Questi valori rappresentano la media e la deviazione standard della gaussiana distribution, non il dataset totale. 1.96 deviazioni standard avrebbero posto un limite al limite del 95% di confidenza (p ≤ 0,05). 2.58 SD darebbe il 99%, e del 3,3 SD 99,9%. La soglia deve essere determinata individualmente per ogni esperimento replica in quanto può variare.
8 Analisi dei dati III:. Unire dataset Replica
Nota: Per essere sicuri di identificare partner di interazione di un esperimento a tendina tipico SILAC dovrebbe idealmente essere eseguita tre volte, con campioni etichettati medi e pesanti acceso per una delle ripetizioni per il controllo per effetto dei media sui risultati. Una proteina che mostra come interagire in almeno due dei tre esperimenti, con il campione 'switched'-media idealmente rappresenta uno di questi, è un'interazione alta fiducia.
- Tornare al file Excel, creare una nuova scheda denominata 'combinato'. Colonna Label A 'adesione' e copiare tutti i numeri di adesione da ogni of singoli esperimenti in questa singola colonna.
- Selezionare la scheda 'Dati', e poi l'opzione 'Rimuovi duplicati.
- Creare colonne per i rapporti SILAC, e il rapporto di variabilità da ogni esperimento, nonché per la proteina di nome / descrizione di colonna.
- Nella scheda descrizione, utilizzare la formula vlookup per raccogliere la descrizione di ogni numero di accesso. Trascinare la formula della colonna per riempire la descrizione di proteine. La formula CERCA.VERT è: "= CERCA.VERT (AccessionNo, WheretoLook, ColumnsAccross, False)", dove: AccessionNo è $ aX con X essendo il numero di riga WheretoLook è la 'scheda Experiment Name' $ a $ 2:.! $ Y $ Z dove Y e Z sono il pezzo in basso a destra dei dati sul foglio di calcolo si fa riferimento. ColumnsAccross è il numero di colonne di fronte al numero di adesioneber nella colonna A un valore desiderato compreso.
Nota: Ad esempio, se il numero adesione è nella colonna A, ed i dati di interesse risiede nella colonna E. Il valore qui sarebbe 5 (colonna A vale 1). Come numeri di accesso sono stati raggruppati nel passaggio b, è più probabile che vlookup non otterrà tutti i nomi richiesti da un esperimento. Dove restituisce N / A, modificare la formula per ottenere la descrizione di ogni esperimento a turno finché tutte le descrizioni sono acquisiti. - Per evidenziare proteine interagenti nel dataset combinato, selezionare ciascuna colonna rapporto singolarmente e fare clic sulla scheda Home, seguita da Formattazione condizionale> evidenziare le celle regole> Altre regole.
- Seleziona stile 'classico'. Formatta solo le celle che contengono 'value cellulare', 'maggiore o uguale a'. Nella casella, digitare il valore di deviazione standard 1.96, e fare clic su 'OK'.
- Valutare la variabilità rapporto per ogni interazione positiva. Se sottraendo var%ESPONSABILITÀ potrebbe cadere un 'colpo' al di sotto del valore di soglia, questa deve essere trattata con cautela.
Nota: Questo rappresenta il modo coerente i rapporti individuali di ciascun peptide che insieme costituiscono rapporto SILAC 'una delle proteine sono, ed è dato come percentuale. Quando la variabilità rapporto viene preso in considerazione e una proteina dà un rapporto SILAC sopra la soglia, questa proteina rappresenta un successo. Se la variabilità rapporto fornisce una gamma che scende sotto la soglia, il 'colpo' può rappresentare un contaminante. - Ripetere questa operazione per ciascuno dei risultati colonne e confronta attraverso esperimenti. Proteine evidenziati identificate in due o più esperimenti rappresentano un'interazione alta fiducia.
Nota: a seconda del database utilizzato per assegnare i numeri di adesione di proteine, in esperimenti diversi di una proteina può essere stato identificato sotto un diverso, o anche più numeri di adesione. E 'importante controllare questo al fine di garantire che le proteine non vengano accidentalmente omessi.
Risultati
In un tipico esperimento SILAC pulldown, la stragrande maggioranza delle proteine identificate (> 90%) rappresentano contaminanti così come le proteine di legame non specifico alla matrice di affinità e ciò è illustrato in Figura 2B, anche quando i protocolli di lavaggio rimuovere una maggioranza di contaminanti citoplasmatici come GAPDH (Figura 2A). Tuttavia, il raggruppamento di proteine non si lega specificamente in una distribuzione normale permette proteine che si legano ad una proteina di interesse deve essere distinto come questi hanno campione / rapporti finti superiori rispetto allo sfondo. Mentre contaminanti dovrebbero teoricamente raggrupparsi intorno a un rapporto di registro 2 SILAC di 0, questo non è necessariamente il caso, un esempio di questo è dato in Figura 3. Possibili ragioni di questo includono l'etichettatura SILAC imperfetta delle cellule, caricare volumi disuguali o concentrazioni di lisato su le perle anti-GFP, la perdita accidentale di perline durante la purificazione o disparità mixzione dei campioni al termine della procedura di purificazione 3. Tuttavia, supponendo che i dati vengono analizzati in una soglia deviazione standard dalla media dei contaminanti normalmente distribuiti, cambiamenti minori nella centratura dei dati non devono pregiudicare la qualità dei risultati.
Quando si confrontano le differenze nelle interazioni proteina tra due proteine correlate, una situazione simile si può verificare se una proteina di interesse è prodotto a livelli superiori all'interno delle cellule di un secondo (a causa di variazioni di efficienza di trasfezione, o una proprietà intrinseca della proteina o mRNA) . Alcune variazioni nell'espressione (ad esempio, la Figura 2A) possono essere corretti per analizzando il rapporto SILAC per questi due campioni. In questo esempio, questi sarebbero i campioni GFP-eIF4AI e GFP-eIF4AII. Analizzando il rapporto 4AI/4AII SILAC come discusso nella sezione 7, è possibile identificare proteine la cui legame varia significativamente tra isoforme.
In Figura 4, una rappresentazione delle proteine-eIF4A legame identificate in uno degli esperimenti condotti replica è mostrato che illustra la copertura del complesso fattore di iniziazione questo protocollo raggiunto. I quozienti più elevati erano tipicamente osservati con eIF4G, che si lega direttamente al eIF4A, con rapporti inferiori per eIF4E, che si lega a eIF4G in un sito lontano dal sito eIF4A vincolante. Rapporti più bassi sono stati osservati per i membri del complesso eIF3. Tuttavia, questo dimostra chiaramente che l'esperimento soddisfacente identificato partner di legame sia dirette e indirette di eIF4AI e II. Come ci si potrebbe aspettare da loro identità elevata di sequenza 6, proteine: interazioni proteina è apparso in gran parte conservata tra le due isoforme di questo sistema sperimentale 7, 8. Una selezione di alcune delle proteine interagenti sono riportati nella tabella 2, che illustra il formato dei dati.| Intestazione colonna | Descrizione |
| Accessione | Visualizza il numero di accesso per la sequenza |
| Copertura | Percentuale della sequenza proteica coperta dai peptidi identificati |
| ♯ PSM | Peptide partita spettrale |
| ♯ peptidi | Numero totale di peptidi unici identificati per una proteina |
| ♯ AA | La lunghezza di una proteina di aminoacidi |
| MW (Da) | Il peso molecolare di una proteina in Dalton. Esclude modifiche |
| calc. PI | Il punto isoelettrico teorica di una proteina |
| Punteggio | Il punteggio totale di una proteina (che rappresenta la somma del indivcolonne sonore peptide idual). Il punteggio esatto richiesto per rilevanza varierà tra esperimenti. Una struttura di MS di solito applicare un tasso di falsi scoperta cutoff 5%. |
| Sequenza | La sequenza di aminoacidi che costituiscono la proteina |
| Rapporto | L'intensità relativa dei peptidi in un campione marcato denominata, rispetto ad un secondo campione marcato |
| Conte Rapporto | Il numero di rapporti peptidici che sono stati utilizzati per calcolare l'un dato rapporto proteina |
| Rapporto di variabilità (%) | La variabilità dei rapporti individuali peptidi utilizzati per calcolare un dato rapporto proteina |
| Descrizione | Il nome della proteina |
Tabella 1. Intestazioni di colonna standard da una relazione Proteome Discoverer.Sebbene le informazioni utili possono essere acquisite da tutte queste colonne, quelli critici per questa analisi sono mostrati in grassetto.
| Accessione | Peptidi | 4AI/Mock | 4AII/Mock | Nome | Analisi SILAC |
| A8K7F6 | 21 | 100 | 1 | eIF4AI | Proteina 'Bait' |
| Q14240 | 22 | 0.01 | 90,855 | eIF4AII | |
| G5E9S1 | 25 | 47,575 | 30.53 | eIF4GI | Interazione proteine |
| Q59GJ0 | 5 | 11,778 | 10,619 | eIF4GII60; | |
| P06730 | 3 | 7.22 | 7.57 | eIF4E | |
| Q5T6W5 | 4 | 0.685 | 0,646 | hnRNPK | Contaminanti non si lega specificamente |
| P62805 | 1 | 0,531 | 0,498 | Istone H4 | |
| H6VRG2 | 18 | - | - | Cheratina-1 | Contaminanti ambientali |
| P35527 | 11 | 0.01 | 0.01 | Cheratina-1 citoscheletro 9 |
Tabella 2. Dati tipici di un esperimento SILAC immunoprecipitazione. Dare esempio i dati per una proteina di interesse / esca (peptidi alti, elevato rapporto), proteine che interagiscono con una proteina di interesse (peptidi alto / basso, alto rapporto), non si lega specificamente proteine (/ basso PEPT altaidi, il rapporto scende sotto soglia - in questo esperimento 0.96), e contaminanti ambientali (spesso peptidi alti, rapporto negativo / sotto la soglia).

Figura 1. Piano sperimentale. Innanzitutto le cellule sono coltivate in mezzi privi arginina e lisina e sostituiti con isotopo stabile etichettati arginina e lisina per 2 settimane (1). (2) Le cellule vengono seminate in 10 centimetri 2 piatti e transitoriamente trasfettate con plasmidi codificanti per GFP (Mock) o proteine di fusione GFP (Campioni). (3) Le cellule vengono lisate e le proteine GFP o GFP fusione sono immunoprecipitati da lisati cellulari. (4) I campioni sono combinati in un rapporto di 1:1 e sottoposti ad analisi LC-MS/MS. I dati vengono poi analizzati per rimuovere basso contenuto proteico fiducia identcifiche e selezionare un livello di proteina arricchimento corrispondente alle proteine interagenti originali.
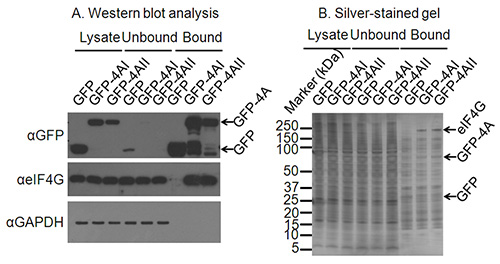
Figura 2. Conferma condizioni di immunoprecipitazione adeguate. A) Analisi Western blot di lisati cellulari, così come le frazioni liberi e legati dalla immunoprecipitazione conferma espressione e immunoprecipitazione della proteina di interesse. Un western blot contro GAPDH conferma esaurimento delle proteine non interagenti e un ulteriore western blot un noto socio interagente di eIF4A conferma l'immunoprecipitazione successo delle proteine leganti la proteina di interesse. B) Per il partner interagenti di una proteina di interesse non sono noti, un gel-argento colorato può confermare immunoprecipitazione di proteine che interagiscono. Su questo argento-macchia bande di gel ed per GFP e GFP-eIF4AI/II sono chiare, e una banda migrazione al formato corretto per eIF4G è presente solo nelle corsie GFP-4AI/II-bound e non sulla corsia di controllo GFP.

Figura 3. Rappresentante dei risultati. Istogramma mostra la distribuzione dei rapporti di proteine da una ripetizione di (A) GFP-eIF4AI o (B) GFP-eIF4AII discesa. L'1.96 standard di cutoff deviazione è contrassegnato da una linea tratteggiata. Proteine interagenti rientrano i contaminanti normalmente distribuiti sono evidenti da ~ 0,25 e 1 in (A), e ~ 0.3-1.5 in (B).
les/ftp_upload/51656/51656fig4.jpg "/>
Figura 4. Identificazione delle eIF membri complessi da un singolo replica di un esperimento SILAC IP. Proteine in verde sono stati la proteina di interesse utilizzato per la discesa Interazione proteine sono ombreggiate dal rosso al bianco secondo il login 2 rapporto SILAC nel PI SILAC con il rosso essendo la proteina più abbondante per l'analisi e il bianco è il cutoff 1,96 SD. Le proteine in grigio non sono stati identificati in questa analisi.
Discussione
La strategia tendina SILAC qui descritto rappresenta un mezzo molto sensibili e potenti di rilevazione nuova proteina: interazioni proteiche, e inoltre permette la discriminazione rapida e semplice dei modelli vincolanti alterati tra i campioni strettamente connessi di interesse. In questo esempio, questa tecnica è stata utilizzata per studiare la proteina: proteina dei eIF4AI e eIF4AII proteine 6. A conoscenza dell'autore, questo è il primo studio in letteratura sfruttando l'utilità di proteomica SILAC indagare dell'interattoma cellulare di questi due isoforme di eIF4A.
L'approccio sopra descritto utilizza una GFP-tag e anti-GFP perline 9, 10 e quindi modifiche possono essere necessarie per ottenere questo approccio da utilizzare per una specifica proteina di interesse, per esempio se la variabile è posto N-o C-terminale di una proteina. Ove possibile, le macchie occidentali o test funzionali dovrebberoessere eseguita per rilevare legame di un noto proteina partner di interazione. Se una proteina non tollerare fusione con un tag GFP, altra marcatura o strategie a tendina sono stati applicati a pulldowns SILAC utilizzando sia gli altri tag (FLAG 11, biotina 12, STREP (propri dati, inedito)), oppure utilizzando anticorpi primari contro una proteina di interesse dove siRNA knockdown della proteina bersaglio fornisce un campione di controllo 13. Tali esperimenti sono stati descritti altrove in letteratura, ma in breve, punto 1 e passi 5,4-8 verrebbero applicate come sopra, con passaggi 2-5,3 modificati come appropriato per il sistema di espressione / tendina di scelta utilizzano gli ingressi uguali di proteine, come descritto nella passi 2,4-2,5. Poiché le fasi di quantificazione consentono proteine di legame non specifici per essere rimossi a livello di analisi, si raccomanda di omettere pre-incubazione con sfere di controllo, o lavaggi di sali per preservare proteina bassa affinità: interazioni proteina con una proteina di interesse . A ma nucleasiy essere inclusi o omessi da questo protocollo secondo le specifiche di un particolare esperimento. Ad esempio: come le proteine utilizzate in questo metodo sono RNA elicasi, RNasi cocktail è stato incluso nel protocollo per rimuovere interazioni indirette mediate attraverso l'RNA (passo 3.2). In alcuni casi, tuttavia, ci potrebbe essere beneficiano di esecuzione esperimenti paralleli con e senza nucleasi per identificare interazioni dipendenti acidi nucleici.
All'interno di questo protocollo, la commutazione di 'medium' e campioni "pesanti" negli esperimenti replicati si raccomanda di controllare la variazione introdotta da differenze nei media SILAC o la crescita delle cellule. Un controllo alternativo comporta la commutazione sequenziale di tutti e tre ('luce', 'medium', e 'pesante') media in esperimenti di replica. Mentre questo approccio è potenzialmente più rigoroso, aumenta la complessità dell'analisi, come in almeno una replica, una proteina di interesse will essere prodotto in 'leggeri' cellule marcate ed è quindi necessario distinguere tra proteine costantemente identificati in campioni 'leggeri' (contaminanti ambientali come cheratine), e quelli che sono arricchiti solo in campioni 'leggeri' quando si lega ad una proteina di interesse.
Mentre l'uso di dati quantificazione permette di discriminazione specifici da interazioni non specifiche attraverso l'uso di una soglia, inevitabilmente alcune interazioni originali può essere scartata. L'approccio di cui sopra è un metodo semplice e rapido per identificare proteine: interazioni proteiche che possono essere facilmente tentato da ricercatori con esperienza precedente con la spettrometria di massa, o l'analisi di grandi proteine: proteine set di dati di interazione. Per gli usi più questo è più che sufficiente per identificare nuove proteine di interesse. Ulteriori modifiche di questo approccio per contribuire a ridurre questa perdita di dati sono descritte altrove in letteratura e comprendono l'utilizzo di un prbiblioteca frequenza otein se noto proteine contaminanti per un insieme specifico di parametri sperimentali (linea cellulare, matrice perlina, condizioni di buffer) può essere escluso 10, 14. Tuttavia, a seconda dei parametri sperimentali, tuttavia, può essere necessario eseguire una serie di esperimenti di controllo per generare un proteoma tallone e questo può quindi aumentare sia il costo e la complessità di questo esperimento. Ulteriori informazioni su questa tecnica è disponibile sul sito www.peptracker.co.uk 14.
Va inoltre osservato che il protocollo sopra descritto comporta la miscelazione di campioni diverso etichettati al termine del processo di immunoprecipitazione (definito un Mixing dopo purificazione - esperimento SILAC MAP). Questo viene fatto come proteina: proteina si verificano in un determinato equilibrio 15. Va notato che altri gruppi hanno combinato questo approccio MAP SILAC descritto in questo protocollo con incubazione dei campioniprima pulldown (Purificazione Dopo miscelazione - PAM SILAC) per periodi di tempo diversi (20 min a 2 ore sono stati utilizzati in letteratura) 15, 16. Sulla base di quanto rapidamente un rapporto proteina scende verso 1:1 è possibile indagare qualitativamente affinità di legame e di definire proteine come proteine interagenti stabili o dinamici 15.
In sintesi, pulldowns SILAC rappresentano un mezzo molto potente per identificare proteine che interagiscono con una determinata proteina di interesse, in un ambiente fisiologicamente rilevanti. La tecnica può essere facilmente adattato ad una serie di strategie di purificazione diverse, permettendo la sua applicazione a qualsiasi proteina di interesse. Quantificazione dei risultati semplifica enormemente l'identificazione delle interazioni autentiche, e permette il rilassamento delle condizioni di buffer stringenti utilizzati per eliminare leganti non specifici, e conserva quindi le interazioni a bassa affinità. Come fino a tre campioni possono essere confrontati nel suddettostrategia, la tecnica ha chiari punti di forza in confronto differenze nel legame proteico tra diverse isoforme proteiche, proteine mutanti, o l'effetto degli inibitori farmacologici. Come intere fette di gel sono analizzati piuttosto che singole bande che macchia da Coomassie, il numero di proteine identificate ad alta fiducia sono in genere superiori a quelli individuati in un GST / TAP-pulldown standard e sperimentatore pregiudizi nella scelta proteine di interesse viene rimosso. La tecnica confronta quindi molto favorevolmente con altre tecniche comunemente utilizzate utilizzate nella identificazione di nuove proteine-interazioni (lievito 2-ibridi, GST / His o TAP pulldowns).
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari in competizione.
Riconoscimenti
Questo lavoro è stato sostenuto da sovvenzioni dal Wellcome Trust e BBSRC a IG. IG è un Wellcome Senior Fellow.
Materiali
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium (DMEM) | Dundee Cell Products | LM010 | DMEM lacking Arginine and Lysine. (and containing L-glutamine) |
| Dialysed FBS (10 kDa cutoff) 500 ml | Dundee Cell Products | DS1003 | |
| Arginine (R0) 25 g | Sigma-Aldrich | A8094 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R6) 0.5 g | Cambridge Isotope Labs | CLM-2265 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R10) 0.5 g | Cambridge Isotope Labs | CNLM-539 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Lysine (K0) 25 g | Sigma-Aldrich | L8662 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K4) 0.5 g | Cambridge Isotope Labs | DLM-2640 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K8) 0.5 g | Cambridge Isotope Labs | CNLM-291 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Penicillin/streptomycin, Liquid. 100 ml | Gibco | 15140-122 | |
| Lipofectamine 2000 transfection reagent | Invitrogen | 11668-027 | |
| GFP-trap Agarose (500 μl resin) | Chromotek | gta-20 | |
| 5x SDS Sample loading buffer | Fisher Scientific | PN39000 | The use of purchased rather than homemade sample buffer is recommended to minimize keratin contamination. |
| Protease inhibitor cocktail set III | Calbiochem | 539134 | |
| 1.7 ml prelubricated tubes | Costar | 3207 | |
| BCA protein assay kit (1 L) | Pierce | 23225 | |
| Tris (Trizma) | Sigma-Aldrich | T1503 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E6758 | |
| Sodium chloride | Sigma-Aldrich | S3014 | |
| Rnase cocktail | Ambion | AM2286 | |
| NP-40 alternative | Millipore | 492016 |
Riferimenti
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem Soc Trans. 38, 875-878 (2010).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), (2012).
- Trinkle-Mulcahy, L. Resolving protein interactions and complexes by affinity purification followed by label-based quantitative mass spectrometry. Proteomics. 12, 1623-1638 (2012).
- Emmott, E., et al. The cellular interactome of the coronavirus infectious bronchitis virus nucleocapsid protein and functional implications for virus biology. J Virol. 87, 9486-9500 (2013).
- Li, W., Belsham, G. J., Proud, C. G. Eukaryotic initiation factors 4A (eIF4A) and 4G (eIF4G) mutually interact in a 1:1 ratio in vivo. J Biol Chem. 276, 29111-29115 (2001).
- Nielsen, P. J., Trachsel, H. The mouse protein synthesis initiation factor 4A gene family includes two related functional genes which are differentially expressed. EMBO J. 7, 2097-2105 (1988).
- Galicia-Vazquez, G., Cencic, R., Robert, F., Agenor, A. Q., Pelletier, J. A cellular response linking eIF4AI activity to eIF4AII transcription. RNA. 18, 1373-1384 (2012).
- Zakowicz, H., et al. Mutational analysis of the DEAD-box RNA helicase eIF4AII characterizes its interaction with transformation suppressor Pdcd4 and eIF4GI. RNA. 11, 261-274 (2005).
- Rothbauer, U., et al. A versatile nanotrap for biochemical and functional studies with fluorescent fusion proteins. Mol Cell Proteomics. 7, 282-289 (2008).
- Trinkle-Mulcahy, L., et al. Identifying specific protein interaction partners using quantitative mass spectrometry and bead proteomes. J Cell Biol. 183, 223-239 (2008).
- Dobreva, I., Fielding, A., Foster, L. J., Dedhar, S. Mapping the integrin-linked kinase interactome using SILAC. J Proteome Res. 7, 1740-1749 (2008).
- Mittler, G., Butter, F., Mann, M. A SILAC-based DNA protein interaction screen that identifies candidate binding proteins to functional DNA elements. Genome research. 19, 284-293 (2009).
- Selbach, M., Mann, M. Protein interaction screening by quantitative immunoprecipitation combined with knockdown QUICK. Nat Methods. 3, 981-983 (2006).
- Boulon, S., et al. Establishment of a protein frequency library and its application in the reliable identification of specific protein interaction partners. Mol Cell Proteomics. 9, 861-879 (2010).
- Wang, X., Huang, L. Identifying dynamic interactors of protein complexes by quantitative mass spectrometry. Mol Cell Proteomics. 7, 46-57 (2008).
- Fang, L., et al. Characterization of the human COP9 signalosome complex using affinity purification and mass spectrometry. J Proteome Res. 7, 4914-4925 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon