Method Article
Identificação de parceiros de interação de proteínas em células de mamíferos utilizando SILAC-imunoprecipitação quantitativos Proteômica
Neste Artigo
Resumo
Experimentos de imunoprecipitação SILAC representar um poderoso meio para descobrir nova proteína: interações protéicas. Ao permitir que a quantificação relativa exacta da abundância de proteína em ambas as amostras de controlo e de ensaio, os verdadeiros interacções podem ser facilmente distinguidos dos contaminantes experimentais, e interacções de baixa afinidade preservado através da utilização de condições de tampão menos rigorosas.
Resumo
Proteômica quantitativos combinados com purificação de imunoafinidade, SILAC imunoprecipitação, representam um poderoso meio para a descoberta de nova proteína: interações protéicas. Ao permitir que a quantificação relativa exacta da abundância de proteínas tanto em amostras de controlo e de ensaio, os verdadeiros interacções podem ser facilmente distinguidos dos contaminantes experimentais. Interacções de baixa afinidade podem ser preservadas por meio da utilização de condições de tampão menos rigorosas e ficar prontamente identificável. Este protocolo descreve a marcação de células de cultura de tecidos com isótopos estáveis aminoácidos marcados, transfecção e imunoprecipitação de uma afinidade marcado proteína de interesse, seguido pela preparação para a apresentação de uma instalação de espectrometria de massa. Este protocolo, em seguida, discute como analisar e interpretar os dados retornados do espectrômetro de massa, a fim de identificar parceiros celulares que interagem com uma proteína de interesse. Como um exemplo desta técnica é aplicada a identificar proteínas de ligação aos fatores de iniciação eucarióticos tradução: eIF4AI e eIF4AII.
Introdução
Um passo essencial para a compreensão da função das proteínas é a identificação de proteínas que interagem relevantes. Quando essas proteínas são desconhecidos, há uma série de técnicas disponíveis, cada um com as suas próprias vantagens e desvantagens. Estes incluem o sistema de leveduras duplamente híbrido, utilizando ensaios de pulldown proteína recombinante, bem como a purificação por afinidade em tandem ou TAP-marcação 1, 2.
Uma adição mais recente destas técnicas é a combinação de purificação por afinidade de uma proteína de interesse a partir de uma linha celular de mamífero relevante, seguido por espectrometria de massa de análise quantitativa utilizando rotulagem isótopo estável de aminoácidos em cultura de células (SILAC) 3. Isto tem vantagens sobre a levedura abordagem de dois híbridos em que a localização celular e modificações pós-tradução não são perturbados, assim como as vantagens sobre os tradicionais, em que é uma abordagem quantitativa do que qualitativa permitindo ao utilizador a rea-marcação TAPdily distinguir não especificamente proteínas que interagem e contaminantes, de factores do hospedeiro que se ligam especificamente. Além disso, como a amostra é tipicamente analisados inteiro, em vez de bandas de proteínas individuais como, proteínas de interesse não são mascarados por semelhante a migração de proteínas em gel, nem eles tipicamente têm de estar presentes em níveis suficientes para ser visível após a coloração, levando a aumento do número de proteínas de confiança identificados 4.
Para demonstrar esta técnica, fusões GFP do fator de iniciação da tradução eucariótica intimamente relacionado eIF4AI e eIF4AII que partes sobre identidade de aminoácidos de 90% foram investigados por SILAC-imunoprecipitação proteômica quantitativa. EIF4AI humano e II foram clonados em pEGFP-C1 para formar uma proteína de fusão onde a GFP é fundido com o terminal N de elF4A. Para evitar a necessidade de se gerar linhagens de células estáveis a transfecção transiente foi usado para entregar estas construções para células 293T de isótopos estáveis rotulados.
As células foram marcadas em primeiro lugar durante duas semanas em meio de cultura de células SILAC, seguido por transfecção de plasmídeo de ADN que codifica uma proteína de interesse. As células foram então lisadas, a concentração de proteína normalizada, e quantidades iguais de lisado purificado por afinidade em agarose de anti-GFP. Quantidades iguais de eluato foram então combinados e submetidos à análise de LC-MS/MS. Os resultados desta análise são então processadas para identificar proteínas de elevada confiança: interacções de proteína (Figura 1).
SILAC imunoprecipitação permite a identificação não só de interações diretas, mas também baixa afinidade ou interações indiretas com complexos de proteína 4. Usando este sistema, eIF4AI e II imunoprecipitações permitiu a identificação reprodutível e confiável do parceiro de ligação primária eIF4G (isoformas I / II e III) 5, assim como as interacções indirectas com a eIF4E, e os vários componentes do complexo eIF3.
Protocolo
1. Geração e Passaging de linhas celulares marcadas com SILAC
Nota: o uso de Tripsina-EDTA deve ser evitada em todas as fases de passaging e preparação de amostras para análises experimentais como a tripsina pode conter aminoácidos não marcados, o que levaria a marcação incompleta de amostras.
- Para preparar um frasco de meios SILAC adicionar uma alíquota de 0,5 ml de uma arginina apropriadamente SILAC-marcado (84 mg / ml em PBS) e lisina (146 mg / ml em PBS) a um frasco de 500 ml de Arg / Lys DMEM livre (contendo L-glutamina).
- Em seguida, adicionar 50 ml de FBS dialisado e 5 mL de penicilina / estreptomicina a garrafa de 500 ml.
Nota: as células HEK 293T (ATCC), devem ser mantidas em meio DMEM sem arginina e lisina e suplementada com luz (R0K0), médio (R6K4) ou pesado (R10K8) aminoácidos, soro fetal de bovino dialisado (corte de 10 kDa), e penicilina / estreptomicina. As células devem ser mantidas em meio para um mínimo de cinco divisões celulares para assegurar a completarotulagem. Na maioria dos casos, as células são prontamente marcado no ≥ 2 semanas. - Para dividir células para passaging, as células devem ser desalojado do monocamada por bater o frasco. As alternativas incluem a utilização de raspadores de células ou por utilização, tampão de dissociação celular com base em PBS livre de enzima.
Nota: Para conservar a mídia, as células devem ser repicadas em frascos T25 e número de células maiores gerado apenas imediatamente antes de um experimento. - 24 horas antes da transfecção de células, as sementes de 3,5 x 10 6 células marcadas com SILAC em um único prato de 10 cm2 para cada condição experimental em investigação.
2. Transfecção de células marcadas com construções pEGFP-fusão
- Remova a mídia das células e substituí-lo com 9 ml de livre de antibiótico SILAC DMEM mídia (leve, médio ou pesado).
Nota: Os antibióticos não deverá ser adicionado ao meio, uma vez que podem interferir com a eficácia de reagentes de transfecção à base de lipossomas. - Prepare uma mistura de 10 mg do plasmídeo apropriado (epEGFP-C1, pEGFP-eIF4A-I, pEGFP-eIF4A-II) em 500 mL de antibiótico-livre SILAC DMEM e misture com 500 mL de DMEM SILAC sem antibiótico contendo 10 ul de reagente de transfecção (por exemplo, lipofectamina 2000). Misture a reação completamente pipetando para cima e para baixo várias vezes.
- Incubar a mistura de reacção à temperatura ambiente durante 20 min e em seguida adicionar gota a gota para a monocamada de células. Balance a placa suavemente de um lado para o outro.
- Incubar as células transfectadas a 37 ° C e 10% de CO 2 durante 24 horas.
Nota: Se a expressão de uma proteína de interesse resulta em qualquer toxicidade aparente, então pode ser necessário para reduzir a quantidade de plasmídeo transfectado e / ou a duração do período subsequente expressão.
3. Colheita lisados celulares
- Células colheita do prato em PBS gelado usando um raspador de células. Recolher as células por centrifugação a 220 xg, a 476; C durante 5 min. Lave as células uma outra 3x em 10 ml de PBS gelado.
- Ressuspender o sedimento celular em 200 ul de tampão de lise celular (10 mM de TrisCl / pH 7,5, NaCl 150 mM, EDTA 0,5 mM, NP40 a 0,5%) contendo recentemente adicionado cocktail de inibidores de protease de III na concentração de RNase e 1x cocktail (opcional) em 5 ul por ml.
Nota: Para as proteínas com ácido nucleico conhecida actividade de ligação, pode ser necessário adicionar nucleases para o lisado antes da precipitação. Nos casos em que é adicionado a nuclease, as amostras devem ser incubadas em gelo durante 30 minutos em gelo, com pipetando a cada 10 min. Extração de ácidos nucleicos totais a partir de uma pequena fração da amostra e análise por eletroforese em gel de agarose pode testar a eficácia da nuclease. - Centrifugar as amostras a 13.000 xg, 4 ° C durante 10 min reter o sobrenadante como o ligado celular solúvel.
- A concentração do ligado celular devem ser avaliadas por ensaio BCA de acordo com as instruções do fabricante.
- Utilize tampão de lise contendo mistura de inibidores de protease de III para normalizar a concentração de proteína num volume final de 500 ul.
- Ajustar o volume para 1 ml com a adição de 500 ul de tampão de diluição (10 mM de TrisCl / pH 7,5, NaCl 150 mM, EDTA 0,5 mM) contendo mistura de inibidores de protease de III na concentração final de 1x. Uma alíquota de 50 ul da amostra deve ser retida como a entrada de amostra e o lisado mantido em gelo enquanto se prepara os grânulos de anti-GFP (por exemplo, GFP-armadilha).
Nota: os rendimentos variam tipicamente entre 1-3,5 mg de proteína na final de 1 ml de amostra. Embora os tampões acima são adequados para muitas proteínas com interesse, para os outros, pode ser necessário modificar os componentes do tampão para garantir a proteína isco é solubilizado e para manter as interacções proteína-proteína. Alterações possíveis incluem o agente tampão (fosfato, HEPES), a concentração de sal (150-500 mM), a escolha do detergente, ou outros aditivos.
4. Ligação para Anti-GContas PF
- Resumidamente vortex a suspensão do grânulo para voltar a suspender as contas. Usando uma pipeta de ponta de 200 ul com a extremidade cortada, transferir 25 ul de pérolas por amostra para um tubo fresco.
Nota: O usuário deve preparar as contas para um único experimento SILAC como mastermix para minimizar a variação de amostra-a-amostra. - Para cada 25 ul de suspensão de grânulo, adiciona 20 volumes (1500 mL por 75 pasta ul) de tampão de diluição, e centrifuga-se as esferas a 2700 xg durante 5 min. Em seguida, lavar as contas umas 2x em 20 volumes de tampão de diluição.
- Adicionar 100 ul de tampão de diluição de por 25 ul de suspensão de esferas. Utilizando uma ponta de 200 ul com a extremidade cortada, transferir 85 ul desta suspensão de ressuspenso para cada uma das amostras marcadas com SILAC do passo 3.6.
- Incubar as amostras com os grânulos num rotor, a 4 ° C durante 2 horas.
5. Lavar roupa, eluição e Preparação de Amostras para Análise de MS
- Amostras centrifugar a 2.700 xg, 46; C durante 5 min. 50 ul do sobrenadante deve ser retida como a amostra não ligada, com o restante do sobrenadante descartado.
- 1 ml de tampão de diluição deve ser adicionada a cada tubo para ressuspender as esferas e a amostra centrifugada a 2700 xg, a 4 ° C durante 5 min. O sobrenadante deve ser rejeitado. Este deve ser realizado duas vezes.
- Eluir a proteína a partir das pérolas por meio da adição de 50 ul de tampão de carregamento 2 x SDS, e aquecimento a 95 ° C durante 10 min. Sedimentar as pérolas por centrifugação a 2700 xg durante 2 min a 4 ° C
- Reter o sobrenadante em tubos pré-lubrificadas, onde ele pode ser, em seguida, armazenados a -80 ° C até ser necessário. Para a submissão a uma instalação de espectrometria de massa, misture as amostras rotuladas 01:01:01 (por exemplo, 10 ml de cada) e enviar a amostra mista.
Nota: Neste ponto, as amostras podem ser testadas por western blot,-coloração com prata ou por outros meios para testar a interacção com os parceiros de ligação conhecidos / desconhecidos. Um exemplo é dado na figuraure 2 que mostra a ligação específica de um parceiro conhecido interacção - eIF4G por western blot, e o aparecimento de bandas de coloração de prata presentes em amostras suspensos a partir de uma proteína de interesse, mas não é uma amostra de controlo. - Enviar amostra para análise LC-MS/MS.
Nota: uma vez convencido de que uma proteína de interesse é marcado parceiros de interação, volumes iguais de cada amostra marcada (leve, médio e pesado) são combinados e submetidos para análise LC-MS/MS com sucesso vinculativo. É usual para apresentar 30 ul total de uma amostra para a análise de IP. Isso implicaria misturar 10 ml de amostra Light-rotulados com 10 ml de amostras pesadas marcado com 10 ml marcado médio e para dar um total de 30 mL.
6 Análise de Dados I:. Compreender os resultados, e Remoção de Identificações de baixa confiança
Nota: A lista de títulos de coluna retornados pelo software Proteome Discoverer é dada na Tabela 1 Dif.software ferentes (por exemplo, MSQuant, MaxQuant) voltará diferentes posições, no entanto, apenas um subconjunto destes é necessária para a análise e estes são comuns aos diferentes pacotes de software. Os dados devem incluir sempre um número de acesso para cada proteína identificada, relação de comparação de cada relação de amostra (luz vs médio, leve vs pesado, médio vs pesado etc), o número de peptídeos únicos identificados, e alguma forma de taxa de falsos positivos ou indicação confiança.
- Antes de atravessar os dados, primeiro copie os dados brutos para uma nova planilha. A partir desta planilha remover todas as colunas, exceto aquelas que dão o número de acesso, número de peptídeos exclusivos, índices que comparam amostras, relação de variabilidade, bem como a descrição da proteína. Se experimentos réplicas foram realizadas, estas devem ser combinados em um único arquivo de Excel, com cada experiência que aparece em uma guia separada.
Nota: Os dados de baixa confiança inclui proteínas identificadas por apenas um único péptido original, e aqueles waqui quantificação não foi possível. - Use a função de 'tipo' Excels 'para ordenar os dados pelo número de peptídeos e remover as entradas para as proteínas que faltam mais de um peptídeo. Depois classificar por relação e remover as proteínas que não possuem relações SILAC (proteínas não quantificadas).
- Converta índices SILAC a um log de dois valores usando a fórmula: '= log (Taxa SILAC, 2)', onde 'Razão SILAC' é substituído pelo identificador da célula.
Observação: Como rácio SILAC resultados em todos os dados para as proteínas que mostram uma redução na abundância sendo restrito aos valores entre 0 e 1, que é habitual para converter uma relação a uma relação SILAC SILAC registo 2, como isso significa que as proteínas, tanto aumentada ou diminuída em uma amostra são representados em uma escala logarítmica, onde 2 ou -2 representa um aumento de 4 vezes ou diminuição da abundância, e 3 ou -3 um aumento ou diminuição na abundância, respectivamente de 9 vezes. Após essa transformação, os dados devem atender a uma Gaussian distribuição centrada em torno de um rácio de log 2 SILAC de 0. Um exemplo disto é dado na Figura 3. - Criar novas colunas no arquivo excel e calcular log 2 proporções SILAC para todas as amostras / colunas simulados. Para a conversão de uma relação falsa / amostra a uma relação amostra / simulada, usar a fórmula: "= 1/ratio"
7 Análise de Dados II:. Seleção Interações grande confiança para Estudo
- Abrir Graphpad Prism, selecione Novo> Tabela de Dados e Gráfico ... 'coluna' Selecione na lista no lado esquerdo da janela, e selecione os dados Digite / Importação> Digite os valores de réplicas, empilhados em opções de colunas. Pressione 'Criar'.
- Selecione e copie um determinado log 2 Amostra / Mock SILAC coluna proporção da planilha Excel para o novo arquivo Prism.
- Clique no menu drop-down 'Inserir' e selecione 'Nova análise ". Sob análise coluna selecione 'Distribuição de freqüência '. Manter as opções padrão, clique em 'OK'.
Nota: Este passo gera um histograma que ilustra o número de proteínas identificadas numa dada proporção. Isso deve formar uma distribuição de Gauss. - Na pasta 'Resultados' uma nova seção "Histograma" terá sido gerado. Selecione a seção de distribuição de freqüência.
- Clique no menu drop-down 'Inserir' e selecione Novo análise> XY> regressão não-linear (ajuste de curva). Clique em 'distribuição de Gauss' e clique em OK.
Nota: Este passo encaixa uma curva aos dados da distribuição de frequências geradas na etapa 7.3, obtendo-se os valores que podem ser posteriormente utilizadas para calcular os limiares de significância. - Na janela de resultados que aparece, a média eo desvio-padrão são dadas. Gerar um limite, adicionando 1,96 desvios-padrão da média.
Nota: Estes valores representam as médias e desvios-padrão da dist Gaussianribution, não o conjunto de dados total. 1,96 desvios padrão iria colocar um limite no limite de confiança de 95% (p ≤ 0,05). 2,58 SD daria 99%, e 3,3 SD 99,9%. O limite deve ser determinado individualmente para cada experimento réplica, pois pode variar.
8 Análise de Dados III:. Mesclando Datasets Replica
Nota: Para ter a certeza na identificação de parceiros de interação um SILAC suspenso experiência típica idealmente deve ser realizado três vezes, com o meio e as amostras pesadas rotulados comutada para uma das repetições para o controle de qualquer efeito da mídia sobre os resultados. Uma proteína que se mostra como interagindo em pelo menos duas das três experiências, com o 'amostra switched'-mídia idealmente representando um destes, é uma interacção de alta confiança.
- Retornar para o arquivo Excel, criar uma nova guia chamada 'Combinado'. Coluna Etiqueta A 'adesão' e copie todos os números de acesso de cada um of as experiências individuais em esta única coluna.
- Selecione a guia 'Dados', e em seguida a opção "remover duplicatas '.
- Criar colunas para as relações SILAC e variabilidade proporção de cada experimento, bem como para o nome / descrição da coluna de proteína.
- Na aba descrição, usar a fórmula PROCV para coletar a descrição de cada número de acesso. Arrastar a fórmula para baixo na coluna para preencher na descrição da proteína. A fórmula PROCV é: "= PROCV (AccessionNo, WheretoLook, ColumnsAccross, False)", onde: AccessionNo é de R $ aX com X sendo o número da linha WheretoLook é o "guia Experiment Name '$ A $ 2:.! $ Y $ Z, onde Y e Z são a parte inferior direita de dados sobre a planilha que está sendo referenciado. ColumnsAccross é o número de colunas em frente ao núme adesãober na coluna A um valor desejado se encontra.
Nota: Por exemplo, se o número de acesso é na coluna A, e os dados de interesse situa-se na coluna E. O valor aqui seria de 5 (coluna A conta como 1). Como os números de adesão foram reunidas no passo b, o mais provável é que não vlookup obter todos os nomes requeridos de uma experiência. Quando ele retorna N / D, modificar a fórmula para se obter a descrição de cada experiência, por sua vez, até todas as descrições são adquiridas. - Para destacar a interação de proteínas no conjunto de dados combinados, selecione cada coluna individualmente relação e clique na guia Início, seguido por regras células Formatação Condicional> Destaque> Mais regras.
- Selecione o estilo 'clássico'. Formatar apenas células que contêm 'value celular', 'Maior ou Igual ". Na caixa, digite o valor do desvio padrão de 1,96 e clique em 'OK'.
- Avaliar a variabilidade proporção para cada interação positiva. Se subtraindo o% varESPONSABILIDADE cairia um 'hit' abaixo do valor limite, isso deve ser tratado com cautela.
Nota: Este representa o quão consistente os índices individuais para cada peptídeo que, juntos, compõem uma proporção SILAC 'as proteínas são, e é dada como uma porcentagem. Quando a relação de variabilidade é tida em conta e uma proteína dá uma proporção SILAC acima do limiar, a proteína representa uma batida. Se a variabilidade proporção dá um intervalo que fica abaixo do limiar, a 'batida' pode representar um contaminante. - Repita esse procedimento para cada uma das colunas de resultados e comparar através de experimentos. Destaque proteínas identificadas em dois ou mais experimentos representam uma interação de alta confiança.
Nota: Dependendo do banco de dados utilizado para atribuir números de adesão de proteínas, em diferentes experimentos uma proteína pode ter sido identificado com um nome diferente, ou até mesmo vários números de adesão. É importante verificar isso para garantir que as proteínas não são acidentalmente omitido.
Resultados
Numa experiência típica SILAC suspenso, a grande maioria das proteínas identificadas (> 90%) representam os contaminantes, assim como as proteínas de ligação não específica para a matriz de afinidade, e que é ilustrado na Figura 2B, mesmo quando os protocolos de lavagem remove a maioria dos contaminantes citoplasmáticos tais como GAPDH (Figura 2A). No entanto, a agregação de proteínas não se ligar especificamente a uma distribuição normal, permite que as proteínas que se ligam especificamente a uma proteína de interesse a ser distinguida como estas têm maiores proporções de amostra / simulacro do que o fundo. Embora contaminantes deve teoricamente aglomerar em torno de um rácio de log 2 SILAC de 0, este não é necessariamente o caso, um exemplo disto é dado na Figura 3. Possíveis razões para isto incluem a rotulagem SILAC imperfeita de células, carregando os volumes ou concentrações desiguais de lisado para as contas anti-GFP, a perda acidental de contas durante a purificação ou mistura desigualção de amostras no final do processo de purificação de 3. No entanto, assumindo que os dados são analisados com base em um limite de desvio padrão da média dos contaminantes normalmente distribuídos, pequenas mudanças na centragem dos dados não deve afectar a qualidade dos resultados obtidos.
Ao comparar as diferenças nas interacções proteína entre duas proteínas relacionadas, uma situação semelhante pode ocorrer quando uma proteína de interesse é produzida a níveis mais elevados do que as células no interior de uma segunda (ou devido à variação na eficiência de transfecção, ou uma propriedade intrínseca da proteína ou do ARNm) . Alguma variação na expressão (por exemplo, a Figura 2A) pode ser corrigida por analisar a relação SILAC para estas duas amostras. Neste exemplo, estas seriam as amostras GFP-eIF4AI e GFP-eIF4AII. Ao analisar a relação 4AI/4AII SILAC como discutido na seção 7, é possível identificar proteínas cuja ligação varia significativamente entre as isoformas.
Na Figura 4, uma representação de uma das proteínas de ligação de elF4A identificados em uma das experiências realizadas réplicas é mostrado ilustrando a cobertura do complexo do factor de iniciação deste protocolo alcançado. As maiores proporções foram tipicamente observados com eIF4G, que se liga diretamente à eIF4A, com rácios mais baixos para eIF4E, que se liga a eIF4G em um local longe do local de ligação eIF4A. Foram observados índices mais baixos para os membros do complexo eIF3. No entanto, isso demonstra claramente que o experimento satisfatoriamente identificados parceiros de ligação entre diretos e indiretos da eIF4AI e II. Como pode ser esperado a partir da sua identidade elevada sequência 6, proteína: interacções proteína pareceu muito conservada entre as duas isoformas, neste sistema experimental, 7, 8. Uma selecção de algumas das proteínas que interagem são apresentados na Tabela 2, que ilustra o formato de dados.| Título de Coluna | Descrição |
| Adesão | Exibe o número de acesso para a seqüência |
| Cobertura | Proporção da sequência da proteína abrangida pelos péptidos identificados |
| ♯ PSM | Peptide jogo espectral |
| ♯ peptídeos | O número total de péptidos únicos identificados para uma proteína |
| ♯ AAs | O comprimento de uma proteína em aminoácidos |
| MW (Da) | O peso molecular de uma proteína em Daltons. Exclui modificações |
| cale. PI | O ponto isoeléctrico de uma proteína teórico |
| Contagem | A pontuação total de uma proteína (o qual representa a soma dos indivpontuações peptídicos idual). A pontuação exata necessária para significância irá variar entre os experimentos. Uma instalação de MS normalmente aplicar um corte de taxa de falsa descoberta de 5%. |
| Seqüência | A sequência de aminoácidos que constituem a proteína |
| Relação | A intensidade relativa dos péptidos em uma amostra marcada chamado, em comparação com uma segunda amostra marcada |
| Contagem Relação | O número de relações de péptidos que foram utilizados para calcular a uma dada razão de proteínas |
| Variabilidade Ratio (%) | A variabilidade dos rácios de péptidos individuais usados para calcular uma determinada proporção de proteínas |
| Descrição | O nome da proteína |
Tabela 1. Títulos de coluna padrão de um relatório Proteome Discoverer.Embora a informação útil pode ser adquirida a partir de todas estas colunas, aqueles crítica para esta análise são apresentados em negrito.
| Adesão | Peptídeos | 4AI/Mock | 4AII/Mock | Nome | Análise SILAC |
| A8K7F6 | 21 | 100 | 1 | eIF4AI | Proteína 'Bait' |
| Q14240 | 22 | 0,01 | 90,855 | eIF4AII | |
| G5E9S1 | 25 | 47,575 | 30.53 | eIF4GI | Interagindo proteínas |
| Q59GJ0 | 5 | 11.778 | 10.619 | eIF4GII60; | |
| P06730 | 3 | 7,22 | 7,57 | eIF4E | |
| Q5T6W5 | 4 | 0,685 | 0,646 | hnRNPK | Contaminantes não se ligam especificamente |
| P62805 | 1 | 0,531 | 0,498 | Histona H4 | |
| H6VRG2 | 18 | - | - | Queratina-1 | Contaminantes ambientais |
| P35527 | 11 | 0,01 | 0,01 | Queratina-1 do citoesqueleto 9 |
Dados típicos de um experimento de SILAC imunoprecipitação. Dando exemplos de dados para uma proteína de interesse / isca (peptídeos, alta proporção), proteínas que interagem com a proteína de interesse (baixa peptídeos alto /, relação elevada), não especificamente de ligação Tabela 2. proteínas (baixo Pept / altaides, a relação cai abaixo de cutoff - neste experimento 0,96), e contaminantes ambientais (muitas vezes peptídeos altos, relação negativa / abaixo do limite).

Figura 1. Plano Experimental. Em primeiro lugar as células são cultivadas em meio sem arginina e lisina, e substituídos com isótopos estáveis marcado arginina e lisina durante 2 semanas (1). (2) As células são semeadas em pratos de 10 cm2 e transitoriamente transfectadas com plasmídeos que codificam para GFP (Mock) ou proteínas de fusão de GFP (amostras). (3) As células são lisadas e as proteínas de fusão de GFP ou GFP são imunoprecipitada a partir de lisados celulares. (4) As amostras são combinadas numa proporção de 1:1 e submetido a análise de LC-MS/MS. Os dados são então analisados para remover baixo ident proteína confiançaficações e para selecionar um nível de enriquecimento de proteínas correspondentes a proteínas que interagem genuínos.
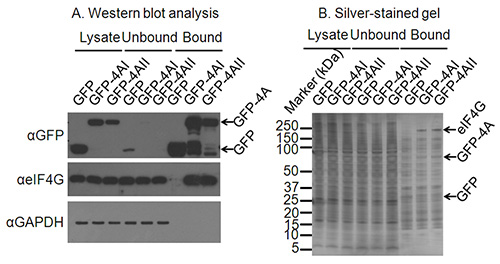
Figura 2. Confirmando condições adequadas de imunoprecipitação. A) análise de transferência de Western de lisados de células, assim como as fracções não ligadas e ligadas do imunoprecipitação confirma expressão e imunoprecipitação da proteína de interesse. Um Western blot contra GAPDH confirma a depleção de proteínas não interagem e mais uma western blot de um parceiro de interacção conhecida de elF4A confirma a imunoprecipitação bem sucedida das proteínas de ligação para a proteína de interesse. B) Quando não são conhecidos interagindo parceiros de uma proteína de interesse, um gel corado pela prata pode confirmar imunoprecipitação de proteínas que interagem. Nesta prata-mancha bandas de gel ed para GFP e GFP-eIF4AI/II são claras, e uma banda de migração no tamanho correto para eIF4G está presente apenas nas pistas GFP-4AI/II-bound e não na pista controle GFP.

Figura 3. Resultados representativos. Histograma mostrando a distribuição de proporções de proteína a partir de uma repetição de (A) GFP-eIF4AI ou (B) GFP-eIF4AII suspenso. O corte de 1,96 desvio padrão é marcado com uma linha tracejada. As proteínas que interagem abrangidos os contaminantes normalmente distribuídos são evidentes a partir de ~ 0,25 e 1 em (A), e ~ 0,3-1,5 em (B).
les/ftp_upload/51656/51656fig4.jpg "/>
Figura 4. Identificação dos membros da FEI complexos a partir de uma única réplica de uma experiência SILAC IP. Proteínas em verde foram a proteína de interesse utilizada para o suspenso Interagindo proteínas são sombreadas de vermelho para branco de acordo com a relação de log 2 SILAC no PI SILAC com vermelho sendo a proteína mais abundante na análise, e sendo o ponto de corte branco 1,96 DP. Proteínas sombreados em cinza não foram identificadas nesta análise.
Discussão
A estratégia suspenso SILAC descrito aqui representa um meio muito sensíveis e poderosos de detectar nova proteína: interações proteína, e, além disso, permite que a discriminação rápida e simples de padrões de ligação entre as amostras alteradas estreitamente relacionadas de interesse. Neste exemplo, esta técnica foi utilizada para investigar a relação proteína: interacções proteicas das proteínas eIF4AI e eIF4AII 6. Para o conhecimento do autor, este é o primeiro estudo na literatura explorando a utilidade da proteômica SILAC para investigar o interactome celular destas duas isoformas de eIF4A.
A abordagem descrita acima utiliza um GFP-tag e anti-GFP grânulos 9, 10 e, por conseguinte, as modificações podem ser necessárias para permitir que esta abordagem para ser utilizado para uma proteína específica de interesse, por exemplo, se a marca é colocada na extremidade N-ou C-terminal de uma proteína. Sempre que possível, Western blot ou ensaios funcionais devemser realizados para detectar a ligação de um parceiro de interacção proteína conhecida. Se uma proteína não tolerar fusão com a tag GFP, outra marcação ou estratégias suspensos foram aplicadas a pulldowns SILAC usando as duas outras tags (Flag 11, Biotina 12, STREP (dados próprios, não publicado)), ou usando anticorpos primários contra uma proteína de interesse onde siRNA knockdown da proteína-alvo fornece uma amostra de controlo 13. Tais experiências têm sido descritos em outros lugares na literatura, mas em breve, passo 1, e as etapas de 5,4-8 seria aplicada como acima, com passos 2-5,3 modificados conforme apropriado para o sistema de expressão / suspenso de escolha da utilização de factores de proteínas iguais como descrito em os passos 2,4-2,5. À medida que as fases de quantificação permitir proteínas de ligação não específicas a serem removidos no nível de análise, recomenda-se omitir a pré-incubação com esferas de controlo, ou lavagens elevados de sal, a fim de preservar a proteína de baixa afinidade: interacções de proteína com uma proteína de interesse . A ma nucleasey ser incluído ou omitido a partir deste protocolo de acordo com as especificidades de uma experiência particular. Por exemplo: como as proteínas utilizadas neste método são as helicases de RNA, RNase cocktail foi incluído no protocolo para remover interacções indirectas mediadas através do ARN (passo 3.2). Em alguns casos, no entanto, não pode ser beneficiar realizando experimentos paralelos com e sem nuclease para identificar ácidos nucléicos interações dependentes.
Dentro deste protocolo, a troca de amostras "pesados" em experiências idênticas, "médio" e é recomendado para controlar a variação introduzida por diferenças nos media SILAC ou crescimento celular. Um controle alternativo envolve a troca sequencial de todos os três ('light', 'médio' e 'pesado') de mídia em experimentos de réplica. Enquanto esta abordagem é potencialmente mais rigorosas, que faz aumentar a complexidade da análise, tal como em, pelo menos, uma réplica, uma proteína de interesse wdoente ser produzida em células marcadas "light" e por isso é necessário fazer a distinção entre proteínas consistentemente identificados em amostras de "light" (contaminantes ambientais, como a queratina), e aqueles que só são enriquecidas em amostras "light", quando ligado a uma proteína de interesse.
Embora o uso de dados permite a discriminação de quantificação específicos do de interacções não específicas através da utilização de um limiar, inevitavelmente algumas interacções genuínas pode ser descartado. A abordagem acima é uma abordagem simples e rápido para identificação de proteína: interações proteína que pode ser facilmente tentados por pesquisadores com nenhuma experiência anterior com espectrometria de massa, ou de análise de grandes conjuntos de dados de interação proteína: proteína. Para a maioria das utilizações este é mais do que suficiente para a identificação de novas proteínas de interesse. Outras modificações para essa abordagem para ajudar a reduzir a perda de dados são descritos em outro lugar na literatura e incluem o uso de um prbiblioteca freqüência otein onde conhecido proteínas contaminantes para um conjunto específico de parâmetros experimentais (linha celular, matriz talão, condições buffer) podem ser excluídos 10, 14. No entanto, dependendo das condições experimentais particulares, pode ser necessário executar uma série de experiências de controlo para gerar um proteoma do grânulo e, por conseguinte, este pode aumentar tanto o custo e complexidade do experimento. Mais informações sobre esta técnica está disponível no site da www.peptracker.co.uk 14.
Deve também notar-se que o protocolo descrito acima envolve a mistura de amostras marcadas de forma diferente no final do processo de imunoprecipitação (denominada uma Mistura Após Purificação - MAP SILAC experimento). Isto é feito como proteína: interacções de proteínas ocorrer num determinado equilíbrio 15. Deve notar-se que outros grupos combinaram esta abordagem MAP SILAC descrito neste protocolo com a incubação das amostrasantes de suspenso (Purificação Após mistura - PAM SILAC) para diferentes períodos de tempo (de 20 min a 2 h foram utilizados na literatura) 15, 16. Com base em como rapidamente uma proporção de proteína de gotas em relação de 1:1, é possível investigar qualitativamente afinidades de ligação e para definir as proteínas como as proteínas que interagem estáveis ou dinâmicos 15.
Em resumo, pulldowns SILAC representar um meio muito poderoso de identificação de proteínas que interagem com uma dada proteína de interesse, em um ambiente fisiologicamente relevante. A técnica pode ser muito facilmente adaptado a um certo número de diferentes estratégias de purificação, o que permite a sua aplicação a qualquer proteína de interesse. A quantificação dos resultados simplifica grandemente a identificação de interacções genuínos, e permite o relaxamento das condições do tampão utilizado para remover rigorosas ligantes não específicos, e assim preserva as interacções de baixa afinidade. Como até três amostras pode ser comparado, em que precedeestratégia, a técnica tem pontos fortes claras na comparação diferenças na proteína de ligação entre as diferentes isoformas de proteínas, proteínas mutantes, ou o efeito de inibidores farmacológicos. Como fatias inteiras de gel são analisados em vez de bandas individuais que mancha de Coomassie, o número de proteínas identificadas em alta confiança são normalmente mais elevados do que aqueles identificados em uma GST / TAP-suspenso padrão, e experimentador viés na seleção de proteínas de interesse é removido. Assim, a técnica compara muito favoravelmente com outras técnicas comumente usados utilizados na identificação de novos proteína-interações (levedura 2-híbridos, GST / His ou TAP Pulldowns).
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Este trabalho foi financiado por doações do Wellcome Trust e BBSRC a IG. IG é um Wellcome Senior Fellow.
Materiais
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium (DMEM) | Dundee Cell Products | LM010 | DMEM lacking Arginine and Lysine. (and containing L-glutamine) |
| Dialysed FBS (10 kDa cutoff) 500 ml | Dundee Cell Products | DS1003 | |
| Arginine (R0) 25 g | Sigma-Aldrich | A8094 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R6) 0.5 g | Cambridge Isotope Labs | CLM-2265 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Arginine (R10) 0.5 g | Cambridge Isotope Labs | CNLM-539 | Resuspend 0.5 g in 6 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 84 mg/ml stock) |
| Lysine (K0) 25 g | Sigma-Aldrich | L8662 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K4) 0.5 g | Cambridge Isotope Labs | DLM-2640 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Lysine (K8) 0.5 g | Cambridge Isotope Labs | CNLM-291 | Resuspend 0.5 g in 3.5 ml PBS, filter sterilize, and freeze in 0.5 ml aliquots (gives a 146 mg/ml stock) |
| Penicillin/streptomycin, Liquid. 100 ml | Gibco | 15140-122 | |
| Lipofectamine 2000 transfection reagent | Invitrogen | 11668-027 | |
| GFP-trap Agarose (500 μl resin) | Chromotek | gta-20 | |
| 5x SDS Sample loading buffer | Fisher Scientific | PN39000 | The use of purchased rather than homemade sample buffer is recommended to minimize keratin contamination. |
| Protease inhibitor cocktail set III | Calbiochem | 539134 | |
| 1.7 ml prelubricated tubes | Costar | 3207 | |
| BCA protein assay kit (1 L) | Pierce | 23225 | |
| Tris (Trizma) | Sigma-Aldrich | T1503 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E6758 | |
| Sodium chloride | Sigma-Aldrich | S3014 | |
| Rnase cocktail | Ambion | AM2286 | |
| NP-40 alternative | Millipore | 492016 |
Referências
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem Soc Trans. 38, 875-878 (2010).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), (2012).
- Trinkle-Mulcahy, L. Resolving protein interactions and complexes by affinity purification followed by label-based quantitative mass spectrometry. Proteomics. 12, 1623-1638 (2012).
- Emmott, E., et al. The cellular interactome of the coronavirus infectious bronchitis virus nucleocapsid protein and functional implications for virus biology. J Virol. 87, 9486-9500 (2013).
- Li, W., Belsham, G. J., Proud, C. G. Eukaryotic initiation factors 4A (eIF4A) and 4G (eIF4G) mutually interact in a 1:1 ratio in vivo. J Biol Chem. 276, 29111-29115 (2001).
- Nielsen, P. J., Trachsel, H. The mouse protein synthesis initiation factor 4A gene family includes two related functional genes which are differentially expressed. EMBO J. 7, 2097-2105 (1988).
- Galicia-Vazquez, G., Cencic, R., Robert, F., Agenor, A. Q., Pelletier, J. A cellular response linking eIF4AI activity to eIF4AII transcription. RNA. 18, 1373-1384 (2012).
- Zakowicz, H., et al. Mutational analysis of the DEAD-box RNA helicase eIF4AII characterizes its interaction with transformation suppressor Pdcd4 and eIF4GI. RNA. 11, 261-274 (2005).
- Rothbauer, U., et al. A versatile nanotrap for biochemical and functional studies with fluorescent fusion proteins. Mol Cell Proteomics. 7, 282-289 (2008).
- Trinkle-Mulcahy, L., et al. Identifying specific protein interaction partners using quantitative mass spectrometry and bead proteomes. J Cell Biol. 183, 223-239 (2008).
- Dobreva, I., Fielding, A., Foster, L. J., Dedhar, S. Mapping the integrin-linked kinase interactome using SILAC. J Proteome Res. 7, 1740-1749 (2008).
- Mittler, G., Butter, F., Mann, M. A SILAC-based DNA protein interaction screen that identifies candidate binding proteins to functional DNA elements. Genome research. 19, 284-293 (2009).
- Selbach, M., Mann, M. Protein interaction screening by quantitative immunoprecipitation combined with knockdown QUICK. Nat Methods. 3, 981-983 (2006).
- Boulon, S., et al. Establishment of a protein frequency library and its application in the reliable identification of specific protein interaction partners. Mol Cell Proteomics. 9, 861-879 (2010).
- Wang, X., Huang, L. Identifying dynamic interactors of protein complexes by quantitative mass spectrometry. Mol Cell Proteomics. 7, 46-57 (2008).
- Fang, L., et al. Characterization of the human COP9 signalosome complex using affinity purification and mass spectrometry. J Proteome Res. 7, 4914-4925 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados