Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un ELISA basado Encuadernación y método Competencia determinación rápida interacciones ligando-receptor
En este artículo
Resumen
The presented protocols describe two enzyme-linked immunosorbent assay (ELISA) based techniques for the rapid investigation of ligand-receptor interactions: The first assay allows the determination of dissociation constant between ligand and receptor. The second assay enables a rapid screening of blocking peptides for ligand-receptor interactions.
Resumen
A comprehensive understanding of signaling pathways requires detailed knowledge regarding ligand-receptor interaction. This article describes two fast and reliable point-by-point protocols of enzyme-linked immunosorbent assays (ELISAs) for the investigation of ligand-receptor interactions: the direct ligand-receptor interaction assay (LRA) and the competition LRA. As a case study, the ELISA based analysis of the interaction between different lambda interferons (IFNLs) and the alpha subunit of their receptor (IL28RA) is presented: the direct LRA is used for the determination of dissociation constants (KD values) between receptor and IFN ligands, and the competition LRA for the determination of the inhibitory capacity of an oligopeptide, which was designed to compete with the IFNLs at their receptor binding site. Analytical steps to estimate KD and half maximal inhibitory concentration (IC50) values are described. Finally, the discussion highlights advantages and disadvantages of the presented method and how the results enable a better molecular understanding of ligand-receptor interactions.
Introducción
Una comprensión completa de las vías de señalización requiere un conocimiento detallado acerca de la interacción ligando-receptor. La mayoría de los métodos para evaluar la interacción de un ligando particular con su receptor específico son caros, intensiva consume tiempo, trabajo y requieren equipo y la experiencia 1 específico.
Este artículo describe dos protocolos rápidos y fiables punto por punto para investigar la interacción ligando-receptor basado en una enzima ensayo inmunoenzimático (ELISA): el ensayo de interacción ligando-receptor directo (LRA) y el LRA competencia. ELISA es una técnica altamente sensible, específica y fácilmente disponible, que se utiliza de forma rutinaria en casi todos los laboratorios. ELISA se puede realizar y adaptada de varias maneras. Los protocolos presentados están optimizados para la investigación de la interacción entre diferentes interferones lambda (infls) y su receptor.
El LRA directa permite una quantificatien de unión ligando-receptor con respecto a la concentración de ligando y por lo tanto produce una curva de unión. El uso de un modelo apropiado para la interacción ligando-receptor, los datos pueden ser analizados para estimar la constante de disociación (K D).
En el protocolo presentado, la ecuación de Hill de uso común se aplica al modelo de la unión de ligando-receptor. Aunque otros métodos tales como la tecnología de resonancia de plasmón de superficie 2,3 permiten la determinación de las afinidades de unión entre dos proteínas, esta tecnología es a menudo un trabajo intensivo, costoso y requiere un equipo especial de laboratorio.
El LRA competencia permite el cribado de péptidos inhibidores de: la unión ligando-receptor se cuantifica con respecto a la concentración de péptido. Esto produce una curva de dosis-respuesta que describe el efecto inhibitorio del péptido. Los datos pueden ser analizados para estimar la media máxima concentración inhibitoria (IC 50 ) del péptido de bloqueo.
Ambos protocolos de ELISA son fáciles de usar y pueden adaptarse a una amplia gama de temas de investigación. Las proteínas recombinantes de cualquier tipo se pueden utilizar para determinar de forma fiable y rápido de las partes de interacción. Además, el LRA competencia puede ser utilizado para determinar los sitios de interacción crítica de ligandos y receptores mediante el uso de péptidos de bloqueo, que están diseñados para imitar el ligando o el receptor. Si el péptido de bloqueo muestra la inhibición eficiente y específica, el péptido se encuentra en un sitio crítico interacción del ligando (si los compuestos peptídicos miméticos del receptor) o del ligando (si los compuestos peptídicos miméticos el ligando).
El primer protocolo describe la determinación K D valor de diferentes infls y la subunidad alfa de su receptor, es decir, el receptor de la interleucina-28 (IL28RA) utilizando el directo LRA. A continuación, el segundo protocolo se muestra cómo determinar la capacidad de un péptido largo de 20 amino ácido parainhibir las interacciones INFL-IL28RA. El péptido está diseñado para competir con IFNLs en su sitio de unión al receptor y por lo tanto permite una comprensión molecular de la interacción. Además, este péptido puede ser usado para bloquear IL28RA en experimentos in vitro para determinar el impacto sobre los efectos de señalización corriente abajo 4.
Protocolo
1. Preparación de los reactivos
- Para preparar tampón de recubrimiento de carbonato, se disuelven 0,36 g de Na 2 CO 3 y 0,84 g de NaHCO3 en 100 ml de agua destilada; filtro estéril de la memoria intermedia mediante el uso de un vacío impulsado 0,22 micras polietersulfona (PES) filtro de membrana y se almacena a temperatura ambiente hasta su uso.
- Preparar la solución de lavado mediante la adición de 0,05% v / v de Tween 20 en tampón fosfato salino (PBS).
- Preparar un 5% albúmina de suero bovino (BSA) (solución de bloqueo) en solución PBS disolviendo 5 g de BSA (≥98%) en 100 ml de PBS y se almacena a 4 ° C.
- Recombinante del receptor, los ligandos y los péptidos de bloqueo
- Reconstitutethe subunidad recombinante humana receptor de la interleucina alfa (IL28RA) y ligandos recombinantes con cola de histidina de IFN humano (IFNL1-3) de acuerdo con las instrucciones del fabricante y almacenar a -80 ° C. Sintetizar el bloqueo de péptidos y se utiliza como se ha descrito previamente 4. Utilice PBS para preparar diferentes concentraciones de ligandos y péptidos para uso en los ensayos.
- Para preparar el anticuerpo primario, se diluye 6x Su anticuerpo monoclonal de ratón en PBS con 0,1% de BSA en dilución 1: 1000. Para preparar el anticuerpo secundario, se diluye con peroxidasa de rábano (HRP) conjugado de cabra anti-IgG de ratón (H + L) en PBS con 0,1% de BSA en dilución 1: 10.000.
- Preparar la solución de TMB mediante la mezcla de los reactivos A y B de acuerdo con las instrucciones del fabricante.
- Preparar la solución de parada añadiendo 5 N de ácido sulfúrico (H 2 SO 4) en agua destilada y se almacena a temperatura ambiente.
2. inmunoabsorción ligado a enzimas (ELISA Ensayos)
NOTA: La interacción directa ligando-receptor ELISA (directa LRA, Figura 1) se puede utilizar para medir la constante de disociación del receptor-ligando (K D), como una medida de la afinidad de unión receptor-ligando. La interacción competencia ligando-receptor ELISA (LRA competencia, la Figura 2) todosOWS de detección de péptidos (y otros compuestos de bloqueo), que actúan para interferir con la interacción entre ligando y receptor. El protocolo básico que fue previamente publicada el 5 ha optimizado aún más.
NOTA: En ambos ELISA métodos utilizan pipeta multicanal para añadir soluciones a los pocillos de la placa de 96 pocillos en cada paso. En decantación solución o etapas de lavado, tirar las soluciones directamente en el fregadero.
- Ligando-receptor-Interacción directa de ensayo (directa LRA)
NOTA: Para ver una ilustración del flujo de trabajo (ver Figura 1).- Placa de recubrimiento con receptor recombinante
- Diluir el receptor recombinante en tampón de carbonato a una concentración final de 100 ng / l. recubrir los pocillos de placa de microtitulación de 96 pocillos con una concentración de receptor fija (100 ng / l) pipeteando 100 l a cada pocillo usando una pipeta multicanal. Excluir paredes exteriores de la placa para evitar así artefacto borde. Cubrir la placa con una tapa e incubar laplaca a 4 ° CO / N.
- El bloqueo y la adición de ligandos
- Al día siguiente, retire la solución de recubrimiento por la inclinación de la placa contra el fregadero y lavar la placa 3 veces con solución de lavado (PBS + 0,05% v v Tween / 20).
- Bloquear los sitios de unión al receptor libres en la placa recubierta con 200 l de solución de BSA 5% a cada pocillo usando una pipeta multicanal y se incuba la placa durante 2 horas a RT.
- Descartar la solución de bloqueo (vea el paso 2.1.2.1.) Y lavar la placa 3 veces con solución de lavado.
- Preparar el recombinante marcada con His ligandos a diferentes concentraciones (por ejemplo, 8 g / ml, 4 mg / ml, 2 mg / ml, 1 mg / ml, 0,5 g / ml, 0.25 g / ml, 0,125 g / ml, 0,063 g / ml, 0,031 mg / ml, 0,0 mg / ml) en PBS. Agregue sólo PBS en los pocillos de blanco.
- Añadir 100 l de cada concentración de ligando a los pocillos por duplicado y se incuba la placa durante 2 horas a RT permitiendo receptor-ligandoInteracción.
- La incubación con anticuerpos
- Después de la incubación con los ligandos, lavar la placa 3 veces con solución de lavado.
- Pipetear 100 l de su anti-solución primaria anticuerpo monoclonal de ratón (1: 1000) a cada pocillo.
- Se incuba la placa a temperatura ambiente durante 2 h; Después de la incubación, desechar la solución de anticuerpos (véase el paso 2.1.2.1.) y lavar la placa 3 veces con solución de lavado.
- Añadir 100 l de HRP de cabra acoplados solución de anticuerpo secundario anti-IgG de ratón (1: 10.000) a cada pocillo. Incubar la placa durante 45 min a TA.
- Descartar la solución de anticuerpo (véase el paso 2.1.2.1.) Y lavar la placa 3 veces con solución de lavado.
- La adición de sustrato y el Desarrollo
- Llevar las soluciones de sustrato TMB a TA y preparar la solución de sustrato TMB A y B en la proporción de 1: 1. Añadir 100 l de sustrato recién preparada a cada pocillo y mantener la placa a temperatura ambiente durante 15-30 min. Después de suficientes col o el desarrollo de añadir la solución de parada 50 l.
- La lectura de la placa y Análisis de Datos
NOTA: El protocolo descrito se basa en la suposición de que la señal medida se eleva de unión específica. Puede ser que sea necesario para estimar la contribución de la unión no específica a la señal, pero esto está fuera del ámbito de aplicación de este protocolo.- Leer la absorbancia (densidad óptica, OD) directamente a 450 nm.
- Restar la señal de fondo a partir de los valores de DO medidos y normalizarlos. Transformar todos los valores de la concentración de ligando a escala logarítmica (base 10, log 10).
- Representar gráficamente los normalizado y corrección de fondo a los valores de DO (eje Y, corresponde a la fracción de sitios de unión del receptor de ocupados) frente al logaritmo de la concentración de ligando (eje X, ingrese 10 escala).
- Para estimar el valor KD, ajustar los datos a la siguiente forma de la ecuación de Hill:
tp_upload / 53575 / 53575eq1.jpg "/>
NOTA: Aquí Y denota la fracción de sitios de unión de los receptores ocupados e Y max la unión máxima; [L] indica la concentración de ligando libre y el coeficiente de Hill. Si sólo hay un sitio de unión para el ligando, el coeficiente de Hill es n = 1. Para sistemas con sitio de unión a más de un ligando, la unión exposiciones cooperatividad positiva si n> 1, cooperatividad negativa si n <1 y no cooperatividad si n = 1. la constante de disociación microscópico se denomina y corresponde a la mitad de la máxima concentración efectiva CE 50 6. La constante de disociación aparente es Kd = (KD) n. En el caso más simple, donde n = 1, la constante de disociación corresponde a la concentración de ligando a la que la mitad de los sitios de unión del receptor están ocupados y K d = K D. Este modelo supone la acción de masas de unión en condiciones de equilibrio, así como que sólo una pequeña fracción deel ligando añadido se une al receptor, es decir, [L] >> [RL].
- Placa de recubrimiento con receptor recombinante
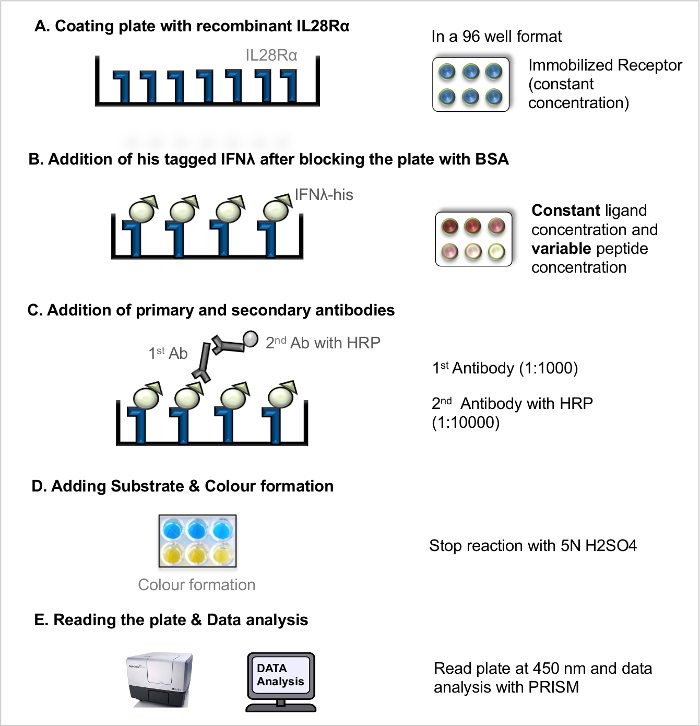
Figura 1. Un ensayo directo ligando-receptor de la interacción (directa LRA). Protocolo paso a paso para LRA directa. Por favor, haga clic aquí para ver una versión más grande de esta figura.
- Competencia ligando-receptor-Interacción de ensayo (la competencia LRA)
NOTA:. Para una ilustración del flujo de trabajo ver Figura 2 El procedimiento de concurso LRA sigue los mismos pasos que el LRA directa (recubrimiento de la placa, de anticuerpos de incubación, el desarrollo de la placa) a excepción de los cambios importantes en el ligando y péptidos etapa de adición. controles negativos apropiados son esenciales para este ensayo. En un estudio de cribado anterior 4, el péptido bloqueo revueltos no mostró efectos antagónicos.- El bloqueo - La adición de ligandos y Péptidos de bloqueo
- Al día siguiente, se retira la solución de recubrimiento y lavar la placa (véase 2.1.2.1).
- Bloquear la placa revestida por la adición de 200 l de solución de BSA 5% a cada pocillo y se incuba la placa durante 2 horas a RT.
- Preparar los ligandos recombinantes con cola de histidina (IFNL1-3) a una concentración fija (2x-20 ng / ml) en PBS.
- Preparar el péptido de bloqueo (véase la tabla 3) con diferentes concentraciones que variaban de 10 nM a 100 mM en PBS para garantizar una curva de dosis-respuesta.
NOTA: Esto permite la posterior determinación del valor IC50 para el bloqueo péptido. En pocillos de control, añadir la concentración de ligando solamente fija sin péptido para obtener el máximo (100%) de unión. En el espacio en blanco, agregar sólo PBS sin ligando o péptido. - Añadir 50 l de los ligandos (IFNL1-3) y 50 l de cada peconcentración ptide a los pocillos en los duplicados.
- Incubar la placa durante 2 horas a RT.
- La lectura de la placa de datos y análisis
NOTA: El protocolo descrito se basa en la suposición de que la señal medida se eleva de unión específica. Puede ser que sea necesario para estimar la contribución de la unión no específica a la señal, pero esto está fuera del ámbito de aplicación de este protocolo.- Leer la absorbancia (densidad óptica, OD) directamente a 450 nm.
- Restar la señal de fondo a partir de los valores de DO medidos y normalizarlos. Transformar todos los valores de la concentración de péptido a escala logarítmica (base 10, log 10).
- Representar gráficamente los normalizado y corrección de fondo a los valores de DO (eje Y, corresponde a la fracción de sitios de unión del receptor de ocupados) frente al logaritmo de la concentración de ligando (eje X, ingrese 10 escala).
- Para estimar el valor IC 50, ajustar los datos a la ecuación siguiente:
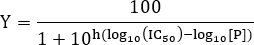
NOTA: Aquí [P] es la concentración de péptido y la pendiente de Hill. La pendiente de Hill describe la pendiente de la curva dosis-respuesta. El IC 50 corresponde a la concentración de inhibidor a la que se observó 50% de inhibición de la unión entre ligando y receptor.
- El bloqueo - La adición de ligandos y Péptidos de bloqueo

Figura 2. Competencia ensayo ligando-receptor de la interacción (la competencia LRA). Protocolo paso a paso para la competencia LRA. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Las constantes de disociación entre INFL1-3 y su subunidad alfa del receptor de IL28RA se determinaron utilizando el directo LRA. Los resultados se muestran en la Figura 3: La fracción de sitios de unión ocupados se representa frente al logaritmo de la respectiva concentración de IFN. La representación de Scatchard de los datos se muestra en la esquina inferior derecha. Los resultados ilustran que el LRA directa produce una curva de unión, que puede ser analizada m...
Discusión
ELISA es un estándar y un método bien establecido para muchos laboratorios. Hemos modificado y mejorado aún más un método publicado previamente 5,7. El protocolo demostrado paso a paso muestra cómo se puede utilizar de una manera sencilla para determinar los valores K D de interacciones ligando-receptor. Además, el IC50 de un péptido de bloqueo que interfiere con la interacción ligando-receptor puede ser determinada.
Las principales ventajas son la configuraci?...
Divulgaciones
A.E. was supported by a research grants from the "SNSF Ambizione Score" program (PZ00P3_154709), "Forschungsfond, Förderung strategischer Projekte" University of Basel, Stiftungsinfektionskrankheiten Basel, and Bangeter Rhyner Stiftung. J.L. acknowledges support by an iPhD fellowship of the SystemsX.ch initiative in systems biology program (9th call).
Agradecimientos
We thank Prof. J. Stelling (Department of Biosystems Science and Engineering, ETH Zurich and Swiss Institute for Bioinformatics, Basel, Switzerland) for his critical review of the manuscript.
Materiales
| Name | Company | Catalog Number | Comments |
| Nunc-Immunoplate (F96 Maxi sorp) | Thermo Scientific | 442404 | ELISA plate |
| Sodium carbonate (Na2CO3) | Merck | 497-19-8 | For ELISA plate coating buffer |
| Sodium hydrogen carbomnate(NaHCO3) | Merck | 144-55-8 | For ELISA plate coating buffer |
| Bovine Serum Albumin (BSA) | Sigma | A7030-100G | 5% BSA in PBS for Blocking |
| rhIL-28Rα/IFNλR1 | R&D systems | 5260-MR | Recombinant human interlukin-28 Receptor alpha |
| rhIL-29/IFNλ1 | R&D systems | 1598-IL/CF | Recombinant human interlukin-29/Carrier free/C-terminal 10-His tag |
| rhIL-28A/IFNλ2 | R&D systems | 1587-IL/CF | Recombinant human interlukin-28A/Carrier free/C-terminal 6-His tag |
| rhIL-28B/IFNλ3 | R&D systems | 5259-IL/CF | Recombinant human interlukin-28B/Carrier free/C-terminal 6-His tag |
| 6x His Monoclonal antibody (Mouse) | Clontech | 631212 | Primary antiboy to capture His tagged Ligands |
| Goat anti-Mouse igG (H+L) | Jackson Immuno Research | 115-035-166 | Horseradish Peroxidase conjucated secondary antibody |
| BDoptEIA TMB reagent set | BD Biosciences | 555214 | ELISA - TMB substrate solution |
| Sulfuric acid (H2SO4) | Fulka | 84720 | 5 N H2SO4 (Enzyme reaction stop solution) |
| Synergy/H1 - Microplate reader | BioTeK | ELISA plate reader |
Referencias
- Schneider, P., Willen, L., Smulski, C. R. Tools and techniques to study ligand-receptor interactions and receptor activation by TNF superfamily members. Methods in enzymology. 545, 103-125 (2014).
- Rossi, G., et al. Biosensor analysis of anti-citrullinated protein/peptide antibody affinity. Analytical biochemistry. 465, 96-101 (2014).
- van der Merwe, P. A., Barclay, A. N. Analysis of cell-adhesion molecule interactions using surface plasmon resonance. Curr Opin Immunol. 8, 257-261 (1996).
- Egli, A., et al. IL-28B is a key regulator of B- and T-cell vaccine responses against influenza. PLoS Pathog. 10, e1004556 (2014).
- Rosenbluh, J., et al. Positively charged peptides can interact with each other, as revealed by solid phase binding assays. Analytical biochemistry. 352, 157-168 (2006).
- Goutelle, S., et al. The Hill equation: a review of its capabilities in pharmacological modelling. Fundamental & clinical pharmacology. 22, 633-648 (2008).
- Levin, A., et al. Peptides derived from HIV-1 integrase that bind Rev stimulate viral genome integration. PLoS One. 4, e4155 (2009).
- Egli, A., Santer, M. D., O'Shea, D., Tyrrell, D. L., Houghton, M. The impact of the interferon-lambda family on the innate and adaptive immune response to viral infections. Emerging infectious diseases. , e51 (2014).
- Gad, H. H., Hamming, O. J., Hartmann, R. The structure of human interferon lambda and what it has taught us. J Interferon Cytokine Res. 30, 565-571 (2010).
- Folch, B., Rooman, M., Dehouck, Y. Thermostability of salt bridges versus hydrophobic interactions in proteins probed by statistical potentials. Journal of chemical information and modeling. 48, 119-127 (2008).
- Yuzlenko, O., Lazaridis, T. Interactions between ionizable amino acid side chains at a lipid bilayer-water interface. The journal of physical chemistry. B. 115, 13674-13684 (2011).
- Tissot, A. C., Vuilleumier, S., Fersht, A. R. Importance of two buried salt bridges in the stability and folding pathway of barnase. Biochemistry. 35, 6786-6794 (1996).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados