Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un ELISA à base de liaison et la méthode de la concurrence de déterminer rapidement interactions ligand-récepteur
Dans cet article
Résumé
The presented protocols describe two enzyme-linked immunosorbent assay (ELISA) based techniques for the rapid investigation of ligand-receptor interactions: The first assay allows the determination of dissociation constant between ligand and receptor. The second assay enables a rapid screening of blocking peptides for ligand-receptor interactions.
Résumé
A comprehensive understanding of signaling pathways requires detailed knowledge regarding ligand-receptor interaction. This article describes two fast and reliable point-by-point protocols of enzyme-linked immunosorbent assays (ELISAs) for the investigation of ligand-receptor interactions: the direct ligand-receptor interaction assay (LRA) and the competition LRA. As a case study, the ELISA based analysis of the interaction between different lambda interferons (IFNLs) and the alpha subunit of their receptor (IL28RA) is presented: the direct LRA is used for the determination of dissociation constants (KD values) between receptor and IFN ligands, and the competition LRA for the determination of the inhibitory capacity of an oligopeptide, which was designed to compete with the IFNLs at their receptor binding site. Analytical steps to estimate KD and half maximal inhibitory concentration (IC50) values are described. Finally, the discussion highlights advantages and disadvantages of the presented method and how the results enable a better molecular understanding of ligand-receptor interactions.
Introduction
Une compréhension approfondie des voies de signalisation nécessite des connaissances détaillées sur l'interaction ligand-récepteur. La plupart des méthodes d'évaluation de l'interaction d'un ligand particulier avec son récepteur spécifique sont chers, beaucoup de temps, de main - d'œuvre et nécessitent des équipements et de l' expertise 1 spécifique.
Cet article décrit deux protocoles rapides et fiables point par point pour étudier l'interaction ligand-récepteur basé sur une enzyme linked immunosorbent assay (ELISA): le ligand-récepteur directe interaction dosage (LRA) et la LRA de la concurrence. ELISA est une technique très sensible, spécifique et facilement disponibles, couramment utilisés dans presque tous les laboratoires. ELISA peut être réalisée et adaptée de diverses façons. Les protocoles présentés sont optimisés pour l'étude de l'interaction entre les différents interférons lambda (de INFLs) et leur récepteur.
La LRA directe permet une quantificatile ligand de liaison au récepteur par rapport à la concentration de ligand et on obtient ainsi une courbe de liaison. En utilisant un modèle approprié pour l'interaction ligand-récepteur, les données peuvent être analysées pour estimer la constante de dissociation (K D).
Dans le protocole présenté, l'équation de Hill couramment utilisée est appliquée au modèle de la liaison ligand-récepteur. Bien que d' autres méthodes telles que la technologie de résonance plasmonique de surface 2,3 permettent de déterminer les affinités de liaison entre les deux protéines, cette technologie est souvent un travail intensif, coûteux et nécessite un équipement de laboratoire spécial.
La LRA de la concurrence permet le criblage de peptides inhibiteurs: La liaison ligand-récepteur est quantifiée par rapport à la concentration de peptide. On obtient ainsi une courbe dose-réponse décrivant l'effet inhibiteur du peptide. Les données peuvent être analysées pour estimer la concentration inhibitrice à demi maximale (IC50 ) du peptide de blocage.
Les deux protocoles ELISA sont faciles à utiliser et peuvent être adaptés à un large éventail de questions de recherche. Les protéines recombinantes de toute nature peuvent être utilisés pour déterminer de manière fiable et rapide des pièces d'interaction. En outre, la LRA de la compétition peut être utilisée pour déterminer des sites d'interaction critiques des ligands et des récepteurs en utilisant des peptides de blocage, qui sont conçus pour simuler le ligand ou le récepteur. Si le peptide bloquant montre l'inhibition efficace et spécifique, le peptide occupe un site critique de l'interaction du ligand (si le peptide imite le récepteur) ou du ligand (si le peptide imite le ligand).
Le premier protocole décrit la détermination de la valeur K D de différents INFLs et la sous - unité alpha de leur récepteur, soit le récepteur de l' interleukine-28 (IL28RA) en utilisant la LRT directe. Ensuite, le second protocole indique comment déterminer la capacité d'un peptide long de 20 acides aminés àinhiber les interactions Infl-IL28RA. Le peptide a été conçu pour rivaliser avec IFNLs à leur site de liaison du récepteur et permet ainsi une bonne compréhension de l'interaction moléculaire. En outre, ce peptide peut être utilisé pour bloquer IL28RA dans des expériences in vitro pour déterminer l'impact sur les effets de signalisation en aval 4.
Protocole
1. Préparation des réactifs
- Pour préparer un tampon de revêtement de carbonate, on dissout 0,36 g de Na 2 CO 3 et 0,84 g de NaHCO3 dans 100 ml d'eau distillée; filtre stérile le tampon en utilisant un vide poussé de 0,22 um polyéthersulfone (PES) filtre à membrane et stocker à température ambiante jusqu'à utilisation.
- Préparer une solution de lavage par addition de 0,05% v / v de Tween 20 dans du tampon phosphate salin (PBS).
- Préparer un 5% de sérum albumine bovine (BSA) (solution de blocage) dans une solution de PBS en dissolvant 5 g de BSA (≥98%) dans 100 ml de PBS et conserver à 4 ° C.
- Recombinant Receptor, Ligands et Peptides de blocage
- Reconstitutethe sous-unité recombinante humaine récepteur de l'interleukine alpha (IL28RA) et ligands His-tagged recombinantes d'IFN humain (IFNL1-3) selon les instructions et magasin du fabricant à -80 ° C. Synthétiser peptides bloquants et utilisé comme décrit précédemment 4. Utiliser du PBS pour préparer diverses concentrations de ligands et des peptides destinés à être utilisés dans les essais.
- Pour préparer l'anticorps primaire, diluer 6x son anticorps monoclonal de souris dans du PBS avec 0,1% de BSA à 1: 1000 dilution. Pour préparer l'anticorps secondaire, on dilue la peroxydase de raifort (HRP) conjugué chèvre anti-souris IgG (H + L) dans du PBS avec 0,1% de SAB à 1: 10 000 dilution.
- Préparer une solution de TMB en mélangeant les réactifs A et B selon les instructions du fabricant.
- Préparer une solution d'arrêt en ajoutant 5 N d' acide sulfurique (H 2 SO 4) dans l' eau distillée et conserver à la température ambiante.
2. immuno-enzymatique (ELISA Assays)
NOTE: L'interaction ligand-récepteur ELISA direct (LRA directe, figure 1) peut être utilisé pour mesurer la dissociation du récepteur-ligand constante (K D), en tant que mesure de l'affinité de liaison au récepteur-ligand. L'interaction compétition ligand-récepteur ELISA (compétition LRA, Figure 2) toutOWS criblage de peptides (et d'autres composés bloquants), qui agissent de manière à interférer avec l'interaction entre le ligand et le récepteur. Le protocole de base qui a été précédemment publié 5 a été optimisée.
NOTE: Dans les deux ELISA méthodes utilisent une pipette multicanaux pour ajouter des solutions aux puits de plaque de 96 puits dans chaque étape. Dans transvaser de solution ou des étapes de lavage, jeter les solutions directement dans l'évier.
- Direct ligand-récepteur-Interaction Assay (LRA direct)
NOTE: Pour une illustration du flux de travail (voir la figure 1).- Plaque de revêtement avec récepteur recombinant
- On dilue le récepteur recombinant dans un tampon carbonate à une concentration finale de 100 ng / ul. puits Manteau de 96 puits microplaque avec la concentration du récepteur fixe (100 ng / ul) par pipetage 100 pi à chaque puits à l'aide d'une pipette multicanaux. Exclure les murs extérieurs de la plaque pour éviter ainsi l'artefact de pointe. Couvrir la plaque avec un couvercle et laisser incuber laplaque à 4 ° CO / N.
- L'addition de blocage et Ligands
- Le lendemain, retirer la solution de revêtement en inclinant la plaque contre l'évier et laver la plaque 3 fois avec une solution de lavage (PBS + 0,05% v / v de Tween 20).
- Bloquer les sites de liaison aux récepteurs libres dans la plaque revêtue en utilisant 200 pi de solution de BSA à 5% à chaque puits à l'aide d'une pipette multicanaux et incuber la plaque pendant 2 heures à la température ambiante.
- Jeter la solution de blocage (voir l'étape 2.1.2.1.) Et laver la plaque 3 fois avec une solution de lavage.
- Préparer la recombinante marquée par His ligands à des concentrations différentes (par exemple, 8 pg / ml, 4 ng / ml, 2 pg / ml, 1 pg / ml, 0,5 pg / ml, 0,25 pg / ml, 0,125 ng / mL, 0,063 pg / ml 0,031 ug / ml, 0,0 pg / ml) dans du PBS. Ajouter seulement PBS dans les puits vides.
- Ajouter 100 ul de chaque concentration de ligand dans les puits en double et on incube la plaque pendant 2 heures à température ambiante permettant de récepteur-ligandinteraction.
- Incubation avec des anticorps
- Après incubation avec les ligands, laver la plaque 3 fois avec une solution de lavage.
- Introduire à la pipette 100 ul d'anticorps anti-His solution primaire monoclonal de souris d'anticorps (1: 1000) dans chaque puits.
- Incuber la plaque à température ambiante pendant 2 h; après incubation, jeter la solution d'anticorps (voir étape 2.1.2.1.) et laver la plaque 3 fois avec une solution de lavage.
- Ajouter 100 ul de HRP de chèvre couplés solution d'anticorps secondaire anti-IgG de souris (1: 10000) dans chaque puits. Laisser incuber la plaque pendant 45 min à température ambiante.
- Jeter la solution d'anticorps (voir étape 2.1.2.1.) Et laver la plaque 3 fois avec une solution de lavage.
- Ajout du substrat et le développement
- Apporter les solutions de substrat TMB à la température ambiante et préparer TMB solution substrat A et B au rapport 1: 1. Ajouter 100 pi fraîchement préparés substrat à chaque puits et maintenir la plaque à température ambiante pendant 15-30 min. Après col suffisante ou le développement ajouter une solution d'arrêt de 50 pi.
- La lecture de la plaque et analyse des données
NOTE: Le protocole décrit est basé sur l'hypothèse que le signal mesuré augmente de liaison spécifique. Il pourrait être nécessaire d'estimer la contribution de la liaison non spécifique au signal, mais cela est hors de la portée de ce protocole.- Lire l'absorbance (densité optique, OD) directement à 450 nm.
- Soustraire le signal de fond à partir des valeurs de DO mesurées et les normaliser. Transformer toutes les valeurs de la concentration de ligand à l' échelle logarithmique (base 10, log 10).
- Terrain normalisé et le fond corrigé des valeurs de DO (axe Y, correspond à la fraction des sites occupés de liaison au récepteur) par rapport au logarithme de la concentration de ligand (axe X, log 10 échelle).
- Pour estimer la valeur K D, ajuster les données à la forme de l'équation de Hill suivante:
tp_upload / 53575 / 53575eq1.jpg "/>
NOTE: Ici Y représente la fraction des sites occupés des récepteurs de liaison et Y max la liaison de maximale; [L] désigne la concentration en ligand libre et le coefficient de Hill. S'il n'y a qu'un seul site de liaison pour le ligand, le coefficient de Hill est n = 1. Pour les systèmes avec le site de liaison plus d'un ligand, la présente liaison coopérativité positive si n> 1, coopérativité négative si n <1 et aucune coopérativité si n = 1. la constante de dissociation microscopique est appelée et correspond à la moitié maximale de CE50 concentration efficace 6. La constante de dissociation apparente Kd = a (K D) n. Dans le cas le plus simple où n = 1, la dissociation constante correspond à la concentration de ligand à laquelle la moitié des sites de liaison des récepteurs sont occupés et Kd = Kd. Ce modèle suppose la liaison dans des conditions d'équilibre, ainsi que seule une petite fraction d'action de massele ligand ajouté est lié au récepteur, à savoir, [L] >> [RL].
- Plaque de revêtement avec récepteur recombinant
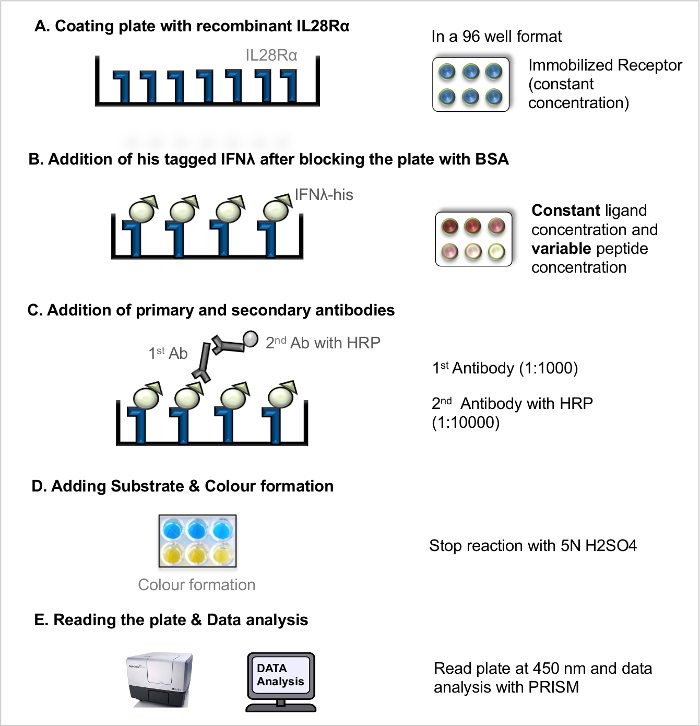
Figure 1. Direct ligand-récepteur-interaction dosage (LRA direct). Protocole étape par étape pour la LRA direct. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
- Concurrence Ligand-Receptor-Interaction Assay (Concours LRA)
NOTE:. Pour une illustration du flux de travail voir Figure 2 La procédure de concurrence de la LRA suit les mêmes étapes que la LRA directe (revêtement de la plaque, l' anticorps d' incubation, le développement de la plaque) , sauf pour des changements importants dans le ligand et peptides étape d'addition. contrôles négatifs appropriés sont essentiels pour ce dosage. Dans une étude de dépistage précédente 4, le peptide bloquant brouillé n'a pas montré d' effets antagonistes.- Blocage - Ajout de Ligands et Peptides de blocage
- Le lendemain, retirer la solution de revêtement et laver la plaque (voir 2.1.2.1).
- Bloquer la plaque revêtue par addition de 200 pl de solution de BSA à 5% à chaque puits et on incube la plaque pendant 2 heures à température ambiante.
- Préparer les ligands recombinantes His-tagged (IFNL1-3) à une concentration fixe (2x-20 ng / ml) dans du PBS.
- Préparer le peptide de blocage (voir tableau 3) à différentes concentrations allant de 10 nM à 100 pM dans du PBS pour garantir une courbe dose-réponse.
REMARQUE: Cela permet une détermination ultérieure de la valeur de CI50 pour le peptide de blocage. Dans les puits témoins, ajouter la concentration de ligand ne fixe sans peptide pour obtenir le maximum (100%) de liaison. Dans l'ébauche, ajouter seulement PBS sans ligand ou peptide. - Ajouter 50 ul des ligands (IFNL1-3) et 50 ul de chaque peconcentration ptide aux puits en double.
- Incuber la plaque pendant 2 heures à température ambiante.
- La lecture de la plaque et les données d'analyse
NOTE: Le protocole décrit est basé sur l'hypothèse que le signal mesuré augmente de liaison spécifique. Il pourrait être nécessaire d'estimer la contribution de la liaison non spécifique au signal, mais cela est hors de la portée de ce protocole.- Lire l'absorbance (densité optique, OD) directement à 450 nm.
- Soustraire le signal de fond à partir des valeurs de DO mesurées et les normaliser. Transformer toutes les valeurs de la concentration de peptide à l' échelle logarithmique (base 10, log 10).
- Terrain normalisé et le fond corrigé des valeurs de DO (axe Y, correspond à la fraction des sites occupés de liaison au récepteur) par rapport au logarithme de la concentration de ligand (axe X, log 10 échelle).
- Pour estimer la valeur IC 50, ajuster les données à l'équation suivante:
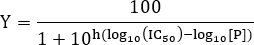
Remarque: ici [P] est la concentration du peptide et la pente de Hill. La pente de Hill décrit la pente de la courbe dose-réponse. L'IC 50 correspondant à la concentration d'inhibiteur à laquelle 50% d' inhibition de la liaison entre le ligand et le récepteur est observée.
- Blocage - Ajout de Ligands et Peptides de blocage

Figure 2. Concurrence ligand-récepteur interaction test (compétition LRA). Protocole étape par étape pour la compétition LRA. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Résultats
Les constantes de dissociation entre INFL1-3 et de leur sous-unité du récepteur alpha IL28RA ont été déterminées par la LRA directe. Les résultats sont présentés sur la figure 3: La fraction des sites de liaison occupés est tracée en fonction du logarithme de la concentration d' IFN respectif. L'intrigue Scatchard des données est affiché dans le coin inférieur droit. Les résultats montrent que la LRA directe donne une courbe de liaison, qui peut en...
Discussion
ELISA est une norme et une méthode bien établie pour de nombreux laboratoires. Nous avons en outre modifié et amélioré une méthode publiée précédemment 5,7. Le protocole montré étape par étape montre comment il peut être utilisé d'une manière simple pour déterminer les valeurs Kd interactions ligand-récepteur. En outre, la CI50 d'un peptide de blocage qui interfère avec l'interaction ligand-récepteur peut être déterminée.
Les principaux...
Déclarations de divulgation
A.E. was supported by a research grants from the "SNSF Ambizione Score" program (PZ00P3_154709), "Forschungsfond, Förderung strategischer Projekte" University of Basel, Stiftungsinfektionskrankheiten Basel, and Bangeter Rhyner Stiftung. J.L. acknowledges support by an iPhD fellowship of the SystemsX.ch initiative in systems biology program (9th call).
Remerciements
We thank Prof. J. Stelling (Department of Biosystems Science and Engineering, ETH Zurich and Swiss Institute for Bioinformatics, Basel, Switzerland) for his critical review of the manuscript.
matériels
| Name | Company | Catalog Number | Comments |
| Nunc-Immunoplate (F96 Maxi sorp) | Thermo Scientific | 442404 | ELISA plate |
| Sodium carbonate (Na2CO3) | Merck | 497-19-8 | For ELISA plate coating buffer |
| Sodium hydrogen carbomnate(NaHCO3) | Merck | 144-55-8 | For ELISA plate coating buffer |
| Bovine Serum Albumin (BSA) | Sigma | A7030-100G | 5% BSA in PBS for Blocking |
| rhIL-28Rα/IFNλR1 | R&D systems | 5260-MR | Recombinant human interlukin-28 Receptor alpha |
| rhIL-29/IFNλ1 | R&D systems | 1598-IL/CF | Recombinant human interlukin-29/Carrier free/C-terminal 10-His tag |
| rhIL-28A/IFNλ2 | R&D systems | 1587-IL/CF | Recombinant human interlukin-28A/Carrier free/C-terminal 6-His tag |
| rhIL-28B/IFNλ3 | R&D systems | 5259-IL/CF | Recombinant human interlukin-28B/Carrier free/C-terminal 6-His tag |
| 6x His Monoclonal antibody (Mouse) | Clontech | 631212 | Primary antiboy to capture His tagged Ligands |
| Goat anti-Mouse igG (H+L) | Jackson Immuno Research | 115-035-166 | Horseradish Peroxidase conjucated secondary antibody |
| BDoptEIA TMB reagent set | BD Biosciences | 555214 | ELISA - TMB substrate solution |
| Sulfuric acid (H2SO4) | Fulka | 84720 | 5 N H2SO4 (Enzyme reaction stop solution) |
| Synergy/H1 - Microplate reader | BioTeK | ELISA plate reader |
Références
- Schneider, P., Willen, L., Smulski, C. R. Tools and techniques to study ligand-receptor interactions and receptor activation by TNF superfamily members. Methods in enzymology. 545, 103-125 (2014).
- Rossi, G., et al. Biosensor analysis of anti-citrullinated protein/peptide antibody affinity. Analytical biochemistry. 465, 96-101 (2014).
- van der Merwe, P. A., Barclay, A. N. Analysis of cell-adhesion molecule interactions using surface plasmon resonance. Curr Opin Immunol. 8, 257-261 (1996).
- Egli, A., et al. IL-28B is a key regulator of B- and T-cell vaccine responses against influenza. PLoS Pathog. 10, e1004556 (2014).
- Rosenbluh, J., et al. Positively charged peptides can interact with each other, as revealed by solid phase binding assays. Analytical biochemistry. 352, 157-168 (2006).
- Goutelle, S., et al. The Hill equation: a review of its capabilities in pharmacological modelling. Fundamental & clinical pharmacology. 22, 633-648 (2008).
- Levin, A., et al. Peptides derived from HIV-1 integrase that bind Rev stimulate viral genome integration. PLoS One. 4, e4155 (2009).
- Egli, A., Santer, M. D., O'Shea, D., Tyrrell, D. L., Houghton, M. The impact of the interferon-lambda family on the innate and adaptive immune response to viral infections. Emerging infectious diseases. , e51 (2014).
- Gad, H. H., Hamming, O. J., Hartmann, R. The structure of human interferon lambda and what it has taught us. J Interferon Cytokine Res. 30, 565-571 (2010).
- Folch, B., Rooman, M., Dehouck, Y. Thermostability of salt bridges versus hydrophobic interactions in proteins probed by statistical potentials. Journal of chemical information and modeling. 48, 119-127 (2008).
- Yuzlenko, O., Lazaridis, T. Interactions between ionizable amino acid side chains at a lipid bilayer-water interface. The journal of physical chemistry. B. 115, 13674-13684 (2011).
- Tissot, A. C., Vuilleumier, S., Fersht, A. R. Importance of two buried salt bridges in the stability and folding pathway of barnase. Biochemistry. 35, 6786-6794 (1996).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon