Method Article
Detección ultrasensible de biomarcadores utilizando una impresión Molecular basado Biosensor capacitiva
En este artículo
Resumen
Aquí, presentamos un protocolo para la detección y cuantificación de moléculas abundantes bajas en soluciones complejas usando molecular imprinting en combinación con un biosensor de capacitancia.
Resumen
La capacidad para detectar y cuantificar biomoléculas en soluciones complejas siempre ha sido codiciada dentro de la ciencia natural; se utiliza para la detección de biomarcadores, contaminantes y otras moléculas de interés. Una técnica comúnmente utilizada para este propósito es la enzima-ligado del inmunosorbente ensayo (ELISA), donde a menudo un anticuerpo está dirigido hacia una molécula objetivo específico, y un segundo anticuerpo marcado se utiliza para la detección del anticuerpo primario, permitiendo la cuantificación absoluta de la biomolécula bajo estudio. Sin embargo, el uso de anticuerpos como elementos de reconocimiento limita la robustez del método; como la necesidad de la utilización de moléculas marcadas. Para superar estas limitaciones, se ha implementado impresión molecular, creación de sitios de reconocimiento artificial complementaria a la molécula de la plantilla y obsoleting la necesidad del uso de anticuerpos para el enlace inicial. Además, para mayor sensibilidad, el anticuerpo secundario marcado puede reemplazarse por biosensores confiando en la capacidad para la cuantificación de la molécula objetivo. En este protocolo, se describe un método para rápidamente y sin etiqueta de detectar y cuantificar biomoléculas baja abundante (virus y proteínas) en muestras complejas, con una sensibilidad que es significativamente mejor que los sistemas de detección utilizados como el ELISA. Todo esto es mediado por impresión molecular en combinación con un biosensor de capacitancia.
Introducción
La cuantificación de biomoléculas se utiliza en muchos campos de investigación dentro de la ciencia, empleando métodos como el radioinmunoanálisis (RIA) o ELISA1. Algunos de estos métodos requieren un etiquetado reactivo como un radioisótopo o enzima marcado antígeno anticuerpo, que hace que requieren mucho trabajo y tiempo con procedimientos complejos2. Además, la robustez, la selectividad y la sensibilidad de estos métodos no son suficientes para todos los análisis; en particular no son suficientes cuando nanogramo cantidades necesitan ser analizados, en lugar de pictograma cantidades3. Para ello, biosensores han ganado considerable interés4,5, en particular en combinación con impresión molecular para una mayor robustez.
Impresión molecular se basa en la creación de cavidades polimerizando monómeros funcionales alrededor de la plantilla6, creación de sitios de reconocimiento artificial que recuerdan perfectamente a la plantilla7. Esta técnica se ha utilizado para varias aplicaciones, incluyendo sistemas de entrega de fármacos y la separación analítica, sino también como elementos de bioreconocimiento en biosensores8,9,10. Sin embargo, todavía hay algunas dificultades en el diseño de polímeros molecularmente impresos (MIPs) para las plantillas de macromoléculas como proteínas y células de11,12. Debido a esto, muchos investigadores se han centrado en impresión la proteína de la plantilla directamente sobre un substrato, creando así una superficie que será reconocida por la proteína blanco del12. Esta técnica de recubrimiento utilizada para emplear las cavidades reconocimiento asambleas incluyendo proteínas y grandes moléculas se llama capas impresión13,14. El procedimiento general del método depende de la polimerización entre dos superficies - un sello de plantilla y un soporte de polímero - que la plantilla es adsorbida sobre una superficie de15,16y en contacto con el superficie tratada con monómero. De esta forma, se forma una película delgada de polímero sobre el soporte mediante polimerización UV. Finalmente, se retira la plantilla, dejando cavidades específicas de plantilla en la superficie del electrodo impreso. Este método tiene algunas ventajas como una pérdida de la actividad reducida de la molécula del impresa, así como que requiere cantidades muy pequeñas de moléculas de plantilla para la impresión de procesan16,17. Así, se pueden crear estas superficies rentables, estables, sensibles y selectivas en la superficie del sensor, dirigidos a cualquier plantilla de elección del usuario.
El biosensor se puede utilizar para la detección de proteínas solo y biomacromoléculas mucho más grandes, incluyendo virus. Un grupo específico de virus ganando interés reciente es el bacteriófago, que es un virus que infecta bacterias. Detección rápida y sensible de los bacteriófagos es importante durante biotecnológica y biofarmacéutica procesos para determinar las infecciones de los cultivos bacterianos con bacteriófagos18. El ensayo biológico más utilizado para la detección de bacteriófagos es el método de agar de doble capa de19, que es laborioso y consume tiempo. Se hicieron varios intentos para desarrollar nuevas herramientas de diagnóstico para virus (incluidos los bacteriófagos) como fuerza atómica (AFM) la microscopia20, interferometría21, electroquímica22sensor sistema23, 24. mucho trabajo se ha centrado en biosensores debido a sus ventajas como ser fáciles de operar, muy sensible y capaz de medición en tiempo real15,25. Un tipo específico de biosensor se basa en cambios en la capacitancia. Estos biosensores capacitivos son los sensores electroquímicos que medir los cambios en las propiedades del dieléctricas cuando un analito se interactúa con un elemento de biorreconocimiento sobre la superficie del sensor, causando una disminución en la capacitancia2,4 . Capacitivas biosensores se han utilizado para la detección de varios analitos como antígenos, anticuerpos, proteínas, metales pesados iones6,26,27,28. Estos tipos de biosensores tienen muchas ventajas como la rapidez inherente, alta sensibilidad, simplicidad, bajo costo, fácil manipulación y medición en tiempo real sin etiquetado29.
El método descrito en este documento está dirigido a permitir la detección y cuantificación de biomoléculas baja abundante en muestras muy complejas, sin la necesidad de utilizar ningún etiquetado. En particular, la técnica es más útil en el rango del picogramo atto de biomoléculas, donde otros instrumentos comercialmente existentes fallan cuantificar con precisión su objetivo.
Protocolo
1. modificación de la cubierta de cristal desliza (plantilla de estampillas)
- Para limpiar el vidrio cubreobjetos, sumerja secuencialmente en 10 mL de HCl 1.0 M, agua desionizada y 1,0 M de NaOH, respectivamente, durante 10 minutos en cada paso en un limpiador ultrasónico a temperatura ambiente.
- Seque el cubreobjetos de vidrio con gas nitrógeno.
Nota: El cubreobjetos se secan por evaporación bajo corriente de nitrógeno gaseoso.
- Seque el cubreobjetos de vidrio con gas nitrógeno.
- Sumerja el cubreobjetos limpios y secos en el 10% (v/v), 10 mL de solución de 3-amino-propil-trietoxisilano (APTES) en etanol durante 1 h para introducir a grupos amino en la superficie del vidrio de cubierta, a temperatura ambiente.
- Enjuague el cubreobjetos con agua desionizada.
- Seque el cubreobjetos con gas nitrógeno.
- Sumerja el cubreobjetos APTES modificado en 5% (v/v), 10 mL de solución de glutaraldehído en 10 mM de tampón fosfato (pH 7,4) por 2 h para activar los grupos aminos en la superficie, a temperatura ambiente6,15.
- Enjuague el cubreobjetos con 10 mM de tampón fosfato (pH 7,4) para quitar glutaraldehído al exceso de la superficie.
- Seque el cubreobjetos con gas nitrógeno.
- Preparar 1,0 mL de solución de plantilla (proteína/bacteriófago) de 10 mM de tampón fosfato (pH 7,4) en concentración de 0,1 mg/mL.
Nota: Disolver 0.1 mg de proteína en 1,0 mL de tampón fosfato (10 mM, pH 7,4). Si es necesario, un espectrofotómetro (280 nm) puede utilizarse para determinar la concentración de proteína.- 200 μL de esta solución de plantilla sobre el cubreobjetos modificadas de la gota e incubar a 4 ° C durante la noche.
Nota: La solución sobrante de plantilla se puede utilizar para preparar 1-2 más sellos de la plantilla para ser utilizado en los estudios de caracterización. - Enjuague el cubreobjetos con 10 mM de tampón fosfato (pH 7,4) con el fin de quitar la plantilla de independiente de la superficie.
Nota: Para enjuagar los electrodos, lavarlos con tampón fosfato (10 mM, pH 7.4) durante 30 s. - Seque el cubreobjetos con gas nitrógeno.
Nota: El cubreobjetos deben almacenarse a 4 ° C hasta su uso en el paso de la polimerización.
- 200 μL de esta solución de plantilla sobre el cubreobjetos modificadas de la gota e incubar a 4 ° C durante la noche.
2. modificación de electrodos de oro capacitivos
- Para limpiar los electrodos, sumerja los electrodos en un vaso de precipitados pequeño, que contiene 5 mL de etanol (70%), agua desionizada, acetona, agua desionizada, solución ácida Piraña (3:1, H2SO4: H2O2, v/v) y desionizada agua, respectivamente por 10 min en cada paso, en un limpiador ultrasónico a temperatura ambiente.
- Secar los electrodos con gas nitrógeno.
- Para llevar a cabo la electropolymerization de tiramina, preparar 8 mL de solución de tiramina de 10 mM en 10 mM fosfato tampón (pH 7.4) que contiene etanol (2 mL)15.
Nota: El volumen total de la solución de tiramina debe ser 8 mL en total incluyendo 2 mL de etanol.- Realizar análisis voltamétricos cíclicas (15 ciclos) en esta solución utilizando un potenciostato que cubre un potencial rango de 0 - 1,5 V (Ag/AgCl) y una frecuencia de barrido de 50 mV/s30.
- Enjuague los electrodos con agua desionizada.
- Secar los electrodos con gas nitrógeno.
- Sumerja los electrodos en una solución que contiene cloruro de acryloyl de 30 mM y 30 mM trimetilamina en tolueno (Vtotal = 5 mL) a temperatura ambiente durante la noche6,15,30.
- Enjuague los electrodos con agua desionizada.
Nota: Para asegurar una mejor remoción de acryloyl residuos de cloruro y trimetilamina, también es posible lavar la superficie con NaOH después de agua desionizada. - Secar los electrodos con gas nitrógeno.
- Enjuague los electrodos con agua desionizada.
3. elaboración de plantilla impresa capacitivos electrodos de oro
-
Preparación de bacteriófago impresos electrodos capacitivos de oro
- Antes de la polimerización, preparar una solución de monómero que contiene monómeros (acrilamida de N-hidroximetil) y vinculante (Dimetacrilato de polietilenglicol 400) en una proporción de 1:5 (mol/mol) en 1 mL de tampón fosfato 10 mM (pH 7,4).
Nota: La solución de monómero que contiene monómero y vinculante puede utilizarse en diferentes proporciones, o los tipos de monómero y vinculante pueden ser cambiados según la plantilla para la optimización de la interacción específica. - Añadir 1 mg de foto-iniciador en esta solución.
Nota: Si la polimerización se realiza bajo luz UV, entonces el foto iniciador debe usarse para iniciar la polimerización. Si la polimerización se realiza por polimerización de radicales libres, se debe cambiar el tipo de iniciador. - Pipetear 1,5 μl de esta solución sobre la superficie de oro del electrodo de oro modificado.
Nota: La superficie del electrodo de oro se muestra esquemáticamente en la figura 1. - Llevar el sello de la plantilla en contacto con la solución del monómero en la parte superior de la superficie del electrodo de oro.
- Iniciar la polimerización UV (365 nm, 400 W) y continuar durante 15 minutos31.
Nota: Polimerización UV se realiza dentro de un gabinete de enfriamiento que se encuentra a-25 ° C antes de iniciar la polimerización. A continuación, se enciende la luz de UV sistema de curado y la polimerización se continúa durante 15 minutos antes de apagar la luz UV sistema de curado. - Quitar el sello de la plantilla de la superficie mediante el uso de fórceps.
Nota: Al quitar el sello de la plantilla de la superficie, puede dañarse la película polimérica en la superficie. Por lo tanto, el sello debe retirarse de la superficie muy cuidadosamente y lentamente sin usar la fuerza. - Enjuagar la superficie del electrodo con agua desionizada y secarla con gas nitrógeno.
- Sumerja los electrodos en 1 mL de 10 mM 1-dodecanethiol preparado en etanol por 20 min para cubrir agujeros en la superficie del electrodo.
- Enjuague los electrodos con agua desionizada y secar los electrodos con gas nitrógeno.
- Antes de la polimerización, preparar una solución de monómero que contiene monómeros (acrilamida de N-hidroximetil) y vinculante (Dimetacrilato de polietilenglicol 400) en una proporción de 1:5 (mol/mol) en 1 mL de tampón fosfato 10 mM (pH 7,4).
-
Preparación de la proteína impresos electrodos capacitivos de oro
- Antes de la polimerización, preparar una solución de monómero que contiene monómeros (acrilamida: 54 mg; N-hydroxymethylacrylamide: 140 μl; N-isopropylacrylamide: 85,6 mg) y vinculante (Metilenbisacrilamida: 9,5 mg) en 820 μl de agua ultra pura30,32.
- Preparar 5% (v/v) N, N, N', N'-tetramethylethyldiamine (TEMED) en agua ultra pura.
- Añadir 20 μl de la solución TEMED en la solución de monómero y purgar con gas de nitrógeno durante 5 minutos.
- Preparar el persulfato de amonio 10% (p/v) (APS) en agua ultra pura.
- Añadir 20 μl de la solución de la APS en la solución de monómero.
- Pipetear 1,5 μl de la solución del monómero en el electrodo de oro modificado.
- Llevar el sello de la plantilla en contacto con la superficie tratada con monómero.
- Iniciar la polimerización a temperatura ambiente y continuar durante 5 h.
Nota: Para la preparación de electrodos de oro impresa de proteína, en lugar de UV-polimerización, polimerización de radicales libres a temperatura ambiente (25 ° C) se realiza utilizando APS-TEMED como el catalizador iniciador. - Retire el sello de la plantilla de la superficie cuidadosamente con unas pinzas.
- Enjuague el electrodo con agua desionizada y seque con gas nitrógeno.
- Sumerja los electrodos en 1 mL de 10 mM 1-dodecanethiol preparado en etanol por 20 min para cubrir agujeros en la superficie del electrodo.
- Enjuague los electrodos con agua desionizada y secar los electrodos con gas nitrógeno.
4. Caracterización de la superficie del electrodo con microscopía electrónica de barrido (SEM)
- Montar a las muestras en los soportes de aluminio con cinta adhesiva carbono.
- Recubrir los electrodos con 10 nm paladio y oro.
- Examinar los electrodos con SEM.
5. en tiempo real medidas capacitivas con plantilla imprimen capacitivos electrodos de oro
- Inserte los electrodos de oro capacitivos impresos en la celda electroquímica de flujo integrada a un biosensor capacitivo.
- Preparar 100 mL de tampón de regeneración (glicina-HCl 25 mM, pH 2.5, incluyendo 50 mM Tween-20) y 1 L de tampón de corriente (para bacteriófago impresos sistema: fosfato 10 mM, pH 7,4; para el sistema impreso de proteína: 50 mM Tris-HCl, pH 7,4).
- Iniciar el análisis con la inyección de búfer de regeneración para regenerar el sistema y el buffer corriente para volver a equilibrar el sistema durante 25 minutos.
- Preparar soluciones de plantilla estándar en la gama de la concentración deseada en el almacenador intermediario de funcionamiento.
Nota: Primero preparar un balance de la solución de plantilla disolviendo 0,1 mg de proteína, o aproximadamente 108 pfu bacteriófagos, en 1,0 mL de tampón de fosfato (fosfato 10 mM, pH 7,4). Luego, preparar las soluciones estándar para la curva de calibración poniendo diez diluciones secuenciales de 10 veces de la solución. Estas soluciones se analizarán con el biosensor capacitivo en paso 5.5. Concentración de la proteína puede medirse utilizando un espectrofotómetro (280 nm). Para medir la concentración de bacteriófago, puede utilizarse un método de doble capa de agar, que se describe en detalles anteriormente19. - Inyecte 250 μl de estas soluciones estándar secuencialmente en el sistema en óptimas condiciones (caudal: 100 μl/min, temperatura: 25 ° C).
Nota: En esta aplicación, las proteína estándar se prepararon las soluciones en el intervalo de concentraciones de 1.0 x 10-4 - 1.0 x 10-14, mientras que las concentraciones de bacteriófago en la gama 1.0 x 101 - 1.0 x 105 pfu/mL. Las soluciones fueron colocadas en la válvula de inyección y secuencialmente se inyecta en el sistema, inyectando las muestras en triplicado a través de la bomba de jeringa sola y la válvula selectora. La disminución de la capacitancia después de la inyección de las soluciones patrón derivadas de la Unión de la plantilla a las cavidades impresas se controla automáticamente por el software del instrumento.
Resultados
Siguiendo el protocolo, según el esquema en la figura 1, un electrodo de oro desnudo a ser impreso con una plantilla, que representa la estructura de un biomacromolecule. Este electrodo se puede aplicar en un biosensor capacitivo (figura 2), permitiendo la aplicación estable de una plantilla sobre el electrodo y la medición de los cambios en capacitancia sobre la Unión de la plantilla.
Una representación esquemática del biosensor capacitiva se muestra en la figura 2. La bomba de centris, que se encarga de inyección continua del corriente tampón (fosfato 10 mM, pH 7,4) y el búfer de regeneración (glicina-HCl 25 mM, pH 2.5) durante la regeneración en la célula de flujo, puede verse claramente en la figura. La célula de flujo consiste en trabajo, referencia y electrodos de contador. La válvula de inyección se compone de soluciones estándar de la proteína/del bacteriófago, que pasan por el desgaseador primero y luego se inyectan secuencialmente en el sistema. En cuanto a las soluciones de alcanzan el electrodo de trabajo insertado en la celda de flujo, el resultado es monitoreado en tiempo real. Los valores de capacitancia pueden ser registrados siguiendo el sensorgrams en la pantalla del ordenador.
Figura 3 y figura 4 representan las diferencias entre la superficie de electrodos de oro desnudos e impresos. Este paso de caracterización es importante garantizar que existen cavidades poliméricas, vistas como rugosidad en la superficie del electrodo después de impresión. Aparte de SEM, también hay otros métodos de caracterización como AFM, mediciones del ángulo de contacto, Elipsometría, etc., que pueden utilizarse para caracterizar la superficie después de la impresión. De esta forma, se puede asegurar que el proceso de impresión es acertado y que las cavidades de la plantilla se forman en la superficie. Dentro de estas cavidades, puede enlazar la plantilla con afinidad y especificidad alta.
Después de la inyección de las soluciones estándar en el sistema capacitivo, un promedio de las cinco últimas lecturas fue calculado automáticamente por el software, y se obtuvieron las gráficas de calibración trazando el cambio en capacitancia vs la concentración de la analito. La disminución de la capacitancia registrada surgió de la Unión de la plantilla. El más moléculas que se unen al electrodo de oro superficial, cuanto mayor sea la reducción de la capacidad total, según el principio general de medición capacitiva. Figura 5 y figura 6 muestran que con el aumento de concentración del analito, la ΔC aumenta como se esperaba. El rango dinámico (entre los cuales es el intervalo de concentraciones donde el sistema es útil para la detección de un objetivo específico) y el límite de detección (LOD) pueden ser evaluados mediante el análisis de estos gráficos. Según la figura 6, el biosensor capacitiva bacteriófago impreso puede detectar bacteriófagos en el rango de concentración de 101 - 105 pfu/mL, con un valor LOD de 10 pfu/mL en este estudio. Figura 5 y figura 6 también destaca la necesidad de medir la curva de calibración en el mismo intervalo requerido para la concentración de la plantilla se supone, puesto que la linealidad de la regresión puede variar con las concentraciones ( Figura 5), o tienen diferentes pendientes (figura 6). También hay que señalar que debido a las bajas concentraciones utilizado, el sistema es muy sensible a las fluctuaciones (turbidez en la muestra, guinda, etc.), y por lo tanto, se recomienda realizar por lo menos triplicado para reducir la posibilidad de incluir valores atípicos . Por la misma razón, la desviación estándar puede ser bastante significativa para las muestras muy diluidas, como se ve en la figura 6.
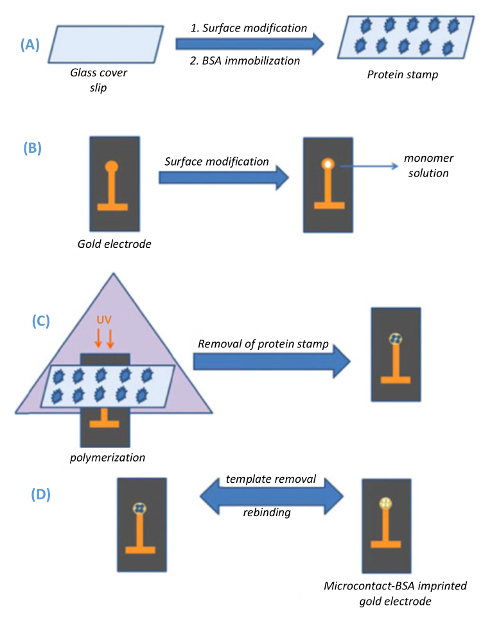
Figura 1 . Representación esquemática de las capas de impresión método. (A) preparación de los cubreobjetos de vidrio (estampillas de plantilla), (B) preparación de los electrodos capacitivos de oro, impresión de capas (C) de la plantilla sobre la superficie del electrodo de oro mediante polimerización UV y (D) retiro de plantilla de la superficie del electrodo (reproducido de Ertürk et al., biotecnología informes 2014 (3): 65-72 con permiso). Haga clic aquí para ver una versión más grande de esta figura.
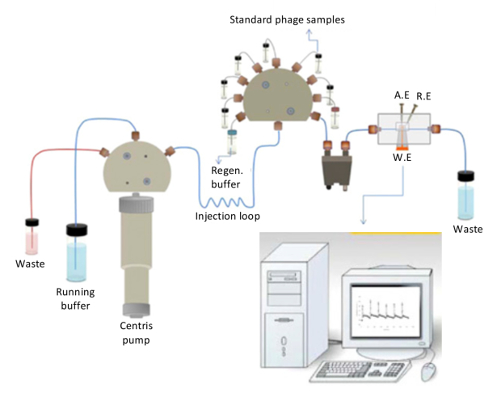
Figura 2. Representación esquemática del biosensor capacitivo. La disposición general del biosensor capacitivo utilizado en este estudio (reproducido de Ertürk et al., biotecnología informes 2014 (3): 65-72 con permiso). Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Microscopía electrónica de la proteína impresos electrodos. Imágenes de SEM de (A) un electrodo desnudo de oro (barra de escala = 20 μm), y (B) una proteína impreso Electrodo capacitivo de oro (barra de escala = 10 μm). Haga clic aquí para ver una versión más grande de esta figura.

Figura 4. Microscopía electrónica del bacteriófago impresos electrodos. Imágenes de SEM de un electrodo desnudo de oro (barra de escala = 20 μm) (A), y grabada de un bacteriófago Electrodo capacitivo de oro en diferentes aumentos (6600 x, barra de escala = 10 μm) (B) y 11, 500 X, escala de la barra = 5 μm) (C); las flechas denotan bacteriófagos adheridos). Haga clic aquí para ver una versión más grande de esta figura.
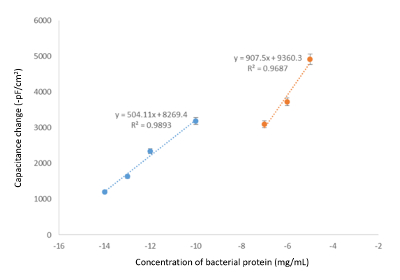
Figura 5. Efecto de la composición del tampón para gráficos de calibración. Curva de calibración que muestra el cambio en capacitancia vs concentración de proteína en condiciones óptimas (almacenador intermediario de funcionamiento: 50 mM Tris-HCl, pH 7,4; Búfer de regeneración: 25 mM glicina-HCl, pH 2.5 incluyendo 50 mM Tween-20; Caudal: 100 μl/min, volumen de muestra: 250 μl; T: 25 ° C). Haga clic aquí para ver una versión más grande de esta figura.

Figura 6. Curva de calibración representativos para grandes biomacromoléculas. Curva de calibración que muestra el cambio en capacitancia vs concentración de bacteriófago en óptimas condiciones (almacenador intermediario de funcionamiento: 10 mM fosfato, pH 7,4; Búfer de regeneración: 25 mM glicina-HCl, pH 2.5 incluyendo 50 mM Tween-20; Caudal: 100 μl/min, volumen de muestra: 250 μl; T: 25 ° C) por favor haga clic aquí para ver una versión más grande de esta figura.
Discusión
Cuando este método se lleva a cabo, hay algunos pasos críticos que deben ser considerados siguiendo el protocolo. Un paso crítico es el paso de limpieza con solución ácida de piraña. Paso 2.1 debe no ser más de 10 minutos. La solución de la plantilla de paso 1.4 no debe exceder 0,1 mg/mL, ya que estos valores se han optimizado previamente. Análisis cíclico voltametric no deben exceder 15 ciclos para obtener el grueso óptimo. Paso 3.1.3, 1,5 μl es un valor optimizado. Este valor no debe ser mayor para este tipo de electrodo. Si el sistema de curado de UV tiene una potencia de 400 W, la polimerización debe realizarse por un máximo de 10-15 minutos. Tan pronto como el APS (iniciador) se agrega a la solución después de TEMED, el paso posterior se debe realizar muy rápidamente para evitar la polimerización inmediata (paso 3.2.5).
Uno de los pasos más importantes es la eliminación de la estampilla de la plantilla de la superficie después de la polimerización UV. Si este paso no se realiza correctamente, existe el riesgo de que la película polimérica en la superficie del electrodo puede ser eliminado con el sello. Por lo tanto, se recomienda sumergir el electrodo y el sello de la proteína en la parte superior en una solución de agua después de la polimerización, a continuación, retire el sello muy lentamente y con cuidado la superficie (paso 3.1.6).
Basado en la plantilla utilizada, modificaciones en el tipo y la proporción de monómeros utilizados (monómero funcional y vinculante) pueden evaluarse en términos de generar mayor sensibilidad. Esto debe determinarse empíricamente. Además, la afinidad de unión de la molécula de la plantilla generalmente implica varias interacciones diferentes simultáneamente. Por lo tanto, esto puede llevar a problemas durante los pasos de regeneración. Si la plantilla dependiente no se suelta de la superficie correctamente, esto puede influir en la reutilización del electrodo para su posterior análisis. Estas afinidades multipuntos también pueden resultar del enlace a través de las interacciones más débiles. En tales sistemas, fijación no específica puede tener lugar, que puede influir negativamente en la selectividad del sistema16. Estas son comunes y generales, limitaciones del método.
Aparte de estas limitaciones específicas, hay muchas ventajas importantes del método discutido sobre los métodos existentes. Mientras que la medición fluorométrica, RIAs y ELISA es muy sensibles, requieren el uso de material marcado (plantilla o detector), mientras que el biosensor es totalmente etiqueta libre. Estos métodos también son más costosos y lentos. Un biosensor permite síntesis rápida y paralela de MIPs en diversas composiciones en el mismo tiempo16. Ya que sólo unos pocos microlitros de solución de monómero se requiere para la preparación, el método es conveniente cuando se utiliza monómeros limitadas caros o no. Electrodos de MIP más, solos pueden utilizarse para aproximadamente 80 análisis sin una disminución significativa en el rendimiento, que es significativamente mayor que otros de los métodos existentes30. Los métodos existentes también sufren, en diversos grados, de baja sensibilidad y selectividad, mientras que el método descrito permite la detección y cuantificación de moléculas en el rango de pM con alta selectividad.
Debido a la rentabilidad, la facilidad de operación del instrumento y la detección sensible en tiempo real en un corto tiempo en comparación con los métodos existentes, los biosensores son muy prometedor punto de sistemas de detección de cuidado bajo condiciones de campo; por ejemplo, para monitoreo del medio ambiente y para aplicaciones en los países en desarrollo. En muchas aplicaciones en el diagnóstico de la enfermedad, la detección en tiempo real, sensible, selectiva y rápida de un biomarcador en una mezcla compleja como suero es necesario15,25. Aquí, biosensores son superiores a los métodos existentes, en particular, debido a su robustez y sensibilidad. Específicamente para la detección de agentes infecciosos, bacteriófagos son considerados recientemente como elemento de biorreconocimiento alternativos para biosensores debido a su anfitrión bacterias especificidad33,34,35. Reemplazo de anticuerpos con bacteriófagos es muy prometedor para reducir el costo y aumentar la estabilidad aún más36. Este sistema permitirá también la detección y cuantificación de fagos específicos en el medio ambiente y de muestras clínicas. Debido a la prevalencia de bacteriófagos y su capacidad de transducir las bacterias con genes de resistencia a los antibióticos37,38, este método puede ser valioso estudiar la propagación de bacterias resistentes.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
María Baumgarten (IQ plataforma de biotecnología, infección de medicina, Universidad de Lund) es reconocido por realizar y brindar análisis de micrografías. Este trabajo fue apoyado por subvenciones de investigación sueco el Consejo Formas (2017-00100) como parte de la tercera articulación programación iniciativa europea llamada resistencia antimicrobiana (JPIAMR) "Dinámica de transmisión". Los fundadores no tenían ningún papel en el diseño del estudio, interpretación, escritura, preparación del manuscrito, la decisión de presentar o toma de decisiones para publicar el trabajo.
Materiales
| Name | Company | Catalog Number | Comments |
| Glass Cover slips | ThermoFisher | 102222 | protein stamp |
| HCl | Sigma-Aldrich | H1758-500ML | cleaning |
| NaOH | Sigma-Aldrich | 72068-100ML | cleaning |
| Ultrasonic cleaner | Branson Ultrasonic | BRANSONIC M1800- E | cleaning |
| 3-amino-propyl-triethoxysilane (APTES) | Sigma-Aldrich | A3648-100ML | modification |
| EtOH | Sigma-Aldrich | 1009836010 | rinsing/cleaning |
| glutaraldehyde | Sigma-Aldrich | G5882-100ML | cross-linker |
| acetone | Sigma-Aldrich | 34850-1L-M | cleaning |
| H2SO4 | Sigma-Aldrich | 339741-100ML | piranha solution |
| H2O2 | Sigma-Aldrich | H1009-500ML | piranha solution |
| tyramine | Sigma-Aldrich | T90344-5G | modification |
| CompactStat | Ivium Technologies | CompactStat.h: 30mA@10V/3MHz | potentiostat |
| Platinum Counter Electrode Kit | Equilabrium | AFCTR5 | potentiostat |
| Reference Electrode | Equilabrium | RREF0021 | potentiostat |
| acryloyl chloride | EMD Millipore | 8.00826.0100 | modification |
| triethylamine | EMD Millipore | 8.08352.0100 | modification |
| toluene | Sigma-Aldrich | 244511-100ML | modification |
| N-hydroxymethyl acrylamide | Sigma-Aldrich | 245801-100G | functional monomer |
| poly ethylene glycol-400-dimethacrylate | Sigma-Aldrich | 409510-250ML | cross-linker |
| 2-Hydroxy-4′-(2-hydroxyethoxy)-2-methylpropiophenone | Sigma-Aldrich | 410896-50G | functional monomer |
| UV polymerizator | Dymax | Dymax 5000ECE | UV-polymerization |
| forceps | Sigma-Aldrich | Z168777-1EA | consumable |
| 1-dodecanethiol | Sigma-Aldrich | 471364-100ML | blocking agent |
| acrylamide | Sigma-Aldrich | A3553-100G | functional monomer |
| N-hydroxymethylacrylamide | Sigma-Aldrich | 245801-100G | functional monomer |
| N-isopropylacrylamide | Sigma-Aldrich | 415324-50G | functional monomer |
| methylenebisacrylamide | Sigma-Aldrich | 146072-500G | cross-linking monomer |
| N,N,N',N'-tetrametyhlethyldiamine (TEMED) | Sigma-Aldrich | T9281-25ML | catalyst |
| ammonium persulphate | Sigma-Aldrich | A3678-25G | initiator |
| Capacitive biosensor | CapSenze | Equipment | |
| Glycine | Merck | 1042011000 | regeneration buffer |
| Tween-20 | Sigma-Aldrich | P9416-50ML | regeneration buffer |
| Trizma base | Sigma-Aldrich | 93352-1KG | running buffer |
| Na2HPO4 • 2H2O | Calbiochem | 567547-1KG | running buffer |
| NaH2PO4 • 2H2O | Calbiochem | 567549-1KG | running buffer |
| DELPHI correlative light and electron microscope | Phenom-World | equipment | |
| Capacitive gold electrodes | CapSenze Biosystems | consumables | |
| 2,2'-azobis(2-methypropionitrile) | Sigma-Aldrich | 441090-25G | photo-initiator |
| CapSenze Smart Software | CapSenze Biosystems | software program |
Referencias
- Lin, T. Y., Hu, C. H., Chou, T. C. Determination of albumin concentration by MIP-QCM sensor. Biosensors and Bioelectronics. 20 (1), 75-81 (2004).
- Berggren, C., Bjarnason, B., Johansson, G. Capacitive Biosensors. Electroanalysis. 13 (3), 173-180 (2001).
- Zhang, S., Garcia-D'Angeli, A., Brennan, J. P., Huo, Q. Predicting detection limits of enzyme-linked immunosorbent assay (ELISA) and bioanalytical techniques in general. The Analyst. 139 (2), 439-445 (2014).
- Mattiasson, B., Teeparuksapun, K., Hedström, M. Immunochemical binding assays for detection and quantification of trace impurities in biotechnological production. Trends in Biotechnology. 28 (1), 20-27 (2010).
- Limbut, W., Hedström, M., Thavarungkul, P., Kanatharana, P., Mattiasson, B. Capacitive biosensor for detection of endotoxin. Analytical and Bioanalytical Chemistry. 389 (2), 517-525 (2007).
- Ertürk, G., Berillo, D., Hedström, M., Mattiasson, B. Microcontact-BSA imprinted capacitive biosensor for real-time, sensitive and selective detection of BSA. Biotechnology Reports. 3, 65-72 (2014).
- Arshady, R., Mosbach, K. Synthesis of substrate-selective polymers by host-guest polymerization. Macromolecular Chemistry and Physics. 182 (2), 687-692 (1981).
- Andersson, L. I. Molecular imprinting: developments and applications in the analytical chemistry field. Journal of chromatography. B, Biomedical sciences and applications. 745 (1), 3-13 (2000).
- Li, X., Husson, S. M. Two-dimensional molecular imprinting approach to produce optical biosensor recognition elements. Langmuir: the ACS Journal of Surfaces and Colloids. 22 (23), 9658-9663 (2006).
- Alexander, C., Davidson, L., Hayes, W. Imprinted polymers: artificial molecular recognition materials with applications in synthesis and catalysis. Tetrahedron. 59 (12), 2025-2057 (2003).
- Kryscio, D. R., Peppas, N. A. Surface imprinted thin polymer film systems with selective recognition for bovine serum albumin. Analytica Chimica Acta. 718, 109-115 (2012).
- Inerowicz, H. D., Howell, S., Regnier, F. E., Reifenberger, R. Multiprotein immunoassay arrays fabricated by microcontact printing. Langmuir. 18 (13), 5263-5268 (2002).
- Lin, H. Y., Hsu, C. Y., Thomas, J. L., Wang, S. E., Chen, H. C., Chou, T. C. The microcontact imprinting of proteins: The effect of cross-linking monomers for lysozyme, ribonuclease A and myoglobin. Biosensors and Bioelectronics. 22 (4), 534-543 (2006).
- Liao, P. C., Tyan, Y. C., Wang, C. Y., Hsu, J. F., Chou, T. C., Lin, H. Y. Assessing the binding selectivity of molecularly imprinted polymer artificial antibodies by mass spectrometry-based profiling system. Journal of Biomedical Materials Research Part A. 91 (2), 597-604 (2009).
- Ertürk, G., Hedström, M., Tümer, M. A., Denizli, A., Mattiasson, B. Real-time prostate-specific antigen detection with prostate-specific antigen imprinted capacitive biosensors. Analytica Chimica Acta. 891, 120-129 (2015).
- Ertürk, G., Mattiasson, B. From imprinting to microcontact imprinting-A new tool to increase selectivity in analytical devices. Journal of Chromatography. B, Analytical Technologies in the Biomedical and Life Sciences. 1021, 30-44 (2016).
- Ertürk, G., Mattiasson, B. Molecular Imprinting: The Creation of Biorecognition Imprints on Biosensor Surfaces. Advanced Molecularly Imprinting Materials. , 523-560 (2017).
- Janczuk-Richter, M., et al. Long-period fiber grating sensor for detection of viruses. Sensors and Actuators B: Chemical. 250, 32-38 (2017).
- Kropinski, A. M., Mazzocco, A., Waddell, T. E., Lingohr, E., Johnson, R. P. Enumeration of bacteriophages by double agar overlay plaque assay. Methods in Molecular Biology. 501, 69-76 (2009).
- Meillan, M., et al. Self-assembled monolayer for AFM measurements of Tobacco Mosaic Virus (TMV) at the atomic level. RSC Adv. 4 (23), 11927 (2014).
- Ymeti, A., et al. Fast, ultrasensitive virus detection using a Young interferometer sensor. Nano Letters. 7 (2), 394-397 (2007).
- Wei, Y., Wong, L. P., Toh, C. -. S. Fuel cell virus sensor using virus capture within antibody-coated nanochannels. Analytical Chemistry. 85 (3), 1350-1357 (2013).
- Guliy, O. I., et al. Immunodetection of bacteriophages by a piezoelectric resonator with lateral electric field. Applied biochemistry and microbiology. 52 (4), 457-463 (2016).
- Reta, N., Michelmore, A., Saint, C., Prieto-Simón, B., Voelcker, N. H. Porous silicon membrane-modified electrodes for label-free voltammetric detection of MS2 bacteriophage. Biosensors & Bioelectronics. 80, 47-53 (2016).
- Ertürk, G., Özen, H., Tümer, M. A., Mattiasson, B., Denizli, A. Microcontact imprinting based surface plasmon resonance (SPR) biosensor for real-time and ultrasensitive detection of prostate specific antigen (PSA) from clinical samples. Sensors and Actuators B: Chemical. 224, 823-832 (2016).
- Lebogang, L., Mattiasson, B., Hedström, M. Capacitive sensing of microcystin variants of Microcystis aeruginosa using a gold immunoelectrode modified with antibodies, gold nanoparticles and polytyramine. Microchimica Acta. 181 (9-10), 1009-1017 (2014).
- Loyprasert, S., Hedström, M., Thavarungkul, P., Kanatharana, P., Mattiasson, B. Sub-attomolar detection of cholera toxin using a label-free capacitive immunosensor. Biosensors & Bioelectronics. 25 (8), 1977-1983 (2010).
- Teeparuksapun, K., Hedström, M., Wong, E. Y., Tang, S., Hewlett, I. K., Mattiasson, B. Ultrasensitive detection of HIV-1 p24 antigen using nanofunctionalized surfaces in a capacitive immunosensor. Analytical Chemistry. 82 (20), 8406-8411 (2010).
- Labib, M., Hedström, M., Amin, M., Mattiasson, B. A capacitive biosensor for detection of staphylococcal enterotoxin B. Analytical and Bioanalytical Chemistry. 393 (5), 1539-1544 (2009).
- Ertürk, G., Hedström, M., Mattiasson, B. A sensitive and real-time assay of trypsin by using molecular imprinting-based capacitive biosensor. Biosensors & Bioelectronics. 86, 557-565 (2016).
- Ertürk, G., Uzun, L., Tümer, M. A., Say, R., Denizli, A. Fab fragments imprinted SPR biosensor for real-time human immunoglobulin G detection. Biosensors & Bioelectronics. 28 (1), 97-104 (2011).
- El-Sharif, H. F., Phan, Q. T., Reddy, S. M. Enhanced selectivity of hydrogel-based molecularly imprinted polymers (HydroMIPs) following buffer conditioning. Analytica Chimica Acta. 809, 155-161 (2014).
- Wang, F., et al. Detection of Salmonella Typhimurium on Spinach Using Phage-Based Magnetoelastic Biosensors. Sensors (Basel, Switzerland). 17 (2), (2017).
- Chin, B. A., et al. Rapid detection of small quantities of specific bacteria using phage-based wireless biosensors. 2016 10th International Conference on Sensing Technology (ICST). , 1-5 (2016).
- Park, M. -. K., Chin, B. A. Novel Approach of a Phage-Based Magnetoelastic Biosensor for the Detection of Salmonella enterica serovar Typhimurium in Soil. Journal of Microbiology and Biotechnology. 26 (12), 2051-2059 (2016).
- Mack, J. D., Yehualaeshet, T., Park, M. -. K., Tameru, B., Samuel, T., Chin, B. A. Phage-Based Biosensor and Optimization of Surface Blocking Agents to Detect Salmonella typhimurium on Romaine Lettuce. Journal of food safety. 37 (2), 12299 (2017).
- Colomer-Lluch, M., Jofre, J., Muniesa, M. Antibiotic resistance genes in the bacteriophage DNA fraction of environmental samples. Plos One. 6 (3), 17549 (2011).
- Lood, R., Ertürk, G., Mattiasson, B. Revisiting antibiotic resistance spreading in wastewater treatment plants - bacteriophages as a much neglected potential transmission vehicle. Frontiers in microbiology. 8, (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados