Method Article
Ultrasensibile rilevazione di biomarcatori utilizzando un Imprinting molecolare basato biosensore capacitivo
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per la rilevazione e la quantificazione delle molecole abbondanti bassi in soluzioni complesse utilizzando molecolare imprinting in combinazione con un biosensore di capacitanza.
Abstract
La capacità di rilevare e quantificare biomolecole in soluzioni complesse è sempre stato molto ricercata all'interno delle scienze naturali; utilizzato per l'individuazione di biomarcatori, contaminanti e altre molecole di interesse. Una tecnica comunemente utilizzata per questo scopo è l'enzima-collegata dell'immunosorbente Assay (ELISA), dove spesso un anticorpo è diretto verso una molecola specifica dell'obiettivo, e un secondo anticorpo marcato viene utilizzato per la rilevazione dell'anticorpo primario, consentendo la Quantificazione assoluta di biomolecole in fase di studio. Tuttavia, l'utilizzo di anticorpi come elementi di riconoscimento limita la robustezza del metodo; così come l'esigenza di usare molecole con etichettate. Per superare queste limitazioni, imprinting molecolare è stata implementata, creazione di siti di riconoscimento artificiale complementari alla molecola del modello e alla necessità dell'uso di anticorpi per l'associazione iniziale. Ulteriormente, per ancora più elevata sensibilità, l'anticorpo secondario marcato può essere sostituita da biosensori affidarsi la capacità per la quantificazione della molecola target. In questo protocollo, descriviamo un metodo per rapidamente e privo di etichetta rilevare e quantificare basso-abbondanti biomolecole (proteine e virus) in campioni complessi, con una sensibilità che è significativamente migliore rispetto ai sistemi di rilevamento comunemente usati come l'ELISA. Tutto questo è mediato dall'imprinting molecolare in combinazione con un biosensore di capacitanza.
Introduzione
La quantificazione delle biomolecole è utilizzata in molti campi di ricerca diversi all'interno delle scienze, che impiegano metodi come la radioimmunoanalisi (RIA) o ELISA1. Alcuni di questi metodi richiedono un etichettati reagente come un radioisotopo o enzima etichettato anticorpi e antigeni, che li rende laborioso e dispendioso con procedure complesse2. Ulteriormente, la robustezza, la selettività e la sensibilità di questi metodi non sono sufficienti per tutte le analisi; in particolare non sono sufficienti quando attogram quantitativi devono essere analizzati, piuttosto che di pittogramma quantità3. Per questo scopo, biosensori hanno guadagnato notevole interesse4,5, in particolare in combinazione con imprinting molecolare per una maggiore robustezza.
Imprinting molecolare si basa sulla creazione di cavità di polimerizzazione di monomeri funzionali intorno al modello6, creazione di siti di riconoscimento artificiale che assomigliano perfettamente il modello7. Questa tecnica è stata utilizzata per diverse applicazioni, tra cui sistemi di drug delivery e separazione analitica, ma anche come elementi di presta in biosensori8,9,10. Tuttavia, ci sono ancora alcune difficoltà nella progettazione di stampati molecolare Polimeri (MIPs) per i modelli macromolecolari come proteine e cellule11,12. A causa di questo, molti ricercatori si sono concentrati sull'imprinting la proteina modello direttamente su un substrato, creando così una superficie che verrà riconosciuta dalla proteina bersaglio12. Questa tecnica di rivestimento di superficie utilizzata per impiegare cavità di riconoscimento per assembly tra cui proteine e molecole di grandi dimensioni viene chiamata microcontatto imprinting13,14. La procedura generale del metodo dipende la polimerizzazione tra due superfici - un timbro di modello e un supporto di polimero - su cui il modello è adsorbito su una superficie di15,16e portato in contatto con il superficie trattata monomero. In questo modo, un film polimerico sottile è formato sul supporto tramite polimerizzazione UV. Infine, il modello viene rimosso, lasciando dietro di sé il cavità specifiche modello sulla superficie dell'elettrodo impresso. Questo metodo presenta alcuni vantaggi, tra cui una perdita ridotta attività della molecola impressa, anche come che richiedono piccole quantità di molecole di modello per l'imprinting elaborare16,17. Così, queste superfici conveniente, stabile, sensibile e selettive possono essere create sulle superfici sensore, targeting per qualsiasi modello di scelta dell'utente.
Il biosensore può essere utilizzato per la rilevazione di singole proteine e biomacromolecole molto più grandi, compresi i virus. Un gruppo specifico di virus guadagnando interesse recente è il batteriofago, che è un virus che infetta i batteri. Rilevamento rapido e sensibile dei batteriofagi è importante durante biotecnologiche e biofarmaceutica processi al fine di determinare le infezioni di colture batteriche con batteriofagi18. Il saggio biologico più comunemente utilizzato per il rilevamento del batteriofago è il doppio strato agar metodo19, che è laboriosa e richiede tempo. Parecchi tentativi sono stati fatti per sviluppare nuovi strumenti diagnostici per virus (compresi i batteriofagi) come forza atomica (AFM) la microscopia20, interferometria21, elettrochimica22e sensore sistema23, 24. un sacco di lavoro è stato incentrato su biosensori a causa di loro vantaggi come essendo facile da usare, altamente sensibile e capace di misurazione in tempo reale15,25. Un tipo specifico di biosensore è basato su variazioni di capacità. Questi biosensori capacitivi sono i sensori elettrochimici che misurano i cambiamenti nelle proprietà del dielettrica quando un analita interagisce con un elemento inoltre sulla superficie del sensore, causando una diminuzione nella capacità2,4 . Capacitivi biosensori sono stati utilizzati per la rilevazione di vari analiti come antigeni, anticorpi, proteine e metalli pesanti ioni6,26,27,28. Questi tipi di biosensori hanno molti vantaggi come intrinseca rapidità, alta sensibilità, semplicità, basso costo, facile manipolazione e misura in tempo reale senza etichettatura29.
Il metodo descritto nel presente documento mira a consentire l'individuazione e la quantificazione di basso-abbondanti biomolecole in campioni altamente complessi, senza la necessità dell'utilizzo di qualsiasi etichettatura. In particolare, la tecnica è più utile nella gamma atto-picogrammo di biomolecole, dove altri strumenti commercialmente esistenti non riescono a quantificare con precisione la loro destinazione.
Protocollo
1. modifica del coprioggetto in vetro (modello francobolli)
- Per pulire il vetro vetrini coprioggetto, immergerli in sequenza in 10 mL di HCl 1,0 M, acqua deionizzata e 1,0 M di NaOH, rispettivamente, per 10 min in ogni passaggio in un pulitore ad ultrasuoni a temperatura ambiente.
- Asciugare il vetro vetrini coprioggetto con gas azoto.
Nota: I vetrini coprioggetto sono asciugati da evaporazione sotto un flusso di azoto gassoso.
- Asciugare il vetro vetrini coprioggetto con gas azoto.
- Immergere i vetrini puliti e asciugati in 10% (v/v), 10 mL di soluzione di 3-ammino-propil-trietossisilano (APTES) in etanolo per 1 h per introdurre gruppi amminici sulla superficie del vetro di copertura, a temperatura ambiente.
- Sciacquare i vetrini con acqua deionizzata.
- Asciugare i vetrini con gas azoto.
- Immergere i vetrini APTES-modificato nel 5% (v/v), 10 mL di soluzione di glutaraldeide in tampone fosfato 10 mM (pH 7.4) per 2 h per attivare i gruppi amminici sulla superficie, a temperatura ambiente6,15.
- Sciacquare i vetrini con 10 mM di tampone fosfato (pH 7,4) al fine di rimuovere glutaraldeide in eccesso dalla superficie.
- Asciugare i vetrini con gas azoto.
- Preparare 1,0 mL di soluzione di modello (proteina/del batteriofago) in tampone fosfato 10 mM (pH 7.4) in concentrazione di 0,1 mg/mL.
Nota: Sciogliere 0,1 mg di proteina in 1,0 mL di tampone fosfato (10 mM, pH 7.4). Se necessario, uno spettrofotometro (280 nm) può essere utilizzato per determinare la concentrazione di proteina.- Goccia di 200 µ l di questa soluzione di modello sul coprioggetto modificate e incubare a 4 ° C durante la notte.
Nota: La soluzione del modello in eccesso possa essere utilizzata per preparare 1-2 Altri Stamp di modello a essere utilizzata negli studi di caratterizzazione. - Sciacquare i vetrini con 10 mM di tampone fosfato (pH 7,4) al fine di rimuovere il modello non associato dalla superficie.
Nota: Per sciacquare gli elettrodi, lavarli con tampone fosfato (10 mM, pH 7.4) per 30 s. - Asciugare i vetrini con gas azoto.
Nota: Le polizze di copertura deve essere conservate a 4 ° C fino a quando non vengono utilizzati in fase di polimerizzazione.
- Goccia di 200 µ l di questa soluzione di modello sul coprioggetto modificate e incubare a 4 ° C durante la notte.
2. modifica delle sonde capacitive di oro
- Per pulire gli elettrodi, immergere gli elettrodi in un becher da piccolo, in sequenza, contenente 5 mL di etanolo (70%), deionizzata acqua, acetone, acqua deionizzata, soluzione piranha acide (3:1, H2SO4: H2O2, v/v) e deionizzata acqua, rispettivamente per 10 min in ogni passo, in un pulitore ad ultrasuoni a temperatura ambiente.
- Asciugare gli elettrodi con gas azoto.
- Per poter eseguire l'elettropolimerizzazione di tiramina, preparare 8 mL di soluzione di tiramina di 10 mM in 10 mM fosfato tampone (pH 7,4) contenente etanolo (2 mL)15.
Nota: Il volume totale della soluzione tiramina deve essere 8 mL in totale di cui 2 mL di etanolo.- Eseguire analisi voltammetriche ciclici (15 cicli) in questa soluzione utilizzando un potenziostato coprendo un potenziale gamma di 0 - 1.5 V (Ag/AgCl) e una velocità di scansione di 50 mV/s30.
- Sciacquare gli elettrodi con acqua deionizzata.
- Asciugare gli elettrodi con gas azoto.
- Immergere gli elettrodi in una soluzione contenente cloruro di acriloil di 30 mM e 30 mM trimetilammina in toluene (Vtotale = 5 mL) a temperatura ambiente durante la notte6,15,30.
- Sciacquare gli elettrodi con acqua deionizzata.
Nota: Per garantire una migliore rimozione delle non reagito acriloil residui di cloruro e trimetilammina, inoltre è possibile lavare la superficie con NaOH dopo acqua deionizzata. - Asciugare gli elettrodi con gas azoto.
- Sciacquare gli elettrodi con acqua deionizzata.
3. preparazione del modello impresso capacitive di oro
-
Preparazione del batteriofago impresso capacitive di oro
- Prima della polimerizzazione, preparare una soluzione di monomero contenente monomero (N-idrossimetil acrilammide) e cross-linker (polietilene glicole-400-dimetacrilato) in un rapporto di 1:5 (mol/mol) in 1 mL di tampone fosfato 10 mM (pH 7.4).
Nota: La soluzione di monomero contenente monomero e cross-linker può essere utilizzata in diversi rapporti, o i tipi del monomero e cross-linker possono essere cambiati secondo il modello per l'ottimizzazione dell'interazione specifico. - Aggiungere 1 mg di foto-iniziatore in questa soluzione.
Nota: Se la polimerizzazione avviene sotto luce UV, l'iniziatore di foto deve essere utilizzato per avviare la polimerizzazione. Se la polimerizzazione avviene mediante polimerizzazione radicalica, il tipo dell'iniziatore deve essere modificato. - Pipettare 1,5 µ l di questa soluzione sulla superficie dell'oro dell'elettrodo d'oro modificata.
Nota: La superficie dell'oro dell'elettrodo è rappresentata schematicamente nella Figura 1. - Portare il timbro di modello a contatto con la soluzione di monomero nella parte superiore la superficie dell'elettrodo d'oro.
- Avviare la polimerizzazione UV (365 nm, 400 W) e proseguire per 15 min31.
Nota: Polimerizzazione UV viene eseguita all'interno di un armadietto di raffreddamento che è impostato a-25 ° C prima dell'inizio della polimerizzazione. Quindi, il sistema di polimerizzazione UV è acceso e la polimerizzazione è continuata per 15 min prima di spegnere il sistema di polimerizzazione UV. - Rimuovere il timbro modello dalla superficie usando il forcipe.
Nota: Durante la rimozione del timbro modello dalla superficie, il film polimerico sulla superficie potrebbe essere danneggiato. Pertanto, il timbro deve essere rimosso dalla superficie molto attentamente e lentamente senza usare la forza. - Sciacquare la superficie dell'elettrodo con acqua deionizzata e asciugare con gas azoto.
- Immergere gli elettrodi in 1 mL di 10 mM 1-dodecanethiol preparato in etanolo per 20 min al fine di coprire i fori sulla superficie dell'elettrodo.
- Sciacquare gli elettrodi con acqua deionizzata e asciugare gli elettrodi con gas azoto.
- Prima della polimerizzazione, preparare una soluzione di monomero contenente monomero (N-idrossimetil acrilammide) e cross-linker (polietilene glicole-400-dimetacrilato) in un rapporto di 1:5 (mol/mol) in 1 mL di tampone fosfato 10 mM (pH 7.4).
-
Preparazione della proteina impresso capacitive di oro
- Prima della polimerizzazione, preparare una soluzione di monomero contenente monomeri (acrilammide: 54 mg; N-hydroxymethylacrylamide: 140 µ l; N-isopropylacrylamide: 85,6 mg) e cross-linker (methylenebisacrylamide: 9,5 mg) in 820 µ l di acqua ultra-pura30,32.
- Preparare 5% (v/v) N, N, N', N'-tetramethylethyldiamine (TEMED) in acqua ultra-pura.
- Aggiungere 20 µ l della soluzione TEMED nella soluzione di monomero e spurgo con azoto gassoso per 5 min.
- Preparare 10% (p/v) ammonio persolfato (APS) in acqua ultra-pura.
- Aggiungere 20 µ l della soluzione APS nella soluzione di monomero.
- Pipettare 1,5 µ l di soluzione di monomero sulla superficie dell'elettrodo d'oro modificate.
- Portare il timbro di modello a contatto con la superficie trattata monomero.
- Avviare la polimerizzazione a temperatura ambiente e continuare per 5 h.
Nota: Per la preparazione di elettrodi d'oro impresso proteina, invece di UV-polimerizzazione, polimerizzazione radicalica a temperatura ambiente (25 ° C) viene eseguita utilizzando APS-TEMED come il catalizzatore di iniziatore. - Rimuovere con attenzione il timbro modello dalla superficie usando il forcipe.
- Sciacquare l'elettrodo con acqua deionizzata e asciugare con gas azoto.
- Immergere gli elettrodi in 1 mL di 10 mM 1-dodecanethiol preparato in etanolo per 20 min al fine di coprire i fori sulla superficie dell'elettrodo.
- Sciacquare gli elettrodi con acqua deionizzata e asciugare gli elettrodi con gas azoto.
4. caratterizzazione della superficie dell'elettrodo con microscopia elettronica (SEM)
- Montare gli esemplari su supporti in alluminio con nastro adesivo carbonio.
- Rivestire gli elettrodi con 10 nm palladium/oro.
- Esaminare gli elettrodi con SEM
5. in tempo reale misurazioni capacitivi con modello impresso capacitive di oro
- Inserire gli elettrodi d'oro capacitivi impressi nella cella elettrochimica flusso integrata di un biosensore capacitivo.
- Preparare 100 mL di buffer di rigenerazione (25 mM glicina-HCl, pH 2.5, includendo 50mm Tween-20) e 1 L in esecuzione buffer (per batteriofago impresso sistema: fosfato 10 mM, pH 7.4; per sistema impresso proteina: 50 mM Tris-HCl, pH 7.4).
- Avviare l'analisi con l'iniezione del buffer di rigenerazione per rigenerare il sistema e in esecuzione buffer per riequilibrare il sistema per 25 min.
- Preparare soluzioni modello standard nella gamma di concentrazione desiderata in tampone di corsa.
Nota: in primo luogo preparare un brodo della soluzione del modello sciogliendo 0,1 mg di proteina, o circa 108 pfu batteriofagi, in 1,0 mL di tampone fosfato (fosfato 10 mM, pH 7.4). Quindi, preparare soluzioni standard per la curva di taratura facendo dieci diluizioni sequenziale 10 volte dalla soluzione di riserva. Queste soluzioni saranno analizzate con il biosensore capacitivo al punto 5.5. Concentrazione di proteine può essere misurata mediante uno spettrofotometro (280 nm). Al fine di misurare la concentrazione di batteriofago, può essere utilizzato un metodo di agar di doppio strato, che è descritto in precedenza Dettagli19. - Iniettare 250 µ l di queste soluzioni standard in sequenza per il sistema in condizioni ottimali (portata: 100 µ l/min, temperatura: 25 ° C).
Nota: In questa applicazione, le soluzioni standard della proteina sono state preparate nella gamma di concentrazione di 1,0 x 10-4 - 1,0 x 10-14, mentre le concentrazioni di batteriofago erano nella gamma 1.0 x 101 - 1.0 x 105 pfu/mL. Le soluzioni erano collocate nella valvola di iniezione e in modo sequenziale iniettate nel sistema, iniettando i campioni in triplici copie attraverso la pompa a siringa singola e la valvola multivie. La diminuzione della capacità dopo l'iniezione di soluzioni standard derivanti da un'associazione del modello per le cavità impresse è controllata automaticamente dal software dello strumento.
Risultati
Seguendo il protocollo, seguendo lo schema in Figura 1, nudo elettrodo d'oro sarà improntato con un modello, che rappresentano la struttura di un biomacromolecule. Questo elettrodo può essere applicato in un biosensore capacitivo (Figura 2), consentendo l'applicazione stabile di un modello sull'elettrodo e la misurazione delle variazioni della capacità all'associazione del modello.
Una rappresentazione schematica del biosensore capacitivo è illustrata nella Figura 2. La pompa centris, che è responsabile per iniezione continua del buffer in esecuzione (fosfato 10 mM, pH 7.4) e il buffer di rigenerazione (25 mM glicina-HCl, pH 2.5) durante la rigenerazione in cella di flusso, può essere visto chiaramente nella figura. La cella di flusso è costituito da lavoro, riferimento ed elettrodi contatore. La valvola di iniezione è composta da soluzioni standard della proteina/del batteriofago, che passano attraverso il degassificatore prima e poi iniettato in sequenza nel sistema. Appena le soluzioni raggiungono l'elettrodo di lavoro inserito nella cella di flusso, il risultato è monitorato in tempo reale. I valori di capacità possono essere registrati seguendo la sensorgrams sullo schermo del computer.
Figura 3 e Figura 4 raffigurano le differenze tra la superficie del nudo e impressi elettrodi d'oro. Questo passaggio di caratterizzazione è importante assicurare che ci sono cavità polimerici, visto come rugosità sulla superficie dell'elettrodo dopo imprinting. Oltre a SEM, ci sono anche altri metodi di caratterizzazione tra cui AFM, misure di angolo di contatto, ellissometria ecc., che può essere usato per caratterizzare la superficie dopo imprinting. In questo modo, è possibile garantire che il processo di imprinting è successo e che modello nelle cavità si formata sulla superficie. All'interno di queste cavità, il modello possibile associare con affinità e alta specificità.
Dopo l'iniezione delle soluzioni standard nel sistema capacitivo, una media delle ultime cinque letture è stata calcolata automaticamente dal software, e i grafici di calibrazione sono stati ottenuti tracciando il cambiamento nella capacità contro la concentrazione dei analita. La diminuzione della capacità registrati è sorto da un'associazione del modello. Le altre molecole che si legano all'elettrodo d'oro in superficie, maggiore la riduzione di capacità totale, secondo il principio generale di misurazione capacitivi. Figura 5 e Figura 6 indicano che con l'aumento della concentrazione dell'analita, la ΔC aumenta come previsto. La gamma dinamica (fra cui è l'intervallo di concentrazione dove il sistema è utile per l'individuazione di un target specifico) e il limite di rilevabilità (LOD) può essere valutate analizzando questi grafici. Secondo Figura 6, il biosensore capacitivo batteriofago improntato in grado di rilevare i batteriofagi nella gamma di concentrazione di 101 - 105 pfu/mL, con un valore LOD di pfu/mL 10 in questo studio. Si suppone che sia Figura 5 e Figura 6 anche evidenziare la necessità di misurare la curva di taratura nello stesso intervallo richiesto per la concentrazione di modello, poichè la linearità della regressione può variare sopra le concentrazioni ( Figura 5), o hanno diverse piste (Figura 6). Si deve inoltre osservare che, a causa delle basse concentrazioni utilizzate, il sistema è abbastanza sensibile alle fluttuazioni (torbidezza nel campione, progetto di aria, ecc.) e di conseguenza, si consiglia di eseguire almeno triplici copie per ridurre il potenziale di valori anomali, tra cui . Per lo stesso motivo, la deviazione standard può essere abbastanza significativa per campioni altamente diluiti, come si vede nella Figura 6.
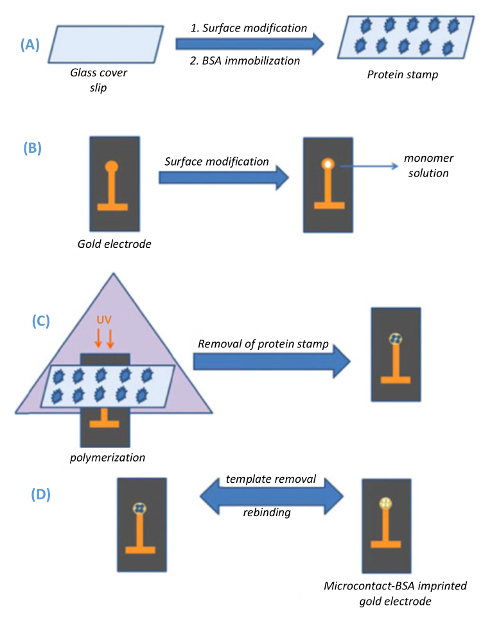
Figura 1 . Rappresentazione schematica di microcontatto imprinting metodo. (A) preparazione dei vetro vetrini (francobolli di modello), (B) preparazione degli elettrodi d'oro capacitivi, (C) microcontatto imprinting del modello sulla superficie dell'elettrodo d'oro tramite polimerizzazione UV e (D) rimozione del modello dalla superficie dell'elettrodo (riprodotto da Ertürk et al., biotecnologia rapporti 2014 (3): 65-72 con il permesso). Clicca qui per visualizzare una versione più grande di questa figura.
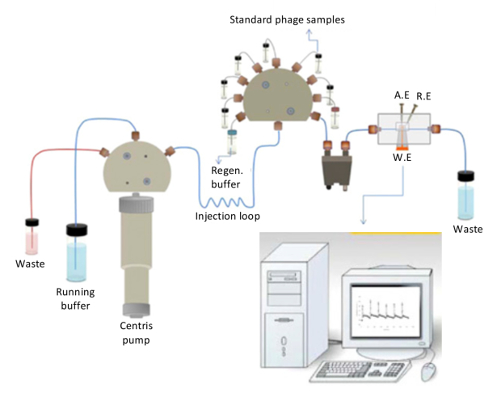
Nella figura 2. Rappresentazione schematica del biosensore capacitivo. Il layout generale del biosensore capacitivo utilizzato in questo studio (riprodotto da Ertürk et al., biotecnologia rapporti 2014 (3): 65-72 con il permesso). Clicca qui per visualizzare una versione più grande di questa figura.

Nella figura 3. Scansione microscopia elettronica della proteina impresso elettrodi. Immagini di SEM di (A) nudo elettrodo d'oro (barra della scala = 20 µm), e (B), una proteina impresso capacitiva di oro (barra della scala = 10 µm). Clicca qui per visualizzare una versione più grande di questa figura.

Nella figura 4. Scansione microscopia elettronica del batteriofago impresso elettrodi. Immagini di SEM di un elettrodo d'oro nudo (barra della scala = 20 µm) (A), e un batteriofago impresso capacitiva di oro in diversi livelli di ingrandimento (6600 x, barra della scala = 10 µm) (B) e 11, 500 X, scala bar = 5 µm) (C); le frecce indicano aderiti batteriofagi). Clicca qui per visualizzare una versione più grande di questa figura.
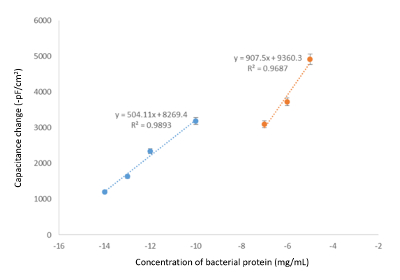
Nella figura 5. Effetto della composizione di buffer per grafici di taratura. Grafico di taratura mostrando il cambiamento nella capacità vs una concentrazione nella proteina in condizioni ottimali (Buffer di esecuzione: 50 mM Tris-HCl, pH 7.4; Buffer di rigenerazione: 25 mM glicina-HCl, pH 2.5 includendo 50mm Tween-20; Portata: 100 µ l/min, volume del campione: 250 µ l; T: 25 ° C). Clicca qui per visualizzare una versione più grande di questa figura.

Nella figura 6. Curva di calibrazione rappresentativo per grandi biomacromolecules. Grafico di taratura che mostra la variazione della capacità vs concentrazione batteriofago in condizioni ottimali (Running Buffer: 10 mM fosfato, pH 7.4; Buffer di rigenerazione: 25 mM glicina-HCl, pH 2.5 includendo 50mm Tween-20; Portata: 100 µ l/min, volume del campione: 250 µ l; T: 25 ° C) Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Quando questo metodo viene eseguito, ci sono alcuni passaggi critici che devono essere considerati mentre seguendo il protocollo. Un passaggio fondamentale è la fase di pulizia con soluzione acida piranha. Fase 2.1 non deve essere più di 10 min. La soluzione del modello per passo 1.4 non deve superare 0,1 mg/mL, dato che questi valori sono stati ottimizzati in precedenza. Voltametric ciclico scansioni non devono superare 15 cicli al fine di ottenere lo spessore ottimale. Per passo 3.1.3, 1,5 µ l è un valore ottimizzato. Questo valore non deve essere più alto per questo specifico tipo di elettrodo. Se il sistema di polimerizzazione UV ha una potenza di 400 W, la polimerizzazione deve essere eseguita per un massimo di 10-15 min. Non appena l'APS (iniziatore) viene aggiunto alla soluzione dopo TEMED, il passo successivo deve essere eseguito molto rapidamente per evitare immediata polimerizzazione (punto 3.2.5).
Una delle fasi più critiche è la rimozione del timbro modello dalla superficie dopo polimerizzazione UV. Se questo passaggio non viene eseguito correttamente, c'è il rischio che il film polimerico sulla superficie dell'elettrodo può essere rimosso con il timbro. Pertanto, si consiglia di immergere l'elettrodo e il timbro della proteina sulla parte superiore in una soluzione di acqua dopo la polimerizzazione, quindi rimuovere il timbro molto lentamente e con attenzione dalla superficie (punto 3.1.6).
In base al modello utilizzato, modifiche nel tipo e nel rapporto di monomeri usati (monomero funzionale e cross-linker) possono essere valutate in termini di generazione di sensibilità. Questo deve essere determinato empiricamente. Ulteriormente, il legame di affinità della molecola stampo solitamente coinvolge numerose interazioni differenti contemporaneamente. Di conseguenza, questo può portare a problemi durante la procedura di rigenerazione. Se il modello associato non viene rilasciato correttamente dalla superficie, questo può influenzare la possibilità di riutilizzo dell'elettrodo per ulteriori analisi. Queste affinità multipunto può anche derivare da associazione tramite interazioni più deboli. In tali sistemi, il legame non specifico può aver luogo, che possa influenzare negativamente la selettività del sistema16. Si tratta di comune e generale, limiti del metodo.
Oltre a queste limitazioni specifiche, ci sono molti vantaggi significativi del metodo discusso rispetto ai metodi esistenti. Mentre RIAs, Elisa e fluorometriche misurazioni sono molto sensibili, richiedono l'utilizzo di materiale con etichettato (modello o rivelatore), mentre il biosensore è completamente privo di etichetta. Questi metodi sono anche più costosi e che richiede tempo. Un approccio di biosensore consente sintesi rapida, parallela di MIPs in diverse composizioni al tempo stesso16. Poiché solo pochi microlitri di soluzione di monomero è necessaria per la preparazione, il metodo è conveniente quando si utilizza costosi o altrimenti limitati monomeri. Elettrodi di MIP più ulteriormente, singoli possono essere utilizzati per circa 80 analisi senza una significativa diminuzione delle prestazioni, che è significativamente superiore a altri metodi esistenti30. I metodi esistenti anche soffrono, in vari gradi, di bassa sensibilità e selettività, mentre il metodo descritto consente una rilevazione e quantificazione delle molecole nella gamma pM con elevata selettività.
A causa del rapporto costo-efficacia, la facilità di funzionamento dello strumento e la rilevazione in tempo reale e sensibile in tempi brevi rispetto ai metodi esistenti, i biosensori sono molto promettente punto di sistemi di rilevazione di cura in condizioni di campo; ad esempio, per il monitoraggio ambientale e per applicazioni nei paesi in via di sviluppo. In molte applicazioni all'interno di diagnosi della malattia, in tempo reale, sensibile, selettivo e rapido rilevamento di un biomarcatore in una miscela complessa come siero è richiesto15,25. Qui, i biosensori sono superiori a metodi esistenti, in particolare, grazie alla loro robustezza e sensibilità. In particolare per la rilevazione di agenti infettivi, batteriofagi recentemente sono considerati come elemento alternativo presta per biosensori a causa del loro ospite batteri specificità33,34,35. Sostituzione degli anticorpi con batteriofagi è molto promettente al fine di ridurre i costi e aumentare la stabilità anche altri36. Tale sistema permetterà anche per la rilevazione e la quantificazione dei fagi specifici nell'ambiente e da campioni clinici. A causa della prevalenza di batteriofagi e la loro capacità di trasdurre batteri con resistenza antibiotica geni37,38, tale metodo può essere utile studiare la diffusione di batteri resistenti.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Maria Baumgarten (piattaforma biotecnologia IQ, infezione medicina, Università di Lund) è riconosciuto per eseguire e fornire scansione micrografi elettronici. Questo lavoro è stato sostenuto da sovvenzioni da The Swedish Research Consiglio Formas (2017-00100) come parte dell'europeo terza programmazione iniziativa congiunta su chiamata resistenza antimicrobica (JPIAMR) "Dinamica di trasmissione". I finanziatori non avevano alcun ruolo nel disegno dello studio, interpretazione, scrittura, preparazione del manoscritto, decisione di inviare, o la decisione di pubblicare il lavoro.
Materiali
| Name | Company | Catalog Number | Comments |
| Glass Cover slips | ThermoFisher | 102222 | protein stamp |
| HCl | Sigma-Aldrich | H1758-500ML | cleaning |
| NaOH | Sigma-Aldrich | 72068-100ML | cleaning |
| Ultrasonic cleaner | Branson Ultrasonic | BRANSONIC M1800- E | cleaning |
| 3-amino-propyl-triethoxysilane (APTES) | Sigma-Aldrich | A3648-100ML | modification |
| EtOH | Sigma-Aldrich | 1009836010 | rinsing/cleaning |
| glutaraldehyde | Sigma-Aldrich | G5882-100ML | cross-linker |
| acetone | Sigma-Aldrich | 34850-1L-M | cleaning |
| H2SO4 | Sigma-Aldrich | 339741-100ML | piranha solution |
| H2O2 | Sigma-Aldrich | H1009-500ML | piranha solution |
| tyramine | Sigma-Aldrich | T90344-5G | modification |
| CompactStat | Ivium Technologies | CompactStat.h: 30mA@10V/3MHz | potentiostat |
| Platinum Counter Electrode Kit | Equilabrium | AFCTR5 | potentiostat |
| Reference Electrode | Equilabrium | RREF0021 | potentiostat |
| acryloyl chloride | EMD Millipore | 8.00826.0100 | modification |
| triethylamine | EMD Millipore | 8.08352.0100 | modification |
| toluene | Sigma-Aldrich | 244511-100ML | modification |
| N-hydroxymethyl acrylamide | Sigma-Aldrich | 245801-100G | functional monomer |
| poly ethylene glycol-400-dimethacrylate | Sigma-Aldrich | 409510-250ML | cross-linker |
| 2-Hydroxy-4′-(2-hydroxyethoxy)-2-methylpropiophenone | Sigma-Aldrich | 410896-50G | functional monomer |
| UV polymerizator | Dymax | Dymax 5000ECE | UV-polymerization |
| forceps | Sigma-Aldrich | Z168777-1EA | consumable |
| 1-dodecanethiol | Sigma-Aldrich | 471364-100ML | blocking agent |
| acrylamide | Sigma-Aldrich | A3553-100G | functional monomer |
| N-hydroxymethylacrylamide | Sigma-Aldrich | 245801-100G | functional monomer |
| N-isopropylacrylamide | Sigma-Aldrich | 415324-50G | functional monomer |
| methylenebisacrylamide | Sigma-Aldrich | 146072-500G | cross-linking monomer |
| N,N,N',N'-tetrametyhlethyldiamine (TEMED) | Sigma-Aldrich | T9281-25ML | catalyst |
| ammonium persulphate | Sigma-Aldrich | A3678-25G | initiator |
| Capacitive biosensor | CapSenze | Equipment | |
| Glycine | Merck | 1042011000 | regeneration buffer |
| Tween-20 | Sigma-Aldrich | P9416-50ML | regeneration buffer |
| Trizma base | Sigma-Aldrich | 93352-1KG | running buffer |
| Na2HPO4 • 2H2O | Calbiochem | 567547-1KG | running buffer |
| NaH2PO4 • 2H2O | Calbiochem | 567549-1KG | running buffer |
| DELPHI correlative light and electron microscope | Phenom-World | equipment | |
| Capacitive gold electrodes | CapSenze Biosystems | consumables | |
| 2,2'-azobis(2-methypropionitrile) | Sigma-Aldrich | 441090-25G | photo-initiator |
| CapSenze Smart Software | CapSenze Biosystems | software program |
Riferimenti
- Lin, T. Y., Hu, C. H., Chou, T. C. Determination of albumin concentration by MIP-QCM sensor. Biosensors and Bioelectronics. 20 (1), 75-81 (2004).
- Berggren, C., Bjarnason, B., Johansson, G. Capacitive Biosensors. Electroanalysis. 13 (3), 173-180 (2001).
- Zhang, S., Garcia-D'Angeli, A., Brennan, J. P., Huo, Q. Predicting detection limits of enzyme-linked immunosorbent assay (ELISA) and bioanalytical techniques in general. The Analyst. 139 (2), 439-445 (2014).
- Mattiasson, B., Teeparuksapun, K., Hedström, M. Immunochemical binding assays for detection and quantification of trace impurities in biotechnological production. Trends in Biotechnology. 28 (1), 20-27 (2010).
- Limbut, W., Hedström, M., Thavarungkul, P., Kanatharana, P., Mattiasson, B. Capacitive biosensor for detection of endotoxin. Analytical and Bioanalytical Chemistry. 389 (2), 517-525 (2007).
- Ertürk, G., Berillo, D., Hedström, M., Mattiasson, B. Microcontact-BSA imprinted capacitive biosensor for real-time, sensitive and selective detection of BSA. Biotechnology Reports. 3, 65-72 (2014).
- Arshady, R., Mosbach, K. Synthesis of substrate-selective polymers by host-guest polymerization. Macromolecular Chemistry and Physics. 182 (2), 687-692 (1981).
- Andersson, L. I. Molecular imprinting: developments and applications in the analytical chemistry field. Journal of chromatography. B, Biomedical sciences and applications. 745 (1), 3-13 (2000).
- Li, X., Husson, S. M. Two-dimensional molecular imprinting approach to produce optical biosensor recognition elements. Langmuir: the ACS Journal of Surfaces and Colloids. 22 (23), 9658-9663 (2006).
- Alexander, C., Davidson, L., Hayes, W. Imprinted polymers: artificial molecular recognition materials with applications in synthesis and catalysis. Tetrahedron. 59 (12), 2025-2057 (2003).
- Kryscio, D. R., Peppas, N. A. Surface imprinted thin polymer film systems with selective recognition for bovine serum albumin. Analytica Chimica Acta. 718, 109-115 (2012).
- Inerowicz, H. D., Howell, S., Regnier, F. E., Reifenberger, R. Multiprotein immunoassay arrays fabricated by microcontact printing. Langmuir. 18 (13), 5263-5268 (2002).
- Lin, H. Y., Hsu, C. Y., Thomas, J. L., Wang, S. E., Chen, H. C., Chou, T. C. The microcontact imprinting of proteins: The effect of cross-linking monomers for lysozyme, ribonuclease A and myoglobin. Biosensors and Bioelectronics. 22 (4), 534-543 (2006).
- Liao, P. C., Tyan, Y. C., Wang, C. Y., Hsu, J. F., Chou, T. C., Lin, H. Y. Assessing the binding selectivity of molecularly imprinted polymer artificial antibodies by mass spectrometry-based profiling system. Journal of Biomedical Materials Research Part A. 91 (2), 597-604 (2009).
- Ertürk, G., Hedström, M., Tümer, M. A., Denizli, A., Mattiasson, B. Real-time prostate-specific antigen detection with prostate-specific antigen imprinted capacitive biosensors. Analytica Chimica Acta. 891, 120-129 (2015).
- Ertürk, G., Mattiasson, B. From imprinting to microcontact imprinting-A new tool to increase selectivity in analytical devices. Journal of Chromatography. B, Analytical Technologies in the Biomedical and Life Sciences. 1021, 30-44 (2016).
- Ertürk, G., Mattiasson, B. Molecular Imprinting: The Creation of Biorecognition Imprints on Biosensor Surfaces. Advanced Molecularly Imprinting Materials. , 523-560 (2017).
- Janczuk-Richter, M., et al. Long-period fiber grating sensor for detection of viruses. Sensors and Actuators B: Chemical. 250, 32-38 (2017).
- Kropinski, A. M., Mazzocco, A., Waddell, T. E., Lingohr, E., Johnson, R. P. Enumeration of bacteriophages by double agar overlay plaque assay. Methods in Molecular Biology. 501, 69-76 (2009).
- Meillan, M., et al. Self-assembled monolayer for AFM measurements of Tobacco Mosaic Virus (TMV) at the atomic level. RSC Adv. 4 (23), 11927 (2014).
- Ymeti, A., et al. Fast, ultrasensitive virus detection using a Young interferometer sensor. Nano Letters. 7 (2), 394-397 (2007).
- Wei, Y., Wong, L. P., Toh, C. -. S. Fuel cell virus sensor using virus capture within antibody-coated nanochannels. Analytical Chemistry. 85 (3), 1350-1357 (2013).
- Guliy, O. I., et al. Immunodetection of bacteriophages by a piezoelectric resonator with lateral electric field. Applied biochemistry and microbiology. 52 (4), 457-463 (2016).
- Reta, N., Michelmore, A., Saint, C., Prieto-Simón, B., Voelcker, N. H. Porous silicon membrane-modified electrodes for label-free voltammetric detection of MS2 bacteriophage. Biosensors & Bioelectronics. 80, 47-53 (2016).
- Ertürk, G., Özen, H., Tümer, M. A., Mattiasson, B., Denizli, A. Microcontact imprinting based surface plasmon resonance (SPR) biosensor for real-time and ultrasensitive detection of prostate specific antigen (PSA) from clinical samples. Sensors and Actuators B: Chemical. 224, 823-832 (2016).
- Lebogang, L., Mattiasson, B., Hedström, M. Capacitive sensing of microcystin variants of Microcystis aeruginosa using a gold immunoelectrode modified with antibodies, gold nanoparticles and polytyramine. Microchimica Acta. 181 (9-10), 1009-1017 (2014).
- Loyprasert, S., Hedström, M., Thavarungkul, P., Kanatharana, P., Mattiasson, B. Sub-attomolar detection of cholera toxin using a label-free capacitive immunosensor. Biosensors & Bioelectronics. 25 (8), 1977-1983 (2010).
- Teeparuksapun, K., Hedström, M., Wong, E. Y., Tang, S., Hewlett, I. K., Mattiasson, B. Ultrasensitive detection of HIV-1 p24 antigen using nanofunctionalized surfaces in a capacitive immunosensor. Analytical Chemistry. 82 (20), 8406-8411 (2010).
- Labib, M., Hedström, M., Amin, M., Mattiasson, B. A capacitive biosensor for detection of staphylococcal enterotoxin B. Analytical and Bioanalytical Chemistry. 393 (5), 1539-1544 (2009).
- Ertürk, G., Hedström, M., Mattiasson, B. A sensitive and real-time assay of trypsin by using molecular imprinting-based capacitive biosensor. Biosensors & Bioelectronics. 86, 557-565 (2016).
- Ertürk, G., Uzun, L., Tümer, M. A., Say, R., Denizli, A. Fab fragments imprinted SPR biosensor for real-time human immunoglobulin G detection. Biosensors & Bioelectronics. 28 (1), 97-104 (2011).
- El-Sharif, H. F., Phan, Q. T., Reddy, S. M. Enhanced selectivity of hydrogel-based molecularly imprinted polymers (HydroMIPs) following buffer conditioning. Analytica Chimica Acta. 809, 155-161 (2014).
- Wang, F., et al. Detection of Salmonella Typhimurium on Spinach Using Phage-Based Magnetoelastic Biosensors. Sensors (Basel, Switzerland). 17 (2), (2017).
- Chin, B. A., et al. Rapid detection of small quantities of specific bacteria using phage-based wireless biosensors. 2016 10th International Conference on Sensing Technology (ICST). , 1-5 (2016).
- Park, M. -. K., Chin, B. A. Novel Approach of a Phage-Based Magnetoelastic Biosensor for the Detection of Salmonella enterica serovar Typhimurium in Soil. Journal of Microbiology and Biotechnology. 26 (12), 2051-2059 (2016).
- Mack, J. D., Yehualaeshet, T., Park, M. -. K., Tameru, B., Samuel, T., Chin, B. A. Phage-Based Biosensor and Optimization of Surface Blocking Agents to Detect Salmonella typhimurium on Romaine Lettuce. Journal of food safety. 37 (2), 12299 (2017).
- Colomer-Lluch, M., Jofre, J., Muniesa, M. Antibiotic resistance genes in the bacteriophage DNA fraction of environmental samples. Plos One. 6 (3), 17549 (2011).
- Lood, R., Ertürk, G., Mattiasson, B. Revisiting antibiotic resistance spreading in wastewater treatment plants - bacteriophages as a much neglected potential transmission vehicle. Frontiers in microbiology. 8, (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon