Method Article
Detecção ultra-sensível de biomarcadores usando um Imprinting Molecular baseado Biosensor capacitivo
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para a detecção e quantificação de baixas moléculas abundantes em soluções complexas usando molecular imprinting em combinação com um biossensor de capacitância.
Resumo
A capacidade de detectar e quantificar biomoléculas em soluções complexas sempre foi muito procurada dentro da ciência natural; sendo usado para a detecção de biomarcadores, contaminantes e outras moléculas de interesse. Uma técnica comumente utilizada para este fim é a enzima-lig da imunoabsorção ensaio (ELISA), onde muitas vezes um anticorpo é direcionado a uma molécula-alvo específico, e um segundo anticorpo marcado é utilizado para a detecção do anticorpo primário, permitindo a quantificação absoluta da biomolécula sob estudo. No entanto, o uso de anticorpos como elementos de reconhecimento limita a robustez do método; como faz a necessidade do uso de moléculas rotuladas. Para superar essas limitações, impressão molecular foi implementado, criação de sites de reconhecimento artificial complementar à molécula de modelo e obsoleting a necessidade do uso de anticorpos para ligação inicial. Além disso, para maior sensibilidade, o anticorpo secundário marcado pode ser substituído por biosensores depender a capacitância para a quantificação da molécula alvo. Neste protocolo, descrevemos um método para rapidamente e sem rótulo detectar e dosar baixo-abundantes biomoléculas (proteínas e vírus) em amostras complexas, com uma sensibilidade que é significativamente melhor do que os sistemas de deteção comumente usados, tais como o ELISA. Isto é tudo mediado por impressão molecular em combinação com um biossensor de capacitância.
Introdução
A quantificação das biomoléculas é usada em muitos campos diferentes de pesquisa dentro da ciência, empregando métodos como o radioimunoensaio (RIA) ou ELISA1. Alguns desses métodos requerem um rotulado reagente como um radioisótopo ou uma enzima chamada anticorpo/antigénio, o que os torna trabalhosa e demorada com procedimentos complexos2. Além disso, a robustez, seletividade e sensibilidade desses métodos não são suficientes para todas as análises; em particular não são suficientes quando quantidades attogram precisam ser analisado, ao invés de pictograma quantidades3. Para este efeito, biosensores ganharam considerável interesse4,5, em especial em combinação com impressão molecular para uma maior robustez.
Impressão molecular baseia-se na criação de cavidades, polimerização de monômeros funcionais em torno do modelo6, criação de sites de reconhecimento artificial que lembram perfeitamente o modelo7. Esta técnica tem sido usada para diversas aplicações, incluindo sistemas de entrega de droga e separação analítica, mas também como elementos biorecognition em biosensores8,9,10. No entanto, existem ainda algumas dificuldades na concepção de polímeros molecularmente impressos (MIPs) para modelos macromoleculares como proteínas e células de11,12. Devido a isto, muitos pesquisadores concentraram-se na impressão a proteína modelo diretamente sobre um substrato, criando assim uma superfície que será reconhecida pela proteína alvo12. Esta técnica de revestimento de superfície usada para empregar cavidades de reconhecimento por grandes moléculas e montagens, incluindo proteínas é chamada microcontact impressão13,14. O procedimento geral do método depende da polimerização entre duas superfícies - um selo de modelo e um suporte de polímero - sobre a qual o modelo é adsorvido em uma superfície de15,16e trouxe em contato com o superfície tratada com monômero. Por esta maneira, uma película fina de polímero é formada com o apoio através de UV-polimerização. Finalmente, o modelo é removido, deixando para trás a cavidades específicas do modelo na superfície do eletrodo impresso. Este método tem algumas vantagens, incluindo uma perda reduzida atividade da molécula imprimida, quantidades muito pequenas, bem como exigindo de moléculas modelo para a impressão processam16,17. Assim, estas superfícies cost-effective, estáveis, sensíveis e seletivas podem ser criados nas superfícies de sensor, visando a qualquer modelo de escolha do usuário.
O biosensor pode ser usado para a detecção de proteínas simples e macromoléculas muito maiores, incluindo vírus. Um grupo específico de vírus ganhando interesse recente é o bacteriófago, que é um vírus que infecta bactérias. Detecção rápida e sensível de bacteriófagos é importante durante biotecnológicas e biofarmacêutica processos a fim de determinar as infecções de culturas bacterianas com bacteriófagos18. O ensaio biológico mais comumente usado para a deteção do bacteriófago é a dupla camada agar método19, que é trabalhoso e demorado. Várias tentativas foram feitas para desenvolver novas ferramentas de diagnóstico para vírus (incluindo bacteriófagos) como força atômica (AFM) a microscopia20, interferometria21, eletroquímica22e sensor sistema23, 24. um monte de trabalho tem sido focado em biosensores devido a suas vantagens como sendo capaz de medição em tempo real,15,25, altamente sensível e fácil de operar. Um tipo específico de biosensor baseia-se em mudanças na capacitância. Estes biosensores capacitivos são os sensores eletroquímicos que medem mudanças nas propriedades dielétricas quando um analito interage com um elemento de biorecognition na superfície do sensor, causando uma diminuição da capacitância2,4 . Biosensores capacitivos têm sido utilizados para a detecção de vários analitos como antígenos, anticorpos, proteínas e heavy metal íons6,26,,27,28. Estes tipos de biosensores têm muitas vantagens como inerente rapidez, alta sensibilidade, simplicidade, baixo custo, fácil manipulação e medição em tempo real sem rotulagem29.
O método descrito neste documento destina-se a permitir a detecção e quantificação de baixo-abundantes biomoléculas em amostras altamente complexas, sem a necessidade de usar qualquer rotulagem. Em particular, a técnica é mais útil na faixa de atto-picograma de biomoléculas, onde outros instrumentos comercialmente existentes não conseguem dosar com precisão seu alvo.
Protocolo
1. modificação de lamínulas de vidro (modelo selos)
- Para limpar as lamínulas de vidro, mergulhe-os sequencialmente em 10 mL de HCl 1,0 M, água deionizada e 1,0 M de NaOH, respectivamente, por 10 min em cada etapa em um líquido de limpeza ultra-sônico à temperatura ambiente.
- Seque as lamínulas de vidro com gás nitrogênio.
Nota: As lamínulas são secas por evaporação sob um fluxo de nitrogênio gasoso.
- Seque as lamínulas de vidro com gás nitrogênio.
- Mergulhe as lamínulas limpas e secas em 10% (v/v), 10 mL da solução de 3-amino-propil-triethoxysilane (APTES) em etanol por 1h introduzir grupos aminoácidos na superfície de vidro, à temperatura ambiente.
- Lave as lamínulas com água desionizada.
- Seque as lamínulas com gás nitrogênio.
- Mergulhe as lamínulas APTES-modificado em 5% (v/v), 10 mL da solução de glutaraldeído em 10 mM de tampão fosfato (pH 7,4) por 2 h a fim de ativar os grupos aminoácidos na superfície, a temperatura ambiente6,15.
- Lave as lamínulas com 10 mM de tampão fosfato (pH 7,4) a fim de remover o excesso glutaraldeído da superfície.
- Seque as lamínulas com gás nitrogênio.
- Prepare 1,0 mL de solução de modelo (proteína/bacteriófago) em 10 mM de tampão fosfato (pH 7,4) na concentração de 0,1 mg/mL.
Nota: Dissolva 0,1 mg de proteína em 1,0 mL de tampão fosfato (10 mM, pH 7,4). Se necessário, um espectrofotômetro (280 nm) pode ser usado para determinar a concentração de proteína.- Largue a 200 µ l desta solução de modelo para as lamínulas modificadas e incubar a 4 ° C durante a noite.
Nota: A solução do modelo em excesso pode ser usada para preparar 1-2 mais selos do modelo a ser usado nos estudos de caracterização. - Lave as lamínulas com 10 mM de tampão fosfato (pH 7,4) a fim de remover o modelo desvinculado da superfície.
Nota: Para enxaguar os eléctrodos, lave-os com tampão fosfato (10 mM, pH 7,4) por 30 s. - Seque as lamínulas com gás nitrogênio.
Nota: As lamínulas devem ser armazenadas a 4 ° C até que eles são usados na etapa de polimerização.
- Largue a 200 µ l desta solução de modelo para as lamínulas modificadas e incubar a 4 ° C durante a noite.
2. modificação de eletrodos de ouro capacitivos
- Para limpar os eletrodos, mergulhar os eléctrodos num copo pequeno, sequencialmente, contendo 5 mL de etanol (70%), água deionizada água, acetona, água desionizada, solução ácida piranha (3:1, H2SO4: H2O2, v/v) e deionizada água, respectivamente, para 10 min em cada etapa, em um líquido de limpeza ultra-sônico à temperatura ambiente.
- Seque os eléctrodos com gás nitrogênio.
- Para executar o electropolymerization de tiramina, prepare-se 8 mL de solução de tiramina de 10 mM em 10 mM fosfato tampão (pH 7,4) contendo etanol (2 mL)15.
Nota: O volume total da solução de tiramina deve ser 8 mL no total incluindo 2 mL de etanol.- Executar varreduras voltamétricos cíclicas (15 ciclos) nesta solução usando um potentiostat cobrindo um potencial intervalo de 0 - 1.5 V (Ag/AgCl) e uma taxa de varredura de 50 mV/s30.
- Lave os eletrodos com água desionizada.
- Seque os eléctrodos com gás nitrogênio.
- Mergulhar os eléctrodos em uma solução contendo cloreto de acryloyl de 30 mM e 30 mM trimetilamina em tolueno (Vtotal = 5 mL) à temperatura ambiente durante a noite,6,15,30.
- Lave os eletrodos com água desionizada.
Nota: Para garantir uma melhor remoção de acryloyl não tenha reagido resíduos cloreto e trimetilamina, também é possível lavar a superfície com NaOH após água desionizada. - Seque os eléctrodos com gás nitrogênio.
- Lave os eletrodos com água desionizada.
3. preparação do modelo de impresso capacitivos eletrodos de ouro
-
Preparação do bacteriófago impresso capacitivos eletrodos de ouro
- Antes da polimerização, prepare uma solução de monómero que contém monômero (N-hidroximetil acrilamida) e cross-linker (polietileno glicol-400-Dimetacrilato) na proporção de 1:5 em 1 mL de 10 mM de tampão fosfato (pH 7,4) (mol/mol).
Nota: A solução de monómero que contém monômero e cross-linker pode ser usada em diferentes proporções, ou os tipos do monômero e cross-linker podem ser alterados de acordo com o modelo para otimização da interação específica. - Adicione 1 mg de foto-iniciador para esta solução.
Nota: Se a polimerização é realizada sob a luz UV, então o foto-iniciador deve ser usado para iniciar a polimerização. Se a polimerização é realizada pela polimerização do radical livre, o tipo de iniciador deve ser alterado. - Pipete 1,5 µ l desta solução para a superfície de ouro do eletrodo ouro modificado.
Nota: A superfície de ouro do eléctrodo é mostrada esquematicamente na Figura 1. - Trazer o carimbo modelo em contacto com a solução de monômero na parte superior da superfície do eletrodo de ouro.
- Iniciar a polimerização UV (365 nm, 400 W) e continue por 15 min31.
Nota: A polimerização UV é executada dentro de um armário de refrigeração, que é definido para-25 ° C antes de iniciar a polimerização. Então, a sistema de cura a luz UV é ativada e a polimerização é continuada por 15 min antes de desligar a sistema de fotopolimerização de luz UV. - Remova o selo de modelo da superfície com o uso de fórceps.
Nota: Ao remover o selo de modelo da superfície, a película polimérica na superfície pode ser danificada. Portanto, o carimbo deve ser removido da superfície com muito cuidado e lentamente sem usar força. - Enxaguar a superfície do eletrodo com água deionizada e seque-o com gás nitrogênio.
- Mergulhe os eléctrodos em 1 mL de 10 mM 1-dodecanethiol preparado em etanol por 20 min para cobrir furos na superfície do eletrodo.
- Lave os eletrodos com água desionizada e seque os eléctrodos com gás nitrogênio.
- Antes da polimerização, prepare uma solução de monómero que contém monômero (N-hidroximetil acrilamida) e cross-linker (polietileno glicol-400-Dimetacrilato) na proporção de 1:5 em 1 mL de 10 mM de tampão fosfato (pH 7,4) (mol/mol).
-
Preparação de proteína impresso capacitivos eletrodos de ouro
- Antes da polimerização, preparar uma solução de monômero contendo monômeros (acrilamida: 54 mg; N-hydroxymethylacrylamide: 140 µ l; N-isopropylacrylamide: mg 85,6) e cross-linker (methylenebisacrylamide: 9,5 mg) em 820 µ l de água ultra pura30,32.
- Prepare-se 5% (v/v) N, N, N', N'-tetramethylethyldiamine (TEMED) em água ultra pura.
- Adicionar 20 µ l da solução TEMED para a solução de monômero e purga com gás de nitrogênio por 5 min.
- Prepare-se 10% (p/v) persulfato de amónio (APS) em água ultra pura.
- Adicione 20 µ l da solução de APS para a solução de monômero.
- Pipete 1,5 µ l da solução de monômero para a superfície do eletrodo de ouro modificada.
- Trazer o carimbo modelo em contacto com a superfície tratada com monômero.
- Iniciar a polimerização à temperatura ambiente e continue por 5 h.
Nota: Para a preparação de proteína-impresso eletrodos de ouro, em vez de UV-polimerização, radicais livres polimerização à temperatura ambiente (25 ° C) é executada usando APS-TEMED como iniciador-catalisador. - Remova o selo de modelo da superfície cuidadosamente usando fórceps.
- Lave o eletrodo com água deionizada e seque com gás nitrogênio.
- Mergulhe os eléctrodos em 1 mL de 10 mM 1-dodecanethiol preparado em etanol por 20 min para cobrir furos na superfície do eletrodo.
- Lave os eletrodos com água desionizada e seque os eléctrodos com gás nitrogênio.
4. Caracterização da superfície do eletrodo com microscopia eletrônica (SEM)
- Monte os espécimes nos suportes de alumínio com fita adesiva de carbono.
- Revesti os eléctrodos com 10 nm de paládio/ouro.
- Examinar os eléctrodos com SEM.
5. medições capacitivas em tempo real com modelo impresso capacitivos eletrodos de ouro
- Inserir os eletrodos de ouro capacitivos imprimidos a célula eletroquímica fluxo integrada para um biossensor capacitivo.
- Preparar 100 mL de buffers de regeneração (25mm glicina-HCl, pH 2.5, incluindo 50mm Tween-20) e execução 1 L (para bacteriófago impresso sistema: fosfato 10 mM, pH 7,4; para o sistema de proteína-impresso: 50 mM Tris-HCl, pH 7,4).
- Inicie a análise com a injeção de buffer de regeneração para regenerar o sistema e execução buffer para re-equilibrar o sistema por 25 min.
- Prepare soluções de modelo padrão na faixa de concentração desejada em reserva de marcha.
Nota: Primeiro prepare um estoque da solução modelo dissolvendo 0,1 mg de proteína, ou aproximadamente 108 pfu bacteriófagos, em 1,0 mL de tampão de fosfato (fosfato 10 mM, pH 7,4). Então, prepare-se das soluções-padrão para a curva de calibração, fazendo dez diluições de 10 vezes sequenciais de solução-mãe. Essas soluções serão analisadas com o biosensor capacitivo na etapa 5.5. Concentração de proteína pode ser medida usando um espectrofotômetro (280 nm). Para medir a concentração do bacteriófago, pode ser usado um método de ágar de dupla camada, que é descrito em detalhes anteriormente19. - Injectar 250 µ l destas soluções padrão sequencialmente para o sistema em ótimas condições (taxa de fluxo: 100 µ l/min, temperatura: 25 ° C).
Nota: Nesta aplicação, as soluções de proteína padrão foram preparadas na faixa de concentração de 1,0 x 10-4 - 1,0 x 10-14, enquanto as concentrações de bacteriófago estavam no intervalo de 1.0 x 101 - 1,0 x 105 pfu/mL. As soluções foram colocadas na válvula de injeção e sequencialmente injetadas no sistema, injetando as amostras em triplica através da bomba de seringa única e a válvula multiporta. A diminuição da capacidade após injetar as soluções padrão decorrentes da vinculação do modelo das cavidades impressa é monitorada automaticamente pelo software do instrumento.
Resultados
Seguindo o protocolo, de acordo com o esquema na Figura 1, um eletrodo de ouro nua será gravado com um modelo, que representa a estrutura de um biomacromolecule. Este eletrodo pode ser aplicado em um biossensor capacitivo (Figura 2), permitindo que o aplicativo estável de um modelo para o eletrodo e a medição de mudanças na capacitância mediante vinculação do modelo.
Uma representação esquemática do biosensor capacitivo é mostrada na Figura 2. A bomba de centris, que é responsável pela injeção contínua de buffer de execução (fosfato 10 mM, pH 7,4) e o buffer de regeneração (25mm glicina-HCl, pH 2.5) durante a regeneração dentro da célula de fluxo, pode ser vista claramente na figura. A célula de fluxo consiste em trabalhar, referência e eletrodos de contador. A válvula de injeção é composta de soluções padrão de proteína/bacteriófago, que estão de passagem o desgaseificador primeiro e então injetado sequencialmente no sistema. Tão logo as soluções atingem o eletrodo de trabalho inserido na célula de fluxo, o resultado é monitorado em tempo real. Os valores de capacitância podem ser registrados, seguindo o sensorgrams na tela do computador.
Figura 3 e Figura 4 mostram as diferenças entre a superfície dos eléctrodos de ouro desencapados e imprimidas. Esta etapa de caracterização é importante para garantir que existem cavidades poliméricas, vistas como rugosidade na superfície do eléctrodo após a programação. Além de SEM, existem também outros métodos de caracterização incluindo AFM, medições de ângulo de contato, etc., que podem ser usados para caracterizar a superfície após a programação de elipsometria. Desta forma, pode ser assegurado que o processo de impressão é bem sucedido e que as cavidades modelo são formadas na superfície. Dentro dessas cavidades, o modelo pode vincular com afinidade e alta especificidade.
Depois de injectar das soluções-padrão do sistema capacitivo, uma média das cinco últimas leituras foi calculada automaticamente pelo software, e os gráficos de calibração foram obtidos plotando a mudança na capacitância vs. a concentração do analito. A diminuição da capacidade registrada surgiu a ligação do modelo. As mais moléculas que se ligam ao eléctrodo de ouro de superfície, maior a redução da capacidade total, de acordo com o princípio geral de medição capacitiva. Figura 5 e Figura 6 mostram que com o aumento da concentração do analito, a ΔC aumenta conforme o esperado. A gama dinâmica (entre os quais é o intervalo de concentração onde o sistema é útil para a detecção de um alvo específico) e o limite de detecção (LOD) podem ser avaliadas através da análise desses gráficos. De acordo com a Figura 6, o biosensor capacitivo do bacteriófago imprimido pode detectar bacteriófagos na faixa de concentração de 101 - 105 pfu/mL, com um valor LOD de pfu/mL 10 neste estudo. Tanto a Figura 5 e Figura 6 também destacar a necessidade de medir a curva de calibração no mesmo intervalo necessário para a concentração de modelo deve, desde que a linearidade da regressão pode variar mais as concentrações ( Figura 5), ou ter inclinações diferentes (Figura 6). Também note que devido as baixas concentrações utilizadas, o sistema é bastante sensível às flutuações (turbidez da amostra, projecto de ar, etc.), e portanto, é recomendável executar pelo menos triplica para reduzir o potencial de outliers, incluindo . Pela mesma razão, o desvio-padrão pode ser bastante significativo para amostras altamente diluídas, como visto na Figura 6.
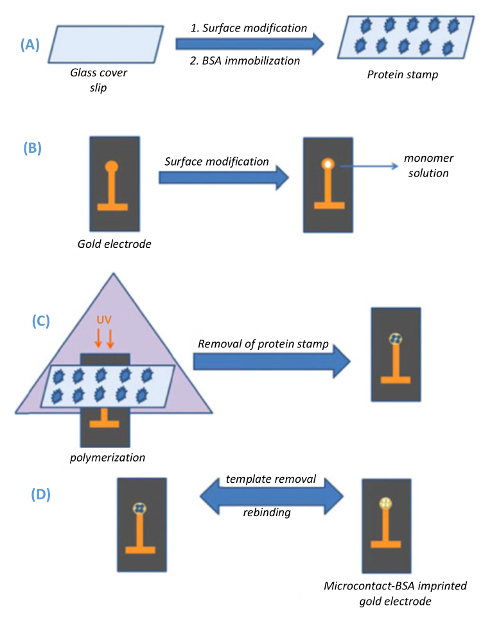
Figura 1 . Representação esquemática do método de impressão microcontact. (A) preparação de lamínulas de vidro (selos de modelo), (B) preparação dos eléctrodos de ouro capacitivos, (C) microcontact impressão do modelo para a superfície do eletrodo de ouro através de UV-polimerização e (D) remoção do modelo da superfície do eletrodo (reproduzido do Ertürk et al, biotecnologia relatórios 2014 (3): 65-72, com permissão). Clique aqui para ver uma versão maior desta figura.
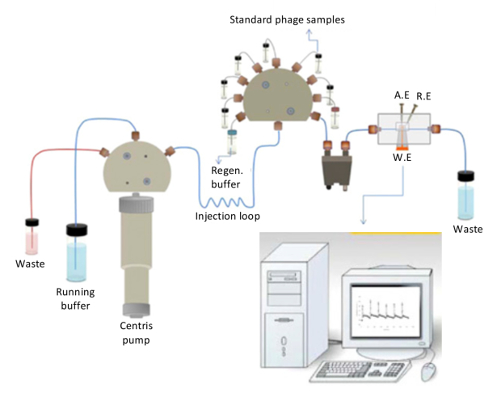
Figura 2. Representação esquemática do biosensor capacitiva. O layout geral do biosensor capacitivo utilizado neste estudo (reproduzido do Ertürk et al, biotecnologia relatórios 2014 (3): 65-72, com permissão). Clique aqui para ver uma versão maior desta figura.

Figura 3. Microscopia eletrônica de varredura de proteína impresso eletrodos. Imagens SEM do (A), um eletrodo de ouro desencapado (barra de escala = 20 µm), e (B), uma proteína impresso eletrodo capacitivo de ouro (barra de escala = 10 µm). Clique aqui para ver uma versão maior desta figura.

Figura 4. Microscopia eletrônica de varredura do bacteriófago impresso eletrodos. Imagens SEM de um eletrodo de ouro desencapado (barra de escala = 20 µm) (A), e um bacteriófago impresso capacitivo eletrodo de ouro em diferentes ampliações (6600 x, barra de escala = 10 µm) (B) e 11, 500 X, escala bar = 5 µm) (C); as setas denotam aderidos bacteriófagos). Clique aqui para ver uma versão maior desta figura.
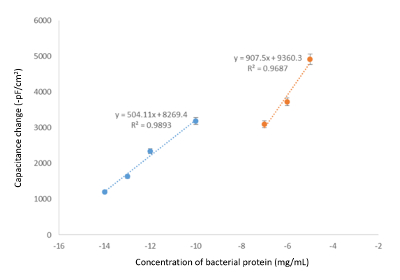
Figura 5. Efeito de composição do tampão para gráficos de calibração. Gráfico de calibração mostrando a mudança na capacitância vs uma concentração de proteína em óptimas condições (executando Buffer: 50 mM Tris-HCl, pH 7,4; Buffer de regeneração: 25mm glicina-HCl, pH 2.5 incluindo 50mm Tween-20; Taxa de fluxo: 100 µ l/min, volume de amostra: 250 µ l; T: 25 ° C). Clique aqui para ver uma versão maior desta figura.

Figura 6. Curva de calibração representante para grandes macromoléculas. Gráfico de calibração que mostra a mudança na capacitância vs concentração do bacteriófago em óptimas condições (executando Buffer: fosfato 10 mM, pH 7,4; Buffer de regeneração: 25mm glicina-HCl, pH 2.5 incluindo 50mm Tween-20; Taxa de fluxo: 100 µ l/min, volume de amostra: 250 µ l; T: 25 ° C) clique aqui para ver uma versão maior desta figura.
Discussão
Quando este método é realizado, existem algumas etapas essenciais que devem ser consideradas ao seguir o protocolo. Um passo crítico é a etapa de limpeza com solução ácida de piranha. Passo 2.1 não deve ser mais de 10 min. A solução de modelo para a etapa 1.4 não deve exceder 0,1 mg/mL, desde que estes valores foram previamente otimizados. Exames titulimétricos cíclica não devem exceder 15 ciclos a fim de obter a espessura ideal. Para etapa 3.1.3, 1,5 µ l é um valor otimizado. Este valor não deve ser mais elevado para este tipo específico de eletrodo. Se o sistema de cura UV tem uma potência de 400 W, a polimerização deve ser executada por um período máximo de 10-15 min. Tão logo o APS (iniciador) é adicionado à solução após TEMED, a etapa subsequente deve ser executada muito rapidamente para evitar a polimerização imediata (passo 3.2.5).
Um dos passos mais importantes é a remoção do carimbo modelo da superfície após a polimerização UV. Se esta etapa não é realizada corretamente, há um risco de que o filme polimérico na superfície do eléctrodo pode ser removido com o carimbo. Portanto, é recomendável para mergulhar o eléctrodo e o carimbo de proteína no topo de uma solução de água após a polimerização e, em seguida, retire o selo muito lentamente e com cuidado a superfície (etapa 3.1.6).
Baseado no modelo utilizado, modificações no tipo e proporção de monômeros usados (monómero funcional e cross-linker) poderão ser avaliadas em termos de geração de maior sensibilidade. Isso deve ser determinado empiricamente. Além disso, a ligação de afinidade da molécula modelo geralmente envolve várias interações diferentes simultaneamente. Portanto, isso pode levar a problemas durante as etapas de regeneração. Se o modelo vinculado não é liberado da superfície corretamente, isso pode influenciar a capacidade de reutilização do eletrodo para posterior análise. Essas afinidades multipontos também podem resultar de ligação através de interações fracas. Em tais sistemas, inespecificas podem ter lugar, que pode influenciar negativamente o a seletividade do sistema16. Estas são comuns e gerais, limitações do método.
Além dessas limitações específicas, existem muitas vantagens significativas do método discutido sobre os métodos existentes. Enquanto RIAs, Elisa e medições fluorométrica são muito sensíveis, eles exigem o uso de material rotulado (modelo ou detector), enquanto o biosensor é completamente rótulo livre. Esses métodos também são mais caros e demorados. Uma abordagem de biosensor permite a síntese rápida, paralelo de MIPs em composições diferentes, o mesmo tempo16. Uma vez que apenas alguns microlitros de solução de monômero é necessária para a preparação, o método é conveniente quando usando caros ou não limitado de monômeros. Além disso, o único eletrodos MIP podem ser usados para aproximadamente 80 análises sem uma diminuição significativa no desempenho, que é significativamente maior do que outros de métodos existentes30. Os métodos existentes também sofrem, em graus variáveis, de baixa sensibilidade e seletividade, enquanto o método descrito permite para detecção e quantificação de moléculas na faixa de pM com alta seletividade.
Devido a relação custo-eficácia, a facilidade de operação do instrumento e a detecção sensível em tempo real em um curto período de tempo em comparação com os métodos existentes, os biosensores são muito promissor ponto-de sistemas de deteção de cuidados sob condições de campo; por exemplo, para monitoramento ambiental e para aplicações em países em desenvolvimento. Em muitas aplicações no diagnóstico da doença, detecção em tempo real, sensível, seletiva e rápida de um biomarcador em uma mistura complexa como o soro é necessário15,25. Aqui, biosensores são superiores aos métodos existentes, em particular, devido à sua sensibilidade e robustez. Especificamente para a detecção de agentes infecciosos, bacteriófagos são recentemente considerados como elemento de biorecognition alternativos para biossensores devido à sua especificidade anfitrião de bactérias33,34,35. Substituição de anticorpos com bacteriófagos é muito promissor, a fim de reduzir o custo e aumentar a estabilidade ainda mais36. Esse sistema também permitirá a detecção e quantificação do fago específico no ambiente e de amostras clínicas. Devido a prevalência de bacteriófagos e sua capacidade de transduce bactérias com genes de resistência aos antibióticos37,38, tal método pode ser valioso para estudar a propagação de bactérias resistentes.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Maria Baumgarten (plataforma de biotecnologia IQ, infecção de medicina, Universidade de Lund) é reconhecido pela execução e fornecer varredura micrografias de elétron. Este trabalho foi apoiado por subsídios de pesquisa Sueco The Conselho Formas (2017-00100) como parte da terceira comum programação iniciativa europeia na chamada de resistência antimicrobiana (JPIAMR) "Dinâmica de transmissão." Os financiadores não tinham qualquer papel no projeto de estudo, interpretação, escrita, preparação do manuscrito, decisão de submeter ou a decisão de publicar o trabalho.
Materiais
| Name | Company | Catalog Number | Comments |
| Glass Cover slips | ThermoFisher | 102222 | protein stamp |
| HCl | Sigma-Aldrich | H1758-500ML | cleaning |
| NaOH | Sigma-Aldrich | 72068-100ML | cleaning |
| Ultrasonic cleaner | Branson Ultrasonic | BRANSONIC M1800- E | cleaning |
| 3-amino-propyl-triethoxysilane (APTES) | Sigma-Aldrich | A3648-100ML | modification |
| EtOH | Sigma-Aldrich | 1009836010 | rinsing/cleaning |
| glutaraldehyde | Sigma-Aldrich | G5882-100ML | cross-linker |
| acetone | Sigma-Aldrich | 34850-1L-M | cleaning |
| H2SO4 | Sigma-Aldrich | 339741-100ML | piranha solution |
| H2O2 | Sigma-Aldrich | H1009-500ML | piranha solution |
| tyramine | Sigma-Aldrich | T90344-5G | modification |
| CompactStat | Ivium Technologies | CompactStat.h: 30mA@10V/3MHz | potentiostat |
| Platinum Counter Electrode Kit | Equilabrium | AFCTR5 | potentiostat |
| Reference Electrode | Equilabrium | RREF0021 | potentiostat |
| acryloyl chloride | EMD Millipore | 8.00826.0100 | modification |
| triethylamine | EMD Millipore | 8.08352.0100 | modification |
| toluene | Sigma-Aldrich | 244511-100ML | modification |
| N-hydroxymethyl acrylamide | Sigma-Aldrich | 245801-100G | functional monomer |
| poly ethylene glycol-400-dimethacrylate | Sigma-Aldrich | 409510-250ML | cross-linker |
| 2-Hydroxy-4′-(2-hydroxyethoxy)-2-methylpropiophenone | Sigma-Aldrich | 410896-50G | functional monomer |
| UV polymerizator | Dymax | Dymax 5000ECE | UV-polymerization |
| forceps | Sigma-Aldrich | Z168777-1EA | consumable |
| 1-dodecanethiol | Sigma-Aldrich | 471364-100ML | blocking agent |
| acrylamide | Sigma-Aldrich | A3553-100G | functional monomer |
| N-hydroxymethylacrylamide | Sigma-Aldrich | 245801-100G | functional monomer |
| N-isopropylacrylamide | Sigma-Aldrich | 415324-50G | functional monomer |
| methylenebisacrylamide | Sigma-Aldrich | 146072-500G | cross-linking monomer |
| N,N,N',N'-tetrametyhlethyldiamine (TEMED) | Sigma-Aldrich | T9281-25ML | catalyst |
| ammonium persulphate | Sigma-Aldrich | A3678-25G | initiator |
| Capacitive biosensor | CapSenze | Equipment | |
| Glycine | Merck | 1042011000 | regeneration buffer |
| Tween-20 | Sigma-Aldrich | P9416-50ML | regeneration buffer |
| Trizma base | Sigma-Aldrich | 93352-1KG | running buffer |
| Na2HPO4 • 2H2O | Calbiochem | 567547-1KG | running buffer |
| NaH2PO4 • 2H2O | Calbiochem | 567549-1KG | running buffer |
| DELPHI correlative light and electron microscope | Phenom-World | equipment | |
| Capacitive gold electrodes | CapSenze Biosystems | consumables | |
| 2,2'-azobis(2-methypropionitrile) | Sigma-Aldrich | 441090-25G | photo-initiator |
| CapSenze Smart Software | CapSenze Biosystems | software program |
Referências
- Lin, T. Y., Hu, C. H., Chou, T. C. Determination of albumin concentration by MIP-QCM sensor. Biosensors and Bioelectronics. 20 (1), 75-81 (2004).
- Berggren, C., Bjarnason, B., Johansson, G. Capacitive Biosensors. Electroanalysis. 13 (3), 173-180 (2001).
- Zhang, S., Garcia-D'Angeli, A., Brennan, J. P., Huo, Q. Predicting detection limits of enzyme-linked immunosorbent assay (ELISA) and bioanalytical techniques in general. The Analyst. 139 (2), 439-445 (2014).
- Mattiasson, B., Teeparuksapun, K., Hedström, M. Immunochemical binding assays for detection and quantification of trace impurities in biotechnological production. Trends in Biotechnology. 28 (1), 20-27 (2010).
- Limbut, W., Hedström, M., Thavarungkul, P., Kanatharana, P., Mattiasson, B. Capacitive biosensor for detection of endotoxin. Analytical and Bioanalytical Chemistry. 389 (2), 517-525 (2007).
- Ertürk, G., Berillo, D., Hedström, M., Mattiasson, B. Microcontact-BSA imprinted capacitive biosensor for real-time, sensitive and selective detection of BSA. Biotechnology Reports. 3, 65-72 (2014).
- Arshady, R., Mosbach, K. Synthesis of substrate-selective polymers by host-guest polymerization. Macromolecular Chemistry and Physics. 182 (2), 687-692 (1981).
- Andersson, L. I. Molecular imprinting: developments and applications in the analytical chemistry field. Journal of chromatography. B, Biomedical sciences and applications. 745 (1), 3-13 (2000).
- Li, X., Husson, S. M. Two-dimensional molecular imprinting approach to produce optical biosensor recognition elements. Langmuir: the ACS Journal of Surfaces and Colloids. 22 (23), 9658-9663 (2006).
- Alexander, C., Davidson, L., Hayes, W. Imprinted polymers: artificial molecular recognition materials with applications in synthesis and catalysis. Tetrahedron. 59 (12), 2025-2057 (2003).
- Kryscio, D. R., Peppas, N. A. Surface imprinted thin polymer film systems with selective recognition for bovine serum albumin. Analytica Chimica Acta. 718, 109-115 (2012).
- Inerowicz, H. D., Howell, S., Regnier, F. E., Reifenberger, R. Multiprotein immunoassay arrays fabricated by microcontact printing. Langmuir. 18 (13), 5263-5268 (2002).
- Lin, H. Y., Hsu, C. Y., Thomas, J. L., Wang, S. E., Chen, H. C., Chou, T. C. The microcontact imprinting of proteins: The effect of cross-linking monomers for lysozyme, ribonuclease A and myoglobin. Biosensors and Bioelectronics. 22 (4), 534-543 (2006).
- Liao, P. C., Tyan, Y. C., Wang, C. Y., Hsu, J. F., Chou, T. C., Lin, H. Y. Assessing the binding selectivity of molecularly imprinted polymer artificial antibodies by mass spectrometry-based profiling system. Journal of Biomedical Materials Research Part A. 91 (2), 597-604 (2009).
- Ertürk, G., Hedström, M., Tümer, M. A., Denizli, A., Mattiasson, B. Real-time prostate-specific antigen detection with prostate-specific antigen imprinted capacitive biosensors. Analytica Chimica Acta. 891, 120-129 (2015).
- Ertürk, G., Mattiasson, B. From imprinting to microcontact imprinting-A new tool to increase selectivity in analytical devices. Journal of Chromatography. B, Analytical Technologies in the Biomedical and Life Sciences. 1021, 30-44 (2016).
- Ertürk, G., Mattiasson, B. Molecular Imprinting: The Creation of Biorecognition Imprints on Biosensor Surfaces. Advanced Molecularly Imprinting Materials. , 523-560 (2017).
- Janczuk-Richter, M., et al. Long-period fiber grating sensor for detection of viruses. Sensors and Actuators B: Chemical. 250, 32-38 (2017).
- Kropinski, A. M., Mazzocco, A., Waddell, T. E., Lingohr, E., Johnson, R. P. Enumeration of bacteriophages by double agar overlay plaque assay. Methods in Molecular Biology. 501, 69-76 (2009).
- Meillan, M., et al. Self-assembled monolayer for AFM measurements of Tobacco Mosaic Virus (TMV) at the atomic level. RSC Adv. 4 (23), 11927 (2014).
- Ymeti, A., et al. Fast, ultrasensitive virus detection using a Young interferometer sensor. Nano Letters. 7 (2), 394-397 (2007).
- Wei, Y., Wong, L. P., Toh, C. -. S. Fuel cell virus sensor using virus capture within antibody-coated nanochannels. Analytical Chemistry. 85 (3), 1350-1357 (2013).
- Guliy, O. I., et al. Immunodetection of bacteriophages by a piezoelectric resonator with lateral electric field. Applied biochemistry and microbiology. 52 (4), 457-463 (2016).
- Reta, N., Michelmore, A., Saint, C., Prieto-Simón, B., Voelcker, N. H. Porous silicon membrane-modified electrodes for label-free voltammetric detection of MS2 bacteriophage. Biosensors & Bioelectronics. 80, 47-53 (2016).
- Ertürk, G., Özen, H., Tümer, M. A., Mattiasson, B., Denizli, A. Microcontact imprinting based surface plasmon resonance (SPR) biosensor for real-time and ultrasensitive detection of prostate specific antigen (PSA) from clinical samples. Sensors and Actuators B: Chemical. 224, 823-832 (2016).
- Lebogang, L., Mattiasson, B., Hedström, M. Capacitive sensing of microcystin variants of Microcystis aeruginosa using a gold immunoelectrode modified with antibodies, gold nanoparticles and polytyramine. Microchimica Acta. 181 (9-10), 1009-1017 (2014).
- Loyprasert, S., Hedström, M., Thavarungkul, P., Kanatharana, P., Mattiasson, B. Sub-attomolar detection of cholera toxin using a label-free capacitive immunosensor. Biosensors & Bioelectronics. 25 (8), 1977-1983 (2010).
- Teeparuksapun, K., Hedström, M., Wong, E. Y., Tang, S., Hewlett, I. K., Mattiasson, B. Ultrasensitive detection of HIV-1 p24 antigen using nanofunctionalized surfaces in a capacitive immunosensor. Analytical Chemistry. 82 (20), 8406-8411 (2010).
- Labib, M., Hedström, M., Amin, M., Mattiasson, B. A capacitive biosensor for detection of staphylococcal enterotoxin B. Analytical and Bioanalytical Chemistry. 393 (5), 1539-1544 (2009).
- Ertürk, G., Hedström, M., Mattiasson, B. A sensitive and real-time assay of trypsin by using molecular imprinting-based capacitive biosensor. Biosensors & Bioelectronics. 86, 557-565 (2016).
- Ertürk, G., Uzun, L., Tümer, M. A., Say, R., Denizli, A. Fab fragments imprinted SPR biosensor for real-time human immunoglobulin G detection. Biosensors & Bioelectronics. 28 (1), 97-104 (2011).
- El-Sharif, H. F., Phan, Q. T., Reddy, S. M. Enhanced selectivity of hydrogel-based molecularly imprinted polymers (HydroMIPs) following buffer conditioning. Analytica Chimica Acta. 809, 155-161 (2014).
- Wang, F., et al. Detection of Salmonella Typhimurium on Spinach Using Phage-Based Magnetoelastic Biosensors. Sensors (Basel, Switzerland). 17 (2), (2017).
- Chin, B. A., et al. Rapid detection of small quantities of specific bacteria using phage-based wireless biosensors. 2016 10th International Conference on Sensing Technology (ICST). , 1-5 (2016).
- Park, M. -. K., Chin, B. A. Novel Approach of a Phage-Based Magnetoelastic Biosensor for the Detection of Salmonella enterica serovar Typhimurium in Soil. Journal of Microbiology and Biotechnology. 26 (12), 2051-2059 (2016).
- Mack, J. D., Yehualaeshet, T., Park, M. -. K., Tameru, B., Samuel, T., Chin, B. A. Phage-Based Biosensor and Optimization of Surface Blocking Agents to Detect Salmonella typhimurium on Romaine Lettuce. Journal of food safety. 37 (2), 12299 (2017).
- Colomer-Lluch, M., Jofre, J., Muniesa, M. Antibiotic resistance genes in the bacteriophage DNA fraction of environmental samples. Plos One. 6 (3), 17549 (2011).
- Lood, R., Ertürk, G., Mattiasson, B. Revisiting antibiotic resistance spreading in wastewater treatment plants - bacteriophages as a much neglected potential transmission vehicle. Frontiers in microbiology. 8, (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados