Method Article
Base de détection ultrasensible des biomarqueurs à l’aide d’une empreinte moléculaire biocapteur capacitif
Dans cet article
Résumé
Nous présentons ici un protocole pour la détection et la quantification des molécules abondantes faibles dans des solutions complexes en utilisant moléculaire en combinaison avec un biocapteur capacité d’impression.
Résumé
La capacité à détecter et à quantifier les biomolécules dans des solutions complexes a toujours été très prisée dans les sciences naturelles ; utilisé pour la détection des biomarqueurs, de contaminants et d’autres molécules d’intérêt. Une technique souvent utilisée à cette fin est l’Enzyme-linked Immunosorbent Assay (ELISA), où souvent un anticorps est dirigé vers une molécule cible spécifique, et un second anticorps marqué est utilisé pour la détection de l’anticorps primaire, permettant la quantification absolue de la biomolécule sous étude. Toutefois, l’utilisation des anticorps en tant qu’éléments de reconnaissance limite la robustesse de la méthode ; comme le fait de la nécessité d’utiliser des molécules marquées. Pour surmonter ces limites, empreinte moléculaire a été implémenté, création de sites de reconnaissance artificiel complémentaire à la molécule modèle et obsolète la nécessité d’utiliser des anticorps pour la liaison initiale. En outre, pour une sensibilité encore plus élevée, l’anticorps secondaire marqué peut être remplacé par biocapteurs en s’appuyant sur la capacité pour la quantification de la molécule cible. Dans le présent protocole, on décrit une méthode pour rapidement et sans étiquette détecter et quantifier la basse-abondant biomolécules (protéines et virus) dans des échantillons complexes, avec une sensibilité qui est nettement mieux que les systèmes de détection couramment utilisés comme l’ELISA. C’est tous médiée par l’empreinte moléculaire en combinaison avec un biocapteur capacitance.
Introduction
La quantification des biomolécules est utilisée dans de nombreux domaines de recherche différents dans la science, en employant des méthodes telles que le dosage radio-immunologique (RIA) ou ELISA1. Certaines de ces méthodes nécessitent une étiquette réactif comme un radio-isotope ou enzyme marqué anticorps/antigène, ce qui les rend fastidieuse et chronophage avec des procédures complexes2. De plus, la robustesse, la sélectivité et la sensibilité de ces méthodes ne sont pas suffisantes pour toutes les analyses ; en particulier, ils ne sont pas suffisantes quand attogram quantités doivent être analysées, plutôt que de pictogramme quantités3. À cette fin, biocapteurs ont acquis un intérêt considérable4,5, en particulier en combinaison avec l’empreinte moléculaire pour une robustesse accrue.
L’empreinte moléculaire repose sur la création de cavités par polymérisation de monomères fonctionnels autour du modèle6, création de sites de reconnaissance artificielles qui ressemblent parfaitement le modèle7. Cette technique a été utilisée pour plusieurs applications, y compris les systèmes de livraison de médicaments et de séparation analytique, mais aussi comme éléments de biocapteurs8,9,10bioreconnaissance. Cependant, il y a encore quelques difficultés dans la conception des polymères (MIPs) pour les modèles macromoléculaires comme protéines et cellules11,12. Pour cette raison, de nombreux chercheurs ont porté imprégnation de la protéine modèle directement sur un substrat, créant ainsi une surface qui sera reconnue par la protéine de cible12. Cette technique de revêtement de surface utilisée pour l’emploi des cavités de reconnaissance pour les grosses molécules et ensembles comprenant des protéines est appelée par microcontact impression13,14. La procédure générale de la méthode dépend de la polymérisation entre deux surfaces - un timbre de modèle et d’un support de polymère - sur laquelle le modèle est adsorbé sur une surface de15,16et mis en contact avec le surface traitée monomère. De cette façon, un film de polymère mince est formé sur le support par UV-polymérisation. Enfin, le modèle est enlevé, laissant derrière lui des cavités de modèle spécifique à la surface de l’électrode imprimée. Cette méthode présente certains avantages, notamment une perte de l’activité réduite de la molécule d’imprimés, ainsi comme exigeant de très petites quantités de molécules de modèle pour l’impression de traitent16,17. Ainsi, ces surfaces rentables, stables, sensibles et sélectifs peuvent être créés sur les surfaces de capteur, axée sur un modèle de choix de l’utilisateur.
Le biocapteur peut être utilisé pour la détection des protéines simples et beaucoup plus grande biomacromolecules, y compris les virus. Un groupe spécifique de virus récent intérêt est le bactériophage, qui est un virus qui infecte les bactéries. Une détection rapide et sensible des bactériophages est importante au cours de biotechnologie et biopharmaceutique processus afin de déterminer les infections de cultures bactériennes avec bactériophages18. Le dosage biologique plus couramment utilisé pour la détection de bactériophage la double couche de gélose méthode19, qui est laborieux et fastidieux. Plusieurs tentatives ont été faites pour développer de nouveaux outils de diagnostic pour les virus (y compris les bactériophages) comme force atomique (AFM) la microscopie20, interférométrie21, électrochimie22et capteur système23, 24. beaucoup de travail a été axé sur les biocapteurs en raison de leurs avantages comme étant capable de mesure en temps réel15,25, très sensible et facile à utiliser. Un certain type de biocapteur est basé sur les changements dans la capacité. Ces biocapteurs capacitifs sont les capteurs électrochimiques qui mesurent les changements dans les propriétés diélectriques lorsqu’un analyte interagit avec un élément de bioreconnaissance sur la surface du capteur, provoquant une diminution de la capacité2,4 . Capacitifs biocapteurs ont été utilisés pour la détection des différents analytes comme antigènes, anticorps, protéines et des ions de métaux lourds6,26,27,28. Ces types de biocapteurs présentent de nombreux avantages comme la rapidité inhérente, haute sensibilité, simplicité, coût bas, facile de manipulation et mesure en temps réel sans étiquetage29.
La méthode décrite ici vise à permettre la détection et la quantification des biomolécules peu abondantes dans des échantillons très complexes, sans avoir besoin d’utiliser n’importe quel étiquetage. En particulier, la technique est très utile dans la gamme atto-picogramme de biomolécules, où autres instruments existants dans le commerce ne parviennent pas à doser avec précision leur cible.
Protocole
1. modification des lamelles de verre (modèle de timbres)
- Pour nettoyer les lamelles de verre, immergez-les dans l’ordre dans 10 mL d’HCl 1.0 M, eau désionisée et 1,0 M de NaOH, respectivement, pendant 10 min à chaque étape dans un nettoyeur à ultrasons à température ambiante.
- Sécher les lamelles de verre à l’azote.
Remarque : Les lamelles sont séchés par évaporation sous un courant d’azote gazeux.
- Sécher les lamelles de verre à l’azote.
- Plongez les lamelles nettoyés et séchés dans 10 % (v/v), 10 mL de solution de 3-amino-propyl-triéthoxysilane (APTES) dans l’éthanol pendant 1 h à introduire des groupes aminés sur la surface du couvercle en verre, à température ambiante.
- Rincer les lamelles à l’eau désionisée.
- Sécher les lamelles à l’azote.
- Plongez les lamelles selon modifiés chez 5 % (v/v), 10 mL de solution de glutaraldéhyde dans un tampon phosphate 10 mM (pH 7,4) pendant 2 h afin d’activer les groupes aminés sur la surface, à température ambiante6,15.
- Rincer les lamelles avec un tampon phosphate 10 mM (pH 7,4) afin de prélever le glutaraldéhyde excédentaire à la surface.
- Sécher les lamelles à l’azote.
- Préparer 1,0 mL de solution de modèle (protéine/bactériophage) dans un tampon phosphate 10 mM (pH 7,4) la concentration de 0,1 mg/mL.
NOTE : Dissoudre 0,1 mg de protéine dans 1,0 mL de tampon phosphate (10 mM, pH 7,4). Si nécessaire, un spectrophotomètre (280 nm) peut être utilisé pour déterminer la concentration de protéine.- Déposer 200 µL de cette solution de modèle sur les lamelles modifiés et incuber une nuit à 4 ° C.
Remarque : La solution de modèle excès peut servir à préparer plus de timbres modèle 1-2 pour être utilisé dans les études de caractérisation. - Rincer les lamelles avec un tampon phosphate 10 mM (pH 7,4) pour supprimer le modèle indépendant de la surface.
Remarque : Pour rincer les électrodes, lavez-les avec un tampon phosphate (10 mM, pH 7,4) pendant 30 s. - Sécher les lamelles à l’azote.
Remarque : Les lamelles doivent être stockés à 4 ° C jusqu'à ce qu’ils sont utilisés à l’étape de polymérisation.
- Déposer 200 µL de cette solution de modèle sur les lamelles modifiés et incuber une nuit à 4 ° C.
2. modification d’or capacitives
- Pour nettoyer les électrodes, plonger les électrodes dans un petit bécher, séquentiellement, contenant 5 mL d’éthanol (70 %), eau désionisée eau, acétone, eau déminéralisée, solution acide piranha (3:1, H2SO4: H2O2, v/v) et déminéralisée eau, respectivement pendant 10 min à chaque étape, dans un nettoyeur à ultrasons à température ambiante.
- Sécher les électrodes à l’azote.
- Afin d’effectuer l’électropolymérisation de la tyramine, préparer 8 mL de solution de tyramine 10 mM 10 mM phosphate tamponné (pH 7,4) contenant de l’éthanol (2 mL)15.
Remarque : Le volume total de la solution de tyramine doit être 8 mL au total dont 2 mL d’éthanol.- Effectuer des analyses de voltampérométrie cycliques (15 cycles) dans cette solution à l’aide d’un potentiostat couvrant un potentiel entre 0 - 1, 5V (Ag/AgCl) et une vitesse de balayage de 50 mV/s30.
- Rincer les électrodes à l’eau désionisée.
- Sécher les électrodes à l’azote.
- Plonger les électrodes dans une solution contenant du chlorure d’acryloyl 30 mM et 30 mM triméthylamine dans le toluène (Vtotal = 5 mL) à la température ambiante la nuit6,15,30.
- Rincer les électrodes à l’eau désionisée.
Remarque : Pour assurer une meilleure élimination des acryloyl n’ayant pas réagi résidus chlorure et la triméthylamine, il est également possible de laver la surface avec du NaOH après l’eau désionisée. - Sécher les électrodes à l’azote.
- Rincer les électrodes à l’eau désionisée.
3. préparation du modèle imprimé or capacitives
-
Préparation du bactériophage imprimé or capacitives
- Avant la polymérisation, préparer une solution de monomère contenant des monomères (N-hydroxyméthyl acrylamide) et court (polyéthylène-glycol-400-diméthacrylate) dans une proportion de 1:5 (mol/mol) dans 1 mL de tampon phosphate de 10 mM (pH 7,4).
Remarque : La solution de monomère contenant des monomères et réticulant peut être utilisée dans différentes proportions, ou les types de monomère et du réticulant peuvent être changés selon le modèle d’optimisation de l’interaction spécifique. - Ajouter 1 mg de photo-initiateur dans cette solution.
Remarque : Si la polymérisation s’effectue sous la lumière UV, puis la photo-initiateur doit servir à initier la polymérisation. Si la polymérisation est réalisée par polymérisation radicalaire, le type de l’initiateur doit être changé. - Distribuer 1,5 µL de cette solution sur la surface de l’électrode d’or mis à jour le d’or.
NOTE : La surface de l’électrode d’or est présentée schématiquement à la Figure 1. - Porter le cachet de modèle en contact avec la solution de monomère sur le dessus de la surface de l’électrode d’or.
- Initier la polymérisation UV (365 nm, 400 W) et continuer pendant 15 min31.
Remarque : La polymérisation UV est effectuée à l’intérieur d’une armoire de refroidissement qui est fixée à-25 ° C avant d’initier la polymérisation. Puis, la lumière UV système de polymérisation est allumée et la polymérisation est maintenue pendant 15 minutes avant d’éteindre la lumière UV système de polymérisation. - Enlever le timbre de modèle de la surface à l’aide de pinces.
Remarque : Lors du retrait du timbre du modèle de la surface, le film polymère sur la surface peut être endommagé. Par conséquent, le timbre doit être retiré de la surface très prudemment et lentement sans recourir à la force. - Rincer la surface de l’électrode à l’eau désionisée et séchez-le à l’azote.
- Plonger les électrodes dans 1 mL de 10 mM 1-dodécanethiol, préparé dans l’éthanol pendant 20 min afin de couvrir les trous d’épingle sur la surface de l’électrode.
- Rincer les électrodes avec de l’eau déminéralisée et sécher les électrodes à l’azote.
- Avant la polymérisation, préparer une solution de monomère contenant des monomères (N-hydroxyméthyl acrylamide) et court (polyéthylène-glycol-400-diméthacrylate) dans une proportion de 1:5 (mol/mol) dans 1 mL de tampon phosphate de 10 mM (pH 7,4).
-
Préparation de la protéine imprimé or capacitives
- Avant la polymérisation, préparer une solution de monomère contenant des monomères (acrylamide : 54 mg ; N-hydroxymethylacrylamide : 140 µL ; N-isopropylacrylamide : 85,6 mg) et court (methylenebisacrylamide : 9,5 mg) dans 820 µL d’eau ultra pure30,32.
- Préparer 5 % (v/v) N, N, N', N'-tetramethylethyldiamine (TEMED) dans l’eau ultra pure.
- Purge à l’azote pendant 5 min et ajouter 20 µL de la solution TEMED dans la solution de monomère.
- Préparer 10 % (p/v) persulfate d’ammonium (APS) dans l’eau ultra pure.
- Ajouter 20 µL de la solution APS dans la solution de monomère.
- Distribuer 1,5 µL de la solution de monomère sur la surface de l’électrode d’or mis à jour le.
- Porter le cachet de modèle en contact avec la surface traitée monomère.
- Commencez la polymérisation à la température ambiante et continuez pendant 5 h.
Remarque : Pour la préparation des électrodes en or protéine-imprimé, au lieu de UV-polymérisation, polymérisation radicalaire à température ambiante (25 ° C) est réalisée en utilisant des APS-TEMED comme l’initiateur du catalyseur. - Retirer soigneusement le timbre de modèle de la surface à l’aide de pinces.
- Rincer l’électrode à l’eau déminéralisée et sécher avec l’azote gazeux.
- Plonger les électrodes dans 1 mL de 10 mM 1-dodécanethiol, préparé dans l’éthanol pendant 20 min afin de couvrir les trous d’épingle sur la surface de l’électrode.
- Rincer les électrodes avec de l’eau déminéralisée et sécher les électrodes à l’azote.
4. caractérisation de la Surface de l’électrode avec la microscopie électronique (MEB)
- Monter les spécimens sur supports en aluminium avec du ruban adhésif carbone.
- Enrober les électrodes avec 10 or/palladium nm.
- Examiner les électrodes avec SEM
5. en temps réel mesures capacitives avec modèle imprimé or capacitives
- Insérez les électrodes en or capacitifs imprimés dans la cellule électrochimique de débit intégrée à un biocapteur capacitif.
- Préparation de 100 mL de buffer de régénération (25 mM glycine-HCl, pH 2,5, y compris de Tween-20 à 50 mM) et 1 L en cours d’exécution (pour bactériophage imprimé système : phosphate 10 mM, pH 7,4 ; système de protéine-imprimé : 50 mM Tris-HCl, pH 7,4).
- Commencer l’analyse par l’injection de tampon de régénération pour régénérer le système et le tampon en cours d’exécution à rééquilibrer le système pendant 25 min.
- Préparer des solutions de modèle standard de la gamme de concentration souhaitée dans le tampon.
NOTE : D’abord préparer un stock de la solution de modèle de dissoudre 0,1 mg de protéine, ou environ 108 pfu bactériophages, à 1,0 mL de tampon de phosphate (phosphate de 10 mM, pH 7,4). Ensuite, préparer les solutions étalons pour la courbe d’étalonnage en faisant dix dilutions 10 fois séquentielles de la solution mère. Ces solutions seront analysées avec le biocapteur capacitif à l’étape 5.5. Concentration de protéine puisse être mesurée à l’aide d’un spectrophotomètre (280 nm). Afin de mesurer la concentration du bactériophage, une méthode de gélose de double couche peut être utilisée, qui est décrite en détails précédemment19. - Injecter 250 µL de ces solutions étalons séquentiellement au système dans des conditions optimales (débit : 100 µL/min, température : 25 ° C).
Remarque : Dans cette application, les solutions standard de protéines ont été préparées dans des concentrations comprises entre 1,0 x 10-4 - 1,0 x 10-14, tandis que les concentrations de bactériophage étaient dans la plage 1,0 x 101 - 1,0 x 105 UFP/mL. Les solutions ont été placées dans la valve d’injection et séquentiellement injectées dans le système, en injectant les échantillons en géométrie à travers la pompe seringue unique et la vanne. La diminution de capacité après l’injection des solutions étalons découlant de la liaison de modèle aux cavités imprimées est contrôlée automatiquement par le logiciel de l’instrument.
Résultats
En suivant le protocole, selon le schéma de la Figure 1, une électrode d’or nue vont être imprimée avec un modèle, qui représente la structure d’un biomacromolécule. Cette électrode peut être appliquée dans un biocapteur capacitif (Figure 2), ce qui permet l’application stable d’un modèle sur l’électrode et la mesure des changements de capacité lors de la liaison du modèle.
Une représentation schématique de la biocapteur capacitif est illustrée à la Figure 2. La pompe de centris, qui est responsable de l’injection continue du tampon en cours d’exécution (phosphate 10 mM, pH 7,4) et le tampon de la régénération (25 mM glycine-HCl, pH 2,5) au cours de la régénération dans la cellule de flux, peut être vu clairement dans la figure. La cellule de flux se compose de travail, référence et électrodes de compteur. La valve d’injection se compose de solutions standard protéine/bactériophage, qui passent par le dégazeur d’abord et puis injectée dans l’ordre dans le système. Dès que les solutions atteignent l’électrode de travail inséré dans la cellule de flux, le résultat est contrôlé en temps réel. Les valeurs de capacité peuvent être inscrits en suivant la sensorgrams sur l’écran de l’ordinateur.
Figure 3 et Figure 4 illustrent les différences entre la surface des électrodes en or nus et imprimés. Cette étape de caractérisation est importante pour s’assurer qu’il existe des cavités polymériques, considérées comme la rugosité de la surface de l’électrode après imprégnation. En dehors de la SEM, il y a aussi des autres méthodes de caractérisation y compris AFM, mesures de l’angle de contact, ellipsométrie etc.qui peut être utilisé pour caractériser la surface après imprégnation. De cette façon, il peut s’assurer que le processus de l’empreinte est réussi et que les cavités de modèle sont forment sur la surface. Dans ces cavités, le modèle peut se lier avec une affinité et une spécificité élevée.
Après l’injection des solutions standards dans le système capacitif, une moyenne des cinq dernières lectures a été calculée automatiquement par le logiciel, et les d’abaques ont été obtenues en traçant la variation de capacité par rapport à la concentration de la analyte. La diminution de la capacité recommandée est née de la liaison du modèle. Les plus de molécules qui se lient à l’électrode d’or de surface, plus la réduction de la capacité totale, selon le principe général de mesure capacitif. Figure 5 et Figure 6 montrent que, avec l’augmentation de la concentration de l’analyte, la ΔC augmente comme prévu. La plage dynamique (entre lesquelles est la plage de concentration où le système est utile pour la détection d’une cible spécifique) et la limite de détection (LD) peut être évalué en analysant ces graphiques. Selon la Figure 6, le biocapteur capacitif bactériophage imprimé peut détecter des bactériophages dans des concentrations comprises entre 101 - 105 UFP/mL, avec une valeur LOD 10 UFP/mL dans cette étude. Les deux Figure 5 et Figure 6 également mettre en évidence la nécessité de mesurer la courbe d’étalonnage dans le même intervalle requis pour que la concentration de modèle est supposé, étant donné que la linéarité de la régression peut varier dans les concentrations ( Figure 5), ou ont des pentes différentes (Figure 6). Il convient également de noter qu’en raison des faibles concentrations utilisées, le système est très sensible aux fluctuations (turbidité dans l’échantillon, tirant d’air, etc.), et par conséquent, il est recommandé d’exécuter au moins réanalysés pour réduire la possibilité d’inclure des valeurs aberrantes . Pour la même raison, l’écart peut être très importante pour les échantillons très dilués, comme on le voit à la Figure 6.
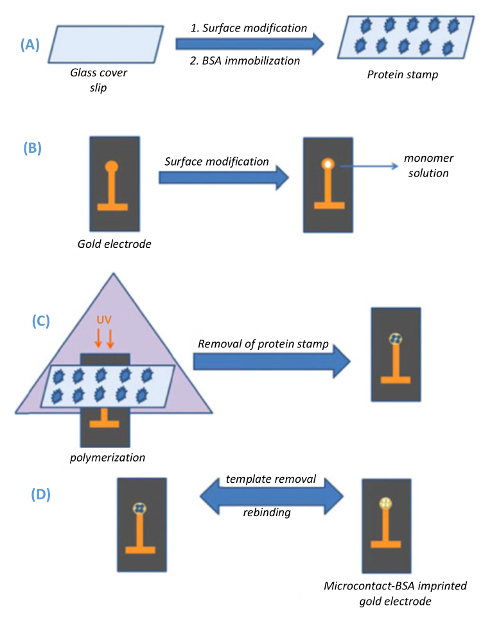
Figure 1 . Représentation schématique de la par microcontact imprégnation méthode. (A) préparer des lamelles de verre (tampons de modèle), (B) préparation des électrodes capacitives or, impression par microcontact (C) du modèle sur la surface de l’électrode d’or via UV-polymérisation et (D) suppression du modèle à partir de la surface de l’électrode (reproduit de Ertürk et coll., biotechnologie rapports 2014 (3) : 65-72 avec autorisation). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
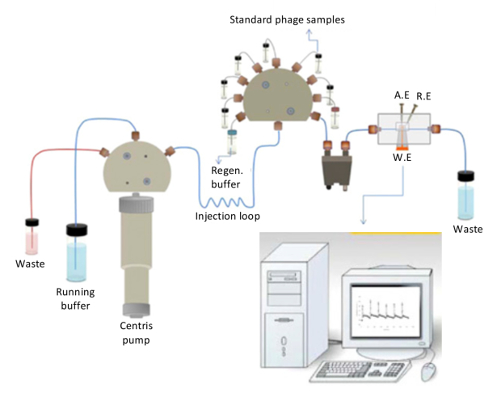
La figure 2. Représentation schématique de la biocapteur capacitif. La disposition générale du biocapteur capacitif utilisée dans cette étude (reproduit de Ertürk et coll., biotechnologie rapports 2014 (3) : 65-72 avec autorisation). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

La figure 3. Imprimé de microscopie électronique de protéine électrodes. Images de SEM de (A) une électrode d’or nue (barre d’échelle = 20 µm), et (B), une protéine imprimé capacitif électrode d’or (Echelle = 10 µm). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

La figure 4. Microscopie électronique du bactériophage imprimé électrodes. Images de SEM d’une électrode d’or nue (barre d’échelle = 20 µm) (A), et un bactériophage imprimé capacitif électrode d’or dans différents grossissements (6600 x, section Echelle = 10 µm) (B) et 11, 500 X, échelle bar = 5 µm) (C) ; flèches indiquent les bactériophages collés). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
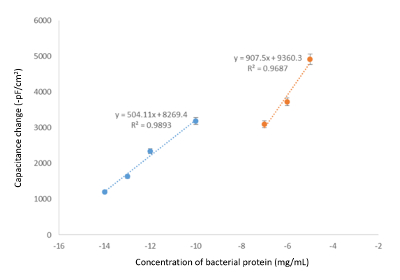
Figure 5. Effet de la composition du tampon pour d’abaques. Courbe d’étalonnage en montrant le changement de capacité par rapport à une concentration de protéines dans des conditions optimales (tampon en cours d’exécution : 50 mM Tris-HCl, pH 7,4 ; Tampon de régénération : 25 mM glycine-HCl, pH 2,5 dont 50 mM Tween-20 ; Débit : 100 µL/min, volume de l’échantillon : 250 µL ; T : 25 ° C). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

La figure 6. Courbe d’étalonnage représentant pour grand biomacromolecules. Courbe d’étalonnage qui illustre la variation de capacité par rapport à la concentration de bactériophage dans des conditions optimales (tampon en cours d’exécution : phosphate 10 mM, pH 7,4 ; Tampon de régénération : 25 mM glycine-HCl, pH 2,5 dont 50 mM Tween-20 ; Débit : 100 µL/min, volume de l’échantillon : 250 µL ; T : 25 ° C) s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Lorsque cette méthode est effectuée, il y a certaines étapes critiques qui doivent être considérés en suivant le protocole. Une étape critique est l’étape de nettoyage avec une solution acide de piranha. Étape 2.1 ne doit pas être plus de 10 min. La solution de modèle pour l’étape 1.4 ne doit pas dépasser 0,1 mg/mL, étant donné que ces valeurs ont été optimisées précédemment. Analyses voltamétriques cyclique ne doivent pas dépasser 15 cycles afin d’obtenir l’épaisseur optimale. Pour l’étape 3.1.3, 1,5 µL est une valeur optimisée. Cette valeur ne doit pas être plus élevée pour ce type spécifique d’électrode. Si le UV système de polymérisation, d’une puissance de 400 W, puis la polymérisation doit être effectuée pour un maximum de 10-15 min. Dès que l’APS (initiateur) est ajouté à la solution, après le TEMED, l’étape suivante doit être effectuée très rapidement pour éviter une polymérisation immédiate (étape 3.2.5).
Une des étapes plus critiques est la suppression du timbre de la surface du modèle après polymérisation UV. Si cette étape n’est pas correctement effectuée, il y a un risque que le film polymère sur la surface de l’électrode peut être enlevé avec du cachet. Par conséquent, il est recommandé de plonger l’électrode et le timbre de protéines sur le dessus dans une solution d’eau après la polymérisation, puis retirez le timbre très lentement et avec précaution la surface (étape 3.1.6).
Basé sur le modèle utilisé, modifications du type et de la proportion de monomères utilisés (monomère fonctionnel et réticulant) peuvent être évaluées en termes de génération d’une sensibilité plus élevée. Elle doit être déterminée empiriquement. En outre, l’affinité de la molécule modèle généralement implique plusieurs interactions différentes simultanément. Par conséquent, cela peut conduire à des problèmes au cours des étapes de la régénération. Si le modèle lié n’est pas libéré de la surface correctement, cela peut influer sur la réutilisation de l’électrode pour une analyse ultérieure. Ces affinités multipoints peuvent aussi résulter d’une liaison via des interactions faibles. Dans de tels systèmes, non-spécifiques peut avoir lieu, qui peut influencer négativement la sélectivité du système16. Voici les limites communes et générales, de la méthode.
En dehors de ces limites spécifiques, il y a plusieurs avantages significatifs de la méthode discuté sur les méthodes existantes. Tandis que RIA, ELISA et mesures fluorimétrique sont très sensibles, elles nécessitent l’utilisation de matériel marqué (modèle ou détecteur), tandis que le biocapteur est complètement étiquette exempt. Ces méthodes sont également plus long et coûteux. Une approche de biocapteurs permet une synthèse rapide, en parallèle de MIPs dans des compositions différentes à la même heure16. Puisque seulement quelques microlitres de la solution de monomère est nécessaire pour la préparation, la méthode est pratique lorsque vous utilisez des monomères limitées chers ou autres. En outre, seul MIP électrodes peuvent être utilisées pour environ 80 analyses sans une baisse significative de la performance, qui est nettement supérieure aux autres actuelle des méthodes30. Méthodes existantes souffrent aussi, à des degrés divers de faible sensibilité et sélectivité, tandis que la méthode décrite permet pour la détection et la quantification des molécules dans la gamme MP à haute sélectivité.
En raison de la rentabilité, la facilité de fonctionnement de l’instrument et la sensibilité de détection en temps réel en peu de temps par rapport aux méthodes existantes, les biocapteurs sont très prometteuses point de systèmes de détection de soins dans des conditions de champ ; par exemple, pour la surveillance de l’environnement et pour des applications dans les pays en développement. Dans de nombreuses applications dans le diagnostic des maladies, une détection en temps réel, sensible, sélective et rapide d’un biomarqueur dans un mélange complexe comme le sérum est obligatoire,15,25. Ici, les biocapteurs sont supérieures aux méthodes existantes, en particulier, en raison de leur robustesse et leur sensibilité. Spécifiquement pour la détection d’agents infectieux, les bactériophages sont récemment considérés comme élément de rechange bioreconnaissance pour les biocapteurs à cause de leur hôte bactéries spécificité33,34,35. Remplacement des anticorps avec des bactériophages est très prometteur pour réduire les coûts et augmenter la stabilité encore36. Un tel système permettra également la détection et la quantification des phages spécifiques dans l’environnement et des échantillons cliniques. En raison de la prévalence des bactériophages et leur capacité à transmettre des bactéries avec des gènes de résistance aux antibiotiques37,38, une telle méthode peut être précieuse étudie la propagation de bactéries résistantes.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Maria Baumgarten (IQ biotechnologie plate-forme, Infection de médecine, Université de Lund) est reconnu pour l’exécution et en fournissant des photographies au microscope électronique de balayage. Ce travail a été soutenu par des subventions de la suédoise recherche Conseil Formas (2017-00100) dans le cadre de l’Initiative troisième européen de programmation conjointe à l’appel, la résistance aux antimicrobiens (JPIAMR) « Dynamique de la Transmission. » Les bailleurs de fonds n’avaient aucun rôle dans la conception de l’étude, de l’interprétation, écriture préparation du manuscrit, décision à soumettre, ou la décision de publier les travaux.
matériels
| Name | Company | Catalog Number | Comments |
| Glass Cover slips | ThermoFisher | 102222 | protein stamp |
| HCl | Sigma-Aldrich | H1758-500ML | cleaning |
| NaOH | Sigma-Aldrich | 72068-100ML | cleaning |
| Ultrasonic cleaner | Branson Ultrasonic | BRANSONIC M1800- E | cleaning |
| 3-amino-propyl-triethoxysilane (APTES) | Sigma-Aldrich | A3648-100ML | modification |
| EtOH | Sigma-Aldrich | 1009836010 | rinsing/cleaning |
| glutaraldehyde | Sigma-Aldrich | G5882-100ML | cross-linker |
| acetone | Sigma-Aldrich | 34850-1L-M | cleaning |
| H2SO4 | Sigma-Aldrich | 339741-100ML | piranha solution |
| H2O2 | Sigma-Aldrich | H1009-500ML | piranha solution |
| tyramine | Sigma-Aldrich | T90344-5G | modification |
| CompactStat | Ivium Technologies | CompactStat.h: 30mA@10V/3MHz | potentiostat |
| Platinum Counter Electrode Kit | Equilabrium | AFCTR5 | potentiostat |
| Reference Electrode | Equilabrium | RREF0021 | potentiostat |
| acryloyl chloride | EMD Millipore | 8.00826.0100 | modification |
| triethylamine | EMD Millipore | 8.08352.0100 | modification |
| toluene | Sigma-Aldrich | 244511-100ML | modification |
| N-hydroxymethyl acrylamide | Sigma-Aldrich | 245801-100G | functional monomer |
| poly ethylene glycol-400-dimethacrylate | Sigma-Aldrich | 409510-250ML | cross-linker |
| 2-Hydroxy-4′-(2-hydroxyethoxy)-2-methylpropiophenone | Sigma-Aldrich | 410896-50G | functional monomer |
| UV polymerizator | Dymax | Dymax 5000ECE | UV-polymerization |
| forceps | Sigma-Aldrich | Z168777-1EA | consumable |
| 1-dodecanethiol | Sigma-Aldrich | 471364-100ML | blocking agent |
| acrylamide | Sigma-Aldrich | A3553-100G | functional monomer |
| N-hydroxymethylacrylamide | Sigma-Aldrich | 245801-100G | functional monomer |
| N-isopropylacrylamide | Sigma-Aldrich | 415324-50G | functional monomer |
| methylenebisacrylamide | Sigma-Aldrich | 146072-500G | cross-linking monomer |
| N,N,N',N'-tetrametyhlethyldiamine (TEMED) | Sigma-Aldrich | T9281-25ML | catalyst |
| ammonium persulphate | Sigma-Aldrich | A3678-25G | initiator |
| Capacitive biosensor | CapSenze | Equipment | |
| Glycine | Merck | 1042011000 | regeneration buffer |
| Tween-20 | Sigma-Aldrich | P9416-50ML | regeneration buffer |
| Trizma base | Sigma-Aldrich | 93352-1KG | running buffer |
| Na2HPO4 • 2H2O | Calbiochem | 567547-1KG | running buffer |
| NaH2PO4 • 2H2O | Calbiochem | 567549-1KG | running buffer |
| DELPHI correlative light and electron microscope | Phenom-World | equipment | |
| Capacitive gold electrodes | CapSenze Biosystems | consumables | |
| 2,2'-azobis(2-methypropionitrile) | Sigma-Aldrich | 441090-25G | photo-initiator |
| CapSenze Smart Software | CapSenze Biosystems | software program |
Références
- Lin, T. Y., Hu, C. H., Chou, T. C. Determination of albumin concentration by MIP-QCM sensor. Biosensors and Bioelectronics. 20 (1), 75-81 (2004).
- Berggren, C., Bjarnason, B., Johansson, G. Capacitive Biosensors. Electroanalysis. 13 (3), 173-180 (2001).
- Zhang, S., Garcia-D'Angeli, A., Brennan, J. P., Huo, Q. Predicting detection limits of enzyme-linked immunosorbent assay (ELISA) and bioanalytical techniques in general. The Analyst. 139 (2), 439-445 (2014).
- Mattiasson, B., Teeparuksapun, K., Hedström, M. Immunochemical binding assays for detection and quantification of trace impurities in biotechnological production. Trends in Biotechnology. 28 (1), 20-27 (2010).
- Limbut, W., Hedström, M., Thavarungkul, P., Kanatharana, P., Mattiasson, B. Capacitive biosensor for detection of endotoxin. Analytical and Bioanalytical Chemistry. 389 (2), 517-525 (2007).
- Ertürk, G., Berillo, D., Hedström, M., Mattiasson, B. Microcontact-BSA imprinted capacitive biosensor for real-time, sensitive and selective detection of BSA. Biotechnology Reports. 3, 65-72 (2014).
- Arshady, R., Mosbach, K. Synthesis of substrate-selective polymers by host-guest polymerization. Macromolecular Chemistry and Physics. 182 (2), 687-692 (1981).
- Andersson, L. I. Molecular imprinting: developments and applications in the analytical chemistry field. Journal of chromatography. B, Biomedical sciences and applications. 745 (1), 3-13 (2000).
- Li, X., Husson, S. M. Two-dimensional molecular imprinting approach to produce optical biosensor recognition elements. Langmuir: the ACS Journal of Surfaces and Colloids. 22 (23), 9658-9663 (2006).
- Alexander, C., Davidson, L., Hayes, W. Imprinted polymers: artificial molecular recognition materials with applications in synthesis and catalysis. Tetrahedron. 59 (12), 2025-2057 (2003).
- Kryscio, D. R., Peppas, N. A. Surface imprinted thin polymer film systems with selective recognition for bovine serum albumin. Analytica Chimica Acta. 718, 109-115 (2012).
- Inerowicz, H. D., Howell, S., Regnier, F. E., Reifenberger, R. Multiprotein immunoassay arrays fabricated by microcontact printing. Langmuir. 18 (13), 5263-5268 (2002).
- Lin, H. Y., Hsu, C. Y., Thomas, J. L., Wang, S. E., Chen, H. C., Chou, T. C. The microcontact imprinting of proteins: The effect of cross-linking monomers for lysozyme, ribonuclease A and myoglobin. Biosensors and Bioelectronics. 22 (4), 534-543 (2006).
- Liao, P. C., Tyan, Y. C., Wang, C. Y., Hsu, J. F., Chou, T. C., Lin, H. Y. Assessing the binding selectivity of molecularly imprinted polymer artificial antibodies by mass spectrometry-based profiling system. Journal of Biomedical Materials Research Part A. 91 (2), 597-604 (2009).
- Ertürk, G., Hedström, M., Tümer, M. A., Denizli, A., Mattiasson, B. Real-time prostate-specific antigen detection with prostate-specific antigen imprinted capacitive biosensors. Analytica Chimica Acta. 891, 120-129 (2015).
- Ertürk, G., Mattiasson, B. From imprinting to microcontact imprinting-A new tool to increase selectivity in analytical devices. Journal of Chromatography. B, Analytical Technologies in the Biomedical and Life Sciences. 1021, 30-44 (2016).
- Ertürk, G., Mattiasson, B. Molecular Imprinting: The Creation of Biorecognition Imprints on Biosensor Surfaces. Advanced Molecularly Imprinting Materials. , 523-560 (2017).
- Janczuk-Richter, M., et al. Long-period fiber grating sensor for detection of viruses. Sensors and Actuators B: Chemical. 250, 32-38 (2017).
- Kropinski, A. M., Mazzocco, A., Waddell, T. E., Lingohr, E., Johnson, R. P. Enumeration of bacteriophages by double agar overlay plaque assay. Methods in Molecular Biology. 501, 69-76 (2009).
- Meillan, M., et al. Self-assembled monolayer for AFM measurements of Tobacco Mosaic Virus (TMV) at the atomic level. RSC Adv. 4 (23), 11927 (2014).
- Ymeti, A., et al. Fast, ultrasensitive virus detection using a Young interferometer sensor. Nano Letters. 7 (2), 394-397 (2007).
- Wei, Y., Wong, L. P., Toh, C. -. S. Fuel cell virus sensor using virus capture within antibody-coated nanochannels. Analytical Chemistry. 85 (3), 1350-1357 (2013).
- Guliy, O. I., et al. Immunodetection of bacteriophages by a piezoelectric resonator with lateral electric field. Applied biochemistry and microbiology. 52 (4), 457-463 (2016).
- Reta, N., Michelmore, A., Saint, C., Prieto-Simón, B., Voelcker, N. H. Porous silicon membrane-modified electrodes for label-free voltammetric detection of MS2 bacteriophage. Biosensors & Bioelectronics. 80, 47-53 (2016).
- Ertürk, G., Özen, H., Tümer, M. A., Mattiasson, B., Denizli, A. Microcontact imprinting based surface plasmon resonance (SPR) biosensor for real-time and ultrasensitive detection of prostate specific antigen (PSA) from clinical samples. Sensors and Actuators B: Chemical. 224, 823-832 (2016).
- Lebogang, L., Mattiasson, B., Hedström, M. Capacitive sensing of microcystin variants of Microcystis aeruginosa using a gold immunoelectrode modified with antibodies, gold nanoparticles and polytyramine. Microchimica Acta. 181 (9-10), 1009-1017 (2014).
- Loyprasert, S., Hedström, M., Thavarungkul, P., Kanatharana, P., Mattiasson, B. Sub-attomolar detection of cholera toxin using a label-free capacitive immunosensor. Biosensors & Bioelectronics. 25 (8), 1977-1983 (2010).
- Teeparuksapun, K., Hedström, M., Wong, E. Y., Tang, S., Hewlett, I. K., Mattiasson, B. Ultrasensitive detection of HIV-1 p24 antigen using nanofunctionalized surfaces in a capacitive immunosensor. Analytical Chemistry. 82 (20), 8406-8411 (2010).
- Labib, M., Hedström, M., Amin, M., Mattiasson, B. A capacitive biosensor for detection of staphylococcal enterotoxin B. Analytical and Bioanalytical Chemistry. 393 (5), 1539-1544 (2009).
- Ertürk, G., Hedström, M., Mattiasson, B. A sensitive and real-time assay of trypsin by using molecular imprinting-based capacitive biosensor. Biosensors & Bioelectronics. 86, 557-565 (2016).
- Ertürk, G., Uzun, L., Tümer, M. A., Say, R., Denizli, A. Fab fragments imprinted SPR biosensor for real-time human immunoglobulin G detection. Biosensors & Bioelectronics. 28 (1), 97-104 (2011).
- El-Sharif, H. F., Phan, Q. T., Reddy, S. M. Enhanced selectivity of hydrogel-based molecularly imprinted polymers (HydroMIPs) following buffer conditioning. Analytica Chimica Acta. 809, 155-161 (2014).
- Wang, F., et al. Detection of Salmonella Typhimurium on Spinach Using Phage-Based Magnetoelastic Biosensors. Sensors (Basel, Switzerland). 17 (2), (2017).
- Chin, B. A., et al. Rapid detection of small quantities of specific bacteria using phage-based wireless biosensors. 2016 10th International Conference on Sensing Technology (ICST). , 1-5 (2016).
- Park, M. -. K., Chin, B. A. Novel Approach of a Phage-Based Magnetoelastic Biosensor for the Detection of Salmonella enterica serovar Typhimurium in Soil. Journal of Microbiology and Biotechnology. 26 (12), 2051-2059 (2016).
- Mack, J. D., Yehualaeshet, T., Park, M. -. K., Tameru, B., Samuel, T., Chin, B. A. Phage-Based Biosensor and Optimization of Surface Blocking Agents to Detect Salmonella typhimurium on Romaine Lettuce. Journal of food safety. 37 (2), 12299 (2017).
- Colomer-Lluch, M., Jofre, J., Muniesa, M. Antibiotic resistance genes in the bacteriophage DNA fraction of environmental samples. Plos One. 6 (3), 17549 (2011).
- Lood, R., Ertürk, G., Mattiasson, B. Revisiting antibiotic resistance spreading in wastewater treatment plants - bacteriophages as a much neglected potential transmission vehicle. Frontiers in microbiology. 8, (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon