Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Longitudinal control morfológico y fisiológico de esferoides tumorales tridimensional mediante tomografía de coherencia óptica
En este artículo
Resumen
Tomografía de coherencia óptica (OCT), una tecnología de imagen tridimensional, fue utilizada para monitorear y caracterizar la cinética de crecimiento de esferoides multicelulares tumor. Cuantificación volumétrica precisa de esferoides tumorales usando un voxel contando el enfoque y la detección de tejido libre de etiqueta muerto en esferoides basado en contraste intrínseco atenuación óptica, fueron demostrados.
Resumen
Esferoides tumorales se han desarrollado como un modelo de cultura (3D) tridimensional de la célula de cáncer investigación y lucha contra el cáncer el descubrimiento de medicamentos. Sin embargo, actualmente, las modalidades de la proyección de imagen de alto rendimiento utilizando detección de campo o fluorescencia brillante, son incapaces de resolver la estructura total 3D de la esferoide de tumor debido a la limitada penetración de la luz, difusión de tintes fluorescentes y profundidad-resolución. Recientemente, nuestro laboratorio demostró el uso de tomografía de coherencia óptica (OCT), no destructiva y sin etiqueta 3D de modalidad, para realizar la caracterización longitudinal de esferoides multicelulares tumor en una placa de 96 pocillos. OCT es capaz de obtener información morfológica y fisiológica 3D de esferoides de tumor creciendo hasta llegar a alrededor de 600 μm de altura. En este artículo, se demuestra un sistema de proyección de imagen de OCT (HT-OCT) alto rendimiento que escanea la placa entera y bien y obtiene datos de OCT 3D de esferoides tumorales automáticamente. Se describen los detalles de las pautas de construcción y sistema de HT-OCT en el protocolo. De los datos de OCT 3D, uno puede visualizar la estructura global del esferoide con 3D prestados y cortes ortogonales, caracterizar la curva de crecimiento longitudinal de la esferoide tumor basado en la información morfológica de tamaño y volumen y controlar el crecimiento de las regiones de muerte celular en el esferoide de tumor basado en contraste óptico atenuación intrínseca. Mostramos que el HT-OCT puede utilizarse como una modalidad de proyección de imagen de alto rendimiento para droga detección, así como caracterizar las muestras biofabricated.
Introducción
El cáncer es la segunda causa de muerte en el mundo1. Desarrollo de fármacos contra el cáncer es de crucial importancia para los pacientes. Sin embargo, se estima que más del 90% de los nuevos fármacos contra el cáncer no en fase de desarrollo debido a la falta de eficacia y toxicidad inesperada en ensayos clínicos2. Parte de la razón puede atribuirse a la utilización de modelos de cultura de célula (2D) de dos dimensiones simple para la detección compuesto, que proporcionan resultados limitados valores predictivos de toxicidad y eficacia compuesto por las siguientes etapas de descubrimiento de drogas2 , 3 , 4. recientemente, tridimensional (3D) tumor esferoide modelos han sido desarrollados para proporcionar datos fisiológicos y farmacológicos clínicamente relevantes de fármaco contra el cáncer descubrimiento3,4,5 ,6,7,8,9,10,11,12,13,14, 15,16,17,18,19,20,21,22,23, 24,25. Puesto que estos esferoides pueden simular propiedades específicas de tejido de tumores en vivo, tales como nutrientes y oxígeno gradiente, hipóxica básico así como drogas resistencia19, el uso de estos modelos puede potencialmente acortar plazos de descubrimiento de drogas, reducir los costos de inversión y traer nuevos medicamentos a los pacientes más con eficacia. Un enfoque crítico para la evaluación de eficacia compuesto en desarrollo del esferoide de tumor 3D es supervisar el crecimiento del esferoide y recidiva en los tratamientos9,26. Para ello, las caracterizaciones cuantitativas de la morfología del tumor, con su diámetro y volumen, con modalidades de imágenes de alta resolución, son imprescindibles.
Modalidades convencionales de la proyección de imagen, como fase contraste7,9,22,24y campo claro y fluorescencia microscopía8,9,16, 18,22 puede proporcionar una medida de diámetro de esferoide pero no puede resolver la estructura general de la esferoide en el espacio 3D. Muchos factores contribuyen a estas limitaciones, incluyendo la penetración de la luz que en el esferoide; difusión de los tintes fluorescentes en el esferoide; emisión de señales fluorescentes de tintes fluorescentes excitados dentro o en la superficie opuesta de la esferoide debido a la fuerte absorción y dispersión; y resolución de profundidad de estas modalidades de imagen. Esto conduce a menudo a una medida incorrecta de volumen. Desarrollo de la base necrótica en esferoides imita necrosis en vivo tumores6,10,15,19,25. Esta característica patológica es probable que en 2D de la célula las culturas19,25,27,28. Con un tamaño de esferoide más de 500 μm de diámetro, una estructura concéntrica de tres capas, como una capa externa de células de la proliferación, una capa intermedia de células quiescentes y un núcleo necrótico, puede observarse en el esferoide6,10 ,15,19,25, debido a la falta de oxígeno y nutrientes. Imágenes de fluorescencia de células vivas y muertas son el acercamiento estándar para identificar el límite de la base necrótica. Sin embargo, una vez más, penetraciones de estos tintes fluorescentes y luz visible obstaculizan el potencial de la sonda en el núcleo necrótico para monitorear su desarrollo en su forma real.
Una alternativa 3D de modalidad, tomografía de coherencia óptica (OCT) es introducida para caracterizar los esferoides tumorales. OCT es una técnica Biomédica de imagen que es capaz de adquirir datos 3D etiqueta-libre, no-destructivo de hasta 1-2 mm profundidad en tejidos biológicos29,30,31,32,33 ,34. OCT emplea interferometría de baja coherencia para detectar señales de back-esparcidas de diferentes profundidades de la muestra y proporciona imágenes profundidad resuelto reconstruidas en resoluciones espaciales de nivel de micrones en direcciones laterales y verticales. OCT ha sido adoptado ampliamente en Oftalmología35,36,37 y angiografía38,39. Estudios previos han utilizado OCT para observar la morfología de in vitro esferoides de tumor en la matriz de la membrana del sótano (p. ej., Matrigel) y evaluar sus respuestas a la terapia fotodinámica40,41. Recientemente, nuestro grupo estableció una plataforma imágenes del OCT de alto rendimiento para sistemáticamente monitorear y cuantificar la cinética de crecimiento de esferoides tumorales 3D en placas de varios pocillos42. Cuantificación volumétrica precisa de esferoides tumorales 3D usando un voxel contando enfoque y detección de tejido necrótico etiqueta-libre en los esferoides basado en contraste intrínseco atenuación óptica fueron demostrados. Este artículo describe los detalles de cómo la plataforma de proyección de imagen de OCT fue construida y utilizada para obtener imágenes 3D de alta resolución de esferoides tumorales. Se describe el paso a paso análisis cuantitativo de la cinética de crecimiento de esferoides tumorales 3D, incluyendo las medidas exactas de volúmenes y diámetro del esferoide. También, se presenta el método de la detección no destructiva de las regiones de tejido necrótico mediante OCT, basado en el contraste de la atenuación óptica intrínseca.
Protocolo
1. preparación de las células
- Obtener líneas de células de un proveedor calificado.
Nota: Verifique que las células de las líneas celulares de interés pueden formar esferoide en los medios de cultivo o con la ayuda de un sustrato (matriz de la membrana basal como Matrigel). Buscar en la literatura9 o realizar una ronda de un pre experimento para una revisión. - Descongelar las células congeladas siguiendo el procedimiento específico suministrado por el proveedor de la línea celular. Un procedimiento general puede encontrarse en otra parte43.
- Las células de 1-2 pasos en frascos de cultivo de 25 cm2 de la cultura. Las células son entonces listas para usar para el cultivo celular 3D.
- Monitorear el estado de salud de las células diariamente y mantenerlos en una incubadora en condiciones estándar (37 ° C, 5% CO2, 95% de humedad). Actualizar los medios de comunicación según sea necesario.
Nota: El medio de cultivo consiste en DMEM (glucosa de 4.5 g/L), 1% antibiótico-antimicótico, 10% suero bovino fetal. Células de subcultivo antes de llegar a confluencia en el frasco de cultivo. Siga la pauta de cultura celular suministrada por el proveedor. Un procedimiento general puede encontrarse elsehwhere44. - Realizar cultivos celulares 3D en placas de varios pocillos basadas en el siguiente protocolo general9.
- Retire los medios de cultivo del frasco de cultivo y lávela con esterilizada tamponada de fosfato salino (PBS, calentado a 37 ° C).
- Resuspender las células añadiendo 1 mL de ácido etilendiaminotetracético de tripsina (EDTA, 0,5%) en el matraz por 3 minutos. Luego, añada los medios de cultivo para diluir la tripsina.
- Transferir la suspensión de células en un tubo de centrífuga de 15 mL y centrifugar durante 5 min a 500 x g y temperatura ambiente.
- Quite el sobrenadante y resuspender las células con 4 mL de medio de cultivo precalentada. Tomar con pipeta una gota de muestra en un hemocitómetro para recuento celular determinar la concentración de células. Diluir las células a una concentración adecuada para la siembra (por ejemplo, 3.000 células/mL).
Nota: Optimizar la concentración celular inicial del esferoide para cada línea celular y cada tipo de placa multi-bien (96-well, 384-bien o 1536-well). - Células de semillas en una placa varios pocillos de fondo redondo fijación ultra baja (ULA). Añadir 200 μL de suspensión de células en cada pocillo a la concentración de 3.000 células/mL para que cada bien tiene cerca de 600 células.
- En RT, centrifugar la placa entera usando un adaptador de placa por 7 min, justo después de la siembra, a una velocidad de 350 x g o la velocidad más baja disponible.
Nota: La centrífuga ayuda a reunir las células para el centro del pozo para facilitar formando un esferoide único y uniforme. El paso de la centrifugadora se realiza sólo una vez al principio para formar los esferoides tumorales. No se repite cuando los esferoides tumorales comienzan a crecer. - Mantener la placa multi-bien a 37 ° C y 5% de CO2 en una incubadora de cultivo y actualizar los medios de cultivo cada 3 días.
Nota: Tiempo de crecimiento puede variar de las condiciones de cultivo 3D diferentes. En nuestro estudio, 3.000 células/mL se utiliza para U-87 MG y HCT 116 líneas celulares en placas de 96 pozos, para que pueda crecer el esferoide a ~ 500 μm en 4\u20127 días para HCT 116 células. Considerar la adición de suplementos de los medios de comunicación y factores de crecimiento para modelos diferentes del esferoide, basado en el general protocolo cultura 3D. - Realizar proyección de imagen de OCT de esferoides tumorales cada días 3\u20124 para un estudio longitudinal de su crecimiento.
Nota: Los puntos de tiempo recomendado para la proyección de imagen de OCT sería día 4, día 7, día 11, día 14, día 18 y el día 21.
2. alto rendimiento OCT plataforma de imágenes
Nota: Véase referenciada trabajo29,30,31,32,33,34 para una revisión exhaustiva de los principios y aplicaciones de PTU. Ver figura 1 y Huang et al. 42 para más detalles del medida OCT sistema utilizado en este estudio de imagen.
- Elegir una fuente de luz banda ancha adecuada para el sistema de OCT para la proyección de imagen de esferoide tumor.
Nota: Aquí, un diodo superluminescent (SLD, figura 1A,B) con una longitud de onda central de ~ 1.320 nm y ~ 110 ancho de banda nm fue utilizado como una fuente de luz de banda ancha. - Construir el brazo de referencia y el brazo de muestra del sistema OCT siguiendo los esquemas (vea la figura 1A,B para más detalles). Vea la Tabla de materiales para obtener una lista de componentes ópticos para construir el sistema de OCT. Asegúrese de que la longitud del camino óptico del brazo de referencia y brazo de muestra son muy igualados.
- Construir el espectrómetro, incluyendo un colimador, una reja, una lente de F-theta y una cámara de barrido de línea (ver figura 1 configuración34) para detalles de diseño del espectrómetro del octubre de forma alternativa, seleccione un espectrómetro comercial que coincide con la longitud de onda central de la fuente de luz. Asegúrese de que el espectrómetro esté alineado correctamente para cubrir el ancho de banda láser toda, para lograr la eficacia de la colección de fotones alta y proporcionar lento lavado-hacia fuera del patrón de interferencia.
- Caracterizar el rendimiento del sistema OCT, incluyendo las siguientes métricas como muestra brazo, total profundidad, dependiente de la profundidad sensibilidad, resolución axial, profundidad de foco y lateral la resolución de la imagen. Colocar un reflector débil (p. ej., un espejo con un filtro de densidad neutra) como muestra para medir la profundidad del foco, dependiente de la profundidad sensibilidad y resolución axial. Coloque un objetivo de USAF resolución prueba tabla como muestra para comprobar la resolución lateral.
Nota: Ver referencias34,45 definiciones de métricas de rendimiento de OCT y protocolos para caracterizar estas métricas45. Vea la tabla 1 para una lista de parámetros medidos por el sistema OCT personalizado utilizado en nuestro estudio. - Seleccione una etapa de traducción motorizados para proporcionar el movimiento horizontal de la placa bien varios esferoides tumorales de imagen en diferentes pozos (ver figura 1B). Uso de una etapa con un recorrido de más de 108 mm x 72 mm para una exploración completa de todos los pozos de la placa y bien. Utilizar una etapa de traducción motorizado 2D o 3D con software de control para permitir la ubicación precisa de cada pozo y automatización del sistema de OCT para la proyección de imagen de alto rendimiento.
- Utilice un adaptador de placa o un sostenedor de la placa de diseño (por la impresión en 3D) para sostener la placa multi-bien en una posición fija.
- Corregir la inclinación y la rotación de la placa multi-bien con un 2D fase y una fase de rotación montado en el escenario traslacional (ver figura 1 D), de inclinación antes de realizar cualquier imagen de OCT para minimizar la variación del plano de enfoque desde diferentes pozos. Utilizar D2 D11, B6, D6, G6 como los pozos guías al seguimiento de su posición relativa en las imágenes de OCT (figura 1A).
- Ajustar la rotación de la placa para asegurar que los bordes de la placa están paralelos a la dirección de movimiento del escenario para que los pozos queden en la misma posición horizontal en las imágenes de OCT (Figura 1E). Ajuste la inclinación de la placa para ser paralelo a la mesa óptica de modo que los pozos siguen en las mismas localizaciones verticales para OCT (Figura 1F) la proyección de imagen.
Nota: Ajuste de la inclinación ángulo de enfoque ayudan a optimizar la calidad de la imagen de OCT para todos los pozos. Sin embargo, las variaciones de la altura de los medios de cultivo en diferentes pozos pueden causar cambios en el trayecto óptico que puede llevar al desenfoque de la imagen del esferoide. Enfoque automático se puede implementar para controlar el plano focal de la proyección de la imagen de OCT para lograr calidad de imagen optimizada. El paso de ajuste no resuelve la mala calidad de imagen de OCT de la esferoide de tumor debido a las siguientes cuestiones: el esferoide descentrado debido a la ubicación inicial de siembra; elevación del esferoide cuando en biofabricated matrices extracelulares; calidad de pobre placa con grandes variaciones de la altura de fondos bien. Control de software adicional con funciones de enfoque automático o uno mismo-alineación puede ser implementada para optimizar el rendimiento del OCT, sistema de imagen. - Utilice un programa informático personalizado para controlar la adquisición de imágenes de OCT y el movimiento de la etapa para recopilar datos de cada uno bien secuencialmente.
3. OCT análisis y procesamiento de esferoides tumorales
- En el día de la proyección de imagen de OCT de esferoides tumorales, quite la placa multi-bien de la incubadora. Transferir la placa multi-bien debajo de la OCT, sistema de imagen. Colóquela sobre el adaptador de la placa.
Nota: Proyección de imagen de OCT de esferoides tumorales puede realizarse con la tapa de placa de poliestireno o desactivar. Sin embargo, las condensaciones de agua en la tapa debido a la evaporación de los pozos pueden afectar la transmisión de la luz y distorsionan la trayectoria de la luz, produciendo menos imágenes de OCT óptima de los esferoides. - Ajuste la altura de la placa moviendo a lo largo de la dirección z de la etapa de la traducción. Mantener la posición del plano focal en ~ 100-200 μm por debajo de la superficie superior de cada esferoide, para minimizar el efecto del no-uniforme depth-wise perfil focal.
- Establecer una adecuada gama de la exploración de OCT (por ejemplo, 1 mm x 1 mm) en el software a la medida para cubrir el esferoide tumor entero según sus etapas de desarrollo. Haga clic en Guardar parámetros para guardar el ajuste.
- Utilice el software personalizado para la adquisición de imágenes en 3D OCT de esferoides tumorales uno por uno para todos los pozos de la placa que contiene esferoides. Haga clic en el botón de vista previa para ver la imagen de previsualización y haga clic en el botón de adquisición para adquirir la imagen de OCT.
Nota: Asegúrese de que los datos del esferoide de OCT se recogen cuando el escenario no está en movimiento. El esferoide está generalmente situado en el centro del U-bien. Sin embargo, el esferoide puede ser cambiado de puesto en los medios de cultivo cuando la etapa de aceleración o desaceleración debido a la inercia de la esferoide en los medios de cultivo. - Proceso 3D OCT conjuntos de datos de esferoides tumorales para generar imágenes estructurales de OCT con un código de procesamiento personalizado de C++. Para un diagrama de flujo del post-processing de los datos de la OCT, vea figura 2A .
Nota: Ver Figura 4A para las imágenes estructurales OCT 3D generadas.- Véase el capítulo 5 de Drexler y Fujimoto34 y Jian et al. 46 para las descripciones de detalle de pasos post-processing de los datos de OCT. Calibrar el tamaño del pixel en las tres dimensiones. Volver a la escala de las imágenes estructurales de OCT en escalas corregidas.
Nota: La distancia en la dirección axial (dirección z) de las imágenes de OCT es una medida de la diferencia de camino óptico entre el brazo de referencia y el brazo de muestra. Así, el índice de refracción de la muestra (n) debe tomarse en consideración cuando esté calibrando el tamaño del pixel en la dirección axial para reescalado. En nuestro estudio, usamos n = 1.37 como el índice de refracción del tumor esferoide42.
- Véase el capítulo 5 de Drexler y Fujimoto34 y Jian et al. 46 para las descripciones de detalle de pasos post-processing de los datos de OCT. Calibrar el tamaño del pixel en las tres dimensiones. Volver a la escala de las imágenes estructurales de OCT en escalas corregidas.
- Generar el collage de imágenes de esferoide utilizando imágenes 2D de la OCT en tres planos XY, XZ y YZ transversales a través el centroide de la esferoide. Ver figura 3-E para la salida del representante de collages de imágenes de esferoide. Realizar el registro de la imagen para todos los esferoides, usando la función MATLAB dftregistration47, para asegurar que los centroides de la esferoide se encuentran aproximadamente en el mismo lugar.
- Obtener la representación 3D de la esferoide usando un software comercial o costumbre.
Nota: Los pasos siguientes muestran cómo obtener la representación 3D de esferoides tumorales usando un software comercial.- Cargar los datos 3D de OCT en el software.
- Haga clic en el panel de Surpass . A continuación, haga clic en Agregar nuevo volumen. Elija el modo de mezcla a utilizar para la representación 3D.
- Ajuste el ángulo de visión arrastrando la imagen con el puntero del ratón.
4. cuantificación morfológica de esferoides tumorales 3D
Nota: Un código personalizado escrito en MATLAB procesa esta cuantificación. Haga clic en el botón Ejecutar para iniciar el proceso. Ver figura 2B para el diagrama de flujo de los pasos de cuantificación morfológica de esferoides.
- Cuantificar esferoide diámetro, altura y diámetro base.
- Seleccione imágenes 2D de la OCT en tres transversales planos XY, XZ y YZ que cruzan el centroide de la esferoide.
- Medir el diámetro y la altura del esferoide en los planos XY y XZ, respectivamente.
- Calcular esferoide basada en diámetro volumen con:
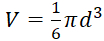 , con una presunción de la forma esférica del tumor.
, con una presunción de la forma esférica del tumor.
- Cuantificar el volumen del voxel-basado del esferoide.
- Aplicar un filtro promediador 3D de datos estructurales de OCT del esferoide para quitar manchas.
- Segmento de esferoides tumorales utilizando el filtro de48 de detección Canny del borde, cuadro por cuadro, con un umbral adecuado separar la región de esferoide tumor desde bien abajo.
- Grupo de voxels conectivo para datos 3D (ver función incorporada: bwconncomp).
- Calcular la distancia media entre cada voxel conectiva en el grupo y el centroide del esferoide (elegido manualmente), para cada grupo. Identificar la región esferoide como el grupo con la media distancia.
- Contar el número de voxels en la región de esferoide y luego multiplicar por el volumen real de un individuo voxel (volumen/voxel), produciendo el volumen total de la esferoide.
5. dead Cell región detección de esferoides tumorales 3D
Nota: En un medio homogéneo, intensidad de back-esparcidas OCT detectado en función de la profundidad ((z)) puede ser descrito por la ley de Beer-Lambert49: 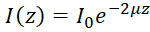 , donde z representa la profundidad, μ es la atenuación óptica coeficiente, y I0 es la intensidad incidente a la muestra. Por lo tanto el coeficiente de atenuación óptica derivada se puede expresar como:
, donde z representa la profundidad, μ es la atenuación óptica coeficiente, y I0 es la intensidad incidente a la muestra. Por lo tanto el coeficiente de atenuación óptica derivada se puede expresar como: 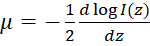 . Desde imágenes de OCT se trazan a menudo en una escala logarítmica, se puede recuperar la pendiente del perfil de intensidad de OCT para obtener el coeficiente de atenuación óptica. Ver figura 2 para un diagrama de flujo de la generación de los mapas de atenuación óptica.
. Desde imágenes de OCT se trazan a menudo en una escala logarítmica, se puede recuperar la pendiente del perfil de intensidad de OCT para obtener el coeficiente de atenuación óptica. Ver figura 2 para un diagrama de flujo de la generación de los mapas de atenuación óptica.
- Realizar la segmentación para eliminar no deseadas regiones fuera del esferoide. Realizar 3D filtro promedio para suprimir el ruido moteado que es inherente a las imágenes de OCT.
- Pixel-wise obtener coeficientes de atenuación óptica por lineal el perfil de intensidad escala log OCT sobre cierta gama de la profundidad (ventana móvil), extracto de su pendiente y la pendiente se multiplican por -1/2.
Nota: El coeficiente de atenuación en cada voxel dentro de la región segmentada esferoide se calcula basado en la pendiente del perfil de intensidad de OCT en una ventana de profundidad 10-voxel (~ 40 μm en profundidad), con el vóxel situado en el centro de la ventana. - Aplicar los métodos anteriormente (pasos 5.1 y 5.2) para cada exploración axial en un marco y cada marco en un conjunto de datos 3D que contiene la región segmentada esferoide hasta que se calculan los coeficientes de atenuación óptica para todos los vóxeles de la región de esferoide segmentado.
- Llevar a cabo la umbralización binaria para destacar la región de alta atenuación.
Nota: Ver Huang et al. 42 para la determinación del umbral de la región de alta atenuación con análisis del histograma. - Destacar el mapa binarizada atenuación óptica en la imagen original a la región de muertos de la célula (fusión) de la etiqueta. Generar la imagen 3D renderizado del mapa de atenuación mezclado para visualizar la distribución 3D de la región de la muerte celular.
6. histología e inmunohistoquímica
Nota: La histología y la inmunohistoquímica (IHQ) manchados imágenes de esferoides tumorales se obtienen para correlacionar con los resultados de OCT correspondientes.
- En puntos de tiempo seleccionado, seleccione esferoides de tumor de 1-2 en la placa varios pocillos histología y tinción IHQ. Utilice una pipeta con la punta de la pipeta de 1 mL para transferir la esferoide del pozo a un tubo de centrífuga de 1.5 mL.
Nota: Cortar la punta de la pipeta de 1 mL antes de la transferencia para asegurarse de que la abertura de la punta es más grande que el tamaño de la esferoide del tumor para evitar dañar la estructura del esferoide. - Recoger cada esferoide de tumor en un tubo de microcentrífuga de 1,5 mL solo con formaldehído al 10% y solución de 48 h.
- Realizar los procesos IHC e histología para cada esferoide, utilizando parafina estándar técnicas de incrustación.
Nota: La mancha 5 secciones μm de espesor de esferoides tumorales de hematoxilina y eosina (H & E) y terminal deoxynucleotidyl transferase dUTP nick end etiquetado detección de apoptosis (TUNEL). Una contratinción de hematoxilina se aplica al TUNEL. Un escáner de diapositivas digital fue utilizado para analizar la muestra teñida y obtener alta resolución histológica e imágenes IHC.
Resultados
Alto rendimiento proyección de imagen de tomografía de coherencia óptica de esferoides en una placa de 96 pocillos
Figura 3 muestra el resultado de HT-OCT la exploración de una placa de 96 pocillos con HCT 116 esferoides de tumor el día 3. La exploración secuencial de la placa todo comienza desde el pozo de abajo a la derecha (H12). Figura 3B muestra el diagrama de f...
Discusión
Actividad tumoral es muy importante a su estructura morfológica. Similar a control de curva de crecimiento característica de cultivos celulares 2D, seguimiento de la curva de crecimiento de esferoides tumorales 3D es también un enfoque convencional para caracterizar el comportamiento a largo plazo de crecimiento de esferoide para diferentes líneas celulares. En particular, podemos caracterizar la respuesta de drogas mediante el análisis de degradación del tumor o recrecimiento del tumor directamente reflejada en la...
Divulgaciones
Los autores no divulgar ningún interés concurrentes.
Agradecimientos
Este trabajo fue financiado por la NSF concede IDBR (DBI-1455613), PFI:AIR-TT (PII-1640707), NIH fondo de inicio de becas R21EY026380, R15EB019704 y R01EB025209 y la Universidad de Lehigh.
Materiales
| Name | Company | Catalog Number | Comments |
| Custom Spectral Domain OCT imaging system | Developed in our lab | ||
| Superluminescent Diode (SLD) | Thorlabs | SLD1325 | light source |
| 2×2 single mode fused fiber coupler, 50:50 splitting ratio | AC Photonics | WP13500202B201 | |
| Reference Arm | |||
| Lens Tube | Thorlabs | ||
| Adapter | Thorlabs | ||
| Collimating Lens | Thorlabs | AC080-020-C | |
| Focusing Lens | Thorlabs | ||
| Kinematic Mirror Mount | Thorlabs | ||
| Mirror | Thorlabs | ||
| 1D Translational Stage | Thorlabs | ||
| Continuous neutral density filter | Thorlabs | ||
| Pedestrial Post | Thorlabs | ||
| Clamping Fork | Thorlabs | ||
| Sample Arm | |||
| Lens Tube | Thorlabs | ||
| Adapter | Thorlabs | ||
| Collimating Lens | Thorlabs | AC080-020-C | |
| Galvanometer | Thorlabs | ||
| Relay Lens | Thorlabs | AC254-100-C | two Relay lens to make a telescope setup |
| Triangle Mirror Mount | Thorlabs | ||
| Mirror | Thorlabs | ||
| Objective | Mitutoyo | ||
| Pedestrial Post | Thorlabs | ||
| Clamping Fork | Thorlabs | ||
| Polarization Controller | Thorlabs | ||
| 30mm Cage Mount | Thorlabs | ||
| Cage Rod | Thorlabs | ||
| Stage | |||
| 3D motorized translation stage | Beijing Mao Feng Optoelectronics Technology Co., Ltd. | JTH360XY | |
| 2D Tilting Stage | |||
| Rotation Stage | |||
| Plate Holder | 3D printed | ||
| Spectrometer | |||
| Lens Tube | Thorlabs | ||
| Adapter | Thorlabs | ||
| Collimating Lens | Thorlabs | AC080-020-C | |
| Grating | Wasatch | G = 1145 lpmm | |
| F-theta Lens | Thorlabs | FTH-1064-100 | |
| InGaAs Line-scan Camera | Sensor Unlimited | SU1024-LDH2 | |
| Name | Company | Catalog Number | Comments |
| Cell Culture Component | |||
| HCT 116 Cell line | ATCC | CCL-247 | |
| Cell Culture Flask | SPL Life Sciences | 70025 | |
| Pipette | Fisherbrand | 14388100 | |
| Pipette tips | Sorenson Bioscience | 10340 | |
| Gibco GlutaMax DMEM | Thermo Fisher Scientific | 10569044 | |
| Fetal Bovine Serum, certified, US origin | Thermo Fisher Scientific | 16000044 | |
| Antibiotic-Antimycotic (100X) | Thermo Fisher Scientific | 15240062 | |
| Corning 96-well Clear Round Bottom Ultra-Low Attachment Microplate | Corning | 7007 | |
| Gibco PBS, pH 7.4 | Thermo Fisher Scientific | 10010023 | |
| Gibco Trypsin-EDTA (0.5%) | Thermo Fisher Scientific | 15400054 | |
| Forma Series II 3110 Water-Jacketed CO2 Incubators | Thermo Fisher Scientific | 3120 | |
| Gloves | VWR | 89428-750 | |
| Parafilm | Sigma-Aldrich | P7793 | |
| Transfer pipets | Globe Scientific | 138080 | |
| Centrifuge | Eppendorf | 5702 R | To centrifuge the 15 mL tube |
| Centrifuge | NUAIRE | AWEL CF 48-R | To centrifuge the 96-well plate |
| Microscope | Olympus | ||
| Name | Company | Catalog Number | Comments |
| Histology & IHC | |||
| Digital slide scanner | Leica | Aperio AT2 | Obtain high-resolution histological images |
| Histology Service | Histowiz | Request service for histological and immunohistological staining of tumor spheroid | |
| Name | Company | Catalog Number | Comments |
| List of Commerical OCTs | |||
| SD-OCT system | Thorlabs | Telesto Series | |
| SD-OCT system | Wasatch Photonics | WP OCT 1300 nm | |
| Name | Company | Catalog Number | Comments |
| Software for Data Analyses | |||
| Basic Image Analysis | NIH | ImageJ | Fiji also works. |
| 3D Rendering | Thermo Fisher Scientific | Amira | Commercial software. Option 1 |
| 3D Rendering | Bitplane | Imaris | Commercial software. Option 2. Used in the protocol |
| OCT acquisition software | custom developed in C++. | ||
| Stage Control | Beijing Mao Feng Optoelectronics Technology Co., Ltd. | MRC_3 | Incorporated into the custom OCT acquisition code |
| OCT processing software | custom developed in C++. Utilize GPU. Incorporated into the custom OCT acquisition code. | ||
| Morphological and Physiological Analysis | custom developed in MATLAB |
Referencias
- Kola, I., Landis, J. Can the pharmaceutical industry reduce attrition rates?. Nature Reviews Drug Discovery. 3 (8), 711-716 (2004).
- Breslin, S., O'Driscoll, L. Three-dimensional cell culture: the missing link in drug discovery. Drug Discovery Today. 18, 240-249 (2013).
- Hickman, J. A., et al. Three-dimensional models of cancer for pharmacology and cancer cell biology: Capturing tumor complexity in vitro/ex vivo. Biotechnology Journal. 9 (9), 1115-1128 (2014).
- Sutherland, R. M. Cell and environment interactions in tumor microregions: the multicell spheroid model. Science. 240 (4849), 177-184 (1988).
- Mueller-Klieser, W. Three-dimensional cell cultures: from molecular mechanisms to clinical applications. American Journal of Physiology - Cell Physiology. 273, C1109-C1123 (1997).
- Friedrich, J., Seidel, C., Ebner, R., Kunz-Schughart, L. A. Spheroid-based drug screen: considerations and practical approach. Nature Protocols. 4 (3), 309-324 (2009).
- Tung, Y. -. C., et al. High-throughput 3D spheroid culture and drug testing using a 384 hanging drop array. The Analyst. 136 (3), 473-478 (2011).
- Vinci, M., et al. Advances in establishment and analysis of three-dimensional tumor spheroid-based functional assays for target validation and drug evaluation. BMC biology. 10, 29 (2012).
- LaBarbera, D. V., Reid, B. G., Yoo, B. H. The multicellular tumor spheroid model for high-throughput cancer drug discovery. Expert Opinion on Drug Discovery. 7, 819-830 (2012).
- Pampaloni, F., Ansari, N., Stelzer, E. H. K. High-resolution deep imaging of live cellular spheroids with light-sheet-based fluorescence microscopy. Cell and Tissue Research. 352, 161-177 (2013).
- Lovitt, C. J., Shelper, T. B., Avery, V. M. Miniaturized three-dimensional cancer model for drug evaluation. Assay and Drug Development Technologies. 11 (7), 435-448 (2013).
- Wenzel, C., et al. 3D high-content screening for the identification of compounds that target cells in dormant tumor spheroid regions. Experimental Cell Research. 323 (1), 131-143 (2014).
- Astashkina, A., Grainger, D. W. Critical analysis of 3-D organoid in vitro cell culture models for high-throughput drug candidate toxicity assessments. Innovative tissue models for drug discovery and development. 69, 1-18 (2014).
- Edmondson, R., Broglie, J. J., Adcock, A. F., Yang, L. Three-dimensional cell culture systems and their applications in drug discovery and cell-based biosensors. Assay and Drug Development Technologies. 12 (4), 207-218 (2014).
- Gong, X., et al. Generation of multicellular tumor spheroids with microwell-based agarose scaffolds for drug testing. PLoS ONE. 10 (6), e0130348 (2015).
- Hoffmann, O. I., et al. Impact of the spheroid model complexity on drug response. Journal of biotechnology. 205, 14-23 (2015).
- Martinez, N. J., Titus, S. A., Wagner, A. K., Simeonov, A. High-throughput fluorescence imaging approaches for drug discovery using in vitroand in vivothree-dimensional models. Expert Opinion on Drug Discovery. 10, 1347-1361 (2015).
- Nath, S., Devi, G. R. Three-dimensional culture systems in cancer research: Focus on tumor spheroid model. Pharmacology, Therapeutics. 163, 94-108 (2016).
- Li, L., Zhou, Q., Voss, T. C., Quick, K. L., LaBarbera, D. V. High-throughput imaging: Focusing in on drug discovery in 3D. Methods. 96, 97-102 (2016).
- Ham, S. L., Joshi, R., Thakuri, P. S., Tavana, H. Liquid-based three-dimensional tumor models for cancer research and drug discovery. Experimental Biology and Medicine. 241 (9), 939-954 (2016).
- Kessel, S., et al. High-Throughput 3D Tumor Spheroid Screening Method for Cancer Drug Discovery Using Celigo Image Cytometry. Journal of Laboratory Automation. , 2211068216652846 (2016).
- Stock, K., et al. Capturing tumor complexity in vitro: Comparative analysis of 2D and 3D tumor models for drug discovery. Scientific Reports. 6, 28951 (2016).
- Thakuri, P. S., Ham, S. L., Luker, G. D., Tavana, H. Multiparametric analysis of oncology drug screening with aqueous two-phase tumor spheroids. Molecular Pharmaceutics. 13 (11), 3724-3735 (2016).
- Lin, R. Z., Chang, H. Y. Recent advances in three-dimensional multicellular spheroid culture for biomedical research. Biotechnology Journal. 3 (9-10), 1172-1184 (2008).
- Piccinini, F., Tesei, A., Arienti, C., Bevilacqua, A. Cancer multicellular spheroids: Volume assessment from a single 2D projection. Computer Methods and Programs in Biomedicine. 118 (2), 95-106 (2015).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: a systematic approach to enhance the biological relevance of data obtained. Scientific Reports. 6, 19103 (2016).
- Debnath, J., Brugge, J. S. Modelling glandular epithelial cancers in three-dimensional cultures. Nature Reviews Cancer. 5 (9), 675-688 (2005).
- Huang, D., et al. Optical coherence tomography. Science. 254 (5035), 1178-1181 (1991).
- Drexler, W., et al. Optical coherence tomography today: speed, contrast, and multimodality. Journal of Biomedical Optics. 19 (7), 071412 (2014).
- Fujimoto, J., Swanson, E. The development, commercialization, and impact of optical coherence tomography. Investigative Ophthalmology, Visual Science. 57 (9), (2016).
- Vakoc, B. J., Fukumura, D., Jain, R. K., Bouma, B. E. Cancer imaging by optical coherence tomography: preclinical progress and clinical potential. Nature Reviews Cancer. 12 (5), 363-368 (2012).
- Wojtkowski, M. High-speed optical coherence tomography: basics and applications. Applied optics. 49 (16), D30-D61 (2010).
- Drexler, W., Fujimoto, J. G. . Optical coherence tomography: technology and applications. , (2008).
- Geitzenauer, W., Hitzenberger, C. K., Schmidt-Erfurth, U. M. Retinal optical coherence tomography: past, present and future perspectives. British Journal of Ophthalmology. 95 (2), 171 (2011).
- Sakata, L. M., DeLeon-Ortega, J., Sakata, V., Girkin, C. A. Optical coherence tomography of the retina and optic nerve - a review. Clinical, Experimental Ophthalmology. 37 (1), 90-99 (2009).
- van Velthoven, M. E. J., Faber, D. J., Verbraak, F. D., van Leeuwen, T. G., de Smet, M. D. Recent developments in optical coherence tomography for imaging the retina. Progress in Retinal and Eye Research. 26 (1), 57-77 (2007).
- Kashani, A. H., et al. Optical coherence tomography angiography: A comprehensive review of current methods and clinical applications. Progress in Retinal and Eye Research. 60, 66-100 (2017).
- de Carlo, T. E., Romano, A., Waheed, N. K., Duker, J. S. A review of optical coherence tomography angiography (OCTA). International Journal of Retina and Vitreous. 1 (1), 5 (2015).
- Sharma, M., Verma, Y., Rao, K. D., Nair, R., Gupta, P. K. Imaging growth dynamics of tumour spheroids using optical coherence tomography. Biotechnology Letters. 29 (2), 273-278 (2006).
- Jung, Y., Nichols, A. J., Klein, O. J., Roussakis, E., Evans, C. L. Label-Free, Longitudinal Visualization of PDT Response In Vitro with Optical Coherence Tomography. Israel Journal of Chemistry. 52 (8-9), 728-744 (2012).
- Huang, Y., et al. Optical coherence tomography detects necrotic regions and volumetrically quantifies multicellular tumor spheroids. Cancer Research. 77 (21), 6011-6020 (2017).
- Spalteholz, W. . Über das Durchsightigmachen von menschlichen und tierischen Präparaten: nebst Anhang, Über Knochenfärbung. , (1911).
- Dodt, H. -. U., et al. Ultramicroscopy: three-dimensional visualization of neuronal networks in the whole mouse brain. Nature Methods. 4 (4), 331 (2007).
- Leitgeb, R., Hitzenberger, C., Fercher, A. F. Performance of fourier domain vs. time domain optical coherence tomography. Optics express. 11 (8), 889-894 (2003).
- Jian, Y., Wong, K., Sarunic, M. V. . Optical Coherence Tomography and Coherence Domain Optical Methods in Biomedicine XVII. , 85710Z (2013).
- Guizar-Sicairos, M., Thurman, S. T., Fienup, J. R. Efficient subpixel image registration algorithms. Optics Letters. 33 (2), 156-158 (2008).
- Canny, J. A computational approach to edge detection. IEEE Transactions on Pattern Analysis and Machine Intelligence. (6), 679-698 (1986).
- Vermeer, K. A., Mo, J., Weda, J. J. A., Lemij, H. G., de Boer, J. F. Depth-resolved model-based reconstruction of attenuation coefficients in optical coherence tomography. Biomedical Optics Express. 5 (1), 322-337 (2014).
- Klein, T., et al. Multi-MHz retinal OCT. Biomedical Optics Express. 4, 1890-1908 (2013).
- Klein, T., Huber, R. High-speed OCT light sources and systems [Invited]. Biomedical Optics Express. 8 (2), 828-859 (2017).
- Zhou, C., Alex, A., Rasakanthan, J., Ma, Y. Space-division multiplexing optical coherence tomography. Optics Express. 21, 19219-19227 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados