É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Longitudinal de morfológicas e fisiológicas, monitoramento de esferoides Tumor tridimensional usando a tomografia de coerência óptica
Neste Artigo
Resumo
Tomografia de coerência óptica (OCT), uma tecnologia de imagem tridimensional, foi usada para monitorar e caracterizar a cinética de crescimento de esferoides tumor multicelulares. Precisa quantificação volumétrica de esferoides tumor usando um voxel contando a abordagem e deteção de tecido livre de rótulo morto nos esferoides baseado no contraste intrínseco atenuação óptica, foram demonstradas.
Resumo
Esferoides de tumor foram desenvolvidas como um modelo de cultura tridimensional (3D) célula na descoberta de medicamentos de investigação e anti-câncer de câncer. No entanto, atualmente, elevado-throughput modalidades de imagem utilizando a deteção de campo ou fluorescência brilhante, são incapazes de resolver a estrutura geral 3D do spheroid tumor devido à limitada penetração de luz, difusão de corantes fluorescentes e profundidade-resolução. Recentemente, nosso laboratório demonstrou o uso de tomografia de coerência óptica (OCT), um rótulo livre e não-destrutiva de imagem 3D modalidade, para realizar a caracterização longitudinal de esferoides multicelulares tumor em uma placa de 96 poços. OCT foi capaz de obter informações morfológicas e fisiológicas 3D de esferoides de tumor que cresce até cerca de 600 µm de altura. Neste artigo, vamos demonstrar um sistema de imageamento de OCT (HT-OCT) de alto rendimento que verifica a placa toda multi bem e obtém os dados 3D OCT de esferoides tumor automaticamente. Descrevemos os detalhes das orientações de construção e sistema de HT-OCT no protocolo. A partir dos dados de OCT 3D, se pode visualizar a estrutura geral do spheroid com 3D renderizados e fatias ortogonais, caracterizar a curva de crescimento longitudinal do spheroid tumor baseado na informação morfológica de tamanho e volume e monitorar o crescimento de as regiões de mortos-célula no spheroid tumor baseado no contraste óptico atenuação intrínseca. Mostramos que, HT-OCT pode ser usado como uma modalidade de imagem de alto rendimento para droga triagem, bem como a caracterização de amostras de biofabricated.
Introdução
Câncer é a segunda principal causa de morte no mundo1. Desenvolvimento de medicamentos, alvejando câncer é de importância crucial para os pacientes. No entanto, estima-se que mais de 90% das novas drogas anti-câncer falhar na fase de desenvolvimento devido à falta de eficácia e toxicidade inesperada em ensaios clínicos2. Parte do motivo pode ser atribuída ao uso de modelos de cultura simples bidimensional (2D) célula para triagem composta, que fornecem resultados com valores preditivos limitados de compostos eficácia e toxicidade para as fases seguintes da droga descoberta2 , 3 , 4. recentemente, foram desenvolvidos modelos de esferoide tridimensional (3D) tumor para fornecer dados fisiológicos e farmacológicos clinicamente relevantes para drogas anti-câncer descoberta3,4,5 ,6,7,8,9,10,11,12,13,14, 15,16,17,18,19,20,21,22,23, 24,25. Desde que esses esferoides podem imitar propriedades específicas do tecido de tumores no vivo, como nutrientes e oxigênio núcleo gradiente, hipóxico, bem como drogas resistência19, o uso desses modelos pode potencialmente encurtar cronogramas de descoberta de drogas, reduzir os custos de investimento e trazer novos medicamentos para pacientes mais efetivamente. Uma abordagem crítica para avaliar composta eficácia no desenvolvimento de tumor 3D esferoide é monitorar o crescimento de esferoide e recorrência sob tratamentos9,26. Para fazer isso, caracterizações quantitativas da morfologia do tumor, envolvendo o seu diâmetro e volume, com modalidades de imagem de alta resolução, são imperativas.
Modalidades de imagem convencionais, como campo claro, contraste fase7,9,22,de24e fluorescência microscopia8,9,16, 18,22 pode fornecer uma medida do diâmetro do spheroid, mas não é possível resolver a estrutura geral do spheroid em espaço 3D. Muitos fatores contribuem para essas limitações, incluindo penetração da luz sondagem no spheroid; difusão dos corantes fluorescentes para o spheroid; emitindo sinais fluorescentes de corantes fluorescentes animados no interior ou na superfície oposta do spheroid devido à forte absorção e espalhamento; e profundidade-resolução destas modalidades de imagem. Isto conduz frequentemente a uma medida de volume imprecisas. Desenvolvimento do núcleo necrótico em esferoides imita necrose na vivo tumores6,10,15,19,25. Esta característica patológica é improvável reproduzida na célula 2D culturas19,25,,27,28. Com um tamanho de esferoide superior a 500 µm de diâmetro, uma estrutura de três camadas concêntrica, incluindo uma camada exterior de pilhas proliferating, uma camada intermediária de células quiescentes e um núcleo necrótico, pode ser observado no esferoide6,10 ,15,19,25, devido à falta de oxigênio e nutrientes. Imagem latente da fluorescência de pilha de vivo e morto é a abordagem padrão para rotular o limite do núcleo necrótico. No entanto, outra vez, Penetrações tanto destes corantes fluorescentes e luz visível impedem o potencial para sondar sobre o núcleo necrótico para monitorar seu desenvolvimento em sua forma real.
Uma modalidade de imagem de 3D alternativa, tomografia de coerência óptica (OCT) é introduzida para caracterizar os esferoides de tumor. OCT é uma técnica de imagem biomédica que é capaz de adquirir dados 3D etiqueta-livre, não-destrutiva de até 1-2 mm de profundidade em tecidos biológicos29,30,31,32,33 ,34. OCT emprega interferometria de baixa coerência para detectar sinais espalhados por trás de diferentes profundidades da amostra e fornece imagens reconstruídas profundidade-resolvido em nível de mícron resoluções espaciais nas direções laterais e verticais. OCT foi adotado extensamente em oftalmologia35,36,37 e angiografia38,39. Estudos anteriores têm usado a OCT para observar a morfologia do in vitro esferoides de tumor na matriz da membrana basal (por exemplo, Matrigel) e avaliar suas respostas a terapia fotodinâmica40,41. Recentemente, nosso grupo estabeleceu uma plataforma de imagem da OCT do elevado-throughput para sistematicamente monitorar e quantificar a cinética de crescimento de esferoides tumor 3D em placas multi bem42. Precisa quantificação volumétrica de esferoides tumor 3D usando um voxel contando a abordagem e deteção de tecido necrótico rótulo livre nos esferoides baseado no contraste intrínseco atenuação óptica foram demonstradas. Este artigo descreve os detalhes de como a plataforma de imagem OCT foi construída e utilizada para obter imagens 3D de alta resolução de esferoides de tumor. Os passo a passo análises quantitativas da cinética de crescimento de esferoides tumor 3D, incluindo medições precisas de diâmetro esferoide e volumes, é descrita. Além disso, o método da detecção não-destrutiva de regiões do tecido necrosado usando a OCT, baseado no contraste a atenuação óptica intrínseca é apresentado.
Protocolo
1. preparação das células
- Obter linhas de células de um fornecedor qualificado.
Nota: Verifique se as células a partir das linhas de células de interesse podem formar esferoide nos meios de cultura ou com a ajuda de um substrato (matriz de membrana basal como Matrigel). Olhar para a literatura9 ou realizar uma rodada de um pre-experimento de para uma verificação. - Descongele as células congeladas, seguindo o procedimento específico fornecido pelo fornecedor linha celular. Um procedimento geral pode ser encontrado em outro lugar43.
- Cultura de células para 1-2 passagens em frascos de cultura 25 cm2 . As células são então prontas para uso para cultura de células 3D.
- Monitorar o status de saúde das células todos os dias e mantê-las em uma incubadora em condições padrão (37 ° C, 5% de CO2, 95% de umidade). Atualize a mídia conforme necessário.
Nota: O meio de cultura é composto por DMEM (glicose de 4,5 g/L), 1% de antibiótico antimicótico, 10% de soro fetal bovino. Células de subcultura antes que eles atinjam a confluência no frasco de cultura. Siga a orientação de cultura celular fornecida pelo fornecedor. Um procedimento geral pode ser encontrado elsehwhere44. - Realize cultura de células 3D em placas multi bem com base no seguinte protocolo geral9.
- Retire o frasco de cultura com os meios de cultura e lavá-lo com soro fisiológico esterilizado tamponado de fosfato (PBS, aquecido a 37 ° C).
- Ressuspender as células, adicionando 1 mL de ácido etilenodiaminotetracético de tripsina (EDTA, 0,5%) para o balão por 3 min. Em seguida, adicione os meios de cultura para diluir a tripsina.
- Transferi a suspensão de células para um tubo de centrífuga de 15 mL e centrifugar durante 5 min à x 500 g e a temperatura ambiente.
- Remover o sobrenadante e ressuspender as células com 4 mL de meio de cultura previamente aquecido. Pipete uma gota da amostra para um hemocytometer para a contagem de células determinar a concentração de células. Dilua as células de concentração adequada para semeadura (por exemplo, 3.000 células/mL).
Nota: Otimize a concentração inicial de célula do spheroid para cada célula-linha e cada tipo de placa multi bem (96 poços, 384-bem ou bem-1536). - Células de sementes em uma placa multi bem redondo de fixação ultra baixo (ULA). Adicione 200 µ l de suspensão de células em cada poço na concentração de 3.000 células/mL para que cada um, bem, tem cerca de 600 células.
- No RT, centrifugar o prato inteiro usando um adaptador de placa para 7 min, logo após a semeadura, a uma velocidade de 350 x g ou menor velocidade disponível.
Nota: A centrífuga ajuda a reunir as células para o centro do poço para facilitar formando um esferoide único e uniforme. A etapa de centrifugação é executada apenas uma vez no início para formar os esferoides de tumor. Isso não se repetirá quando esferoides o tumor começarem a crescer. - Manter a placa multi bem a 37 ° C e 5% CO2 em uma incubadora de cultura e atualizar os meios de cultura a cada 3 dias.
Nota: O tempo de crescimento pode variar para condições diferentes de cultura 3D. Em nosso estudo, 3.000 células/mL é usado para ambos U-87 MG e HCT 116 linhas de células em placas de 96 poços, para que o spheroid pode crescer a ~ 500 μm em 4\u20127 dias para células 116 HCT. Considere a adição de suplementos de mídia e fatores de crescimento para os modelos diferentes de esferoide, com base no general protocolo de cultura 3D. - Realize imagens de OCT de esferoides de tumor em dias de 3\u20124 para um estudo longitudinal do seu crescimento.
Nota: Os pontos de tempo recomendado para a imagem latente OCT seria dia 4, dia 7, dia 11, dia 14, dia 18 e dia 21.
2. elevado-throughput OCT plataforma de imagem
Nota: Ver referenciado trabalho29,30,31,32,33,34 para uma revisão aprofundada dos princípios e aplicações da OCT. Ver Figura 1 e Huang et al . 42 para obter detalhes da OCT Personalizada sistema utilizado neste estudo de imagem.
- Escolha uma fonte de luz banda larga adequada para o sistema da OCT para a imagem latente de esferoide de tumor.
Nota: Aqui, um diodo superluminescent (SLD, figura 1A,B) com um comprimento de onda central da ~ 1.320 nm e ~ 110 largura de banda nm foi usada como uma fonte de luz de banda larga. - Construa a referência braço e braço de amostra do sistema OCT seguindo os esquemas (ver figura 1A,B para obter detalhes). Consulte a Tabela de materiais para obter uma lista de componentes ópticos para construir o sistema OCT. Certifique-se de que o comprimento do percurso óptico do braço de referência e braço de amostra são estreitamente alinhados.
- Construir o espectrômetro, incluindo um colimador, uma grade, uma lente F-theta e uma câmera de varredura de linha (ver Figura 1 para instalação34) para detalhes de design de espectrômetro de outubro como alternativa, selecione um espectrômetro comercial que corresponde a Centro comprimento de onda da fonte de luz. Certifique-se de que o espectrômetro está alinhado corretamente para cobrir a largura de banda inteira do laser, para alcançar a eficiência de coleta de fótons de alta e fornecer lento lavagem-fora do padrão de interferência.
- Caracteriza o desempenho do sistema OCT, incluindo as seguintes métricas tais como poder de braço de amostra, total profundidade profundidade dependente sensibilidade, resolução axial, profundidade de foco e lateral resolução de imagem. Coloque um refletor fraco (por exemplo, um espelho com um filtro de densidade neutra) como uma amostra para medir a profundidade dependente sensibilidade, resolução axial e a profundidade de foco. Coloque um alvo de gráfico de teste de resolução USAF como amostra para verificar a resolução lateral.
Nota: Ver referências34,45 para definições de métricas de desempenho da OCT e protocolos para caracterizar essas métricas45. Consulte a tabela 1 para obter uma lista de parâmetros medidos para o sistema de OCT personalizado utilizado em nosso estudo. - Selecione uma etapa de tradução motorizado para fornecer o movimento horizontal da placa multi bem de esferoides de tumor de imagem em diferentes poços (ver figura 1B). Use um palco com uma gama de viagens maior que 108 x 72 mm para garantir uma varredura completa de todas as cavidades da placa multi bem. Use um estágio da tradução motorizado 2D ou 3D com software de controle para permitir a localização precisa de cada poço e automação do sistema OCT para a imagem latente de alta produtividade.
- Use um adaptador de placa ou projetar um suporte da placa (por impressão 3D) para segurar a placa multi bem em uma posição fixa.
- Corrigi a inclinação e a rotação da placa multi bem usando um 2D inclinando o palco e uma fase de rotação montada no palco translacional (ver Figura 1 D), antes de realizar qualquer OCT de imagens para minimizar a variação do plano de foco de diferentes poços. Use D2 D11, B6, D6, G6 como orientadores poços ao monitorar suas posições relativas nas imagens OCT (figura 1A).
- Ajuste a rotação da placa para garantir que as bordas da placa são paralelas com a direção do movimento do palco para que os poços permanecem na mesma posição horizontal nas imagens OCT (Figura 1E). Ajuste a inclinação da placa para ser paralela à tabela óptica, para que os poços permanecem nos mesmos locais, verticais para PTU de imagem (Figura 1F).
Nota: O ajuste do ângulo de inclinação e foco ajudam a otimizar a qualidade de imagem da OCT para todos os poços. No entanto, variações da altura de meios de cultura em diferentes poços podem causar alterações no trajecto óptico que pode levar a desfocagem da imagem esferoide. Foco automático pode ser implementado para controlar o plano focal da OCT imaging para alcançar a qualidade de imagem otimizada. O passo de ajuste não resolver má qualidade de imagem de OCT do spheroid tumor devido as seguintes questões: o descentramento esferoide devido à localização de semeadura inicial; elevação de esferoide quando incorporado em matrizes extracelulares de biofabricated; qualidade de placa pobre com grandes variações da altura de fundos bem. Controle de software adicional, com funções de foco automático ou auto alinhamento pode ser implementada para otimizar o desempenho do sistema de imagem PTU. - Use um programa de computador personalizado para controlar a aquisição de imagens da OCT e o movimento de palco para coletar dados de cada bem sequencialmente.
3. out digitalização e processamento de esferoides de Tumor
- No dia da imagem de OCT de esferoides de tumor, leve o prato multi bem da incubadora. Transferi a placa multi bem sob a OCT sistema de imagem. Coloque-o em cima do adaptador de placa.
Nota: Imagem de OCT de esferoides de tumor pode ser realizada com a tampa de poliestireno placa on ou off. No entanto, a condensação de água na tampa devido a evaporação dos poços pode afetar a transmissão de luz e distorcer o trajeto da luz, rendendo menos imagens OCT ideais dos esferoides. - Ajuste a altura da placa, movendo ao longo da z-direção da fase de tradução. Manter a posição de plano focal em ~ 100-200 μm abaixo da superfície superior de cada spheroid, para minimizar o efeito do não-uniforme depth-wise perfil focal.
- Defina um OCT digitalização intervalo adequado (por exemplo, 1 x 1 mm) no software personalizado para cobrir o spheroid tumor inteiro de acordo com seus estágios de desenvolvimento. Clique em Salvar parâmetros para salvar a configuração.
- Use o software personalizado para adquirir imagens em 3D OCT de esferoides tumor um por um, para todos os poços da placa contendo esferoides. Clique no botão Preview para ver a imagem de visualização e clique no botão adquirir para adquirir a imagem da OCT.
Nota: Certifique-se que os dados de esferoide OCT são coletados quando a fase não está em movimento. O spheroid é geralmente localizado no centro do U-fundo bem. No entanto, o esferoide pode ser deslocada nos meios de cultura quando o palco está acelerando ou desacelerando devido a inércia do spheroid nos meios de cultura. - Processo 3D OCT datasets de esferoides de tumor para gerar imagens estruturais de OCT com um código personalizado processamento de C++. Ver Figura 2A para um fluxograma de pós-processamento de dados OCT.
Nota: Ver Figura 4A para 3D OCT estruturais imagens geradas.- Consulte o capítulo 5 de Drexler e Fujimoto34 e Jian et al 46 para descrições de detalhes das etapas de pós-processamento de dados OCT. Calibre o tamanho do pixel em todas as três dimensões. Re-dimensione as imagens estruturais OCT em escalas corrigidas.
Nota: A distância na direção axial (direção z) de imagens OCT é uma medida da diferença entre o braço de referência e o braço de amostra caminho óptico. Assim, o índice de refração da amostra (n) deve ser tomado em consideração quando calibrar o tamanho do pixel na direção axial reescalar. Em nosso estudo, usamos n = 1.37 como o índice de refração do tumor esferoide42.
- Consulte o capítulo 5 de Drexler e Fujimoto34 e Jian et al 46 para descrições de detalhes das etapas de pós-processamento de dados OCT. Calibre o tamanho do pixel em todas as três dimensões. Re-dimensione as imagens estruturais OCT em escalas corrigidas.
- Gere a colagem de imagens de esferoide usando imagens 2D OCT em três planos XY, XZ e YZ transversais através do centroide do spheroid. Ver Figura 3–E para a saída de representante de colagens de imagens de esferoide. Execute o registro de imagem para todos os esferoides, usando o MATLAB função dftregistration47, para garantir que os centroides de todos o spheroid estão localizados aproximadamente no mesmo local.
- Obter a renderização 3D do spheroid usando um software comercial ou personalizado.
Nota: As etapas a seguir mostram como obter a renderização 3D de esferoides tumor usando um software comercial.- Carrega os dados de OCT 3D no software.
- Clique no painel Surpass . Em seguida, clique em Adicionar novo Volume. Escolha o Blend mode para usar para renderização 3D.
- Ajuste o ângulo de visão, arrastando a imagem usando o ponteiro do mouse.
4. quantificação morfológica do Tumor 3D esferoides
Nota: Um código personalizado escrito em MATLAB processa esta quantificação. Clique no botão executar para iniciar o processo. Ver Figura 2B para o fluxograma das etapas de quantificação morfológica dos esferoides.
- Quantificar os volumes baseado em diâmetro, altura e diâmetro de esferoide.
- Selecione imagens 2D OCT em três planos XY, XZ e YZ transversais que cruzam o centroide do spheroid.
- Medir o diâmetro e a altura do spheroid em planos XY e XZ, respectivamente.
- Cálculo baseado no diâmetro esferoide volume usando:
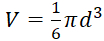 , com a presunção da forma esférica do tumor.
, com a presunção da forma esférica do tumor.
- Quantificar volume de esferoide baseada em voxel.
- Aplica um filtro médio 3D de dados estruturais OCT de esferoide para remover manchas.
- Segmento de esferoides tumor usando o filtro de48 de deteção de borda Canny, quadro a quadro, com um limite adequado, separa a região de esferoide tumor bem fundo.
- Grupo conjuntivo voxels de dados 3D (ver função built-in: bwconncomp).
- Calcule a distância média entre cada voxel conectivo no grupo e o centroide de esferoide (escolhido manualmente), para cada grupo. Identifica a região de esferoide como o grupo com a distância média mínima.
- Contar o número de voxels dentro da região de esferoide e então multiplicar pelo volume real de um voxel individual (volume/voxel), rendendo o volume total do spheroid.
5. mortos-célula região deteção de esferoides Tumor 3D
Nota: Em um meio homogêneo, intensidade de dispersão traseira OCT detectada em função da profundidade (eu(z)) pode ser descrita pela lei de Beer-Lambert49: 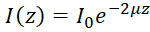 , onde z representa a profundidade, μ é a atenuação óptica coeficiente, e 0 a intensidade incidente à amostra. Portanto, o coeficiente de atenuação óptica derivada pode ser expressa como:
, onde z representa a profundidade, μ é a atenuação óptica coeficiente, e 0 a intensidade incidente à amostra. Portanto, o coeficiente de atenuação óptica derivada pode ser expressa como: 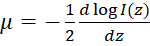 . Desde imagens OCT frequentemente são plotadas em uma escala logarítmica, a inclinação do perfil OCT intensidade pode ser recuperada para derivar o coeficiente de atenuação óptica. Consulte a Figura 2 para um fluxograma da geração de mapas de atenuação óptica.
. Desde imagens OCT frequentemente são plotadas em uma escala logarítmica, a inclinação do perfil OCT intensidade pode ser recuperada para derivar o coeficiente de atenuação óptica. Consulte a Figura 2 para um fluxograma da geração de mapas de atenuação óptica.
- Realize segmentação para remover regiões indesejadas fora o spheroid. Execute 3D filtro médio para suprimir o ruído speckle inerente em imagens da OCT.
- Obter pixel-wise coeficientes de atenuação óptica por linear encaixe o perfil de intensidade-escala logarítmica OCT durante um determinado intervalo de profundidade (janela em movimento), extrair sua encosta e multiplique a inclinação por -1/2.
Nota: O coeficiente de atenuação em cada voxel dentro da região segmentada esferoide é calculado com base na encosta do perfil de intensidade OCT em uma janela de profundidade de 10-voxel (~ 40 μm em profundidade), com o voxel localizado no meio da janela. - Aplica os métodos acima (etapas 5.1 e 5.2) a cada varredura axial em um frame e cada frame em um conjunto de dados 3D contendo região segmentada esferoide até calculam-se os coeficientes de atenuação óptica para todos os voxels da região segmentada esferoide.
- Execute a binária limiarização para destacar a região de alta atenuação.
Nota: Ver Huang et al. 42 para a determinação do limiar da região alta-atenuação usando análise de histograma. - Destaca-se o mapa de atenuação óptica binarized na imagem original para rotular a região mortos-célula (mistura). Gere a imagem em 3D-rendered do mapa para visualizar a distribuição 3D da região mortos-célula atenuação misturado.
6. histologia e imunohistoquímica
Nota: Histologia e imunohistoquímica (IHC) manchado imagens de esferoides de tumor são obtidos para correlacionar com os resultados correspondentes da OCT.
- Nos pontos de tempo selecionado, selecione esferoides de tumor de 1-2 da placa de multi bem para histologia e IHC coloração. Use uma pipeta com as pontas de pipeta de 1 mL para transferir o spheroid do poço para um tubo de centrífuga de 1,5 mL.
Nota: Corte a ponta da pipeta de 1 mL antes da transferência para garantir que a abertura da ponta é maior que o tamanho do spheroid do tumor para não danificar a estrutura do spheroid. - Recolha cada esferoide de tumor em um tubo de microcentrifuga único 1,5 mL cheio com formol 10% e correção para 48 h.
- Execute os processos IHC e histologia para cada esferoide, usando padrão parafina incorporando técnicas.
Nota: Mancha 5 μm espessas seções de esferoides de tumor por hematoxilina e eosina (H & E) e terminal deoxynucleotidyl transferase do dUTP nick fim rotulando a deteção de apoptosis (TUNEL). Um counterstaining de hematoxilina é aplicado ao TUNEL. Um scanner de slides digital foi usado para digitalizar a amostra manchada e obter alta resolução histológica e imagens IHC.
Resultados
Imagem de tomografia computadorizada coerência óptica alto Throughput de esferoides em uma placa de 96 poços
A Figura 3 exibe o resultado de HT-OCT digitalização de uma placa de 96 poços com 116 HCT esferoides de tumor no dia 3. A varredura sequencial da placa inteira começa a partir do poço do canto inferior direito (H12). Figura 3B mostra o fluxograma da implemen...
Discussão
Atividade de tumor é altamente relevante para sua estrutura morfológica. Semelhante à curva de crescimento característica de culturas celulares 2D de monitoramento, acompanhamento da curva de crescimento de esferoides tumor 3D é também uma abordagem convencional para caracterizar o comportamento de crescimento a longo prazo do esferoide para linhas celulares diferentes. Notavelmente, nós pode caracterizar a resposta de drogas através da análise de degradação de tumor ou tumor regrowth reflectida diretamente na...
Divulgações
Os autores não divulgar nenhum interesse de concorrente.
Agradecimentos
Este trabalho foi apoiado pela NSF concede IDBR (DBI-1455613), PFI:AIR-TT (PII-1640707), fundo de inicialização do NIH grants Lehigh University, R15EB019704 e R01EB025209 e R21EY026380.
Materiais
| Name | Company | Catalog Number | Comments |
| Custom Spectral Domain OCT imaging system | Developed in our lab | ||
| Superluminescent Diode (SLD) | Thorlabs | SLD1325 | light source |
| 2×2 single mode fused fiber coupler, 50:50 splitting ratio | AC Photonics | WP13500202B201 | |
| Reference Arm | |||
| Lens Tube | Thorlabs | ||
| Adapter | Thorlabs | ||
| Collimating Lens | Thorlabs | AC080-020-C | |
| Focusing Lens | Thorlabs | ||
| Kinematic Mirror Mount | Thorlabs | ||
| Mirror | Thorlabs | ||
| 1D Translational Stage | Thorlabs | ||
| Continuous neutral density filter | Thorlabs | ||
| Pedestrial Post | Thorlabs | ||
| Clamping Fork | Thorlabs | ||
| Sample Arm | |||
| Lens Tube | Thorlabs | ||
| Adapter | Thorlabs | ||
| Collimating Lens | Thorlabs | AC080-020-C | |
| Galvanometer | Thorlabs | ||
| Relay Lens | Thorlabs | AC254-100-C | two Relay lens to make a telescope setup |
| Triangle Mirror Mount | Thorlabs | ||
| Mirror | Thorlabs | ||
| Objective | Mitutoyo | ||
| Pedestrial Post | Thorlabs | ||
| Clamping Fork | Thorlabs | ||
| Polarization Controller | Thorlabs | ||
| 30mm Cage Mount | Thorlabs | ||
| Cage Rod | Thorlabs | ||
| Stage | |||
| 3D motorized translation stage | Beijing Mao Feng Optoelectronics Technology Co., Ltd. | JTH360XY | |
| 2D Tilting Stage | |||
| Rotation Stage | |||
| Plate Holder | 3D printed | ||
| Spectrometer | |||
| Lens Tube | Thorlabs | ||
| Adapter | Thorlabs | ||
| Collimating Lens | Thorlabs | AC080-020-C | |
| Grating | Wasatch | G = 1145 lpmm | |
| F-theta Lens | Thorlabs | FTH-1064-100 | |
| InGaAs Line-scan Camera | Sensor Unlimited | SU1024-LDH2 | |
| Name | Company | Catalog Number | Comments |
| Cell Culture Component | |||
| HCT 116 Cell line | ATCC | CCL-247 | |
| Cell Culture Flask | SPL Life Sciences | 70025 | |
| Pipette | Fisherbrand | 14388100 | |
| Pipette tips | Sorenson Bioscience | 10340 | |
| Gibco GlutaMax DMEM | Thermo Fisher Scientific | 10569044 | |
| Fetal Bovine Serum, certified, US origin | Thermo Fisher Scientific | 16000044 | |
| Antibiotic-Antimycotic (100X) | Thermo Fisher Scientific | 15240062 | |
| Corning 96-well Clear Round Bottom Ultra-Low Attachment Microplate | Corning | 7007 | |
| Gibco PBS, pH 7.4 | Thermo Fisher Scientific | 10010023 | |
| Gibco Trypsin-EDTA (0.5%) | Thermo Fisher Scientific | 15400054 | |
| Forma Series II 3110 Water-Jacketed CO2 Incubators | Thermo Fisher Scientific | 3120 | |
| Gloves | VWR | 89428-750 | |
| Parafilm | Sigma-Aldrich | P7793 | |
| Transfer pipets | Globe Scientific | 138080 | |
| Centrifuge | Eppendorf | 5702 R | To centrifuge the 15 mL tube |
| Centrifuge | NUAIRE | AWEL CF 48-R | To centrifuge the 96-well plate |
| Microscope | Olympus | ||
| Name | Company | Catalog Number | Comments |
| Histology & IHC | |||
| Digital slide scanner | Leica | Aperio AT2 | Obtain high-resolution histological images |
| Histology Service | Histowiz | Request service for histological and immunohistological staining of tumor spheroid | |
| Name | Company | Catalog Number | Comments |
| List of Commerical OCTs | |||
| SD-OCT system | Thorlabs | Telesto Series | |
| SD-OCT system | Wasatch Photonics | WP OCT 1300 nm | |
| Name | Company | Catalog Number | Comments |
| Software for Data Analyses | |||
| Basic Image Analysis | NIH | ImageJ | Fiji also works. |
| 3D Rendering | Thermo Fisher Scientific | Amira | Commercial software. Option 1 |
| 3D Rendering | Bitplane | Imaris | Commercial software. Option 2. Used in the protocol |
| OCT acquisition software | custom developed in C++. | ||
| Stage Control | Beijing Mao Feng Optoelectronics Technology Co., Ltd. | MRC_3 | Incorporated into the custom OCT acquisition code |
| OCT processing software | custom developed in C++. Utilize GPU. Incorporated into the custom OCT acquisition code. | ||
| Morphological and Physiological Analysis | custom developed in MATLAB |
Referências
- Kola, I., Landis, J. Can the pharmaceutical industry reduce attrition rates?. Nature Reviews Drug Discovery. 3 (8), 711-716 (2004).
- Breslin, S., O'Driscoll, L. Three-dimensional cell culture: the missing link in drug discovery. Drug Discovery Today. 18, 240-249 (2013).
- Hickman, J. A., et al. Three-dimensional models of cancer for pharmacology and cancer cell biology: Capturing tumor complexity in vitro/ex vivo. Biotechnology Journal. 9 (9), 1115-1128 (2014).
- Sutherland, R. M. Cell and environment interactions in tumor microregions: the multicell spheroid model. Science. 240 (4849), 177-184 (1988).
- Mueller-Klieser, W. Three-dimensional cell cultures: from molecular mechanisms to clinical applications. American Journal of Physiology - Cell Physiology. 273, C1109-C1123 (1997).
- Friedrich, J., Seidel, C., Ebner, R., Kunz-Schughart, L. A. Spheroid-based drug screen: considerations and practical approach. Nature Protocols. 4 (3), 309-324 (2009).
- Tung, Y. -. C., et al. High-throughput 3D spheroid culture and drug testing using a 384 hanging drop array. The Analyst. 136 (3), 473-478 (2011).
- Vinci, M., et al. Advances in establishment and analysis of three-dimensional tumor spheroid-based functional assays for target validation and drug evaluation. BMC biology. 10, 29 (2012).
- LaBarbera, D. V., Reid, B. G., Yoo, B. H. The multicellular tumor spheroid model for high-throughput cancer drug discovery. Expert Opinion on Drug Discovery. 7, 819-830 (2012).
- Pampaloni, F., Ansari, N., Stelzer, E. H. K. High-resolution deep imaging of live cellular spheroids with light-sheet-based fluorescence microscopy. Cell and Tissue Research. 352, 161-177 (2013).
- Lovitt, C. J., Shelper, T. B., Avery, V. M. Miniaturized three-dimensional cancer model for drug evaluation. Assay and Drug Development Technologies. 11 (7), 435-448 (2013).
- Wenzel, C., et al. 3D high-content screening for the identification of compounds that target cells in dormant tumor spheroid regions. Experimental Cell Research. 323 (1), 131-143 (2014).
- Astashkina, A., Grainger, D. W. Critical analysis of 3-D organoid in vitro cell culture models for high-throughput drug candidate toxicity assessments. Innovative tissue models for drug discovery and development. 69, 1-18 (2014).
- Edmondson, R., Broglie, J. J., Adcock, A. F., Yang, L. Three-dimensional cell culture systems and their applications in drug discovery and cell-based biosensors. Assay and Drug Development Technologies. 12 (4), 207-218 (2014).
- Gong, X., et al. Generation of multicellular tumor spheroids with microwell-based agarose scaffolds for drug testing. PLoS ONE. 10 (6), e0130348 (2015).
- Hoffmann, O. I., et al. Impact of the spheroid model complexity on drug response. Journal of biotechnology. 205, 14-23 (2015).
- Martinez, N. J., Titus, S. A., Wagner, A. K., Simeonov, A. High-throughput fluorescence imaging approaches for drug discovery using in vitroand in vivothree-dimensional models. Expert Opinion on Drug Discovery. 10, 1347-1361 (2015).
- Nath, S., Devi, G. R. Three-dimensional culture systems in cancer research: Focus on tumor spheroid model. Pharmacology, Therapeutics. 163, 94-108 (2016).
- Li, L., Zhou, Q., Voss, T. C., Quick, K. L., LaBarbera, D. V. High-throughput imaging: Focusing in on drug discovery in 3D. Methods. 96, 97-102 (2016).
- Ham, S. L., Joshi, R., Thakuri, P. S., Tavana, H. Liquid-based three-dimensional tumor models for cancer research and drug discovery. Experimental Biology and Medicine. 241 (9), 939-954 (2016).
- Kessel, S., et al. High-Throughput 3D Tumor Spheroid Screening Method for Cancer Drug Discovery Using Celigo Image Cytometry. Journal of Laboratory Automation. , 2211068216652846 (2016).
- Stock, K., et al. Capturing tumor complexity in vitro: Comparative analysis of 2D and 3D tumor models for drug discovery. Scientific Reports. 6, 28951 (2016).
- Thakuri, P. S., Ham, S. L., Luker, G. D., Tavana, H. Multiparametric analysis of oncology drug screening with aqueous two-phase tumor spheroids. Molecular Pharmaceutics. 13 (11), 3724-3735 (2016).
- Lin, R. Z., Chang, H. Y. Recent advances in three-dimensional multicellular spheroid culture for biomedical research. Biotechnology Journal. 3 (9-10), 1172-1184 (2008).
- Piccinini, F., Tesei, A., Arienti, C., Bevilacqua, A. Cancer multicellular spheroids: Volume assessment from a single 2D projection. Computer Methods and Programs in Biomedicine. 118 (2), 95-106 (2015).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: a systematic approach to enhance the biological relevance of data obtained. Scientific Reports. 6, 19103 (2016).
- Debnath, J., Brugge, J. S. Modelling glandular epithelial cancers in three-dimensional cultures. Nature Reviews Cancer. 5 (9), 675-688 (2005).
- Huang, D., et al. Optical coherence tomography. Science. 254 (5035), 1178-1181 (1991).
- Drexler, W., et al. Optical coherence tomography today: speed, contrast, and multimodality. Journal of Biomedical Optics. 19 (7), 071412 (2014).
- Fujimoto, J., Swanson, E. The development, commercialization, and impact of optical coherence tomography. Investigative Ophthalmology, Visual Science. 57 (9), (2016).
- Vakoc, B. J., Fukumura, D., Jain, R. K., Bouma, B. E. Cancer imaging by optical coherence tomography: preclinical progress and clinical potential. Nature Reviews Cancer. 12 (5), 363-368 (2012).
- Wojtkowski, M. High-speed optical coherence tomography: basics and applications. Applied optics. 49 (16), D30-D61 (2010).
- Drexler, W., Fujimoto, J. G. . Optical coherence tomography: technology and applications. , (2008).
- Geitzenauer, W., Hitzenberger, C. K., Schmidt-Erfurth, U. M. Retinal optical coherence tomography: past, present and future perspectives. British Journal of Ophthalmology. 95 (2), 171 (2011).
- Sakata, L. M., DeLeon-Ortega, J., Sakata, V., Girkin, C. A. Optical coherence tomography of the retina and optic nerve - a review. Clinical, Experimental Ophthalmology. 37 (1), 90-99 (2009).
- van Velthoven, M. E. J., Faber, D. J., Verbraak, F. D., van Leeuwen, T. G., de Smet, M. D. Recent developments in optical coherence tomography for imaging the retina. Progress in Retinal and Eye Research. 26 (1), 57-77 (2007).
- Kashani, A. H., et al. Optical coherence tomography angiography: A comprehensive review of current methods and clinical applications. Progress in Retinal and Eye Research. 60, 66-100 (2017).
- de Carlo, T. E., Romano, A., Waheed, N. K., Duker, J. S. A review of optical coherence tomography angiography (OCTA). International Journal of Retina and Vitreous. 1 (1), 5 (2015).
- Sharma, M., Verma, Y., Rao, K. D., Nair, R., Gupta, P. K. Imaging growth dynamics of tumour spheroids using optical coherence tomography. Biotechnology Letters. 29 (2), 273-278 (2006).
- Jung, Y., Nichols, A. J., Klein, O. J., Roussakis, E., Evans, C. L. Label-Free, Longitudinal Visualization of PDT Response In Vitro with Optical Coherence Tomography. Israel Journal of Chemistry. 52 (8-9), 728-744 (2012).
- Huang, Y., et al. Optical coherence tomography detects necrotic regions and volumetrically quantifies multicellular tumor spheroids. Cancer Research. 77 (21), 6011-6020 (2017).
- Spalteholz, W. . Über das Durchsightigmachen von menschlichen und tierischen Präparaten: nebst Anhang, Über Knochenfärbung. , (1911).
- Dodt, H. -. U., et al. Ultramicroscopy: three-dimensional visualization of neuronal networks in the whole mouse brain. Nature Methods. 4 (4), 331 (2007).
- Leitgeb, R., Hitzenberger, C., Fercher, A. F. Performance of fourier domain vs. time domain optical coherence tomography. Optics express. 11 (8), 889-894 (2003).
- Jian, Y., Wong, K., Sarunic, M. V. . Optical Coherence Tomography and Coherence Domain Optical Methods in Biomedicine XVII. , 85710Z (2013).
- Guizar-Sicairos, M., Thurman, S. T., Fienup, J. R. Efficient subpixel image registration algorithms. Optics Letters. 33 (2), 156-158 (2008).
- Canny, J. A computational approach to edge detection. IEEE Transactions on Pattern Analysis and Machine Intelligence. (6), 679-698 (1986).
- Vermeer, K. A., Mo, J., Weda, J. J. A., Lemij, H. G., de Boer, J. F. Depth-resolved model-based reconstruction of attenuation coefficients in optical coherence tomography. Biomedical Optics Express. 5 (1), 322-337 (2014).
- Klein, T., et al. Multi-MHz retinal OCT. Biomedical Optics Express. 4, 1890-1908 (2013).
- Klein, T., Huber, R. High-speed OCT light sources and systems [Invited]. Biomedical Optics Express. 8 (2), 828-859 (2017).
- Zhou, C., Alex, A., Rasakanthan, J., Ma, Y. Space-division multiplexing optical coherence tomography. Optics Express. 21, 19219-19227 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados