Method Article
Aplicación in vivo del sistema de control remoto para la manipulación de genes endógenos
En este artículo
Resumen
Este protocolo describe los pasos necesarios para generar un modelo de sistema en que se puede regular la transcripción de un gen endógeno de interés condicional en animales vivos o células con mayor lac represor y tet activador de sistemas.
Resumen
Aquí se describe un protocolo para la implantación del sistema de control remoto (Reversible Manipulation of Transcription en Endogenous loci), que permite para el control de la expresión reversibles y regulables de un gen endógeno de interés en la vida del modelo de sistemas. El sistema de control remoto emplea sistemas de activación de mejorada lac de la represión y tet para lograr abajo - o regulación al alza de un gene de la blanco dentro de un sistema biológico único. Represión apretado puede lograrse de flexible encuentra sitios de unión del represor lejos río abajo de un sitio de inicio de transcripción por inhibición de la elongación de la transcripción. Upregulation robusto puede lograrse incrementando la transcripción de un gen endógeno dirigiéndose a activadores transcripcionales de tet al promotor cognado. Este control reversible y armoniosa expresión pueda ser aplicado y retirado varias veces en los organismos. La potencia y la versatilidad del sistema, según lo demostrado por endógeno Dnmt1 , permitirá más precisos análisis funcionales in vivo permitiendo la investigación de funciones de los genes en los distintos niveles de expresión y por la prueba de la reversibilidad de un fenotipo.
Introducción
Knockout genético o enfoques transgénicos han sido medios eficaces para estudiar la función génica en modelos animales. Sin embargo, la regulación de la expresión de estos enfoques es dicotómica (encendido/apagado), no temporal y así no es capaz de revelar el espectro funcional completado de un gen. Condicional Cre/LoxP tecnologías han permitido spatio-temporal inactivación o activación de funciones de los genes, pero su naturaleza dicotómica continúa planteando limitaciones, tales como la mortalidad celular irreversible y1,2 , 3. con el fin de llenar este vacío, condicional precipitación se han desarrollado con tet-regulado shRNA o miRNA4. Sin embargo, los efectos off-target siguen siendo una preocupación para RNAi5 y difíciles de controlar en vivo. Más recientemente, CRISPR/Cas-mediada por tecnologías de control transcripcional han introducido un enfoque más versátil para lograr tanto arriba y desregulación de la expresión génica endógena y demostró sus utilidades6,7 . Sin embargo, la efectividad del control transcripcional CRISPR/Cas-mediada aún es confusa en vivo y la reversibilidad de la basado en KRAB represión queda vista, fuerte represión por parte de KRAB y sus proteínas interactuantes KAP1 ha demostrado inducir 8,9el silenciamiento del gen permanente.
Para hacer frente a estas limitaciones, hemos desarrollado un novedoso sistema de regulación transcripcional capaz de controlar condicionalmente la expresión de genes endógenos en un reversible y manera armonioso en ratones mediante ingeniería transcripcional procariota binario sistemas de regulación10. Procariotas sistemas de regulación transcripcionales binario con ligandos reguladoras, como la lac y tet, han permitido tal reversible y armoniosa expresión control11,12,13, 14. sin embargo, la potencia de represión inadecuada de los sistemas binarios actuales ha impedido su adopción amplia para controlar la expresión de genes endógenos en mamíferos. Hemos desarrollado un sistema de represión de lac mejorado suficientemente potente para la represión de genes endógenos y empleó una nueva estrategia de focalización activadores transcripcionales tet directamente al cognado promotor de un gen endógeno para lograr upregulation robusta (figura 1)10. Con esta tecnología, hemos conseguido casi dos órdenes de magnitud control de expresión del gen Dnmt1 endógeno en un armonioso, inducible y reversible de manera10. Aquí proporcionamos instrucciones detalladas para su aplicación en vivo a otros genes y organismos usando ratones como especie modelo.

Figura 1 : Resumen del sistema de control remoto. La transcripción de un gen endógeno objetivo puede regularse utilizando ingeniería lac represor y tet activador sistemas. El promotor del gen blanco o intrón está diseñado para contener operadores para el represor de LacIGY apretado-que atan o rtactivador TA-M2. R indica Repron (Represión intron), que contiene 12 operadores lac simétrico (S) además un intron de beta-globina conejo parcial. T indica el operador de TTE . El represor o activador está/se expresan de un promotor específico de tejido. La expresión del gen objetivo puede entonces reversible ajustarse al nivel deseado de la expresión administración de IPTG (Isopropil β-D-1-tiogalactopiranósido, un antagonista del LacIGY represor) o doxiciclina (Dox). Esta figura ha sido modificada de Lee et al.10haga clic aquí para ver una versión más grande de esta figura.
Antes de iniciar este protocolo, revisar cuadro 1 para identificar las medidas pertinentes para el control deseado de la expresión génica. Por ejemplo, para diseñar un ratón que permite reversible downregulation de "Gen X", complete las secciones 1, 3 y 4 del siguiente protocolo. También, la tabla 1 resume los componentes necesarios del sistema de control remoto.
| Cambio de expresión deseada | Represión sólo | Activación sólo | Activación y represión |
| Secciones pertinentes del Protocolo de | 1, 3-4 | 2-4 | 1-4 |
| Secuencia de control remoto en Gene de la blanco | Repron ("Intrón de represión"; 12 operadores lac simétrica más un intrón de beta-globina conejo parcial) | Operadores de Tet | Operadores Repron y Tet |
| Ubicación de la secuencia de control remoto | Intrón | Promotor | Intrón y promotor |
| Activador/represor necesario para el Control deseado | LacIGY represor | Activador de rtTA M2 | LacIGY represor y activador rtTA M2 |
| Ligandos reglamentarios | IPTG | Doxiciclina | IPTG o doxiciclina |
Tabla 1: Resumen de componentes de control remoto.
Protocolo
Animales todos los procedimientos se llevaron a cabo con la aprobación del cuidado de Animal institucional y Comité uso (IACUC) de la Universidad de California del sur y el Instituto de investigación de Van Andel y en cumplimiento de la guía para el cuidado y uso de animales de laboratorio de los institutos nacionales de salud15.
1. modificar el gen de interés para la represión por control remoto
- Usando las referencias siguientes, identificar un intrón transcripcionalmente inerte hacia el extremo 5' del gen de interés para la inserción de la secuencia de Repron ("Represión intren «; 12 operadores simétricos lac además un intron de beta-globina conejo parcial). Ten en cuenta de promotores alternativos para el gen de interés y elegir un intrón según las transcripciones para ser controlado (es decir, un intrón compartido por todas las transcripciones o uno específico de transcripción deseada).
Nota: Para los genes sin un intrón o con unos hacia el extremo 3', inserte la secuencia de Repron en el promotor (véase Lee, et al.10 para un procedimiento detallado).- Obtener la secuencia genomic para el gene de interés.
- Navegar por el Browser del genoma de UCSC16,17y seleccione el último proyecto del genoma del ratón (Mouse GRCm38/mm10 en el momento de la publicación), que se encuentra en la pestaña de genomas .
- Introduce el nombre o símbolo del gen de interés en la barra de búsqueda para ver la transcripción para el gen. Haga clic en ir.
- Seleccione la variante deseada transcripción para el gen de interés.
- Haga clic en el símbolo del gen junto a la variante de transcripción de interés (el símbolo de la transcripción anteriormente seleccionada estará en una caja oscura).
- Bajo la bandera de la secuencia y enlaces a herramientas y bases de datos , haga clic en el enlace de la Secuencia genómica .
- Secuencia de recuperación región de opciones, seleccione sólo los exones (5' UTR, CDS y 3' UTR), intrones y el valor por defecto FASTA un registro por gene. Para las Opciones de formato de secuencia, seleccione exones en mayúsculas, todo en minúsculas y Máscara repite a N (para ocultar secuencias repetitivas). Haga clic en Enviar.
- Guarde esta secuencia, conservando la parte superior - y minúsculas formato de un documento o programa que puede ser anotada.
- Evitar la interrupción de las islas CpG, que puede indicar regiones con funciones reguladoras de los genes.
Nota: Aunque un intrón 5' es preferible para la inserción de la secuencia de Repron, el primer intrón puede contener elementos reguladores transcripcionales como las islas de CpG (examinados en este paso) o potenciador elementos (examinados en el paso 1.1.4), que deben evitarse para Repron inserción.- Bajo la bandera de expresión y regulación de los Browser del genoma de UCSC, seleccione Mostrar para la pista de Las islas de CpG y haga clic en Actualizar.
- Zoom en los intrones de 5', haga clic en cada isla de CpG (mostrado en verde en el momento de la publicación si está presente) y seleccione Vista DNA para esta función.
- Después de seleccionar máscara repite n, seleccione obtener ADN para obtener la secuencia de la isla de CpG.
- Superposición de estas secuencias con el archivo original de la secuencia y anotar como regiones intronic para evitar debido a funciones reguladoras de genes posibles.
- Evitar la interrupción de la no-codificación RNAs, en caso de tengan una función reguladora.
- Bajo la bandera de Genes y genes predicciones del Browser del genoma de UCSC, seleccione Mostrar para la pista GENCODE (Ensembl) y haga clic en Actualizar.
- Zoom en los intrones de 5', haga clic en cada RNA no codificante (mostrado en verde en el momento de la publicación si está presente) y obtener la secuencia del ADN de cada una haciendo clic en sus coordenadas cromosómicas.
- En el menú desplegable de Ver , seleccionar el ADNy haga clic en máscara repite a N. Haga clic en obtener ADN.
- Superposición de estas secuencias con el archivo original de la secuencia y anotar como regiones intronic para evitar debido a funciones reguladoras de genes posibles.
- Evitar regiones intronic con firmas potenciador en los tejidos de interés, como H3K4me1, H3K27ac y DNasa I hipersensibilidad18,19,20,21, así como la Unión de CTCF sitios, los cuales regulan potenciador bucle22,23.
- Desplácese al codificar la base de datos24,25y seleccione el icono de experimentos .
- Para el tipo de análisis, seleccione ChIP-seq o DNasa-seqy rellenar el resto de categorías ( Biosample tipo,organismo, etc.) según las células para ser diseñado.
- Después de la selección, sobrevolar los pictogramas en color azul y seleccione el botón Ver resultados lista aparece (pictograma de extremo izquierdo en el momento de la publicación).
- Seleccione los conjuntos de datos para los objetivos de H3K4me1, H3K27ac, DNasa I y CTCF que más se asemejen a las células para ser diseñado.
- Dentro de cada conjunto de datos pertinente, vaya a la sección de archivos , verificar que mm10 (o la versión más reciente del genoma) y UCSC están seleccionadas y haga clic en el botón Visualize .
- Ahora en el Browser del genoma de UCSC y zoom en los intrones 5' del gen de interés, haga click en cada H3K4me1, H3K27ac, DNasa I y pico CTCF en sus respectivos pico anotado.
- Obtener la secuencia del ADN para cada región de pico haciendo clic en sus coordenadas cromosómicas.
- En el menú desplegable de Ver , seleccionar el ADNy haga clic en máscara repite a N. Haga clic en obtener ADN.
- Superposición de estas secuencias con el archivo original de la secuencia y anotar como regiones intronic para evitar debido a funciones reguladoras de genes posibles.
- Evitar ruptura de consenso de secuencias, para no interferir con el adecuado empalme de los exones de empalme.
Nota: Aunque se limitan en gran medida las secuencias necesarias para empalmar a seis bases en el extremo 5' de un intrón26 y 60 bases en los 3' extremo27, es aconsejable seleccionar un intrón grande, considerablemente más amplio márgenes de estos regiones de consenso para reducir al mínimo la probabilidad de afectar empalme. Se recomienda el uso de herramientas de análisis del intrón, como SVM-BPfinder28, para un análisis más minucioso de las posibles secuencias de empalme.
- Obtener la secuencia genomic para el gene de interés.
- Después de identificar una región intronic que cumple con los criterios anteriores (como la ejemplificada por Dnmt1 en datos complementarios), pantalla de la secuencia usando un sgRNA online diseño herramienta para identificar un sgRNA en la región con alta especificidad y predijo puntuaciones de eficiencia.
- Desplácese hasta una herramienta de diseño en línea sgRNA de elección, como CRISPOR29.
- Introduzca la secuencia de la región intronic de interés, especificar el genoma de referencia correspondiente y seleccione el deseado Protospacer adyacentes adorno (PAM). Haga clic en Enviar.
- Ordenar la sgRNAs predicha por cuenta de la especificidad y seleccione uno o más sgRNAs que también tienen una alta eficiencia prevista puntuación29.
- Opcional: Para maximizar la probabilidad de usar un sgRNA eficaz, primero probar la eficiencia de escote de varios sgRNAs de mayor puntaje en un ensayo in vitro de30y proceder con la más eficiente sgRNA en vivo.
- Diseñar una plantilla de ADN que contiene a un PITT (transgénesis orientada basada en inyección Pronuclear) landing pad secuencia, como en la complementaria de la figura 1A, B, flanqueado a ambos lados por brazos de homología base 60 que corresponden a lo sgRNA cortar sitio31 .
Nota: La plataforma de aterrizaje contiene dos heterotípicos loxP sitios (JT15 y Lox2272) y permite inserción dirigida de grandes secuencias a través de un enfoque de dos pasos; Asegúrese de que las uniones de esta inserción no crear sitios de empalme críptico. Alternativamente, puede utilizarse una células madre embrionarias (ESC) - base knock - en estrategia para insertar la secuencia de Repron directamente. - Preparar lo sgRNA Cas9 proteína y la plantilla de ADN (ssDNA) monocatenario para el aterrizaje y microinject en huevos fertilizados (de ratones B6C3F1/J u otra variedad deseada) según protocolos establecidos32,33.
- Pantalla para los ratones con el aterrizaje pad knock-in.
- Diseño de cartillas de la polimerización en cadena complementarias para el locus genómico pero fuera de las regiones dirigidas por los brazos de la homología, como demostrado por Dnmt1 en suplementario Figura 1. Evitar secuencias repetitivas genómicas en el diseño de los cebadores.
- Extraer ADN de clips de cola de los ratones según protocolos establecidos34.
- Uso de PCR y electroforesis para identificar a ratones con una inserción de cojín del aterrizaje del gel.
- Confirmar que el knock-in fue exitosa mediante la secuenciación de los productos PCR.
Nota: Los cambios y canceladuras grandes no deseadas pueden ser introducidos por CRISPR/Cas935, así que cuidado de proyección para la edición de objetivo se recomienda antes de proceder35,36,37.
- Utilizando huevos fertilizados de los ratones de la plataforma de aterrizaje, microinject iCre mRNA38 y un plásmido transgénico que contiene la secuencia de Repron flanqueada por JTZ17 y Lox2272 recombinación sitios31,39,40, 41 según estableció métodos38,42,43.
- Pantalla para los ratones con la inserción de la Repron.
- Inserte el diseño PCR primers complementarios al locus genómico, fuera de la Repron. Evitar secuencias repetitivas genómicas en el diseño de los cebadores.
- Extraer ADN de clips de cola de los ratones según protocolos establecidos34.
- Uso de PCR y electroforesis para identificar a ratones con una inserción Repron del gel.
- Confirmar que el knock-in fue exitosa mediante la secuenciación de los productos PCR.
2. modificar el gen de interés para el upregulation por control remoto
- Con las siguientes pautas, identificar una región en el promotor del gen de interés que es poco probable que perturban la función del promotor sobre la inserción de secuencias de operador TTE . Ten en cuenta de promotores alternativos para el gen de interés y elegir un promotor según las transcripciones para ser controlado (es decir, un promotor compartido por todas las transcripciones o uno específico de transcripción deseada).
- Obtener la secuencia genomic de la promotora de interés.
- Navegar por el Browser del genoma de UCSC16,17y seleccione el último proyecto del genoma del ratón (Mouse GRCm38/mm10 en el momento de la publicación), que se encuentra en la pestaña de genomas .
- Introduce el nombre o símbolo del gen de interés en la barra de búsqueda para ver la transcripción para el gen. Haga clic en ir.
- Seleccione la variante deseada transcripción para el gen de interés.
- Haga clic en el símbolo del gen junto a la variante de transcripción de interés (el símbolo del gene de la transcripción anteriormente seleccionado será en una caja oscura).
- Bajo la bandera de la secuencia y enlaces a herramientas y bases de datos , haga clic en el enlace de la Secuencia genómica .
- Secuencia de recuperación región de opciones, seleccione sólo promotor/Upstream por 1000 bases. Para las Opciones de formato de secuencia, seleccione Máscara repite a N (para ocultar secuencias repetitivas). Haga clic en Enviar.
- Guarde esta secuencia de promotor de un documento o programa que puede ser anotada.
- Seleccionadas regiones libres de sitios de unión de factor de transcripción putativo, como interrupción de estas secuencias pueden alterar la expresión de genes endógenos.
- Navegar por el Browser del genoma de UCSC y abra la versión más reciente del genoma del ratón.
- Por encima de la bandera de mapeo y la secuenciación , seleccione pista hubs.
- Haga clic en el mm10 junto al nombre del centro de JASPAR 2018 TFBS (o la última versión JASPAR).
- Introduce el nombre o símbolo del gen de interés en la barra de búsqueda para ver la transcripción para el gen. Haga clic en ir.
- Seleccione la variante deseada transcripción para el gen de interés.
- Desplácese hacia abajo hasta la bandera JASPAR 2018 TFBS y haga clic en la flecha desplegable para seleccionar la opción de visualización deseada para la pista, como aplasta. Haga clic en Actualizar.
- Zoom en la región del promotor del gen de interés e identificar regiones que son libre (relativamente libre) de sitios de unión del factor de transcripción según la pista JASPAR. Registrar las coordenadas cromosómicas de estas regiones.
- Obtener la secuencia genomic de estas coordenadas cromosómicas escribiendo en la barra de búsqueda y haga clic en ir. En el menú desplegable de Ver , seleccionar el ADNy haga clic en máscara repite a N. Haga clic en obtener ADN.
- Superposición de estas secuencias con el archivo original de la secuencia y anotar como regiones promotor ideal destino debido a la falta de unión del factor de transcripción.
- Dentro de estas secuencias de, seleccione una región promotora perturbable que está aguas arriba pero cerca del sitio de inicio de transcripción del gen de interés.
Nota: Una inserción que está demasiado cerca que del sitio de inicio de transcripción puede incrementar la posibilidad de alterar la actividad de promotor, sino una inserción que es demasiado puede disminuir el nivel de regulación al alza. Insertar secuencias de operador tet 200 basepairs aguas arriba del sitio de inicio de transcripción ha dado lugar a robusto upregulation de los promotores de dos pruebas (véase discusión)10.
- Obtener la secuencia genomic de la promotora de interés.
- Pantalla de la región del promotor seleccionado utilizando una herramienta de diseño en línea sgRNA para identificar un sgRNA en la región con alta especificidad y las puntuaciones de eficiencia prevista.
- Desplácese hasta una herramienta de diseño en línea sgRNA de elección, como CRISPOR29.
- Introduzca la secuencia de la región de interés, especificar el genoma de referencia correspondiente y seleccione el deseado Protospacer adyacentes adorno (PAM). Haga clic en Enviar.
- Ordenar la sgRNAs predicha por cuenta de la especificidad y seleccione uno o más sgRNAs que también tienen una alta eficiencia prevista puntuación29.
- Opcional: Para maximizar la probabilidad de usar un sgRNA eficaz, primero probar la eficiencia de la división de varios sgRNAs en un ensayo in vitro de30y proceder con la más eficiente sgRNA en vivo.
- Diseño de una plantilla de ADN que contienen los operadores tet flanqueados a ambos lados por brazos de homología base 60 que corresponden a lo sgRNA cortar sitio31,33.
Nota: El número de operadores de tet es adaptable; inserción de dos a cuatro secuencias de operador de tet en tándem se ha demostrado previamente para ser eficaz, pero en principio más operadores son deseables para la conducción de la más alta expresión. Alternativamente, una estrategia basada en ESC knock-in se puede utilizar para insertar los operadores TTE .- Opcional: Para la evidencia experimental de que las modificaciones propuestas probablemente no interrumpirá la actividad endógena de transcripcional del gen de interés, copia la secuencia del promotor de ingeniería en un vector de la luciferasa de luciérnaga (como pGL3-Basic), y Comparar su eficacia al promotor original utilizando un ensayo de luciferasa.
- Preparar lo sgRNA Cas9 proteína y la plantilla de ssDNA que contiene las secuencias del operador de tet y microinject en huevos fertilizados (de ratones B6C3F1/J u otra variedad deseada) según protocolos establecidos32,33.
Nota: Debido a la complejidad de la síntesis de una secuencia repetitiva, un enfoque basado en la transcripción in vitro de la transcripción/retroceso recomienda sintetizar una plantilla de ssDNA de un double-stranded DNA (dsDNA) plásmido44. Alternativamente, puede usarse una plantilla de dsDNA para microinyección, pero la eficacia knock-in puede ser reducido45. - Pantalla de ratones knock-in de los operadores de TTE .
- Diseño de cartillas de la polimerización en cadena complementarias para el locus genómico, fuera de las regiones dirigidas por los brazos de homología. Evitar secuencias repetitivas genómicas en el diseño de los cebadores.
- Extraer ADN de clips de cola de los ratones según protocolos establecidos34.
- Utilizar PCR y electroforesis para identificar ratones con la inserción de los operadores de la tet del gel.
- Confirmar que el knock-in fue exitosa mediante la secuenciación de los productos PCR.
Nota: Los cambios y canceladuras grandes no deseadas pueden ser introducidos por CRISPR/Cas935, así que cuidado de proyección para la edición de objetivo se recomienda antes de proceder35,36,37.
3. desarrollar el activador o represor expresando ratones
- Identificar un expresando su firme promotor para los tipos de tejidos o células de interés.
Nota: Una búsqueda de literatura y la base de datos de promotor tejido-específico46 pueden ser útiles para la identificación de un promotor de tal. - Coloque el mayor proporcionado lac represor o tet activador secuencia aguas abajo del promotor deseado para generar una construcción transgénica.
- Producir las líneas de ratón transgénico utilizando procedimientos transgénicos42,43,47. Alternativamente, puede utilizarse un enfoque específico de la transgénesis para evitar efectos de posición y para permitir la inserción de transgenes de la solo-copia31,48.
- Propagar a los fundadores y determinar el nivel de los transgenes en la descendencia de cada fundador y patrón de expresión. Seleccionar líneas con fuerte expresión en el tipo de tejido deseado para cría.
- Los fundadores a los ratones de tipo salvaje de la raza.
- Diseño de primers PCR que detectan la inserción del activador o represor.
- Extraer ADN de clips de cola de la descendencia según protocolos establecidos34.
- Uso de PCR y electroforesis para identificar ratones con la inserción del gel.
- Secuenciación de los productos PCR para confirmar la inserción del transgén.
- Difundir las líneas transgénicas.
- Sacrificar algunos cachorros de cada línea y recoger los tejidos de interés para qRT-PCR, inmunohistoquímica y western blot para analizar la expresión de la gene/proteína de interés en el tipo de tejido blanco.
- Seleccionar las líneas con la expresión más fuerte en los tejidos de interés para su uso en la sección 4.
4. manipular la expresión del gene in vivo
- Se reproducen los ratones con el gen modificado de interés (sección 1 y 2) con los ratones transgénicos de la sección 3 según las prácticas de crianza establecidos49. Para el control de la máxima expresión, se reproducen los ratones a la calidad de homozigoto para el alelo modificado.
- Reversión o ajuste de la represión del gen objetivo, administrar IPTG en el agua potable.
- Determinar experimentalmente la dosis de IPTG utilizar tratar ratones de genotipo apropiado los controles con uno de un rango de dosis (recomendado a partir de rango: 0 – 400 mM IPTG) para por lo menos una semana50,51. Incluyen al menos tres ratones por grupo de tratamiento y seleccionar la edad y sexo de los ratones que son más relevantes para los experimentos futuros previstos. Nota: Las parejas reproductoras de ratones pueden ser tratadas con agua IPTG para proporcionar la exposición del desarrollo de IPTG a la descendencia si se desea13o ratones pueden comenzar el tratamiento cualquier momento después del nacimiento.
- Disolver la masa deseada de IPTG en agua destilada estéril en el día de la administración y revuelva con una barra de agitación durante 5 minutos o hasta que se disuelva completamente.
- Envolver la botella con papel de aluminio y administrar el agua IPTG en un frasco protegido de la luz. Reemplazar dos veces a la semana. Proporcionar la misma fuente de agua a los ratones recibiendo 0 mM IPTG.
- Después de por lo menos una semana, sacrificar a los ratones y analizar la expresión del gen de interés en los tejidos de destino mediante qRT-PCR, inmunohistoquímica y western blot.
- Identificar la dosis que restaura la expresión del gen de interés a la de los controles de tipo salvaje y utilice esta dosis para lograr la expresión normal del gene en futuros experimentos.
- Determinar experimentalmente la dosis de IPTG utilizar tratar ratones de genotipo apropiado los controles con uno de un rango de dosis (recomendado a partir de rango: 0 – 400 mM IPTG) para por lo menos una semana50,51. Incluyen al menos tres ratones por grupo de tratamiento y seleccionar la edad y sexo de los ratones que son más relevantes para los experimentos futuros previstos. Nota: Las parejas reproductoras de ratones pueden ser tratadas con agua IPTG para proporcionar la exposición del desarrollo de IPTG a la descendencia si se desea13o ratones pueden comenzar el tratamiento cualquier momento después del nacimiento.
- Para la inducción del gen upregulation, administrar doxiciclina (Dox) en la dieta.
- Determinar experimentalmente la concentración de Dox para administrar tratamiento de ratones de genotipo apropiado los controles con una de las concentraciones de una gama de Dox (recomendado a partir de rango: 0 – 5000 mg/kg Doxiciclina Hiclato) para por lo menos una semana52 ,53, incluyendo por lo menos tres ratones por grupo de tratamiento. Compra que contengan Dox alimentos de mouse de un vendedor comercial.
Nota: Pares de ratones de cría puede ser tratada con Dox alimentos para proporcionar la exposición del desarrollo de Dox a la descendencia si se desea54,55, o ratones pueden comenzar el tratamiento cualquier momento después del nacimiento. - Vuelva a colocar la dieta una vez por semana. Proporcionar la misma dieta base a ratones recibiendo alimentos libres de Dox.
- Después de por lo menos una semana, sacrificar a los ratones y analizar la expresión del gen de interés en los tejidos de destino mediante qRT-PCR, inmunohistoquímica y western blot.
- Identificar la dosis que la expresión del gen de interés se eleva al nivel deseado y utilice esta dosis para lograr la sobreexpresión del gen en futuros experimentos.
- Determinar experimentalmente la concentración de Dox para administrar tratamiento de ratones de genotipo apropiado los controles con una de las concentraciones de una gama de Dox (recomendado a partir de rango: 0 – 5000 mg/kg Doxiciclina Hiclato) para por lo menos una semana52 ,53, incluyendo por lo menos tres ratones por grupo de tratamiento. Compra que contengan Dox alimentos de mouse de un vendedor comercial.
Resultados
La capacidad de represión del sistema de control remoto ha sido demostrada hasta ahora en dos enfoques diferentes. En el primer enfoque, sitios de unión del represor lac fueron insertados en el endógeno promotor del gen Dnmt1 . En el segundo enfoque, que es recomendado por este protocolo, los sitios de unión del represor fueron insertados en un intrón aguas abajo para evitar el riesgo potencial de afectar la función de promotor de la inserción y de tal modo para simplificar la aplicación del control remoto sistema. Ambos enfoques dio lugar a la represión exitosa (figura 2A, B y Figura 3A-C)10. Dnmt1 expresión fue reprimida al 15% de los niveles no regulados mediante el enfoque basado en el promotor (figura 2A). Esta represión apretada se revirtió de forma dosis-dependiente por el tratamiento de ratones con cantidades variables de IPTG (figura 2A). La represión de Dnmt1 observada fue validada en el nivel de proteína immunostaining (figura 2B). No observamos ninguna diferencia notable en expresión de Dnmt1 entre Dnmt1+ + y Dnmt1LO/LO ratones, confirmando que nuestra inserción de operador lac había interrumpido no promotor normal función10. El enfoque basado en el intrón alcanzadas más de represión del 90% de los operadores situados aguas abajo de varios kilobases del sitio de inicio de transcripción atenuando transcripción elongación (figura 3A, B)10. Este enfoque basado en el intrón fue validado otros siete promotores robustos adicionales (figura 3C). Represión invariablemente apretada logró de todos los promotores probados. No observó ninguna correlación entre los niveles de expresión residual y los puntos fuertes de los promotores, lo que sugiere que la capacidad de represión de nuestro sistema de represión basado en intrón supera la potencia transcripcional de todos los promotores robustos que hemos probado () Figura 3 C).
La capacidad de regulación al alza en vivo del sistema de control remoto también fue demostrada en el gen Dnmt1 . Presentamos dos copias del operador tet en el promotor de Dnmt1 , junto con secuencias de operador lac , para permitir el upregulation o regulación a la baja dependiendo de cual efectoras proteína está presente. Upregulation robusto y desregulación de la expresión de Dnmt1 , cerca de dos órdenes de magnitud (10 a 650%), fueron alcanzados en CES que contiene el alelo modificado de Dnmt1 endógeno (Dnmt1LGT) (figura 4A)10 . Ambas normas fueron completamente reversibles e inducible por tratamientos de IPTG y Dox, respectivamente (figura 4A). A continuación presentamos la modificación de Dnmt1LGT en la línea germinal de ratón para poner a prueba la capacidad de regulación al alza en vivo del sistema de control remoto. Fuerte regulación al alza de Dnmt1 se observó desde el hígado, bazo y riñón, mientras que ningún upregulation detectable en el corazón se observó (figura 4B)7. El patrón de expresión dependiente de ciclo celular de Dnmt1 y la escasez de las células proliferativas en el corazón pueden ser la base de esta observación10,56. Queda por verse si esta limitación se puede superar aumentando el nivel de expresión del activador o el número de sus sitios de Unión.
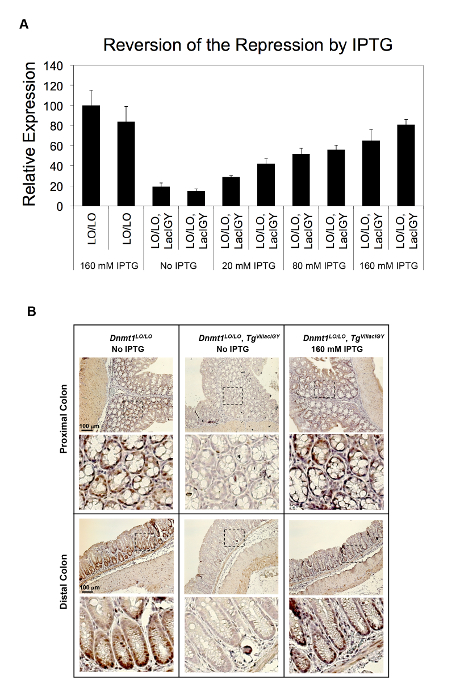
Figura 2 : En vivo represión de Dnmt1 por el represor LacIGY. Ratones (A) con los operadores lac (LO) insertados en el promotor de Dnmt1 , con o sin expresión de LacIGY, fueron tratados con diversas dosis de IPTG. Análisis de expresión de Dnmt1 qRT-PCR muestran la inversión dependiente de la dosis de represión Dnmt1 en vivo por tratamiento de IPTG. Cada barra representa los datos de un ratón diferente. Los datos representan la media ± SEM (n = 3). (B) Immunostaining de proteína Dnmt1 en las criptas colónicas de ratones proporcionó agua potable con o sin 160 mM IPTG durante 3 semanas. Esta figura ha sido modificada de Lee et al.10haga clic aquí para ver una versión más grande de esta figura.
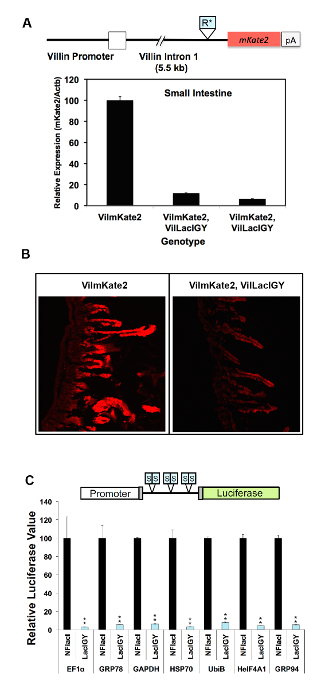
Figura 3 : En vivo y represión en vitro de diversos promotores por el sistema de control remoto. (A) una primera versión de la secuencia de Repron (R *) fue insertado en un intrón aguas abajo del promotor bandido en un ratón transgénico de bandido-mKate2 (VilmKate2). se muestran el análisis de expresión de mKate2 en el intestino de los ratones con o sin el represor LacIGY qRT-PCR. Cada barra representa los datos de un ratón diferente. (B) mKate2 Confocal imágenes del intestino delgado con y sin expresión de LacIGY . Seis operadores de lac simétrico (C) (S) fueron insertados entre varios promotores y un reportero de luciferasa. Plásmidos de represor y Reporteros (50 ng/bien en placa de 96 pocillos) transitoriamente se introdujeron en las células NIH/3T3 en una proporción molar 1:1. Valores de luciferasa fueron evaluado 24 h después de transfección. Estos in vitro datos representan el porcentaje de expresión de luciferasa en LacIGY-expresar las células en relación a los que no funcional LacI (NFlacI). Se utilizaron pruebas T para determinar la significación estadística. Los datos representan la media ± SEM (n = 3). P ≤ 0.05, **P ≤ 0.01. Esta figura ha sido modificada de Lee et al.10haga clic aquí para ver una versión más grande de esta figura.

Figura 4 : Abajo - o upregulation de Dnmt1 expresión in vitro e in vivo. (A) el sistema de control remoto completo fue diseñado en CES cultivadas por gene targeting y electroporación enfoques. Máxima represión de Dnmt1 expresión fue alcanzada con ningún tratamiento mientras que la activación máxima fue alcanzada por el tratamiento de IPTG y Dox. Los datos representan la media ± SEM (n = 3). P ≤ 0.05, **P ≤ 0.01 (de Welch t-tests). (B) activación In vivo de Dnmt1 por el sistema de control remoto, según lo demostrado por inmunotinción de proteína Dnmt1 en varios tejidos de los ratones de control remoto. El alelo de la LGT representa modificación promotor de Dnmt1 contienen sitios de unión de activador de operador y tet de lac . Ratones fueron tratados con una dieta normal o con Dox (Doxiciclina hiclato de 5000 mg/kg) durante un mes. Esta figura ha sido modificada de Lee et al.10haga clic aquí para ver una versión más grande de esta figura.
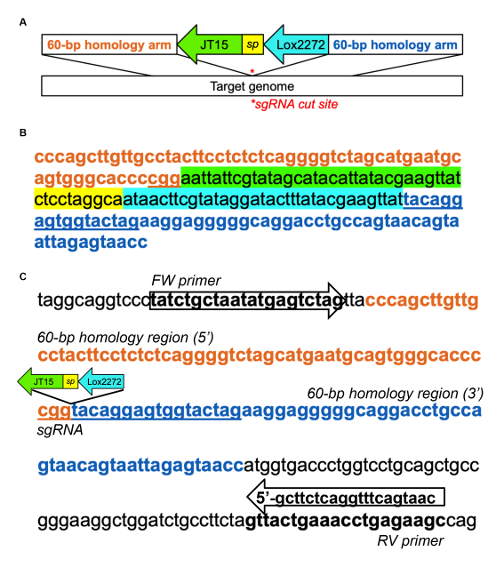
Figura 1 complementaria : Ejemplo de inserción de cojín del aterrizaje en murino Dnmt1 del intrón 1. (A) esquema de plantilla de la DNA para el aterrizaje de la inserción de cojín, adaptado de Quadros et al., (2015)31. Heterotípicos loxP sitios, JT15 y Lox2272, están separados por una secuencia de espaciador corto (sp) y flanqueados a cada lado por 60-bp de DNA que es homólogo a la región genómica de destino. (B) muestra ADN plantilla para inserción de cojín del aterrizaje en el intrón Dnmt1 usando lo sgRNA siguiente: CTAGTACCACTCCTGTACCG (que apunta a la hebra de atrás). La región intronic seleccionada fue bioinformatically por paso 1.1, y el sgRNA fue identificada usando CRISPOR29. (C) ejemplo de diseño de la cartilla PCR para evaluar la inserción de la plataforma de aterrizaje. Cebadores PCR fueron diseñados fuera de los brazos de la homología de la plantilla para confirmar la integración endógena Dnmt1. El amplicon PCR de tipo salvaje es 213 bp; al insertar, se convierte en 291 BP haga clic aquí para ver una versión más grande de esta figura.
Discusión
Un paso crítico y una limitación potencial del sistema de control remoto es el reto asociado con la inserción del represor o sitios de unión del activador sin afectar la expresión del gen objetivo. Nuestro enfoque de represión original, tal como se aplica al gen Dnmt1 , implicó la inserción de sitios de unión de represor lac dentro de regiones transcripcionalmente críticas de un promotor. Para reducir el riesgo de afectar la función de promotor y por lo tanto a mejorar la aplicabilidad general del sistema de control remoto, hemos desarrollado un enfoque basado en el intrón de la represión. La potencia de nuestro sistema mejorado lac permitió reprimir firmemente la transcripción de todos los promotores fuertes, probamos a operadores ubicados cientos a varias kilobases abajo de la transcripción Inicio sitios (figura 3A, C) 10. lo importante, los niveles de represión eran independientes de los puntos fuertes transcripcionales de los promotores (figura 3A – C)10. Esto sugiere que la capacidad de represión de nuestro sistema de represión basado en el intrón supera la fuerza transcripcional de los promotores de la prueba. En este enfoque basado en el intrón, es probable que la represión es mediada a través de la interferencia física entre dos componentes, el mecanismo de elongación de la transcripción y los represores lac del57. Este mecanismo de represión simple y la robustez demostrada del método basado en el intrón pueden representar este enfoque general aplicable a los organismos, tejidos y genes diferentes.
La regulación al alza por el sistema de control remoto requiere las secuencias de enlace transactivator en proximidad con el promotor del gene de la blanco, que entraña un riesgo de afectar la función de promotor. Sin embargo, encontramos que la posición de enlace de secuencias puede ser fuera de la región transcripcionalmente crítica. Dnmt1 y EF1α promotores fueron firme alza de operadores tet encuentra un par de cien bases aguas arriba de la transcripción Inicio sitios10. Esta restricción relajada reduce considerablemente la posibilidad de afectar la función de promotor de un gene de la blanco en ausencia del transactivator. Aumentar el número de enlace de secuencias o uso de transactivators más fuerte podría ayudar a reducir el riesgo al permitir upregulation de sitios más lejos de la página de inicio de transcripción.
Nuestro sistema de control remoto proporciona un elegante control del nivel, el momento y el lugar de la expresión de genes endógenos, lo que permite la reversibilidad de un fenotipo y las consecuencias de los niveles de expresión diferente, que no son fácilmente alcanzables por la prueba tecnologías actuales en vivo gene expresión de control. Es importante tener en cuenta que en la mayoría análisis de expresión del gene, incluido el nuestro, expresión valores representan la media de una población de células entre las que se puede encontrar una variación considerable. Esta heterogeneidad puede influir en procesos celulares de la toma de decisiones, como diferenciación o apoptosis58. Aunque es probable que la precisión de control de expresión del gene puede mejorarse aún más por circuito genético adicional ingeniería59, la potencia observada de nuestro actual sistema permitirá investigación útil de funciones de los genes en muchos contextos biológicos. Además, se espera un alto grado de especificidad de la blanco debido a la complejidad de las secuencias del operador, así como la gran distancia evolutiva entre los mamíferos y las especies originarias de los componentes regulatorios60. Además, líneas de ratón transgénico de represores y activadores pueden ser desarrolladas y empleadas para cualquier gen endógeno. Por ejemplo, se pueden adaptar modelos de ratón de transactivator de tet existentes para lograr la regulación al alza de un gene de la blanco en los tejidos de ratón deseado. Recientemente hemos desarrollado una línea transgénica que puede conducir robusta expresión tejido-específico de nuestro represor lac mayor en múltiples tipos de tejidos cuando se combina con líneas existentes de la Cre por introducción del gen lacIGY en el locus Hipp11 48 bajo el control de un elemento de Lox-parada-Lox (inédito). Esta línea facilitaría sustancialmente la aplicación de tejidos específicos del sistema de control remoto.
Upregulation de gene por el sistema de control remoto ofrece varias ventajas en comparación con el actuales enfoques transgénicos inducibles. No requiere generación de varias líneas transgénicas para probar efectos de posición de la inserción, ya que utiliza el locus endógeno. Además, este enfoque es adecuado para upregulation de los genes con expresión de base fuerte debido a aumenta la expresión de un promotor ya sólido, mientras que los modelos transgénicos dependen de promotores virales mínimas. Por último, el tejido especificidad, control del ciclo celular y que empalman la variantes de un gene de la blanco pueden conservarse al upregulation de nuestro enfoque, que conserva elementos de regulación natural como natural cis-elementos reguladores. El advenimiento de la tecnología de gene targeting CRISPR/Cas-mediada facilitan enormemente la aplicación de esta tecnología en sistemas de diversos modelos.
Divulgaciones
PWL es miembro de la juntas asesora científica de AnchorDx y Progenity, Inc.
Agradecimientos
Agradecemos a la tarde el Dr. Heidi Scrable por su generosa donación de la construcción de gen lacI mamíferos (Mayo Clinic, Rochester, MN), Dr. Daniel Louvard (Instituto Curie, Paris, Francia) para que el promotor de bandido y el Dr. Laurie Jackson-Grusby (Hospital de niños, Boston, MA) por sus contribuciones a las primeras etapas de este desarrollo de la tecnología. Estamos agradecidos por la Dr. Nancy Wu y el Dr. Robert Maxson por su asistencia en la generación de los ratones transgénicos y knockout. Agradecemos a los miembros del laboratorio Laird de discusiones útiles y apoyo. Este trabajo fue financiado por los institutos nacionales de salud [R01 CA75090 DA030325 R01, CA157918 R01 y R01 CA212374 a P.W.L. y 1F31CA213897-01A1 a N.A.V.S].
Materiales
| Name | Company | Catalog Number | Comments |
| B6C3F1/J | The Jackson Laboratory | 100010 | https://www.jax.org/strain/100010 |
| Cas9 Protein | PNA Bio | CP04 | http://www.pnabio.com/products/CRISPR_Cas9.htm?gclid=EAIaIQobChMIsoG8pLL33QIVBr7ACh0naQ4dEAAYAiAAEgKyHvD_BwE |
| CRISPOR | Haeussler et al. 2016 | http://crispor.tefor.net/ | |
| Doxycycline-Containing Mouse Diet | Envigo | Varies by concentration | https://www.envigo.com/products-services/teklad/laboratory-animal-diets/custom-research/doxycycline-diets/ |
| ENCODE Database | Stanford University | https://www.encodeproject.org/ | |
| iCre mRNA synthesis plasmid (pBBI) | Addgene | 65795 | https://www.addgene.org/65795/ |
| IPTG | GoldBio | I2481C | https://www.goldbio.com/search?isSearch=Y&q=iptg |
| pGL3-Basic | Promega | E1751 | https://www.promega.com/products/reporter-assays-and-transfection/reporter-vectors-and-cell-lines/pgl3-luciferase-reporter-vectors/?catNum=E1751 |
| SVM-BPfinder | Regulatory Genomics, Pompeu Fabra University | http://regulatorygenomics.upf.edu/Software/SVM_BP/ | |
| TiProD: Tissue specific promoter Database | Department of Bioinformatics, UMG, University of Göttingen | http://tiprod.bioinf.med.uni-goettingen.de | |
| UCSC Genome Browser | University of California Santa Cruz | https://genome.ucsc.edu/ |
Referencias
- Jackson-Grusby, L., et al. Loss of genomic methylation causes p53-dependent apoptosis and epigenetic deregulation. Nature Genetics. 27 (1), 31-39 (2001).
- David, G., Turner, G. M., Yao, Y., Protopopov, A., DePinho, R. A. mSin3-associated protein, mSds3, is essential for pericentric heterochromatin formation and chromosome segregation in mammalian cells. Genes & Development. 17 (19), 2396-2405 (2003).
- Sumi-Ichinose, C., Ichinose, H., Metzger, D., Chambon, P. SNF2beta-BRG1 is essential for the viability of F9 murine embryonal carcinoma cells. Molecular and Cellular Biology. 17 (10), 5976-5986 (1997).
- Premsrirut, P. K., et al. A rapid and scalable system for studying gene function in mice using conditional RNA interference. Cell. 145 (1), 145-158 (2011).
- Qiu, S., Adema, C. M., Lane, T. A computational study of off-target effects of RNA interference. Nucleic Acids Research. 33 (6), 1834-1847 (2005).
- Gilbert, L. A., et al. Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell. 159 (3), 647-661 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2015).
- Peng, H., Ivanov, A. V., Oh, H. J., Lau, Y. F., Rauscher, F. J. Epigenetic gene silencing by the SRY protein is mediated by a KRAB-O protein that recruits the KAP1 co-repressor machinery. The Journal of Biological Chemistry. 284 (51), 35670-35680 (2009).
- Groner, A. C., et al. KRAB-zinc finger proteins and KAP1 can mediate long-range transcriptional repression through heterochromatin spreading. PLOS Genetics. 6 (3), 1000869 (2010).
- Lee, K. H., Oghamian, S., Park, J. A., Kang, L., Laird, P. W. The REMOTE-control system: a system for reversible and tunable control of endogenous gene expression in mice. Nucleic Acids Research. 45 (21), 12256-12269 (2017).
- Gossen, M., Bonin, A. L., Bujard, H. Control of gene activity in higher eukaryotic cells by prokaryotic regulatory elements. Trends in Biochemical Sciences. 18 (12), 471-475 (1993).
- Hu, M. C., Davidson, N. The inducible lac operator-repressor system is functional in mammalian cells. Cell. 48 (4), 555-566 (1987).
- Cronin, C. A., Gluba, W., Scrable, H. The lac operator-repressor system is functional in the mouse. Genes & Development. 15 (12), 1506-1517 (2001).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118 (2), 401-415 (1993).
- Council, N. R. . Guide for the Care and Use of Laboratory Animals: Eighth Edition. , (2011).
- Casper, J., et al. The UCSC Genome Browser database: 2018 update. Nucleic Acids Research. 46 (1), 762-769 (2018).
- Church, D. M., et al. Lineage-specific biology revealed by a finished genome assembly of the mouse. PLOS Biology. 7 (5), 1000112 (2009).
- Creyghton, M. P., et al. Histone H3K27ac separates active from poised enhancers and predicts developmental state. Proceedings of the National Academy of Sciences of the United States of America. 107 (50), 21931-21936 (2010).
- Heintzman, N. D., et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature. 459 (7243), 108-112 (2009).
- Heintzman, N. D., et al. Distinct and predictive chromatin signatures of transcriptional promoters and enhancers in the human genome. Nature Genetics. 39 (3), 311-318 (2007).
- Rada-Iglesias, A., et al. A unique chromatin signature uncovers early developmental enhancers in humans. Nature. 470 (7333), 279-283 (2011).
- Handoko, L., et al. CTCF-mediated functional chromatin interactome in pluripotent cells. Nature Genetics. 43 (7), 630-638 (2011).
- Splinter, E., et al. CTCF mediates long-range chromatin looping and local histone modification in the beta-globin locus. Genes & Development. 20 (17), 2349-2354 (2006).
- The ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Rosenbloom, K. R., et al. ENCODE data in the UCSC Genome Browser: year 5 update. Nucleic Acids Research. 41, 56-63 (2013).
- Murray, J. I., Voelker, R. B., Henscheid, K. L., Warf, M. B., Berglund, J. A. Identification of motifs that function in the splicing of non-canonical introns. Genome Biology. 9 (6), 97 (2008).
- Taggart, A. J., et al. Large-scale analysis of branchpoint usage across species and cell lines. Genome Research. 27 (4), 639-649 (2017).
- Corvelo, A., Hallegger, M., Smith, C. W., Eyras, E. Genome-wide association between branch point properties and alternative splicing. PLOS Computational Biology. 6 (11), 1001016 (2010).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Grainger, S., et al. CRISPR Guide RNA Validation In Vitro. Zebrafish. 14 (4), 383-386 (2017).
- Quadros, R. M., Harms, D. W., Ohtsuka, M., Gurumurthy, C. B. Insertion of sequences at the original provirus integration site of mouse ROSA26 locus using the CRISPR/Cas9 system. FEBS Open Bio. 5, 191-197 (2015).
- Harms, D. W., et al. Mouse Genome Editing Using the CRISPR/Cas System. Current Protocols in Human Genetics. 83, (2014).
- Miura, H., Quadros, R. M., Gurumurthy, C. B., Ohtsuka, M. Easi-CRISPR for creating knock-in and conditional knockout mouse models using long ssDNA donors. Nature Protocols. 13 (1), 195-215 (2018).
- Laird, P. W., et al. Simplified mammalian DNA isolation procedure. Nucleic Acids Research. 19 (15), 4293 (1991).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Akcakaya, P., et al. In vivo CRISPR editing with no detectable genome-wide off-target mutations. Nature. 561 (7723), 416-419 (2018).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nature Protocols. 13 (11), 2615-2642 (2018).
- Ohtsuka, M., et al. Improvement of pronuclear injection-based targeted transgenesis (PITT) by iCre mRNA-mediated site-specific recombination. Transgenic Research. 22 (4), 873-875 (2013).
- Ohtsuka, M., et al. Pronuclear injection-based mouse targeted transgenesis for reproducible and highly efficient transgene expression. Nucleic Acids Research. 38 (22), 198 (2010).
- Lee, G., Saito, I. Role of nucleotide sequences of loxP spacer region in Cre-mediated recombination. Gene. 216 (1), 55-65 (1998).
- Thomson, J. G., Rucker, E. B., Piedrahita, J. A. Mutational analysis of loxP sites for efficient Cre-mediated insertion into genomic DNA. Genesis. 36 (3), 162-167 (2003).
- Cho, A., Haruyama, N., Kulkarni, A. B. Generation of Transgenic Mice. Current Protocols in Cell Biology. , (2009).
- Pu, X. A., Young, A. P., Kubisch, H. M. Production of Transgenic Mice by Pronuclear Microinjection. Methods in Molecular Biology. 1874, 17-41 (2019).
- Murgha, Y., et al. Combined in vitro transcription and reverse transcription to amplify and label complex synthetic oligonucleotide probe libraries. Biotechniques. 58 (6), 301-307 (2015).
- Quadros, R. M., et al. Easi-CRISPR: a robust method for one-step generation of mice carrying conditional and insertion alleles using long ssDNA donors and CRISPR ribonucleoproteins. Genome Biology. 18 (1), 92 (2017).
- Chen, X., Wu, J. M., Hornischer, K., Kel, A., Wingender, E. TiProD: the Tissue-specific Promoter Database. Nucleic Acids Research. 34, 104-107 (2006).
- Haruyama, N., Cho, A., Kulkarni, A. B. Overview: Engineering transgenic constructs and mice. Current Protocols in Cell Biology. , (2009).
- Tasic, B., et al. Site-specific integrase-mediated transgenesis in mice via pronuclear injection. Proceedings of the National Academy of Sciences of the United States of America. 108 (19), 7902-7907 (2011).
- JoVE Science Education Database. Lab Animal Research. Fundamentals of Breeding and Weaning. Journal of Visualized Experiments. , (2018).
- Wyborski, D. L., DuCoeur, L. C., Short, J. M. Parameters affecting the use of the lac repressor system in eukaryotic cells and transgenic animals. Environmental and Molecular Mutagenesis. 28 (4), 447-458 (1996).
- Wyborski, D. L., Short, J. M. Analysis of inducers of the E.coli lac repressor system in mammalian cells and whole animals. Nucleic Acids Research. 19 (17), 4647-4653 (1991).
- Traykova-Brauch, M., et al. An efficient and versatile system for acute and chronic modulation of renal tubular function in transgenic mice. Nature Medicine. 14 (9), 979-984 (2008).
- Michel, G., Mosser, J., Fauran, F. Serum kinetics of doxycycline polyphosphate in dogs. European Journal of Drug Metabolism and Pharmacokinetics. 4 (1), 43-48 (1979).
- Bertocchi, I., et al. Regulatory functions of limbic Y1 receptors in body weight and anxiety uncovered by conditional knockout and maternal care. Proceedings of the National Academy of Sciences of the United States of America. 108 (48), 19395-19400 (2011).
- Plageman, T. F., Lang, R. A. Generation of an Rx-tTA: TetOp-Cre knock-in mouse line for doxycycline regulated Cre activity in the Rx expression domain. PLOS One. 7 (11), 50426 (2012).
- Mollova, M., et al. Cardiomyocyte proliferation contributes to heart growth in young humans. Proceedings of the National Academy of Sciences of the United States of America. 110 (4), 1446-1451 (2013).
- Ptashne, M. Principles of a switch. Nature Chemical Biology. 7 (8), 484-487 (2011).
- Balazsi, G., van Oudenaarden, A., Collins, J. J. Cellular decision making and biological noise: from microbes to mammals. Cell. 144 (6), 910-925 (2011).
- Nevozhay, D., Zal, T., Balazsi, G. Transferring a synthetic gene circuit from yeast to mammalian cells. Nature Communications. 4, 1451 (2013).
- Labow, M. A., Baim, S. B., Shenk, T., Levine, A. J. Conversion of the lac repressor into an allosterically regulated transcriptional activator for mammalian cells. Molecular and Cellular Biology. 10 (7), 3343-3356 (1990).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados