Method Article
Applicazione in vivo del sistema di controllo remoto per la manipolazione dell'espressione genica endogena
In questo articolo
Riepilogo
Questo protocollo illustra i passaggi necessari per generare un sistema di modello in cui la trascrizione di un gene endogeno di interesse può essere controllata in modo condizionale in animali vivi o celle utilizzando avanzata lac repressore e/o sistemi di attivatore di tet .
Abstract
Qui descriviamo un protocollo per l'implementazione del sistema di controllo remoto (Reversible Modifica of Transcription Endogenous loci), che consente un controllo reversibile e sintonizzabile l'espressione di un sistemi modello gene endogeno di interesse nella vita. Il sistema di controllo remoto utilizza avanzata lac repressione e tet attivazione sistemi per raggiungere giù - o upregulation di un determinato gene all'interno di un unico sistema biologico. Repressione stretto può essere raggiunto da repressore associazione siti flessibilmente situati lontano a valle di un sito di inizio trascrizione inibendo l'allungamento della trascrizione. Upregulation robusto può essere raggiunto aumentando la trascrizione di un gene endogeno prendendo di mira il attivatori trascrizionali tet al promotore cognato. Questo controllo di espressione reversibile e sintonizzabile può essere applicato e ritirato ripetutamente negli organismi. La potenza e la versatilità del sistema, come dimostrato per endogena Dnmt1 qui, vi permetterà più precisare analisi funzionale in vivo consentendo indagini di funzione del gene a vari livelli di espressione e verificando la reversibilità di un fenotipo.
Introduzione
Knockout genetica o approcci transgenici sono stati efficaci mezzi per studiare la funzione del gene in modelli animali. Tuttavia, la regolazione dell'espressione di questi approcci è dicotomica (acceso/spento), non-temporale e quindi non è in grado di rivelare lo spettro completo e funzionale di un gene. Condizionale Cre/LoxP tecnologie hanno permesso spazio-temporali inattivazione o l'attivazione della funzione del gene, ma la loro natura dicotomica continua a porre limitazioni, quali cella letalità e irreversibilità1,2 , 3. al fine di riempire questo vuoto, condizionali atterramento approcci sono stati sviluppati utilizzando tet-regolato shRNA o miRNA4. Tuttavia, fuori bersaglio effetti rimangono una preoccupazione per RNAi5 e sono stato impegnativo per controllare in vivo. Più recentemente, tecnologie di controllo trascrizionale mediata da CRISPR e Cas hanno introdotto un approccio più versatile per raggiungere up - e downregulation dell'espressione del gene endogeno e hanno dimostrato la loro utilità6,7 . Tuttavia, l'efficacia del controllo trascrizionale mediata da CRISPR e Cas è ancora chiaro in vivo e la reversibilità della repressione basata su KRAB rimane per essere visto, come forte repressione da KRAB e sua proteina interagente KAP1 è stato indicato per indurre 8,9il silenziamento genico permanente.
Per risolvere queste limitazioni, abbiamo sviluppato un nuovo sistema di regolamentazione trascrizionale in grado di controllare in modo condizionale espressione genica endogena in un reversibile e sintonizzabile modo nei topi utilizzando progettato procariotica binario trascrizionale sistemi di regolamentazione10. Prokaryotic binari sistemi normativi trascrizionale con ligandi di regolamentazione, come lac e tet, hanno permesso tali reversibile e sintonizzabile espressione controllo11,12,13, 14. Tuttavia, la potenza di repressione inadeguato il sistema binario corrente ha impedito loro l'adozione per controllo dell'espressione genica endogena nei mammiferi. Abbiamo sviluppato un sistema di repressione lac avanzata sufficientemente potente per la repressione di geni endogeni e impiegato una nuova strategia di targeting tet attivatori trascrizionali direttamente al cognate promotore di un gene endogeno per raggiungere upregulation robusto (Figura 1)10. Con questa tecnologia, abbiamo raggiunto quasi due ordini di grandezza controllo di espressione del gene endogeno Dnmt1 in un modo sintonizzabile, viscoelastico e reversibile10. Qui forniamo istruzioni dettagliate per la sua applicazione in vivo ad altri geni e organismi utilizzando topi come una specie di modello.

Figura 1 : Panoramica del sistema di controllo remoto. La trascrizione di un gene target endogeni può essere regolata utilizzando derivati dal lac repressore e sistemi di attivatore di tet . Il promotore del gene bersaglio o introne è progettato per contenere operatori per il repressore LacIGY tight binding e/o rtattivatore TA-M2. R indica Repron (Repression intrin), che contiene 12 operatori lac simmetrica (S) più un introne di beta-globina coniglio parziale. T indica l'operatore di tet . Il repressore e/o attivatore è/sono espressi da un promotore tessuto-specifica. L'espressione del gene dell'obiettivo può quindi essere sintonizzato reversibilmente al livello di espressione desiderata dall'amministrazione di IPTG (isopropyl β-D-1-thiogalactopyranoside, un antagonista del repressore di LacIGY) o doxiciclina (Dox). Questa figura è stata modificata da Lee et al.10fare clic qui per visualizzare una versione più grande di questa figura.
Prima di iniziare questo protocollo, esaminare la tabella 1 per identificare le operazioni rilevanti per il controllo desiderato dell'espressione genica. Ad esempio, per progettare un mouse che consente reversibile downregulation di "Gene X", completare sezioni 1, 3 e 4 del sotto protocollo. La tabella 1 riassume anche i componenti necessari del sistema di controllo remoto.
| Cambiamento di espressione desiderata | Repressione solo | Attivazione solo | Sia di repressione e di attivazione |
| Sezioni pertinenti del protocollo | 1, 3-4 | 2-4 | 1-4 |
| Telecomando sequenza necessaria nel Gene Target | Repron ("Introne repressione"; 12 operatori lac simmetrico più un introne di beta-globina coniglio parziale) | Tet all'operatore | All'operatore Repron e Tet |
| Posizione di sequenza telecomando | Introne | Promotore | Introne & Promoter |
| Attivatore/repressore necessari per controllo desiderato | LacIGY repressore | Attivatore di rtTA-M2 | LacIGY repressore e attivatore rtTA-M2 |
| Ligandi normativi | IPTG | Doxiciclina | IPTG e/o doxiciclina |
Tabella 1: Panoramica di componenti di controllo remoto.
Protocollo
Tutte le procedure di animali sono stati condotti con l'approvazione del istituzionale Animal Care e uso Committee (IACUC) della University of Southern California e l'Istituto di ricerca di Van Andel e in conformità con la guida per la cura e l'uso di animali da laboratorio dagli istituti nazionali di salute15.
1. modificare il gene di interesse per la repressione di telecomando
- Utilizzando le linee guida di seguito, identificare un introne trascrizionalmente inerte verso l'estremità 5' del gene di interesse per l'inserimento della sequenza Repron ("Repression intrsu"; 12 operatori lac simmetrica plus un introne di beta-globina coniglio parziale). Essere consapevoli dei promotori alternativi per il gene di interesse e scegliere un introne secondo il transcript(s) essere controllato (cioè un introne condiviso da tutte le trascrizioni o uno specifico per una trascrizione desiderata).
Nota: Per i geni con quelli verso l'estremità 3' o senza un introne, inserire la sequenza di Repron nel promotore (Vedi Lee, et al.10 per una procedura dettagliata).- Ottenere la sequenza genomic per il gene di interesse.
- Passare al UCSC Genome Browser16,17e selezionare l'ultima bozza del genoma del topo (Mouse GRCm38/mm10 al momento della pubblicazione), che è attualmente sotto la scheda di genomi .
- Immettere il nome o il simbolo del gene di interesse nella barra di ricerca per visualizzare le trascrizioni per il gene. Fare clic su Vai.
- Selezionare la variante desiderata trascrizione per il gene di interesse.
- Clicca sul simbolo del gene accanto la variante di trascrizione di interesse (il simbolo della trascrizione precedentemente selezionato sarà in una scatola scuro).
- Sotto la bandiera di sequenza e collegamenti a strumenti e database , fare clic sul collegamento di Sequenza genomica .
- Per le Opzioni di sequenza recupero regione, selezionare solo esoni (5' UTR, CD e 3' UTR), introni e il valore predefinito FASTA un record per ciascun gene. Per le Opzioni di formattazione di sequenza, selezionare esoni in lettere maiuscole, tutto il resto in minuscolo e Maschera si ripete a N (per nascondere le sequenze ripetute). Quindi fare clic su Invia.
- Salvare questa sequenza, preservando il superiore - e formattazione, in un documento o un programma che può essere annotata in minuscolo.
- Evitare l'interruzione delle isole di CpG, che possono indicare le regioni con funzione regolatrice del gene.
Nota: Anche se un introne 5' è preferibile per l'inserimento della sequenza Repron, primo introne può contenere elementi regolatori trascrizionali come isole di CpG (esaminate in questo passaggio) o gli elementi enhancer (esaminati nel passaggio 1.1.4), che devono essere evitati per Repron inserimento.- Sotto la bandiera di espressione e la regolazione del UCSC Genome Browser, selezionare Visualizza per la traccia di Isole CpG e fare clic su Aggiorna.
- Zoomando su 5' introni, fare clic su ogni isola di CpG (mostrata in verde al momento della pubblicazione, se presenti) e selezionare Visualizza DNA per questa funzionalità.
- Dopo aver selezionato la maschera si ripete a N, selezionare ottenere DNA per ottenere la sequenza di isola CpG.
- Queste sequenze di sovrapposizione con il file originale di sequenza e annotare questi come regioni introniche per evitare a causa di funzioni di regolamentazione gene possibile.
- Evitare l'interruzione di non-codificazione RNAs, nel caso in cui essi hanno una funzione regolatrice.
- Sotto la bandiera di geni e Gene previsioni del UCSC Genome Browser, selezionare Visualizza per la traccia GENCODE (Ensembl) e fare clic su Aggiorna.
- Zoomando su 5' introni, fare clic su ogni RNA non codificanti (mostrato in verde al momento della pubblicazione, se presenti) e ottenere la sequenza del DNA per ciascuno facendo clic sulle relative coordinate cromosomiche.
- Nel menu a discesa Visualizza , selezionare il DNAe fare clic su maschera ripete a N. Quindi fare clic su ottenere il DNA.
- Queste sequenze di sovrapposizione con il file originale di sequenza e annotare questi come regioni introniche per evitare a causa di funzioni di regolamentazione gene possibile.
- Evitare le regioni introniche con firme enhancer in tessuto d'interesse, quali H3K4me1, H3K27ac e dnasi ho ipersensibilità18,19,20,21, come pure l'associazione CTCF siti, che regolano il rinforzatore looping22,23.
- Passare alla codifica database24,25e selezionare l'icona di esperimenti .
- Per il tipo di analisi, selezionare ChIP-seq o dnasi-seqe popolare le altre categorie (organismo, tipo Biosample, ecc.) secondo le cellule per essere costruito.
- Dopo selezione funzionalità, passa il mouse sopra i pittogrammi in blu e selezionare quello per il quale Visualizzare i risultati come elenco appare (più a sinistra pittogramma al momento della pubblicazione).
- Selezionare il set di dati per gli obiettivi di H3K4me1, H3K27ac, dnasi I e CTCF che corrispondono maggiormente le cellule per essere progettato.
- All'interno di ogni set di dati pertinenti, scorrere fino alla sezione file , verificare che mm10 (o l'ultima versione di genoma) e UCSC siano selezionate e fare clic sul pulsante Visualizza .
- Ora nel Browser genoma UCSC e zoomando su introni 5' per il gene di interesse, cliccate su ogni H3K4me1, H3K27ac, DNasi io e picco CTCF nelle loro piste rispettivo picco con annotazioni.
- Ottenere la sequenza di DNA per ogni area di picco cliccando sulle relative coordinate cromosomiche.
- Nel menu a discesa Visualizza , selezionare il DNAe fare clic su maschera ripete a N. Quindi fare clic su ottenere il DNA.
- Queste sequenze di sovrapposizione con il file originale di sequenza e annotare questi come regioni introniche per evitare a causa di funzioni di regolamentazione gene possibile.
- Evitare interruzioni di consenso d'impionbatura di sequenze, per non interferire con appropriato splicing degli esoni.
Nota: Anche se sequenze necessarie per lo splicing sono essenzialmente limitate a circa sei basi all'estremità 5' di un introne26 e circa 60 basi al 3' finiscono27, si consiglia di selezionare un introne grande, permettendo per considerevolmente più ampi margini da questi regioni di consenso per minimizzare la probabilità di impatto con la giunzione. Strumenti di analisi dell'introne, come SVM-BPfinder28, si consiglia per un'analisi più approfondita dei potenziali splice sequenze.
- Ottenere la sequenza genomic per il gene di interesse.
- Dopo aver identificato una regione intronic che soddisfa i criteri di cui sopra (come esemplificato per Dnmt1 nei dati supplementari), schermo la sequenza utilizzando un sgRNA online progettare strumento per identificare un sgRNA nella regione con alta specificità e predetto gol di efficienza.
- Passare a uno strumento di progettazione sgRNA online di scelta, come ad esempio CRISPOR29.
- Immettere la sequenza della regione intronic di interesse, specificare il genoma di riferimento pertinenti e selezionare il desiderato Protospacer adiacente Motif (PAM). Fate clic su Submit.
- Ordinare il sgRNAs previsto dal punteggio di specificità e selezionare uno o più sgRNAs che hanno anche un elevato rendimento previsto Punteggio29.
- Facoltativo: Per massimizzare la probabilità di utilizzo di un efficace sgRNA, prima testare l'efficienza di clivaggio di diversi sgRNAs massimi in un saggio in vitro30e procedere con il sgRNA più efficiente in vivo.
- Progettare un modello di DNA contenente un PITT (basati su iniezione pronuclei mirati transgenesi) atterraggio sequenza di rilievo, come esemplificato nella Figura complementare 1A, B, fiancheggiato su entrambi i lati da braccia di omologia 60-base che corrispondono ai sgRNA tagliata sito31 .
Nota: La piattaforma di atterraggio contiene due siti eterotipiche loxP (JT15 e Lox2272) e consente l'inserimento mirato di grandi sequenze attraverso un approccio in due fasi; Assicurarsi che le giunzioni di questo inserimento non creano luoghi criptici della giuntura. In alternativa, una cellula staminale embrionale (ESC) - base knock - in strategia utilizzabile per inserire la sequenza di Repron direttamente. - Preparare il sgRNA, Cas9 proteina e il modello singolo filamento di DNA (ssDNA) per la piattaforma di atterraggio e microinject in uova fecondate (da topi B6C3F1/J o altro ceppo desiderato) secondo protocolli stabiliti32,33.
- Schermo per topi con knock-in rilievo di atterraggio.
- Disegnare primers PCR complementari al locus genomico ma fuori delle regioni mirate per le braccia di omologia, come dimostrato per Dnmt1 in complementare figura 1. Evitare sequenze genomic ripetitive quando progetta i primer.
- Estrarre il DNA dalle clip coda dei topi secondo protocolli stabiliti34.
- Utilizzare la PCR ed elettroforesi per identificare i topi con un inserimento di rilievo di atterraggio.
- Confermare che il knock-in è stato successo ordinando i prodotti PCR.
Nota: Le riorganizzazioni e indesiderate grandi omissioni possono essere introdotto da CRISPR/Cas935, così attento screening per l'editing di fuori bersaglio è consigliato prima di procedere35,36,37.
- Utilizzando uova fecondate dai mouse pad atterraggio, microinject iCre mRNA38 e un plasmide transgenico contenente la sequenza di Repron affiancata da JTZ17 e Lox2272 ricombinazione siti31,39,40, 41 secondo stabilito metodi38,42,43.
- Schermo per il mouse con l'inserimento di Repron.
- Progettazione PCR primer complementari al locus genomico, fuori l'inserto Repron. Evitare sequenze genomic ripetitive quando progetta i primer.
- Estrarre il DNA dalle clip coda dei topi secondo protocolli stabiliti34.
- Utilizzare la PCR ed elettroforesi per identificare i topi con un inserimento di Repron.
- Confermare che il knock-in è stato successo ordinando i prodotti PCR.
2. modificare il gene di interesse per il upregulation di telecomando
- Utilizzando le linee guida di seguito, identificare una regione nel promotore del gene di interesse che è improbabile per perturbare la funzione di promotore al momento dell'inserimento delle sequenze di operatore di tet . Essere consapevoli dei promotori alternativi per il gene di interesse e scegliere un promotore secondo il transcript(s) essere controllato (cioè un promotore condiviso da tutte le trascrizioni o uno specifico per una trascrizione desiderata).
- Ottenere la sequenza genomic del promotore di interesse.
- Passare al UCSC Genome Browser16,17e selezionare l'ultima bozza del genoma del topo (Mouse GRCm38/mm10 al momento della pubblicazione), che è attualmente sotto la scheda di genomi .
- Immettere il nome o il simbolo del gene di interesse nella barra di ricerca per visualizzare le trascrizioni per il gene. Fare clic su Vai.
- Selezionare la variante desiderata trascrizione per il gene di interesse.
- Clicca sul simbolo del gene accanto la variante di trascrizione di interesse (la sigla del gene della trascrizione precedentemente selezionata sarà in una scatola scuro).
- Sotto la bandiera di sequenza e collegamenti a strumenti e database , fare clic sul collegamento di Sequenza genomica .
- Per le Opzioni di sequenza recupero regione, selezionare solo promotore/Upstream di 1000 basi. Per le Opzioni di formattazione di sequenza, selezionare Maschera ripete a N (per nascondere le sequenze ripetute). Quindi fare clic su Invia.
- Salvare la sequenza del promotore in un documento o un programma che può essere annotata.
- Seleziona regioni che sono privi di siti di legame di fattore di trascrizione putativo, come interrompere queste sequenze possono alterare l'espressione genica endogena.
- Passare al browser del genoma di UCSC e quindi aprire l'ultima versione di genoma del topo.
- Sopra il banner di mappatura e sequenziamento , selezionare traccia mozzi.
- Fare clic su mm10 accanto al nome del mozzo di JASPAR 2018 TFBS (o l'ultima versione JASPAR).
- Immettere il nome o il simbolo del gene di interesse nella barra di ricerca per visualizzare le trascrizioni per il gene. Fare clic su Vai.
- Selezionare la variante desiderata trascrizione per il gene di interesse.
- Scorrere fino all'insegna di JASPAR 2018 TFBS e fare clic sulla freccia a discesa per selezionare l'opzione di visualizzazione desiderata per la pista, come squish. Fare clic su Aggiorna.
- Zoom nella regione del promotore del gene di interesse e identificare le aree che sono gratuiti (o relativamente libero) di siti di legame del fattore di trascrizione secondo la traccia JASPAR. Registrare le coordinate cromosomiche di queste regioni.
- Ottenere la sequenza genomica di queste cromosomiche coordinate scrivendole nella barra di ricerca e fare clic su Vai. Nel menu a discesa Visualizza , selezionare il DNAe fare clic su maschera ripete a N. Quindi fare clic su ottenere il DNA.
- Queste sequenze di sovrapposizione con il file originale di sequenza e annotare questi come regioni promotore ideale di destinazione a causa della mancanza di legame del fattore di trascrizione.
- All'interno di queste sequenze, selezionare una regione di promotore perturbable che è a Monte, ma vicino al luogo di inizio di trascrizione del gene di interesse.
Nota: Un'inserzione che è troppo vicino al che sito di inizio della trascrizione potrebbe aumentare il rischio di alterare l'attività del promotore, ma un inserimento che è troppo lontano può diminuire il livello di upregulation. Inserimento di sequenze di operatore tet circa 200 basi a Monte del sito di inizio della trascrizione ha provocato robusto upregulation dei due promotori testato (Vedi la discussione)10.
- Ottenere la sequenza genomic del promotore di interesse.
- Schermo della regione promotore selezionato utilizzando uno strumento di progettazione online sgRNA per identificare un sgRNA nella regione con alta specificità e punteggi di efficienza previsti.
- Passare a uno strumento di progettazione sgRNA online di scelta, come ad esempio CRISPOR29.
- Immettere la sequenza della regione di interesse, specificare il genoma di riferimento pertinenti e selezionare il desiderato Protospacer adiacente Motif (PAM). Fate clic su Submit.
- Ordinare il sgRNAs previsto dal punteggio di specificità e selezionare uno o più sgRNAs che hanno anche un elevato rendimento previsto Punteggio29.
- Facoltativo: Per massimizzare la probabilità di utilizzo di un efficace sgRNA, prima testare l'efficienza di clivaggio di diversi sgRNAs in un' analisi in vitro30e procedere con il sgRNA più efficiente in vivo.
- Progettare un modello di DNA contenente operatori tet fiancheggiati su entrambi i lati da braccia di omologia 60-base che corrispondono ai sgRNA tagliata sito31,33.
Nota: Il numero di operatori di tet è personalizzabile; inserimento di due-quattro sequenze di operatore tet in tandem precedentemente è stato indicato per essere efficace, ma in linea di principio più operatori sono desiderabili per una guida più alta espressione. In alternativa, una strategia basata su ESC knock-in può essere utilizzata per inserire gli operatori di tet .- Facoltativo: Per la prova sperimentale che le modifiche proposte probabilmente non disturba l'attività endogena trascrizionale del gene di interesse, clonare la sequenza del promotore ingegnerizzati in un vettore di luciferasi di lucciola (ad esempio pGL3-Basic), e confrontare la sua efficacia al promotore originale utilizzando un'analisi di luciferase.
- Preparare il sgRNA, Cas9 proteina e il modello di ssDNA contenente le sequenze di operatore del tet e microinject in uova fecondate (da topi B6C3F1/J o altro ceppo desiderato) secondo protocolli stabiliti32,33.
Nota: A causa della complessità di sintetizzare una sequenza ripetitiva, un approccio basato su trascrizione in vitro trascrizione/inverso è consigliato per sintetizzare un modello di ssDNA da un doppio filamento del DNA (dsDNA) plasmide44. In alternativa, un modello di dsDNA può essere utilizzato per microiniezione, ma l'efficienza knock-in può essere ridotta45. - Schermo per topi con knock-degli operatori tet .
- Progettazione PCR primer complementari al locus genomico, fuori delle regioni mirati per le braccia di omologia. Evitare sequenze genomic ripetitive quando progetta i primer.
- Estrarre il DNA dalle clip coda dei topi secondo protocolli stabiliti34.
- Utilizzare la PCR ed elettroforesi per identificare i topi con l'inserimento degli operatori di tet .
- Confermare che il knock-in è stato successo ordinando i prodotti PCR.
Nota: Le riorganizzazioni e indesiderate grandi omissioni possono essere introdotto da CRISPR/Cas935, così attento screening per l'editing di fuori bersaglio è consigliato prima di procedere35,36,37.
3. sviluppare attivatore - e/o topi che esprimono repressore
- Identificare un promotore robustamente esprime per i tipi di tessuto o cellula di interesse.
Nota: Una ricerca di letteratura e il tessuto-specifico promotore Database46 può essere utile per identificare un promotore di tale. - Posizionare il repressore fornito avanzata lac o sequenza attivatore tet a valle del promotore desiderato per generare un costrutto transgenico.
- Produrre le righe di topo transgenico utilizzando le procedure standard di transgenici42,43,47. In alternativa, può essere utilizzato un approccio di transgenesi site-specific per evitare effetti di posizione e per consentire la copia singola transgene inserimento31,48.
- Propagare i fondatori e determinare il pattern di espressione e il livello del transgene nella prole di ciascun fondatore. Selezionare righe con robusta espressione nei tipi tessuto previsto per ulteriori d'allevamento.
- Allevare i fondatori di topi selvatici.
- Gli iniettori di PCR che rileveranno l'inserimento di attivatore e/o repressore di design.
- Estrarre il DNA dalle clip coda della prole secondo protocolli stabiliti34.
- Utilizzare la PCR ed elettroforesi per identificare i topi con l'inserimento.
- Confermare l'inserimento del transgene mediante sequenziamento dei prodotti di PCR.
- Propagare le linee transgeniche.
- Sacrificare alcuni cuccioli da ogni riga e raccogliere i tessuti di interesse per qRT-PCR, immunoistochimica e/o western blotting per analizzare l'espressione di gene/della proteina di interesse per i tipi di tessuto di destinazione.
- Selezionare le righe con l'espressione più forte nei tessuti di interesse per l'uso nella sezione 4.
4. modificare l'espressione genica in vivo
- Allevare i topi con il gene modificato di interesse (sezione 1 e/o 2) con i topi transgenici dalla sezione 3 secondo allevamento stabiliti pratiche49. Per controllo di massima espressione, allevare i topi di omozigosi per l'allele modificata.
- Per reversione o regolazione della repressione del gene target, amministrare IPTG nell'acqua potabile.
- Per determinare sperimentalmente la dose di IPTG utilizzare, trattare topi del genotipo appropriato e controlli con uno di una gamma di dosi (consigliato a partire di gamma: 0 – 400 mM IPTG) per almeno una settimana50,51. Includere almeno tre topi per gruppo di trattamento e selezionare l'età e il sesso dei topi che sono più rilevanti per i futuri con gli esperimenti pianificati. Nota: Coppie riproduttrici di topi possono essere trattate con acqua IPTG per fornire l'esposizione nello sviluppo di IPTG alla prole se desiderato13, o topi possono iniziare il trattamento qualsiasi momento dopo la nascita.
- Sciogliere la massa desiderata di IPTG in acqua distillata sterile il giorno di somministrazione e mescolare con un ancoretta per 5 minuti o fino a quando completamente sciolto.
- Avvolgere la bottiglia con un foglio e amministrare l'acqua IPTG in una bottiglia di luce-protetto. Sostituire due volte a settimana. Fornire la stessa fonte di acqua ai topi che ricevono 0 mM IPTG.
- Dopo almeno una settimana, sacrificare i topi e analizzare l'espressione del gene di interesse per il tessuto di destinazione mediante qRT-PCR, immunoistochimica e/o macchiarsi occidentale.
- Identificare la dose che ripristina l'espressione del gene di interesse a quello dei comandi wildtype e utilizzare questa dose per raggiungere espressione normale del gene in futuri esperimenti.
- Per determinare sperimentalmente la dose di IPTG utilizzare, trattare topi del genotipo appropriato e controlli con uno di una gamma di dosi (consigliato a partire di gamma: 0 – 400 mM IPTG) per almeno una settimana50,51. Includere almeno tre topi per gruppo di trattamento e selezionare l'età e il sesso dei topi che sono più rilevanti per i futuri con gli esperimenti pianificati. Nota: Coppie riproduttrici di topi possono essere trattate con acqua IPTG per fornire l'esposizione nello sviluppo di IPTG alla prole se desiderato13, o topi possono iniziare il trattamento qualsiasi momento dopo la nascita.
- Per l'induzione del gene upregulation, somministrare doxiciclina (Dox) nella dieta.
- Per determinare sperimentalmente la concentrazione di Dox per amministrare, trattare topi del genotipo appropriato e controlli con una concentrazione di gamma di Dox (consigliato a partire di gamma: 0-5000 mg/kg Doxycycline Hyclate) per almeno una settimana52 ,53, tra cui almeno tre topi per gruppo di trattamento. Acquistare Dox-contenenti cibo del mouse da un fornitore commerciale.
Nota: Coppie di topi possono essere trattate con Dox cibo per fornire l'esposizione nello sviluppo di Dox alla prole se desiderato54,55, o topi possono iniziare il trattamento qualsiasi momento dopo la nascita. - Sostituire la dieta una volta a settimana. Fornire la stessa dieta base ai topi che ricevono alimenti privi di Dox.
- Dopo almeno una settimana, sacrificare i topi e analizzare l'espressione del gene di interesse per il tessuto di destinazione mediante qRT-PCR, immunoistochimica e/o macchiarsi occidentale.
- Identificare la dose che eleva l'espressione del gene di interesse al livello desiderato e utilizzare questa dose per raggiungere la sovraespressione del gene in futuri esperimenti.
- Per determinare sperimentalmente la concentrazione di Dox per amministrare, trattare topi del genotipo appropriato e controlli con una concentrazione di gamma di Dox (consigliato a partire di gamma: 0-5000 mg/kg Doxycycline Hyclate) per almeno una settimana52 ,53, tra cui almeno tre topi per gruppo di trattamento. Acquistare Dox-contenenti cibo del mouse da un fornitore commerciale.
Risultati
La capacità di repressione del regime di controllo remoto è stata dimostrata in due diversi approcci finora. Nel primo approccio, siti di legame del repressore lac sono stati inseriti al promotore del gene di Dnmt1 endogeno. Nel secondo approccio, che è consigliato dal presente protocollo, i siti di legame del repressore sono stati inseriti in un introne a valle per evitare il rischio di intaccare la funzione di promotore tramite l'inserzione e, quindi, per semplificare l'applicazione del telecomando- sistema. Entrambi gli approcci hanno provocato successo repressione (Figura 2A, B e Figura 3A-C)10. DNMT1 espressione fu repressa al 15% dei livelli non regolamentati utilizzando l'approccio basato su promotore (Figura 2A). Questa repressione stretta è stata invertita in modo dose-dipendente trattando i topi con quantità variabili di IPTG (Figura 2A). La repressione di Dnmt1 osservata è stata convalidata a livello proteico immunostaining (Figura 2B). Non abbiamo osservato alcuna differenza nell'espressione Dnmt1 tra Dnmt1+ + e Dnmt1LO/LO topi, confermando che il nostro inserimento di operatore lac non aveva interrotto promotore normale funzione10. L'approccio basato su introne raggiunto più di 90% di repressione da parte degli operatori si trova parecchi kilobasi a valle del sito di inizio trascrizione attenuando trascrizione allungamento (Figura 3A, B)10. Questo approccio basato su introne è stato ulteriormente convalidato sui sette ulteriori robusti promotori (Figura 3C). Repressione invariabilmente stretto è stato raggiunto da tutti i promotori testati. È stata osservata alcuna correlazione tra i livelli di espressione residua e i punti di forza dei promotori, suggerendo che la capacità di repressione del nostro sistema basato su introne repressione supera la potenza trascrizionale di tutti i promotori robusta abbiamo testato ( Figura 3 C).
La capacità di in vivo upregulation del sistema telecomando inoltre è stata dimostrata sul gene Dnmt1 . Abbiamo introdotto due copie dell'operatore tet nel promotore Dnmt1 , insieme a sequenze di operatore lac , per consentire o upregulation o downregulation a seconda di quale effettrici proteina è presente. Upregulation robusto e downregulation dell'espressione Dnmt1 , vicino a due ordini di grandezza (10% al 650%), sono stati raggiunti in CES che contiene l'allele di Dnmt1 endogeno modificata (Dnmt1LGT) (Figura 4A)10 . Entrambi i regolamenti sono stati completamente reversibile e viscoelastico dai trattamenti IPTG e Dox, rispettivamente (Figura 4A). Successivamente abbiamo introdotto la modifica Dnmt1LGT nella linea germinale del mouse per testare la capacità di in vivo upregulation del sistema di controllo remoto. Forte upregulation di Dnmt1 è stata osservata da fegato, milza e rene, mentre nessun upregulation rilevabile nel cuore è stata osservata (Figura 4B)7. Il modello di espressione del ciclo cellulare-dipendente di Dnmt1 e la scarsità delle cellule proliferative nel cuore può essere alla base di questa osservazione10,56. Resta da vedere se questa limitazione può essere superata aumentando il livello di espressione di attivatore o il numero dei suoi siti di legame.
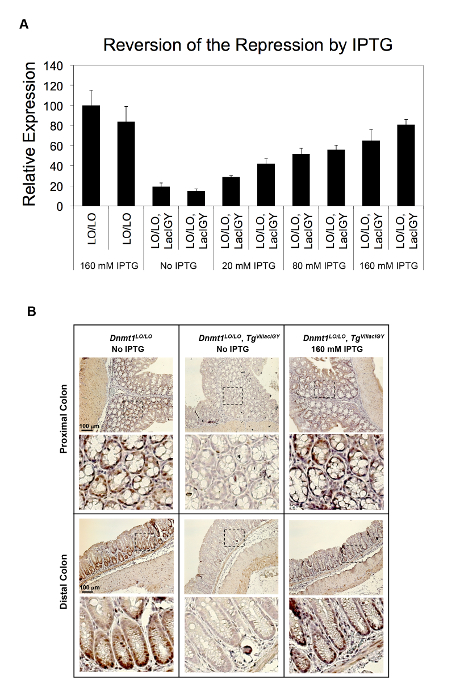
Figura 2 : In vivo repressione di Dnmt1 dal repressore LacIGY. Topi (A), con operatori di lac (LO), inseriti nel promotore Dnmt1 , con o senza espressione di LacIGY, sono stati trattati con varie dosi di IPTG. qRT-PCR analisi di espressione Dnmt1 dimostra l'inversione di dose-dipendente di Dnmt1 repressione in vivo dal trattamento di IPTG. Ogni barra rappresenta dati da un altro mouse. I dati rappresentano la media ± SEM (n = 3). (B) Immunostaining di Dnmt1 proteine nelle cripte coliche dei topi fornito acqua potabile con o senza 160 mM IPTG per 3 settimane. Questa figura è stata modificata da Lee et al.10fare clic qui per visualizzare una versione più grande di questa figura.
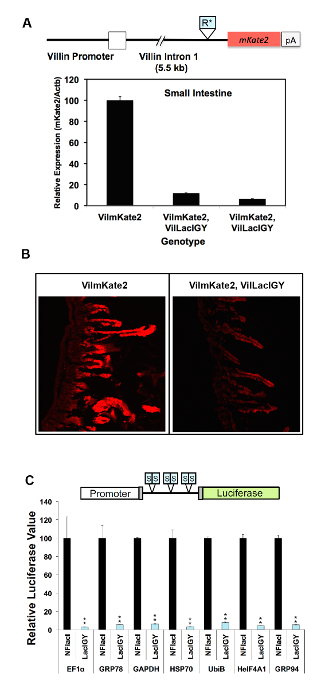
Figura 3 : In vivo e repressione in vitro dei vari promotori dal sistema di controllo remoto. (A) una prima versione della sequenza Repron (R *) è stato inserito in un introne a valle del promotore villina in un topo transgenico villina-mKate2 (VilmKate2). viene visualizzata la finestra di analisi qRT-PCR dell'espressione di mKate2 nell'intestino tenue dei topi con o senza il repressore di LacIGY . Ogni barra rappresenta dati da un altro mouse. (B) mKate2 confocale immagini del piccolo intestino con e senza espressione di LacIGY . (C) sei operatori lac simmetrica (S) sono stati inseriti tra i vari promotori e un reporter di luciferase. Reporter (50 ng/pozzetto in piastra a 96 pozzetti) e repressore plasmidi transitoriamente sono stati introdotti nelle cellule di NIH/3T3 in un rapporto molare 1:1. Valori di luciferasi sono stati valutati 24 h dopo la trasfezione. Questi dati in vitro rappresentano la percentuale di espressione di luciferasi in LacIGY-che esprimono le cellule rispetto a quelli che esprimono non funzionali LacI (NFlacI). T-test sono stati utilizzati per determinare la significatività statistica. I dati rappresentano la media ± SEM (n = 3). P ≤ 0.05, * *P ≤ 0,01. Questa figura è stata modificata da Lee et al.10fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4 : Down - e/o upregulation dell'espressione Dnmt1 in vitro e in vivo. (A) il sistema di controllo remoto completo è stato progettato nel CES colta di gene targeting ed elettroporazione approcci. Massima repressione dell'espressione Dnmt1 è stata realizzata con nessun trattamento mentre attivazione massimale è stato raggiunto dal trattamento di Dox e IPTG. I dati rappresentano la media ± SEM (n = 3). P ≤ 0.05, * *P ≤ 0,01 (di Welch t-test). (B) attivazione In vivo di Dnmt1 dal sistema di controllo remoto, come dimostrato immunostaining di Dnmt1 proteina in vari tessuti dai topi di controllo remoto. L'allele LGT rappresenta modifica promotore di Dnmt1 per contenere i siti di legame di attivatore di operatore e tet di lac . Topi sono stati trattati con una dieta normale o contenenti Dox (5000 mg/kg Doxycycline Hyclate) per un mese. Questa figura è stata modificata da Lee et al.10fare clic qui per visualizzare una versione più grande di questa figura.
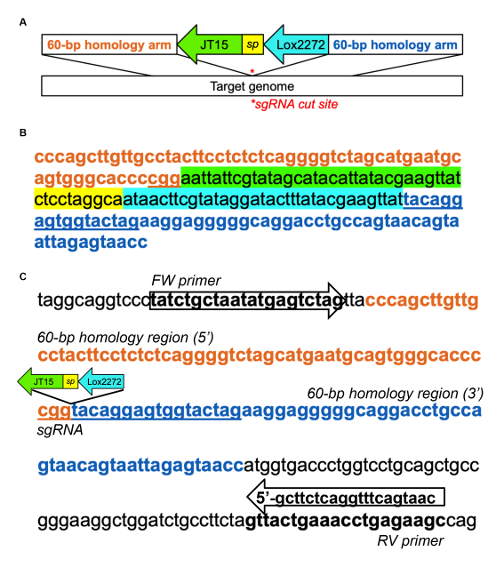
Complementare figura 1 : Esempio di inserimento di rilievo di atterraggio in murino Dnmt1 introne 1. (A) schema del modello di DNA per atterraggio inserimento pad, adattato da Quadros et al (2015)31. Eterotipiche loxP siti, JT15 e Lox2272, sono separati da una sequenza di brevi distanziale (sp) e fiancheggiati da ogni lato da 60-bp di DNA che è omologo alla regione genomica di destinazione. (B) DNA campione modello per atterraggio pad inserimento in introne Dnmt1 utilizzando il seguente sgRNA: CTAGTACCACTCCTGTACCG (che è destinato a filo del inverso). La regione intronic selezionata era bioinformatically informato dal passo 1.1 e il sgRNA è stato identificato usando CRISPOR29. (C) esempio di disegno dell'iniettore di PCR per valutare l'inserimento alla piattaforma di atterraggio. Gli iniettori di PCR sono stati progettati di fuori le braccia di omologia del modello per confermare integrazione endogeno Dnmt1. L'amplicone PCR wildtype ' 213 bp; al momento dell'inserimento, diventa 291 BP. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Un passaggio fondamentale e limitazione potenziale del sistema di controllo remoto è la sfida connessa con l'inserimento di repressore e/o siti di legame di attivatore senza alterare l'espressione genica. Il nostro approccio originale di repressione, come applicata al gene Dnmt1 , coinvolti inserimento di siti di legame di repressore lac all'interno di regioni trascrizionalmente critiche di un promotore. Al fine di ridurre il rischio di intaccare la funzione di promotore e quindi di migliorare l'applicabilità generale del sistema di controllo remoto, abbiamo sviluppato un approccio basato su introne repressione. La potenza del nostro sistema avanzata lac ci ha permesso di strettamente reprimere la trascrizione di tutti i promotori forti abbiamo testato presso operatori situati centinaia di parecchi kilobasi a valle della trascrizione avvia siti (Figura 3A – C) 10. d'importanza, i livelli di repressione erano indipendenti dei punti di forza trascrizionale dei promotori (Figura 3A – C)10. Ciò suggerisce che la capacità di repressione del nostro sistema basato su introne repressione supera la forza trascrizionale dei promotori testati. In questo approccio basato su introne, è probabile che la repressione è mediata attraverso interferenze fisiche tra due componenti, il macchinario di allungamento della trascrizione e il lac repressori57. Questo meccanismo semplice repressione e l'affidabilità dimostrata del metodo basato su introne possono rendere questo approccio generalmente applicabile ad organismi, tessuti e geni differenti.
Il upregulation del sistema di controllo remoto richiede le sequenze di associazione transactivator di essere in prossimità del promotore del gene di destinazione, che comporta un rischio di intaccare la funzione di promotore. Tuttavia, abbiamo trovato che la posizione di sequenze di associazione può essere di fuori della regione critica trascrizionalmente. Promotori sia Dnmt1 ed EF1α erano robustamente sovraregolati da tet operatori si trovano un paio di centinaia di basi a Monte della trascrizione inizio siti10. Questo vincolo rilassato riduce notevolmente la possibilità di pregiudicare la funzione di promotore di un gene bersaglio in assenza del transactivator. Aumentando il numero di associazione sequenze e/o uso di transactivators più forte potrebbe contribuire a ridurre ulteriormente il rischio attivando upregulation da siti più lontano il sito di inizio della trascrizione.
Il nostro sistema di controllo remoto fornisce elegante controllo dei livello, il tempismo e la posizione dell'espressione genica endogena, consentendo per testare la reversibilità di un fenotipo e le conseguenze dei livelli di espressione diversa, che non sono facilmente ottenibili da attuali tecnologie di controllo di espressione genica in vivo. È importante notare che nella maggior parte delle analisi di espressione genica, compreso il nostro, espressione valori rappresentano la media di una popolazione di cellule tra cui considerevole variazione può essere trovata. Questa eterogeneità può influenzare i processi decisionali cellulari, come differenziazione o apoptosi58. Anche se la precisione del controllo di espressione genica potrebbe probabilmente essere ulteriormente migliorata da ulteriori circuito genetico ingegneria59, la potenza osservata del nostro attuale sistema permetterà utile indagine della funzione genica in molti contesti biologici. Inoltre, un elevato grado di specificità di bersaglio è previsto a causa della complessità delle sequenze di operatore, nonché la grande distanza evolutiva tra mammiferi e le specie di origine dei componenti regolatori60. Inoltre, linee di topi transgenici di attivatori e repressori possono essere sviluppate e utilizzate per qualsiasi gene endogeno. Ad esempio, modelli di mouse transactivator tet esistenti possono essere adattati per compire il upregulation di un determinato gene nei tessuti del mouse desiderato. Abbiamo recentemente sviluppato una linea transgenica che può guidare robusta espressione tessuto-specifica del nostro repressore lac rafforzata in diversi tipi di tessuto quando combinato con linee di Cre esistenti introducendo il gene lacIGY nel locus Hipp11 48 sotto il controllo di un elemento di Lox-STOP-Lox (non pubblicato). Questa linea sarebbe sostanzialmente facilitare l'applicazione di tessuto-specifica del sistema di controllo remoto.
Upregulation di gene dal sistema di controllo remoto fornisce diversi vantaggi rispetto alle attuali approcci transgenici inducibili. Non richiede la generazione di più linee transgeniche per testare gli effetti di posizione dell'inserzione, poiché utilizza il locus endogeno. Inoltre, questo approccio è adatto upregulation dei geni con espressione basale forte perché migliora l'espressione da un promotore già robusto, considerando che modelli transgenici convenzionali si basano su minimi promotori virali. Infine, il tessuto specificità, controllo del ciclo cellulare e varianti di geni bersaglio di splicing possono essere conservati su upregulation dal nostro approccio, cui conserva elementi di regolazione naturale come innata cis-elementi regolatori. L'avvento della tecnologia di gene-targeting CRISPR/Cas-mediata agevolerà notevolmente l'applicazione di questa tecnologia in sistemi modello diversi.
Divulgazioni
PWL serve sul Scientific Advisory Boards di AnchorDx e Progenity, Inc.
Riconoscimenti
Ringraziamo il tardo Dr Heidi Scrable per suo generoso dono del costrutto gene lacI mammiferi (Mayo Clinic, Rochester, MN), Dr Daniel Louvard (Institut Curie, Parigi, Francia) per fornire il promotore di villina e Dr Laurie Jackson-Grusby (ospedale pediatrico, Boston, MA) per i suoi contributi alle prime fasi di questo sviluppo di tecnologia. Siamo grati per Dr Nancy Wu e Dr Robert Maxson per la loro assistenza nella generazione di topi transgenici e knockoui. Si ringraziano i membri del laboratorio Laird per utili discussioni e supporto. Questo lavoro è stato supportato dal National Institutes of Health [CA75090 R01, DA030325 R01, CA157918 R01 e R01 CA212374 a p.w.l. e 1F31CA213897-01A1 a N.A.V.S].
Materiali
| Name | Company | Catalog Number | Comments |
| B6C3F1/J | The Jackson Laboratory | 100010 | https://www.jax.org/strain/100010 |
| Cas9 Protein | PNA Bio | CP04 | http://www.pnabio.com/products/CRISPR_Cas9.htm?gclid=EAIaIQobChMIsoG8pLL33QIVBr7ACh0naQ4dEAAYAiAAEgKyHvD_BwE |
| CRISPOR | Haeussler et al. 2016 | http://crispor.tefor.net/ | |
| Doxycycline-Containing Mouse Diet | Envigo | Varies by concentration | https://www.envigo.com/products-services/teklad/laboratory-animal-diets/custom-research/doxycycline-diets/ |
| ENCODE Database | Stanford University | https://www.encodeproject.org/ | |
| iCre mRNA synthesis plasmid (pBBI) | Addgene | 65795 | https://www.addgene.org/65795/ |
| IPTG | GoldBio | I2481C | https://www.goldbio.com/search?isSearch=Y&q=iptg |
| pGL3-Basic | Promega | E1751 | https://www.promega.com/products/reporter-assays-and-transfection/reporter-vectors-and-cell-lines/pgl3-luciferase-reporter-vectors/?catNum=E1751 |
| SVM-BPfinder | Regulatory Genomics, Pompeu Fabra University | http://regulatorygenomics.upf.edu/Software/SVM_BP/ | |
| TiProD: Tissue specific promoter Database | Department of Bioinformatics, UMG, University of Göttingen | http://tiprod.bioinf.med.uni-goettingen.de | |
| UCSC Genome Browser | University of California Santa Cruz | https://genome.ucsc.edu/ |
Riferimenti
- Jackson-Grusby, L., et al. Loss of genomic methylation causes p53-dependent apoptosis and epigenetic deregulation. Nature Genetics. 27 (1), 31-39 (2001).
- David, G., Turner, G. M., Yao, Y., Protopopov, A., DePinho, R. A. mSin3-associated protein, mSds3, is essential for pericentric heterochromatin formation and chromosome segregation in mammalian cells. Genes & Development. 17 (19), 2396-2405 (2003).
- Sumi-Ichinose, C., Ichinose, H., Metzger, D., Chambon, P. SNF2beta-BRG1 is essential for the viability of F9 murine embryonal carcinoma cells. Molecular and Cellular Biology. 17 (10), 5976-5986 (1997).
- Premsrirut, P. K., et al. A rapid and scalable system for studying gene function in mice using conditional RNA interference. Cell. 145 (1), 145-158 (2011).
- Qiu, S., Adema, C. M., Lane, T. A computational study of off-target effects of RNA interference. Nucleic Acids Research. 33 (6), 1834-1847 (2005).
- Gilbert, L. A., et al. Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell. 159 (3), 647-661 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2015).
- Peng, H., Ivanov, A. V., Oh, H. J., Lau, Y. F., Rauscher, F. J. Epigenetic gene silencing by the SRY protein is mediated by a KRAB-O protein that recruits the KAP1 co-repressor machinery. The Journal of Biological Chemistry. 284 (51), 35670-35680 (2009).
- Groner, A. C., et al. KRAB-zinc finger proteins and KAP1 can mediate long-range transcriptional repression through heterochromatin spreading. PLOS Genetics. 6 (3), 1000869 (2010).
- Lee, K. H., Oghamian, S., Park, J. A., Kang, L., Laird, P. W. The REMOTE-control system: a system for reversible and tunable control of endogenous gene expression in mice. Nucleic Acids Research. 45 (21), 12256-12269 (2017).
- Gossen, M., Bonin, A. L., Bujard, H. Control of gene activity in higher eukaryotic cells by prokaryotic regulatory elements. Trends in Biochemical Sciences. 18 (12), 471-475 (1993).
- Hu, M. C., Davidson, N. The inducible lac operator-repressor system is functional in mammalian cells. Cell. 48 (4), 555-566 (1987).
- Cronin, C. A., Gluba, W., Scrable, H. The lac operator-repressor system is functional in the mouse. Genes & Development. 15 (12), 1506-1517 (2001).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118 (2), 401-415 (1993).
- Council, N. R. . Guide for the Care and Use of Laboratory Animals: Eighth Edition. , (2011).
- Casper, J., et al. The UCSC Genome Browser database: 2018 update. Nucleic Acids Research. 46 (1), 762-769 (2018).
- Church, D. M., et al. Lineage-specific biology revealed by a finished genome assembly of the mouse. PLOS Biology. 7 (5), 1000112 (2009).
- Creyghton, M. P., et al. Histone H3K27ac separates active from poised enhancers and predicts developmental state. Proceedings of the National Academy of Sciences of the United States of America. 107 (50), 21931-21936 (2010).
- Heintzman, N. D., et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature. 459 (7243), 108-112 (2009).
- Heintzman, N. D., et al. Distinct and predictive chromatin signatures of transcriptional promoters and enhancers in the human genome. Nature Genetics. 39 (3), 311-318 (2007).
- Rada-Iglesias, A., et al. A unique chromatin signature uncovers early developmental enhancers in humans. Nature. 470 (7333), 279-283 (2011).
- Handoko, L., et al. CTCF-mediated functional chromatin interactome in pluripotent cells. Nature Genetics. 43 (7), 630-638 (2011).
- Splinter, E., et al. CTCF mediates long-range chromatin looping and local histone modification in the beta-globin locus. Genes & Development. 20 (17), 2349-2354 (2006).
- The ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Rosenbloom, K. R., et al. ENCODE data in the UCSC Genome Browser: year 5 update. Nucleic Acids Research. 41, 56-63 (2013).
- Murray, J. I., Voelker, R. B., Henscheid, K. L., Warf, M. B., Berglund, J. A. Identification of motifs that function in the splicing of non-canonical introns. Genome Biology. 9 (6), 97 (2008).
- Taggart, A. J., et al. Large-scale analysis of branchpoint usage across species and cell lines. Genome Research. 27 (4), 639-649 (2017).
- Corvelo, A., Hallegger, M., Smith, C. W., Eyras, E. Genome-wide association between branch point properties and alternative splicing. PLOS Computational Biology. 6 (11), 1001016 (2010).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Grainger, S., et al. CRISPR Guide RNA Validation In Vitro. Zebrafish. 14 (4), 383-386 (2017).
- Quadros, R. M., Harms, D. W., Ohtsuka, M., Gurumurthy, C. B. Insertion of sequences at the original provirus integration site of mouse ROSA26 locus using the CRISPR/Cas9 system. FEBS Open Bio. 5, 191-197 (2015).
- Harms, D. W., et al. Mouse Genome Editing Using the CRISPR/Cas System. Current Protocols in Human Genetics. 83, (2014).
- Miura, H., Quadros, R. M., Gurumurthy, C. B., Ohtsuka, M. Easi-CRISPR for creating knock-in and conditional knockout mouse models using long ssDNA donors. Nature Protocols. 13 (1), 195-215 (2018).
- Laird, P. W., et al. Simplified mammalian DNA isolation procedure. Nucleic Acids Research. 19 (15), 4293 (1991).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Akcakaya, P., et al. In vivo CRISPR editing with no detectable genome-wide off-target mutations. Nature. 561 (7723), 416-419 (2018).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nature Protocols. 13 (11), 2615-2642 (2018).
- Ohtsuka, M., et al. Improvement of pronuclear injection-based targeted transgenesis (PITT) by iCre mRNA-mediated site-specific recombination. Transgenic Research. 22 (4), 873-875 (2013).
- Ohtsuka, M., et al. Pronuclear injection-based mouse targeted transgenesis for reproducible and highly efficient transgene expression. Nucleic Acids Research. 38 (22), 198 (2010).
- Lee, G., Saito, I. Role of nucleotide sequences of loxP spacer region in Cre-mediated recombination. Gene. 216 (1), 55-65 (1998).
- Thomson, J. G., Rucker, E. B., Piedrahita, J. A. Mutational analysis of loxP sites for efficient Cre-mediated insertion into genomic DNA. Genesis. 36 (3), 162-167 (2003).
- Cho, A., Haruyama, N., Kulkarni, A. B. Generation of Transgenic Mice. Current Protocols in Cell Biology. , (2009).
- Pu, X. A., Young, A. P., Kubisch, H. M. Production of Transgenic Mice by Pronuclear Microinjection. Methods in Molecular Biology. 1874, 17-41 (2019).
- Murgha, Y., et al. Combined in vitro transcription and reverse transcription to amplify and label complex synthetic oligonucleotide probe libraries. Biotechniques. 58 (6), 301-307 (2015).
- Quadros, R. M., et al. Easi-CRISPR: a robust method for one-step generation of mice carrying conditional and insertion alleles using long ssDNA donors and CRISPR ribonucleoproteins. Genome Biology. 18 (1), 92 (2017).
- Chen, X., Wu, J. M., Hornischer, K., Kel, A., Wingender, E. TiProD: the Tissue-specific Promoter Database. Nucleic Acids Research. 34, 104-107 (2006).
- Haruyama, N., Cho, A., Kulkarni, A. B. Overview: Engineering transgenic constructs and mice. Current Protocols in Cell Biology. , (2009).
- Tasic, B., et al. Site-specific integrase-mediated transgenesis in mice via pronuclear injection. Proceedings of the National Academy of Sciences of the United States of America. 108 (19), 7902-7907 (2011).
- JoVE Science Education Database. Lab Animal Research. Fundamentals of Breeding and Weaning. Journal of Visualized Experiments. , (2018).
- Wyborski, D. L., DuCoeur, L. C., Short, J. M. Parameters affecting the use of the lac repressor system in eukaryotic cells and transgenic animals. Environmental and Molecular Mutagenesis. 28 (4), 447-458 (1996).
- Wyborski, D. L., Short, J. M. Analysis of inducers of the E.coli lac repressor system in mammalian cells and whole animals. Nucleic Acids Research. 19 (17), 4647-4653 (1991).
- Traykova-Brauch, M., et al. An efficient and versatile system for acute and chronic modulation of renal tubular function in transgenic mice. Nature Medicine. 14 (9), 979-984 (2008).
- Michel, G., Mosser, J., Fauran, F. Serum kinetics of doxycycline polyphosphate in dogs. European Journal of Drug Metabolism and Pharmacokinetics. 4 (1), 43-48 (1979).
- Bertocchi, I., et al. Regulatory functions of limbic Y1 receptors in body weight and anxiety uncovered by conditional knockout and maternal care. Proceedings of the National Academy of Sciences of the United States of America. 108 (48), 19395-19400 (2011).
- Plageman, T. F., Lang, R. A. Generation of an Rx-tTA: TetOp-Cre knock-in mouse line for doxycycline regulated Cre activity in the Rx expression domain. PLOS One. 7 (11), 50426 (2012).
- Mollova, M., et al. Cardiomyocyte proliferation contributes to heart growth in young humans. Proceedings of the National Academy of Sciences of the United States of America. 110 (4), 1446-1451 (2013).
- Ptashne, M. Principles of a switch. Nature Chemical Biology. 7 (8), 484-487 (2011).
- Balazsi, G., van Oudenaarden, A., Collins, J. J. Cellular decision making and biological noise: from microbes to mammals. Cell. 144 (6), 910-925 (2011).
- Nevozhay, D., Zal, T., Balazsi, G. Transferring a synthetic gene circuit from yeast to mammalian cells. Nature Communications. 4, 1451 (2013).
- Labow, M. A., Baim, S. B., Shenk, T., Levine, A. J. Conversion of the lac repressor into an allosterically regulated transcriptional activator for mammalian cells. Molecular and Cellular Biology. 10 (7), 3343-3356 (1990).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon