Method Article
Application in vivo de la système de télécommande pour la Manipulation d’Expression des gènes endogènes
Dans cet article
Résumé
Ce protocole décrit les étapes nécessaires pour générer un modèle de système dans lequel la transcription d’un gène endogène d’intérêt peut être conditionnellement contrôlée dans les animaux vivants ou de cellules à l’aide d’activator, renforcée lac répresseur et tet .
Résumé
Nous décrivons ici un protocole d’application du régime de contrôle à distance (réversible Manipulation of Transcription locus Endogenous), qui permet de contrôler l’expression réversible et ajustable d’un systèmes de modèle endogène gène d’intérêt dans la vie. Le système de contrôle à distance emploie améliorée bac répression et tet systèmes d’activation pour atteindre le bas ou stimulation de l’expression d’un gène cible dans un système biologique unique. Répression serrée peut être atteint de répresseur accepteurs souple situé très en aval d’un site de début de transcription en inhibant l’élongation de la transcription. Upregulation robuste peut être atteint en améliorant la transcription d’un gène endogène en ciblant les activateurs transcriptional tet au promoteur apparenté. Ce contrôle de l’expression réversible et ajustable peut être appliqué et retiré à plusieurs reprises dans les organismes. La puissance et la polyvalence du système, comme l’a démontré pour endogène Dnmt1 ici, permettra aux plus précises analyses fonctionnelles in vivo en permettant l’étude de la fonction du gène à différents niveaux d’expression et en vérifiant la réversibilité d’un phénotype.
Introduction
Masquage génétique ou approches transgéniques ont été un moyen efficace pour étudier la fonction des gènes dans des modèles animaux. Cependant, régulation de l’expression de ces approches est dichotomique (marche/arrêt), non-temporelle et donc n’est pas capable de révéler le spectre fonctionnel complet d’un gène. Conditionnelle Cre/LoxP technologies ont permis l’inactivation spatio-temporelle ou activation de la fonction du gène, mais leur nature dichotomique continue à poser des limites, comme la létalité cellulaire et irréversibilité1,2 , 3. afin de combler ce manque, conditionnels knockdown approches ont été développées à l’aide de tet-régis Sharn ou miRNA4. Cependant, hors cible effets demeurent une préoccupation pour les Arni5 et ont été difficile à contrôler in vivo. Plus récemment, les technologies de contrôle transcriptionnel CRISPR/Cas-mediated ont introduit une approche plus souple pour réaliser aussi bien en amont et une diminution de l’expression des gènes endogènes et démontré leurs utilitaires6,7 . Cependant, l’efficacité du contrôle transcriptionnel CRISPR/Cas-mediated ignore encore in vivo et la réversibilité de répression axées sur le KRAB n’est toujours pas vue, aussi forte répression par KRAB et sa protéine d’interaction KAP1 a été montré pour induire permanente gene silencing8,9.
Afin de pallier ces lacunes, nous avons développé un nouveau système réglementaire transcriptionnel capable de contrôler sous certaines conditions l’expression des gènes endogènes en réversible et accordable manière chez la souris à l’aide d’ingénierie transcriptionnelle procaryote binaire les systèmes de réglementation10. Procaryotes binaires systèmes de régulation transcriptionnelles avec des ligands réglementaires, tels que le lac et tet, ont permis à ce réversible et expression accordable contrôler11,12,13, 14. Cependant, la puissance de répression insuffisante des systèmes binaires actuels a nui à leur adoption généralisée pour le contrôle de l’expression des gènes endogènes chez les mammifères. Nous avons développé un système de répression renforcée lac suffisamment puissant pour la répression des gènes endogènes et employé une stratégie novatrice de ciblage tet transcription activateurs directement au promoteur apparenté d’un gène endogène pour atteindre upregulation robuste (Figure 1)10. Avec cette technologie, nous avons atteint presque deux ordres de grandeur contrôle d’expression du gène endogène Dnmt1 dans une manière accordable, inductible et réversible10. Ici, nous fournissons des instructions détaillées pour son application in vivo à d’autres gènes et les organismes en utilisant la souris comme une espèce de modèle.

Figure 1 : Vue d’ensemble du système télécommande. La transcription d’un gène endogène cible peut être réglée à l’aide machiné lac répresseur et systèmes d’activateur de tet . Le promoteur du gène cible ou l’intron est conçu pour contenir des opérateurs pour le répresseur de LacIGY attachement intime et/ou la rtTA-M2 activator. R indique Repron (Repl intrsur), qui contient 12 exploitants lac symétrique (S) plus un intron de bêta-globine lapin partielle. T indique opérateur tet . Le répresseur et/ou l’activateur est/sont exprimés à partir d’un promoteur spécifique aux tissus. L’expression du gène cible peut alors réversiblement activera le niveau d’expression voulue par l’administration d’IPTG (isopropyl β-D-1-thiogalactopyranoside, un antagoniste de la LacIGY répresseur) ou Doxycycline (Dox). Ce chiffre a été modifié par Lee et al.,10s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Avant de commencer le présent protocole, revue tableau 1 afin d’identifier les mesures pertinentes pour le contrôle souhaité de l’expression génique. Par exemple, pour une souris qui permet la diminution réversible du « Gène X » de l’ingénieur, remplissez les sections 1, 3 et 4 de la sous protocole. Le tableau 1 résume également les composants nécessaires du système de contrôle à distance.
| Changement de l’Expression souhaitée | Seulement la répression | Activation seulement | La répression et l’Activation |
| Sections pertinentes du protocole | 1, 3-4 | 2-4 | 1-4 |
| Séquence de contrôle à distance nécessaire dans le gène cible | Repron (« Intron de répression » ; 12 exploitants symétrique bac plus un intron de bêta-globine lapin partielle) | Tet opérateur (s) | Repron et Tet opérateur (s) |
| Emplacement de la séquence de contrôle à distance | Intron | Promoteur | Intron & promoteur |
| Activator/répresseur nécessaire pour le contrôle souhaité | LacIGY répresseur | rtTA-M2 Activator | LacIGY répresseur et rtTA-M2 Activator |
| Ligands réglementaires | IPTG | Doxycycline | IPTG et/ou Doxycycline |
Tableau 1 : Vue d’ensemble des composants de contrôle à distance.
Protocole
Toutes les procédures d’animaux ont été menées avec l’approbation de l’animalier institutionnel et utilisation Comité (IACUC) de l’University of Southern California et le Van Andel Research Institute et en conformité avec le Guide pour le soin et l’utilisation des animaux de laboratoire des National Institutes of Health,15.
1. modifier le gène d’intérêt pour la répression de la télécommande
- En utilisant les lignes directrices ci-dessous, identifier un intron transcriptionnellement inerte vers l’extrémité 5' du gène d’intérêt pour l’insertion de la séquence Repron ( »Repl intrsur «; 12 opérateurs symétriques bac plus un intron de bêta-globine lapin partielle). Soyez conscient des promoteurs alternatifs pour le gène d’intérêt, puis choisissez un intron selon les relevés de notes à contrôler (c'est-à-dire un intron partagé par toutes les transcriptions ou celui spécifique à une transcription désirée).
Remarque : Pour les gènes avec ceux vers l’extrémité 3' ou sans un intron, insérez la séquence Repron le promoteur (voir Lee, Al.10 pour une procédure détaillée).- Obtenir la séquence génomique pour le gène d’intérêt.
- Accédez à l’UCSC Genome Browser16,17, puis sélectionnez la dernière version du génome de la souris (souris GRCm38/mm10 au moment de la publication), qui est actuellement sous l’onglet de génomes .
- Entrez le nom ou le symbole du gène d’intérêt dans la barre de recherche pour afficher la transcription du gène. Cliquez sur OK.
- Sélectionner la variante souhaitée de transcription du gène d’intérêt.
- Cliquez sur le symbole de gène à côté de la variante de transcription d’intérêt (le symbole de la transcription précédemment sélectionné sera dans une boîte sombre).
- Sous la bannière de la séquence et des liens vers des outils et bases de données , cliquez sur le lien de la Séquence génomique .
- Pour les Options région d’extraction de séquence, sélectionnez uniquement les Exons (5' UTR, CDS et 3' UTR), Introns et la valeur par défaut FASTA un enregistrement par gène. Pour les Options de mise en forme de séquence, sélectionnez Exons en majuscules, tout le reste en minuscules et Masque répète à N (pour dissimuler les séquences répétitives). Puis cliquez sur soumettre.
- Enregistrez cette séquence, préservant le haut - et minuscules mise en forme, dans un document ou un programme qui peut être annoté.
- Éviter toute interruption des îles de CpG, qui peut indiquer des régions avec fonction de régulation génique.
NOTE : Si un intron en 5' est préférable pour l’insertion de la séquence Repron, le premier intron peut contenir des éléments régulateurs transcriptionnels tels que les îles de CpG (examinés dans cette étape) ou éléments d’enhancer (examinés à l’étape 1.1.4), qui sont à proscrire pour Repron insertion.- Sous la bannière d’Expression et la régulation de l’UCSC Genome Browser, sélectionnez Afficher pour la voie de l’Îles de CpG et cliquez sur actualiser.
- En zoomant sur les introns 5', cliquez sur chaque île de CpG (indiqué en vert au moment de la publication, le cas échéant), puis sélectionnez View ADN pour cette fonctionnalité.
- Après avoir sélectionné le masque se répète à N, sélectionnez obtenir l’ADN pour obtenir la séquence d’île de CpG.
- Ces séquences de superposition avec le fichier original de la séquence et d’annoter ces régions intronic à éviter en raison des fonctions de régulation de gène possible.
- Éviter toute interruption de non-codage RNAs, dans le cas où ils ont une fonction régulatrice.
- Sous la bannière de gènes et les prédictions de gène de l’UCSC Genome Browser, sélectionnez Afficher le morceau GENCODE (Ensembl) et cliquez sur actualiser.
- Zoom sur les 5' introns, clic de chaque ARN non codants (indiqué en vert au moment de la publication, le cas échéant) et obtenir la séquence d’ADN pour chacun en cliquant sur ses coordonnées chromosomiques.
- Dans le menu déroulant Afficher , sélectionnez l’ADNet cliquez sur masque répète à N. Puis cliquez sur obtenir de l’ADN.
- Ces séquences de superposition avec le fichier original de la séquence et d’annoter ces régions intronic à éviter en raison des fonctions de régulation de gène possible.
- Éviter les régions intronic avec signatures de renforceur dans le tissu d’intérêt, tels que H3K4me1, H3K27ac et DNase I hypersensibilité18,19,20,21, ainsi que la fixation de la FCCT sites, qui réglementent le renforceur de bouclage22,23.
- Accédez à la base de données de ENCODE24,25, puis sélectionnez l’icône d’expériences .
- Pour le type de test, sélectionnez ChIP-seq ou DNase-seqet remplir les autres catégories (organisme, type de l’échantillon, etc.) selon les cellules à être conçues.
- Après sélection de composant, survolez les pictogrammes en bleu, puis sélectionnez celui pour lequel Afficher les résultats en tant que liste apparaît (pictogramme le plus à gauche au moment de la publication).
- Sélectionnez les séries de données pour les objectifs de H3K4me1, H3K27ac, DNase I et FCCT qui correspondent plus étroitement les cellules à être conçues.
- Au sein de chaque ensemble de données pertinente, faites défiler jusqu'à la section fichiers , vérifiez que mm10 (ou la dernière version du génome) et UCSC sont sélectionnées, puis cliquez sur le bouton visualiser .
- Maintenant dans l’UCSC Genome Browser et en zoomant sur les introns 5' du gène d’intérêt, cliquez sur chaque H3K4me1, la H3K27ac, la DNase j’ai et pic de la FCCT dans leurs voies respectives pic annoté.
- Obtenir la séquence d’ADN pour chaque région de la crête en cliquant sur ses coordonnées chromosomiques.
- Dans le menu déroulant Afficher , sélectionnez l’ADNet cliquez sur masque répète à N. Puis cliquez sur obtenir de l’ADN.
- Ces séquences de superposition avec le fichier original de la séquence et d’annoter ces régions intronic à éviter en raison des fonctions de régulation de gène possible.
- Éviter toute perturbation du consensus épissage des séquences, pour ne pas gêner l’épissage des exons approprié.
Remarque : Bien que les séquences requises pour l’épissage se limitent en grande partie à environ six bases situées à l’extrémité 5' de l’intron26 et environ 60 bases à la 3' extrémité du27, il est conseillé de sélectionner un grand intron, permettant une plus large marge de ces régions de consensus afin de minimiser la probabilité d’un impact d’épissage. Utilisation des outils d’analyse intron, comme SVM-BPfinder28, est recommandée pour une analyse plus approfondie des séquences d’épissure potentiels.
- Obtenir la séquence génomique pour le gène d’intérêt.
- Après avoir identifié une région intronic que répond aux critères ci-dessus (comme l’a démontré pour Dnmt1 dans les données supplémentaires), écran de la séquence à l’aide d’un sgRNA en ligne conception outil servant à mesurer une sgRNA dans la région avec une grande spécificité et prédit scores de l’efficacité.
- Accédez à un outil de conception en ligne sgRNA de choix, comme CRISPOR29.
- Saisissez la séquence de la région intronic d’intérêt, spécifiez le génome de référence, puis sélectionnez le Motif Adjacent de Protospacer désiré (PAM). Cliquez sur soumettre.
- Trier le sgRNAs prédite par score de spécificité et sélectionnez un ou plusieurs des sgRNAs qui ont également une haute efficacité estimée marquer29.
- Facultatif : Pour maximiser la probabilité d’utiliser un sgRNA efficace, tout d’abord tester l’efficacité de clivage de plusieurs sgRNAs haut score dans un test in vitro de30et procéder à la sgRNA plus efficace in vivo.
- Concevoir un modèle d’ADN contenant un PITT (axée sur l’Injection pronucléaire de transgenèse ciblée) landing pad séquence, comme illustré en Figure supplémentaire 1 a, B, flanqué des deux côtés de bras d’homologie 60-base qui correspondent à le sgRNA site31 de couper .
Remarque : L’aire d’atterrissage comporte deux sites hétérotypiques loxP (JT15 et Lox2272) et permet une insertion ciblée de grandes séquences grâce à une approche en deux étapes ; n’oubliez pas que les jonctions de cette insertion ne créent pas de sites d’épissage cryptique. Par ailleurs, une cellule souche embryonnaire (ESC) - fonction knock - stratégie peut servir à insérer la séquence Repron directement. - Préparer le sgRNA, protéine Cas9 et la matrice d’ADN (ADN simple brin) simple brin à l’aire d’atterrissage et microinject dans des oeufs fécondés (de souris B6C3F1/J ou autre souche désirée) selon les protocoles établis32,,33.
- Écran pour souris avec l’atterrissage touche knock-in.
- Concevoir des amorces PCR complémentaires au locus génomique, mais à l’extérieur les régions ciblées par les bras de l’homologie, comme l’a démontré pour Dnmt1 dans supplémentaire Figure 1. Évitez les séquences génomiques répétées lors de la conception des amorces.
- Extraire l’ADN d’extraits de queue des souris selon les protocoles établis,34.
- Utilisent la PCR et gel électrophorèse pour identifier les souris avec une insertion de tampon d’atterrissage.
- Confirmer que le knock-in a réussi en séquençant les produits PCR.
Remarque : Les réarrangements et délétions importantes non désirées peuvent être introduites par CRISPR/Cas935, si prudemment de dépistage pour l’édition hors-cible est conseillé avant procédure35,36,37.
- À l’aide d’oeufs fécondés des souris pad d’atterrissage, microinject iCre ARNm38 et un plasmide transgénique contenant la séquence Repron flanquée de JTZ17 et Lox2272 recombinaison sites31,39,40, 41 selon établi méthodes38,42,43.
- Écran pour souris avec l’insertion de Repron.
- Concevoir des amorces PCR complémentaires au locus génomique, en dehors de l’insert Repron. Évitez les séquences génomiques répétées lors de la conception des amorces.
- Extraire l’ADN d’extraits de queue des souris selon les protocoles établis,34.
- Utilisent la PCR et l’électrophorèse pour identifier les souris avec une insertion de Repron gel.
- Confirmer que le knock-in a été réussi en séquençant les produits PCR.
2. modifier le gène d’intérêt pour l’upregulation de contrôle à distance
- En utilisant les lignes directrices ci-dessous, indiquez une région dans le promoteur du gène d’intérêt qui est peu susceptible de perturber la fonction de promoteur lors de l’insertion de séquences opérateur tet . Soyez conscient des promoteurs alternatifs pour le gène d’intérêt et choisir un promoteur selon les relevés de notes à contrôler (c'est-à-dire un promoteur partagé par toutes les transcriptions ou un spécifique à une transcription désirée).
- Obtenir la séquence génomique pour le promoteur d’intérêt.
- Accédez à l’UCSC Genome Browser16,17, puis sélectionnez la dernière version du génome de la souris (souris GRCm38/mm10 au moment de la publication), qui est actuellement sous l’onglet de génomes .
- Entrez le nom ou le symbole du gène d’intérêt dans la barre de recherche pour afficher la transcription du gène. Cliquez sur OK.
- Sélectionner la variante souhaitée de transcription du gène d’intérêt.
- Cliquez sur le symbole de gène à côté de la variante de transcription d’intérêt (le symbole du gène de la transcription précédemment sélectionné sera dans une boîte sombre).
- Sous la bannière de la séquence et des liens vers des outils et bases de données , cliquez sur le lien de la Séquence génomique .
- Pour les Options région d’extraction de séquence, sélectionnez uniquement promoteur/Upstream en 1000 bases. Pour les Options de mise en forme de séquence, sélectionnez Masque répète à N (pour dissimuler les séquences répétitives). Puis cliquez sur soumettre.
- Enregistrez cette séquence promoteur dans un document ou un programme qui peut être annoté.
- Certaines régions qui sont libres de sites de fixation de facteur de transcription putatifs, comme interrompre ces séquences peuvent modifier l’expression des gènes endogènes.
- Accédez à l’UCSC Genome Browser et ouvrir la dernière version du génome de la souris.
- Plus haut la bannière de la cartographie et séquençage , sélectionnez suivre les moyeux.
- Cliquez sur mm10 en regard du nom de moyeu de JASPAR 2018 TFBS (ou la dernière version JASPAR).
- Entrez le nom ou le symbole du gène d’intérêt dans la barre de recherche pour afficher la transcription du gène. Cliquez sur OK.
- Sélectionner la variante souhaitée de transcription du gène d’intérêt.
- Faites défiler jusqu'à la bannière JASPAR 2018 TFBS et cliquez sur la flèche de liste déroulante pour sélectionner l’option d’affichage souhaité pour la piste, comme le squish. Cliquez sur actualiser.
- Zoomer sur la région promotrice du gène d’intérêt et de déterminer les régions qui sont libres (ou relativement libres) des sites de fixation de facteurs de transcription selon la piste JASPAR. Enregistrer les coordonnées chromosomiques de ces régions.
- Obtenir la séquence génomique de ces coordonnées chromosomiques en tapant dans la barre de recherche en cliquant sur aller. Dans le menu déroulant Afficher , sélectionnez l’ADNet cliquez sur masque répète à N. Puis cliquez sur obtenir de l’ADN.
- Ces séquences de superposition avec le fichier original de la séquence et d’annoter ces régions promoteur idéal pour cible en raison de l’absence de liaison de facteur de transcription.
- Au sein de ces séquences, sélectionnez une région perturbable promoteur qui est en amont, mais près du site de début de transcription du gène d’intérêt.
Remarque : Une insertion qui est trop proche de que site d’initiation de la transcription peut-être accroître le risque d’entraver l’activité du promoteur, mais une insertion qui est trop loin peut diminuer le niveau de stimulation de l’expression. Insertion de séquences d’opérateur tet a entraîné environ 200 basepairs en amont du site d’initiation de la transcription robuste upregulation des deux promoteurs testés (voir discussion)10.
- Obtenir la séquence génomique pour le promoteur d’intérêt.
- La région du promoteur sélectionné à l’aide d’un outil de conception en ligne sgRNA pour identifier un sgRNA dans la région avec une grande spécificité et l’efficacité prévue des scores de l’écran.
- Accédez à un outil de conception en ligne sgRNA de choix, comme CRISPOR29.
- Saisissez la séquence de la région d’intérêt, spécifiez le génome de référence, puis sélectionnez le Motif Adjacent de Protospacer désiré (PAM). Cliquez sur soumettre.
- Trier le sgRNAs prédite par score de spécificité et sélectionnez un ou plusieurs des sgRNAs qui ont également une haute efficacité estimée marquer29.
- Facultatif : Pour maximiser la probabilité d’utiliser un sgRNA efficace, tout d’abord tester l’efficacité de clivage de plusieurs sgRNAs dans un test in vitro de30et procéder à la sgRNA plus efficace in vivo.
- Concevoir un modèle d’ADN contenant les opérateurs tet , flanqués des deux côtés de bras 60-base homologie qui correspondent à la sgRNA couper site31,33.
Remarque : Le nombre d’opérateurs de tet est personnalisable ; insertion de séquences d’opérateur tet de deux à quatre en tandem a déjà démontrée pour être efficace, mais en principe plusieurs opérateurs sont souhaitables pour la conduite d’une expression plus élevée. Alternativement, une stratégie axée sur les ESC knock-in permet d’insérer les opérateurs tet .- Facultatif : Pour preuves expérimentales que les modifications proposées ne perturberont probablement pas l’activité endogène transcriptionnelle du gène d’intérêt, le clone de la séquence du promoteur machiné dans un vecteur de luciférase firefly (tel que pGL3-Basic), et comparer son efficacité au promoteur initial à un dosage de la luciférase.
- Préparer le sgRNA, Cas9 protéines et l’ADN simple brin modèle contenant les séquences d’opérateur de tet et microinject dans des oeufs fécondés (de souris B6C3F1/J ou autre souche désirée) selon les protocoles établis32,,33.
Remarque : En raison de la complexité de synthétiser une séquence répétitive, une approche axée sur la transcription de transcription in vitro/inverse est recommandée pour synthétiser un modèle d’ADN simple brin d’un double brin (dsDNA) l’ADN plasmide44. Alternativement, un modèle d’ADN double brin sont utilisables pour la microinjection, mais l’efficacité de knock-in peut être réduite de45. - Écran pour souris avec knock-in des opérateurs tet .
- Concevoir des amorces PCR complémentaires au locus génomique, en dehors des régions ciblées par les bras de l’homologie. Évitez les séquences génomiques répétées lors de la conception des amorces.
- Extraire l’ADN d’extraits de queue des souris selon les protocoles établis,34.
- Utilisent la PCR et gel électrophorèse pour identifier les souris avec l’insertion des opérateurs tet .
- Confirmer que le knock-in a réussi en séquençant les produits PCR.
Remarque : Les réarrangements et délétions importantes non désirées peuvent être introduites par CRISPR/Cas935, si prudemment de dépistage pour l’édition hors-cible est conseillé avant procédure35,36,37.
3. développer l’activateur - et/ou les souris exprimant le répresseur
- Identifier un promoteur exprimant de façon robuste pour les ou les types de tissu ou des cellules d’intérêt.
Remarque : Une recherche documentaire et le promoteur de tissu-spécifique de base de données46 peuvent être utiles pour identifier un tel promoteur. - Placez le répresseur améliorés fournis lac ou séquence d’activateur tet en aval du promoteur désiré pour générer une construction transgénique.
- Produire l’ou les lignes des souris transgéniques à l’aide de procédures standard de transgéniques42,43,47. Alternativement, une approche de la transgénèse in situ peut être utilisée afin d’éviter des effets de position et pour permettre la copie unique transgène insertion31,48.
- Propager les fondateurs et déterminer le modèle d’expression et le niveau du transgène chez les descendants de chaque fondateur. Sélectionnez expression robuste s’aligne dans les ou les types de tissus destinés pour l’élevage.
- Se reproduisent les fondateurs à des souris de type sauvage.
- Concevoir des amorces PCR qui permet de détecter l’insertion de l’activateur et/ou le répresseur.
- Extraire l’ADN d’agrafes de la queue de la progéniture selon les protocoles établis,34.
- Utilisent la PCR et gel électrophorèse pour identifier les souris avec l’insertion.
- Confirmer l’insertion du transgène par séquençage des produits PCR.
- Propager les lignées transgéniques.
- Sacrifier quelques chiots de chaque ligne et de recueillir les tissus d’intérêt pour qRT-PCR, immunohistochimie ou éponger occidental pour analyser l’expression de la gène/protéine d’intérêt dans les ou les types de tissus cibles.
- Sélectionnez les lignes avec la plus forte expression dans les tissus d’intérêt pour une utilisation à l’article 4.
4. manipuler l’expression génique in vivo
- Se reproduisent les souris avec le gène modifié d’intérêt (article 1 ou 2) avec la souris transgénique du chapitre 3 selon les pratiques de reproduction établis49. Pour le contrôle de l’expression maximale, se reproduisent les souris à l’homozygotie pour l’allèle modifié.
- Pour le retour ou l’ajustement de la répression du gène cible, administrer IPTG dans l’eau potable.
- Déterminer expérimentalement la dose d’IPTG à utiliser, traiter les souris du génotype approprié et les contrôles avec la gamme de doses (intervalle de départ a recommandé : 0 à 400 mM IPTG) au moins une semaine50,51. Inclure au moins trois souris par groupe de traitement, puis sélectionnez l’âge et le sexe des souris qui sont les plus pertinents pour les expériences futures prévues. NOTE : Couples reproducteurs de souris peuvent être traités avec de l’eau pour fournir l’exposition développementale de l’IPTG à la descendance si désiré13IPTG, ou souris peuvent commencer le traitement de n’importe quel moment après la naissance.
- Dissoudre la masse désirée d’IPTG en eau distillée stérile le jour de l’administration et remuez avec une barre de remuer pendant 5 minutes ou jusqu'à ce que complètement dissous.
- Envelopper le flacon d’aluminium et d’administrer l’eau IPTG dans un flacon de lumière-protégé. Remplacer deux fois par semaine. Fournir la source même de l’eau à des souris recevant 0 mM IPTG.
- Après au moins une semaine, sacrifier les souris et d’analyser l’expression du gène d’intérêt dans le tissu cible à l’aide de qRT-PCR, immunohistochimie ou éponger occidental.
- Identifier la dose qui restaure l’expression du gène d’intérêt pour que des contrôles de type sauvage et utiliser cette dose pour atteindre l’expression normale du gène dans les expériences futures.
- Déterminer expérimentalement la dose d’IPTG à utiliser, traiter les souris du génotype approprié et les contrôles avec la gamme de doses (intervalle de départ a recommandé : 0 à 400 mM IPTG) au moins une semaine50,51. Inclure au moins trois souris par groupe de traitement, puis sélectionnez l’âge et le sexe des souris qui sont les plus pertinents pour les expériences futures prévues. NOTE : Couples reproducteurs de souris peuvent être traités avec de l’eau pour fournir l’exposition développementale de l’IPTG à la descendance si désiré13IPTG, ou souris peuvent commencer le traitement de n’importe quel moment après la naissance.
- Pour l’induction de stimulation de l’expression génique, administrer la Doxycycline (Dox) dans l’alimentation.
- Déterminer expérimentalement la concentration de Dox à administrer, traiter les souris du génotype approprié et contrôles avec l’un d’une gamme de Dox des concentrations (départ plage recommandée : 0 – 5 000 mg/kg de Doxycycline Hyclate) au moins une semaine52 ,53, y compris au moins trois souris par groupe de traitement. Acheter de Dox-contenant la nourriture de souris auprès d’un fournisseur commercial.
NOTE : Couples reproducteurs de souris peuvent être traités avec de la nourriture de Dox à fournissent une exposition du développement de Dox à la descendance si désiré54,55, ou souris peuvent commencer le traitement de n’importe quel moment après la naissance. - Remplacer la diète une fois par semaine. Fournir l’alimentation de base même à des souris recevant des aliments exempts de Dox.
- Après au moins une semaine, sacrifier les souris et d’analyser l’expression du gène d’intérêt dans le tissu cible à l’aide de qRT-PCR, immunohistochimie ou éponger occidental.
- Identifier la dose qui élève l’expression du gène d’intérêt jusqu’au niveau souhaité et utiliser cette dose pour atteindre la surexpression du gène dans les expériences futures.
- Déterminer expérimentalement la concentration de Dox à administrer, traiter les souris du génotype approprié et contrôles avec l’un d’une gamme de Dox des concentrations (départ plage recommandée : 0 – 5 000 mg/kg de Doxycycline Hyclate) au moins une semaine52 ,53, y compris au moins trois souris par groupe de traitement. Acheter de Dox-contenant la nourriture de souris auprès d’un fournisseur commercial.
Résultats
La capacité de répression du système télécommande a été démontrée dans deux approches différentes à ce jour. Dans la première approche, les sites de liaison pour le répresseur lac ont été insérés au promoteur du gène Dnmt1 endogène. Dans la deuxième approche, qui est recommandée par le présent protocole, les sites de liaison du répresseur furent insérées dans un intron en aval pour éviter le risque d’affecter les fonctions de promoteur de l’insertion et donc de simplifier l’application de la télécommande système. Les deux approches a abouti à la répression réussie (Figure 2A, B et la Figure 3 a-C)10. Dnmt1 expression est réprimée à 15 % des niveaux non réglementées en utilisant l’approche axée sur le promoteur (Figure 2A). Cette répression serrée a été renversée de manière dose-dépendante, en traitant des souris avec des quantités variables d’IPTG (Figure 2A). La répression Dnmt1 observée a été validée au niveau protéique par immunomarquage (Figure 2B). Nous n’avons pas observé une différence notable dans l’expression Dnmt1 entre Dnmt1+ / + et souris Dnmt1LO/LO , confirmant que notre insertion d’opérateur lac n'avait pas perturbés promoteur normal fonction10. L’approche axée sur l’intron atteint plus de répression de 90 % des opérateurs situés plusieurs kilobases en aval du site d’initiation de la transcription en atténuant la transcription élongation (Figure 3A, B)10. Cette approche axée sur l’intron a été encore validée sur sept promoteurs robustes supplémentaires (Figure 3C). Répression invariablement serrée a été réalisée à partir de tous les promoteurs testés. Aucune corrélation entre les niveaux d’expression résiduelle et les points forts des promoteurs a été observée, ce qui suggère que la capacité de répression de notre système de répression axées sur l’intron est supérieure à l’activité transcriptionnelle de tous les promoteurs robustes, nous avons testé () Figure 3 ( C).
La capacité de stimulation in vivo de l’expression du système télécommande a aussi été démontrée sur le gène Dnmt1 . Nous avons introduit deux copies de l’opérateur de tet dans le promoteur Dnmt1 , ainsi que des séquences d’opérateur lac , afin de permettre pour une augmentation ou diminution de l’expression selon laquelle effecteur protéine est présente. Upregulation robuste et une diminution d’expression Dnmt1 , à proximité de deux ordres de grandeur (10 % à 650 %), ont été réalisés dans ces contenant l’allèle de Dnmt1 endogène modifié (Dnmt1LGT) (Figure 4A)10 . Les deux règlements ont été entièrement réversibles et inductible par traitements IPTG et Dox, respectivement (Figure 4A). Ensuite, nous avons introduit la modification Dnmt1LGT dans la lignée germinale de souris pour tester la capacité de stimulation in vivo de l’expression du système de contrôle à distance. Forte augmentation de Dnmt1 a été observée de la foie, la rate et le rein, alors qu’aucun upregulation détectable au coeur a été observée (Figure 4B)7. Le modèle d’expression cycle cellulaire dépendante de Dnmt1 et la rareté des cellules prolifératives au coeur pourraient expliquer cette observation10,56. Il reste à savoir si cette restriction peut être surmontée en augmentant le niveau d’expression de l’activateur ou le nombre de ses sites de liaison.
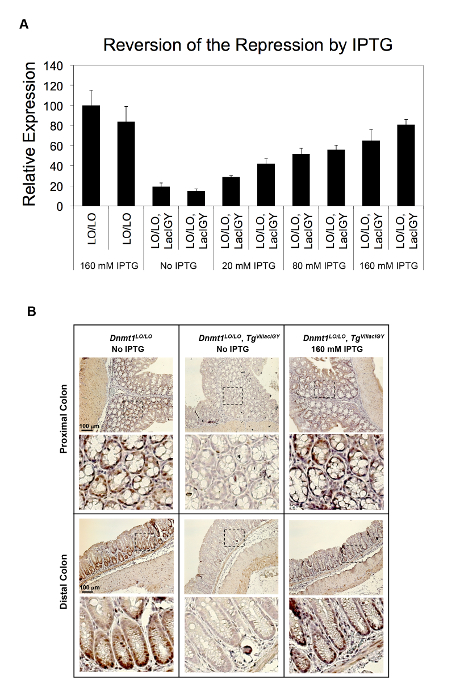
Figure 2 : In vivo répression de Dnmt1 par le répresseur LacIGY. Souris de la (A), avec bac opérateurs (LO), insérées dans le promoteur Dnmt1 , avec ou sans expression de LacIGY, ont été traitées avec des doses différentes d’IPTG. analyse qRT-PCR de Dnmt1 expression montre l’inversion dose-dépendante de la répression Dnmt1 in vivo par le traitement de l’IPTG. Chaque barre représente les données d’une autre souris. Les données représentent la moyenne ± SEM (n = 3). (B) Immunostaining des protéine Dnmt1 dans des cryptes coliques de souris fourni l’eau potable avec ou sans 160 mM IPTG pendant 3 semaines. Ce chiffre a été modifié par Lee et al.,10s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
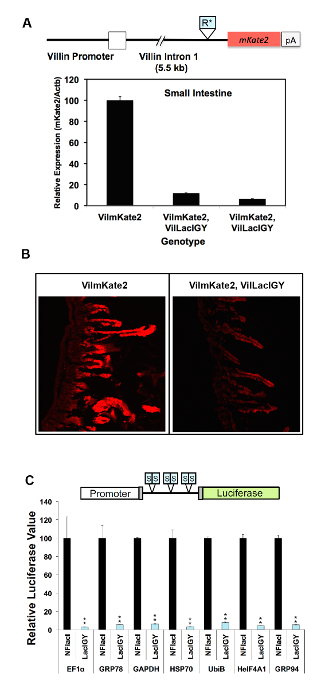
Figure 3 : In vivo et répression in vitro de différents promoteurs par le système de contrôle à distance. (A), une première version de la séquence Repron (R *) a été inséré dans un intron en aval du promoteur Villin dans une souris transgénique Villin-mKate2 (VilmKate2). qRT-PCR analyse d’expression de mKate2 dans l’intestin des souris avec ou sans le répresseur LacIGY est montré. Chaque barre représente les données d’une autre souris. (B) mKate2 confocale images de l’intestin grêle avec et sans expression de LacIGY . (C) Six opérateurs de lac symétrique (S) ont été insérés entre les divers promoteurs et un reporter de la luciférase. Reporters (50 ng par puits en plaque 96 puits) et plasmides répresseur transitoirement ont été introduits de NIH/3 t 3 cellules dans un rapport molaire de 1:1. Luciférase valeurs ont été mises en recouvrement 24h après la transfection. Ces données in vitro représentent le pourcentage d’expression de la luciférase dans LacIGY-exprimant des cellules par rapport à celles exprimant non-fonctionnel LacI (NFlacI). Tests T ont servi à déterminer la signification statistique. Les données représentent la moyenne ± SEM (n = 3). P ≤ 0,05, **P ≤ 0,01. Ce chiffre a été modifié par Lee et al.,10s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 4 : Down - et/ou upregulation de Dnmt1 expression in vitro et in vivo. (A) le système de contrôle à distance complet a été conçu dans ces cultivés par des approches de ciblage et électroporation de gène. Répression maximale d’expression Dnmt1 a été obtenue sans traitement alors que l’activation maximale a été obtenue par traitement des IPTG et Dox. Les données représentent la moyenne ± SEM (n = 3). P ≤ 0,05, **P ≤ 0,01 (Welch t-tests). (B) activation In vivo des Dnmt1 par le système de contrôle à distance, tel que démontré par immunomarquage de protéine Dnmt1 dans divers tissus de souris de contrôle à distance. L’allèle LGT représente modification promoteur de Dnmt1 pour contenir le lac opérateur et tet activateur accepteurs. Souris ont été traitées avec une alimentation normale ou Dox contenant (5 000 mg/kg Doxycycline Hyclate) pendant un mois. Ce chiffre a été modifié par Lee et al.,10s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
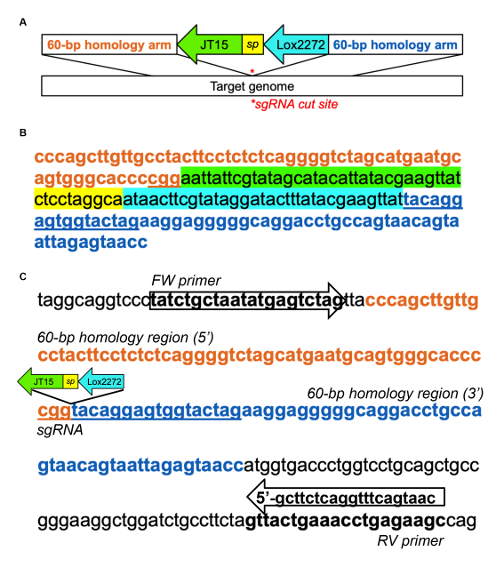
Supplémentaire Figure 1 : Exemple d’atterrissage touche insertion dans murin Dnmt1 intron 1. (A) représentation schématique du modèle de l’ADN pour atterrissage d’insertion de tampon, adaptée de Quadros et coll. (2015)31. Hétérotypiques loxP sites, JT15 et Lox2272, sont séparés par une séquence de l’espaceur court (sp) et flanqués de chaque côté de 60 pb d’ADN qui est homologue à la région génomique de la cible. (B) échantillon ADN modèle d’atterrissage touche insertion dans l’intron Dnmt1 utilisant le sgRNA suivant : CTAGTACCACTCCTGTACCG (qui s’adresse le brin arrière). La région intronic sélectionnée a été bioinformatically par étape 1.1, et le sgRNA a été identifié à l’aide de la CRISPOR29. (C) exemple de conception d’amorces PCR pour évaluer l’insertion de l’aire d’atterrissage. Amorces PCR ont été conçus à l’extérieur les bras de l’homologie du modèle pour confirmer l’intégration endogène Dnmt1. L’amplicon PCR de type sauvage est 213 bp ; lors de l’insertion, il devient 291 BP. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Une étape cruciale et limitation potentielle du système de contrôle à distance est le défi associé à l’insertion du répresseur et/ou sites de liaison activateur sans affecter l’expression des gènes cibles. Notre approche originale de la répression, telle qu’appliquée au gène Dnmt1 , impliqué d’insertion des sites de liaison du répresseur lac au sein des régions transcriptionnellement critiques d’un promoteur. Afin de réduire le risque d’affecter les fonctions de promoteur et ainsi améliorer l’applicabilité générale du système de contrôle à distance, nous avons développé une approche axée sur l’intron de répression. La puissance de notre système amélioré de lac nous a permis de réprimer fermement la transcription de tous les promoteurs fortes nous avons testé à opérateurs situés plusieurs centaines à plusieurs kilobases en aval de la transcription démarrer sites (Figure 3A à C) 10. ce qui est important, les niveaux de répression étaient indépendantes des forces transcriptionnelles des promoteurs (Figure 3A – C)10. Ceci suggère que la capacité de répression de notre système de répression axées sur l’intron dépasse la résistance transcriptionnelle des promoteurs testés. Dans cette approche axée sur les introns, il est probable que la répression est véhiculée par des interférences physiques entre deux composants, le mécanisme d’allongement de transcription et le lac répresseurs57. Ce mécanisme simple de répression et la solidité démontrée de la méthode axée sur les introns peuvent rendre cette approche généralement applicables aux organismes, des tissus et des gènes différents.
La régulation par le système de contrôle à distance nécessite les séquences de liaison transactivateur en proximité avec le promoteur du gène cible, qui entraîne un risque d’affecter les fonctions de promoteur. Cependant, nous avons constaté que la position des séquences de liaison peut être à l’extérieur de la région transcriptionnellement critique. Promoteurs fois Dnmt1 et EF1α ont été solidement sur-exprimés de tet opérateurs situé quelques centaines de bases en amont de la transcription de sites début10. Cette contrainte détendue réduit considérablement le risque d’affecter la fonction de promoteur d’un gène cible en l’absence de la transactivateur. Séquences d’augmenter le nombre de liaison et/ou de l’utilisation des activateurs trans plus forts pourrait aider à réduire davantage les risques en permettant l’augmentation des sites loin du site d’initiation de la transcription.
Notre système de contrôle à distance permet de contrôler élégant le niveau, moment et lieu de l’expression des gènes endogènes, permettant de tester la réversibilité d’un phénotype et les conséquences des niveaux d’expression différents, qui ne sont pas facilement réalisables par technologies actuelles de contrôle expression des gènes in vivo. Il est important de noter que dans la plupart des analyses expression génique, y compris la nôtre, les valeurs de l’expression représentent la moyenne d’une population de cellules parmi lesquels se trouvent des variations considérables. Cette hétérogénéité peut influencer les processus décisionnels cellulaires, tels que la différenciation ou apoptose58. Bien que probablement, la précision du contrôle d’expression de gène pourrait être encore améliorée par des circuits génétiques supplémentaires génie59, l’efficacité observée de notre système actuel permettra à enquête utile de la fonction des gènes dans de nombreux contextes biologiques. En outre, un haut degré de spécificité de cible est attendu en raison de la complexité des séquences opérateur ainsi que la grande distance évolutive entre les mammifères et les espèces originaires des éléments réglementaires60. En outre, lignées de souris transgéniques de répresseurs et activateurs peuvent être développées et employées pour n’importe quel gène endogène. Par exemple, les modèles souris transactivateur tet existants peuvent être adaptés pour accomplir la stimulation de l’expression d’un gène cible dans les tissus de souris désiré. Nous avons récemment développé une lignée transgénique qui permet de piloter robuste expression tissu-spécifique de notre répresseur lac améliorée dans plusieurs types de tissus lorsqu’il est combiné avec les lignes existantes de Cre en introduisant le gène lacIGY dans le locus Hipp11 48 sous le contrôle d’un élément de Lox-STOP-Lox (non publié). Cette ligne faciliterait considérablement l’application de tissu-spécifique du système de contrôle à distance.
Upregulation de gène par le système de contrôle à distance offre plusieurs avantages par rapport aux approches transgéniques inductibles actuelles. Il ne nécessite pas de génération de multiples lignées transgéniques pour tester les effets de position de l’insertion, car il utilise le locus endogène. En outre, cette approche est bien adaptée pour la stimulation de l’expression des gènes avec l’expression de base forte parce qu’elle favorise l’expression d’un promoteur déjà robuste, tandis que les modèles transgéniques conventionnels s’appuient sur des promoteurs viraux minimales. Enfin, le tissu spécificité, contrôle du cycle cellulaire et l’épissage de variantes d’un gène cible peuvent être conservés sur upregulation par notre approche, car il préserve des éléments de régulation naturelle comme innée cis-éléments de régulation. L’avènement de la technologie de ciblage de gène CRISPR/Cas-mediated facilitera grandement l’application de cette technologie dans des systèmes divers modèles.
Déclarations de divulgation
PWL sert des conseils consultatifs scientifiques de AnchorDx et de Progenity, Inc.
Remerciements
Nous remercions la fin Dr Heidi Scrable pour son généreux don de la construction de gène lacI mammifères (Mayo Clinic, Rochester, MN), Dr Daniel Louvard (Institut Curie, Paris, France) pour la fourniture du promoteur Villin et Dr Laurie Jackson-Grusby (hôpital pour enfants, Boston, MA) pour sa contribution aux premiers stades du développement de cette technologie. Nous sommes reconnaissants pour le Dr Nancy Wu et Dr Robert Maxson pour leur aide dans la création de souris transgéniques et knock-out. Nous remercions les membres du laboratoire Laird des discussions utiles et soutien. Ce travail a été soutenu par le National Institutes of Health [R01 CA75090 DA030325 R01, R01 CA157918 et R01 CA212374 à P.W.L. et 1F31CA213897-01 a 1 à N.A.V.S].
matériels
| Name | Company | Catalog Number | Comments |
| B6C3F1/J | The Jackson Laboratory | 100010 | https://www.jax.org/strain/100010 |
| Cas9 Protein | PNA Bio | CP04 | http://www.pnabio.com/products/CRISPR_Cas9.htm?gclid=EAIaIQobChMIsoG8pLL33QIVBr7ACh0naQ4dEAAYAiAAEgKyHvD_BwE |
| CRISPOR | Haeussler et al. 2016 | http://crispor.tefor.net/ | |
| Doxycycline-Containing Mouse Diet | Envigo | Varies by concentration | https://www.envigo.com/products-services/teklad/laboratory-animal-diets/custom-research/doxycycline-diets/ |
| ENCODE Database | Stanford University | https://www.encodeproject.org/ | |
| iCre mRNA synthesis plasmid (pBBI) | Addgene | 65795 | https://www.addgene.org/65795/ |
| IPTG | GoldBio | I2481C | https://www.goldbio.com/search?isSearch=Y&q=iptg |
| pGL3-Basic | Promega | E1751 | https://www.promega.com/products/reporter-assays-and-transfection/reporter-vectors-and-cell-lines/pgl3-luciferase-reporter-vectors/?catNum=E1751 |
| SVM-BPfinder | Regulatory Genomics, Pompeu Fabra University | http://regulatorygenomics.upf.edu/Software/SVM_BP/ | |
| TiProD: Tissue specific promoter Database | Department of Bioinformatics, UMG, University of Göttingen | http://tiprod.bioinf.med.uni-goettingen.de | |
| UCSC Genome Browser | University of California Santa Cruz | https://genome.ucsc.edu/ |
Références
- Jackson-Grusby, L., et al. Loss of genomic methylation causes p53-dependent apoptosis and epigenetic deregulation. Nature Genetics. 27 (1), 31-39 (2001).
- David, G., Turner, G. M., Yao, Y., Protopopov, A., DePinho, R. A. mSin3-associated protein, mSds3, is essential for pericentric heterochromatin formation and chromosome segregation in mammalian cells. Genes & Development. 17 (19), 2396-2405 (2003).
- Sumi-Ichinose, C., Ichinose, H., Metzger, D., Chambon, P. SNF2beta-BRG1 is essential for the viability of F9 murine embryonal carcinoma cells. Molecular and Cellular Biology. 17 (10), 5976-5986 (1997).
- Premsrirut, P. K., et al. A rapid and scalable system for studying gene function in mice using conditional RNA interference. Cell. 145 (1), 145-158 (2011).
- Qiu, S., Adema, C. M., Lane, T. A computational study of off-target effects of RNA interference. Nucleic Acids Research. 33 (6), 1834-1847 (2005).
- Gilbert, L. A., et al. Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell. 159 (3), 647-661 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2015).
- Peng, H., Ivanov, A. V., Oh, H. J., Lau, Y. F., Rauscher, F. J. Epigenetic gene silencing by the SRY protein is mediated by a KRAB-O protein that recruits the KAP1 co-repressor machinery. The Journal of Biological Chemistry. 284 (51), 35670-35680 (2009).
- Groner, A. C., et al. KRAB-zinc finger proteins and KAP1 can mediate long-range transcriptional repression through heterochromatin spreading. PLOS Genetics. 6 (3), 1000869 (2010).
- Lee, K. H., Oghamian, S., Park, J. A., Kang, L., Laird, P. W. The REMOTE-control system: a system for reversible and tunable control of endogenous gene expression in mice. Nucleic Acids Research. 45 (21), 12256-12269 (2017).
- Gossen, M., Bonin, A. L., Bujard, H. Control of gene activity in higher eukaryotic cells by prokaryotic regulatory elements. Trends in Biochemical Sciences. 18 (12), 471-475 (1993).
- Hu, M. C., Davidson, N. The inducible lac operator-repressor system is functional in mammalian cells. Cell. 48 (4), 555-566 (1987).
- Cronin, C. A., Gluba, W., Scrable, H. The lac operator-repressor system is functional in the mouse. Genes & Development. 15 (12), 1506-1517 (2001).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118 (2), 401-415 (1993).
- Council, N. R. . Guide for the Care and Use of Laboratory Animals: Eighth Edition. , (2011).
- Casper, J., et al. The UCSC Genome Browser database: 2018 update. Nucleic Acids Research. 46 (1), 762-769 (2018).
- Church, D. M., et al. Lineage-specific biology revealed by a finished genome assembly of the mouse. PLOS Biology. 7 (5), 1000112 (2009).
- Creyghton, M. P., et al. Histone H3K27ac separates active from poised enhancers and predicts developmental state. Proceedings of the National Academy of Sciences of the United States of America. 107 (50), 21931-21936 (2010).
- Heintzman, N. D., et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature. 459 (7243), 108-112 (2009).
- Heintzman, N. D., et al. Distinct and predictive chromatin signatures of transcriptional promoters and enhancers in the human genome. Nature Genetics. 39 (3), 311-318 (2007).
- Rada-Iglesias, A., et al. A unique chromatin signature uncovers early developmental enhancers in humans. Nature. 470 (7333), 279-283 (2011).
- Handoko, L., et al. CTCF-mediated functional chromatin interactome in pluripotent cells. Nature Genetics. 43 (7), 630-638 (2011).
- Splinter, E., et al. CTCF mediates long-range chromatin looping and local histone modification in the beta-globin locus. Genes & Development. 20 (17), 2349-2354 (2006).
- The ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Rosenbloom, K. R., et al. ENCODE data in the UCSC Genome Browser: year 5 update. Nucleic Acids Research. 41, 56-63 (2013).
- Murray, J. I., Voelker, R. B., Henscheid, K. L., Warf, M. B., Berglund, J. A. Identification of motifs that function in the splicing of non-canonical introns. Genome Biology. 9 (6), 97 (2008).
- Taggart, A. J., et al. Large-scale analysis of branchpoint usage across species and cell lines. Genome Research. 27 (4), 639-649 (2017).
- Corvelo, A., Hallegger, M., Smith, C. W., Eyras, E. Genome-wide association between branch point properties and alternative splicing. PLOS Computational Biology. 6 (11), 1001016 (2010).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Grainger, S., et al. CRISPR Guide RNA Validation In Vitro. Zebrafish. 14 (4), 383-386 (2017).
- Quadros, R. M., Harms, D. W., Ohtsuka, M., Gurumurthy, C. B. Insertion of sequences at the original provirus integration site of mouse ROSA26 locus using the CRISPR/Cas9 system. FEBS Open Bio. 5, 191-197 (2015).
- Harms, D. W., et al. Mouse Genome Editing Using the CRISPR/Cas System. Current Protocols in Human Genetics. 83, (2014).
- Miura, H., Quadros, R. M., Gurumurthy, C. B., Ohtsuka, M. Easi-CRISPR for creating knock-in and conditional knockout mouse models using long ssDNA donors. Nature Protocols. 13 (1), 195-215 (2018).
- Laird, P. W., et al. Simplified mammalian DNA isolation procedure. Nucleic Acids Research. 19 (15), 4293 (1991).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Akcakaya, P., et al. In vivo CRISPR editing with no detectable genome-wide off-target mutations. Nature. 561 (7723), 416-419 (2018).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nature Protocols. 13 (11), 2615-2642 (2018).
- Ohtsuka, M., et al. Improvement of pronuclear injection-based targeted transgenesis (PITT) by iCre mRNA-mediated site-specific recombination. Transgenic Research. 22 (4), 873-875 (2013).
- Ohtsuka, M., et al. Pronuclear injection-based mouse targeted transgenesis for reproducible and highly efficient transgene expression. Nucleic Acids Research. 38 (22), 198 (2010).
- Lee, G., Saito, I. Role of nucleotide sequences of loxP spacer region in Cre-mediated recombination. Gene. 216 (1), 55-65 (1998).
- Thomson, J. G., Rucker, E. B., Piedrahita, J. A. Mutational analysis of loxP sites for efficient Cre-mediated insertion into genomic DNA. Genesis. 36 (3), 162-167 (2003).
- Cho, A., Haruyama, N., Kulkarni, A. B. Generation of Transgenic Mice. Current Protocols in Cell Biology. , (2009).
- Pu, X. A., Young, A. P., Kubisch, H. M. Production of Transgenic Mice by Pronuclear Microinjection. Methods in Molecular Biology. 1874, 17-41 (2019).
- Murgha, Y., et al. Combined in vitro transcription and reverse transcription to amplify and label complex synthetic oligonucleotide probe libraries. Biotechniques. 58 (6), 301-307 (2015).
- Quadros, R. M., et al. Easi-CRISPR: a robust method for one-step generation of mice carrying conditional and insertion alleles using long ssDNA donors and CRISPR ribonucleoproteins. Genome Biology. 18 (1), 92 (2017).
- Chen, X., Wu, J. M., Hornischer, K., Kel, A., Wingender, E. TiProD: the Tissue-specific Promoter Database. Nucleic Acids Research. 34, 104-107 (2006).
- Haruyama, N., Cho, A., Kulkarni, A. B. Overview: Engineering transgenic constructs and mice. Current Protocols in Cell Biology. , (2009).
- Tasic, B., et al. Site-specific integrase-mediated transgenesis in mice via pronuclear injection. Proceedings of the National Academy of Sciences of the United States of America. 108 (19), 7902-7907 (2011).
- JoVE Science Education Database. Lab Animal Research. Fundamentals of Breeding and Weaning. Journal of Visualized Experiments. , (2018).
- Wyborski, D. L., DuCoeur, L. C., Short, J. M. Parameters affecting the use of the lac repressor system in eukaryotic cells and transgenic animals. Environmental and Molecular Mutagenesis. 28 (4), 447-458 (1996).
- Wyborski, D. L., Short, J. M. Analysis of inducers of the E.coli lac repressor system in mammalian cells and whole animals. Nucleic Acids Research. 19 (17), 4647-4653 (1991).
- Traykova-Brauch, M., et al. An efficient and versatile system for acute and chronic modulation of renal tubular function in transgenic mice. Nature Medicine. 14 (9), 979-984 (2008).
- Michel, G., Mosser, J., Fauran, F. Serum kinetics of doxycycline polyphosphate in dogs. European Journal of Drug Metabolism and Pharmacokinetics. 4 (1), 43-48 (1979).
- Bertocchi, I., et al. Regulatory functions of limbic Y1 receptors in body weight and anxiety uncovered by conditional knockout and maternal care. Proceedings of the National Academy of Sciences of the United States of America. 108 (48), 19395-19400 (2011).
- Plageman, T. F., Lang, R. A. Generation of an Rx-tTA: TetOp-Cre knock-in mouse line for doxycycline regulated Cre activity in the Rx expression domain. PLOS One. 7 (11), 50426 (2012).
- Mollova, M., et al. Cardiomyocyte proliferation contributes to heart growth in young humans. Proceedings of the National Academy of Sciences of the United States of America. 110 (4), 1446-1451 (2013).
- Ptashne, M. Principles of a switch. Nature Chemical Biology. 7 (8), 484-487 (2011).
- Balazsi, G., van Oudenaarden, A., Collins, J. J. Cellular decision making and biological noise: from microbes to mammals. Cell. 144 (6), 910-925 (2011).
- Nevozhay, D., Zal, T., Balazsi, G. Transferring a synthetic gene circuit from yeast to mammalian cells. Nature Communications. 4, 1451 (2013).
- Labow, M. A., Baim, S. B., Shenk, T., Levine, A. J. Conversion of the lac repressor into an allosterically regulated transcriptional activator for mammalian cells. Molecular and Cellular Biology. 10 (7), 3343-3356 (1990).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon