Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Generación y caracterización cuantitativa de quistes epiteliales biliares funcionales y polarizados
En este artículo
Resumen
Los sistemas celulares tridimensionales (3D) son modelos relevantes para investigar la organogénesis. Se propone un método basado en hidrogeles para la producción de quistes biliares y su caracterización. Este protocolo desentraña las barreras de la caracterización 3D, con un método sencillo y confiable para evaluar la eficiencia de la formación de quistes, los tamaños y para probar su funcionalidad.
Resumen
Los colangiocitos, las células epiteliales que alinean los conductos biliares en el hígado, supervisan la formación y modificación de la bilis. En los últimos veinte años, en el contexto de las enfermedades hepáticas, han surgido modelos tridimensionales (3D) basados en colangiocitos como quistes, esferoides o estructuras similares a tubos para imitar la topología tisular para la organogénesis, el modelado de enfermedades y los estudios de detección de fármacos. Estas estructuras se han obtenido principalmente mediante la incrustación de colangiocitos en un hidrogel. El propósito principal era estudiar la auto-organización abordando la polaridad epitelial, las propiedades funcionales y morfológicas. Sin embargo, muy pocos estudios se centran en la eficiencia de la formación de quistes. Cuando este es el caso, la eficiencia a menudo se cuantifica a partir de imágenes de un solo plano. Los ensayos funcionales y el análisis estructural se realizan sin representar la heterogeneidad potencial de la distribución de quistes derivada de las heterogeneidades y efectos secundarios de la polimerización de hidrogel. Por lo tanto, el análisis cuantitativo, cuando se hace, no se puede utilizar para la comparación de un artículo a otro. Además, esta metodología no permite comparaciones del potencial de crecimiento 3D de diferentes matrices y tipos de células. Además, no se menciona la solución experimental de problemas para la inmunosumanción de quistes. En este artículo, proporcionamos un método confiable y universal para mostrar que la distribución celular inicial está relacionada con la distribución vertical heterogénea de la formación de quistes. Las células de colangiocito incrustadas en hidrogel se siguen con análisis de pilas Z a lo largo de la profundidad del hidrogel durante el transcurso de 10 días. Con este método, se obtiene una cinética robusta de la eficiencia y el crecimiento de la formación de quistes. También presentamos métodos para evaluar la polaridad del quiste y la función secretora. Por último, se proporcionan consejos adicionales para optimizar los protocolos de inmunosumanidad con el fin de limitar el colapso del quiste para la toma de imágenes. Este enfoque se puede aplicar a otros estudios de cultivo celular 3D, abriendo así las posibilidades de comparar un sistema con otro.
Introducción
En las últimas tres décadas, el campo de la investigación in vitro ha avanzado hacia sistemas de cultivo 3D. Han surgido varios protocolos para el cultivo de células en 3D como esferoides o agregados en presencia o ausencia de un andamio/matriz, en una gota, en agitación, en dispositivos microfluídicos, o flotante1. El uso de métodos de cultivo 3D ha demostrado sus ventajas sobre cultivos tridimensionales (2D), particularmente para células epiteliales, que se demostró que se autoorganizaban en estructuras 3D, llamadas quistes o acini. En este caso, las células forman una monocapa que rodea un lumen, donde las células adquieren su fenotipo epitelial completo con funciones específicas fisiológicas mejoradas2.
Numerosos estudios han contribuido al desarrollo de métodos para la formación de estos organoides epiteliales en matrices naturales. Esto ha permitido recapitular las interacciones in vivo entre células celulares y microambientes, para conseguir el establecimiento y la estabilidad del fenotipo epitelial3,4,5,6,7. Recientemente, y en particular con el objetivo de desarrollar organoides trasplantables y descifrar el requisito de la microambiente para la orquestación del programa epitelial, se han desarrollado hidrogeles sintéticos para mejorar la formación de acini epitelial8,9,10. Desafortunadamente, estos estudios informan sobre datos cualitativos, o presentan métodos de cálculo utilizando referencias internas como la relación de quistes sobre no quistes en un plano 2D8,9,10. Esto impide cualquier comparación entre diferentes estudios en términos de eficiencia, estabilidad o caracterización morfológica y fisiológica de los organoides epiteliales.
La microencapsulación de células epiteliales en cuentas utilizando dispositivos microfluídicos ha permitido resultados cuantitativos y comparativos más realistas. Usando esta tecnología, se formaron organoides de varios tipos de células y se diferenciaron en función de la morfología entre las diferentes estructuras celulares 3D11,12. Sin embargo, esta tecnología no es fácil de trabajar y requiere el uso de salas limpias para producir los dispositivos microfluídicos. Esta tecnología se ha establecido para algunos tipos de hidrogeles pero requiere adaptación técnica para ser aplicado a otros hidrogeles, restringiendo su versatilidad. Por lo tanto, la mayoría de los estudios destinados a desarrollar organoides epiteliales dependen de la incrustación de células epiteliales en un hidrogel a granel. En estos métodos, la alta heterogeneidad de la estructuración de gel y la distribución celular dentro de todo el cultivo 3D a menudo se descuida. Por lo tanto, la mayoría de los análisis se refieren a imágenes 2D individuales, que representan sólo muy aproximadamente la distribución de los diversos objetos celulares en todo el volumen 3D.
Las enfermedades que afectan los conductos biliares, como el colangiocarcinoma, la atresia biliar, la colangitis esclerosante primaria, entre otras, son una de las principales causas de mortalidad y morbilidad. A excepción del trasplante de hígado, no existen tratamientos eficaces para estas afecciones13. Los esfuerzos para investigar la formación de las vías biliares, las causas de la enfermedad y la progresión permitirán el desarrollo de nuevas terapias14.
Se han desarrollado modelos organotípicos biliares de quistes, esferoides o estructuras similares a tubos que utilizan líneas celulares de colangio derivadas de pacientes o normales, diferenciadas o derivadas de progenitores15,16,17,18,19,20. Diversos estudios han recapitulado la polaridad del colangiocito, la expresión de marcadores de colangiocitos, la presencia de cilios, la capacidad secretora y reabsortiva de colangiocitos, y la formación y obstrucción de lúmenes; todas las cuales representan características importantes del fenotipo de colangiocito, morfología y función15,17,19. Otros han reportado mantenimiento de organoides biliares derivados del paciente durante largos períodos de tiempo20. Recientemente, investigamos el papel de las señales bioquímicas y biofísicas en los quistes biliares organogénesis21. Es importante destacar que la patogénesis de la atresia biliar se estudió en esferoides biliares y tubos7,22. Además, las características clave de la colangitis esclerosante primaria como la senescencia del colangiocito, la secreción de citoquinas proinflamatorias, así como el reclutamiento de macrófagos se estudiaron con éxito utilizando esferoides biliares15,20. Sin embargo, todavía se necesitan modelos cuantitativos 3D in vitro reproducibles que modulan fisiológicamente el fenotipo de colangiocito, la fisiología y el microambiente donde se pueden abordar estas cuestiones. Además, sólo unas pocas publicaciones han notificado la eficacia de la formación de quistes21,23. Este es un punto importante a establecer, particularmente al investigar la organogénesis, la causa de la enfermedad y la correlación de las respuestas de los fármacos con la función del colangiocito y la polarización. Además, con diferencias en el andamio/matriz utilizados de protocolo a protocolo, es difícil comparar entre sistemas. Para resolver estos problemas, proponemos un método cuantitativo, confiable y universal para generar quistes biliares que imitan la formación de lúmenes, la polarización de colangiocitos y la función secretora de colangiocito. Es importante destacar que presentamos un análisis sistemático realizado a lo largo del eje Z a través del gel 3D al evaluar con el tiempo, la eficiencia de la formación de quistes, el tamaño, la viabilidad, la polarización y la funcionalidad. Además, utilizamos un hidrogel natural y colangiocitos de ratas normales (NRC), como ejemplo para el protocolo, pero otros hidrogeles naturales o sintéticos, así como células epiteliales podrían utilizarse para la formación de estructuras quísticas 3D.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Generación de quistes
NOTA: Este protocolo se puede realizar con cualquier tipo de hidrogel, si la gelera permite la incrustación de células.
- Recubrimiento de hidrogel
NOTA: El recubrimiento adecuado del hidrogel de la corredera de la cámara es un paso crítico para evitar la formación de capas celulares 2D en la parte inferior del pozo, que podrían interferir con la toma de imágenes de quistes posteriores y afectar el cálculo de la eficiencia de la formación de quistes.- Para garantizar la homogeneidad de la solución de gel, descongele el hidrogel a 4oC durante la noche (O/N).
- Puntas de pipeta precool sobre hielo o O/N a -20 oC y un tobogán de cámara de 8 pozos a -20 oC O/N.
- Coloque el hidrogel y la cámara de 8 pozos deslizarse sobre un cubo de hielo lleno de hielo.
- En un tubo cónico de 15 ml, prepare una solución que contenga un 40% de hidrogel (V/V) en un medio completo NRC frío (ver Tabla de materiales)y coloque el tubo sobre hielo.
- Para cubrir un portaobjetos de cámara, utilizando puntas de pipeta fría, agregue 50 l de solución de hidrogel en el centro de cada pozo, y extienda sobre toda la superficie usando una punta de pipeta, mientras sostiene el portaobjetos de la cámara sobre hielo (Figura 1A).
NOTA: Extienda la solución de hidrogel de la forma más uniforme posible evitando burbujas. - Para polimerizar el hidrogel, incubar el portaobjetos de la cámara durante al menos 15 min a 37oC, 5% CO2.
- Preparación celular
- Calentar el medio completo NRC, la solución salina tamponada con fosfato (PBS) y el ácido tetraacético de trippsina-etilendiamina (tripsina-EDTA) en un baño de agua precalentado a 37 oC.
- Mientras que el hidrogel está polimerizando, asegúrese de que los NRC se cultivan a 70% de confluencia en un matraz recubierto de colágenoT-25 cm 221. Lave las células una vez con 1 PBS precalentado.
- Incubar los NRC con 5 ml de 1x PBS precalentado (para un matraz T-25 cm2) durante 20 min a 37oC, 5% CO2.
NOTA: Este paso, que acorta el tiempo de incubación con trypsin-EDTA es fundamental para conservar las propiedades autoorganizantes de las células. - Desechar el PBS, añadir 1 mL de trippsina-EDTA, e incubar durante 5-10 min a 37 oC, 5% CO2.
- Neutralizar con 4 ml de medio NRC completo precalentado. Recoger y transferir la suspensión celular en un tubo cónico de 15 ml, y girar a 150 x g durante 4 min.
- Deseche el medio y resuspender el pellet celular en 5 ml de medio precalentado.
- Usando un colador celular de 40 m, filtre la solución celular en un tubo cónico de 50 ml y cuente las células.
NOTA: Pasar las células a través de un colador es un paso crítico para que los resultados cuantitativos sean reproducibles, es decir, para obtener un tamaño casi similar de agregados de celda que se va a incrustar.
- Incorporación de la suspensión celular en solución de hidrogel
- Preparar una solución de 1.600 l de hidrogel al 80% (V/V) en medio NRC completo en frío (tubo 1); mantener en el hielo. Diluir 5 x 105 células/ml en 1.600 ml de medio NRC completo en frío (tubo 2) y mantener en hielo.
NOTA: Este paso debe realizarse rápidamente para evitar la polimerización del hidrogel mientras se mezcla con la suspensión celular y para mantener la viabilidad celular. - Para preparar una solución de siembra celular de 2,5 x 105 células/ml en hidrogel al 40% (V/V), mezcle el tubo 1 y el tubo 2. Añadir 400 l de la solución celular en cada pocóter de la corredera de la cámara recubierta de hidrogel evitando burbujas (Figura 1B).
- Mantenga el tobogán de la cámara en una incubadora a 37oC con un 5% de CO2 hasta que cambie el medio.
- Después de 2 días en cultivo, retire 250 l del medio de una esquina de cada pozo, tenga cuidado de no pipetear el hidrogel. A continuación, añada lentamente 250 l del medio de cultivo. Cambie el medio cada 2 días.
NOTA: Minimice el movimiento del portaobjetos de la cámara, especialmente durante la iniciación del quiste.
- Preparar una solución de 1.600 l de hidrogel al 80% (V/V) en medio NRC completo en frío (tubo 1); mantener en el hielo. Diluir 5 x 105 células/ml en 1.600 ml de medio NRC completo en frío (tubo 2) y mantener en hielo.
2. Cuantificación de quistes
- Imágenes de quistes
NOTA: Esta sección debe realizarse rápidamente para no comprometer la viabilidad celular si el microscopio no está equipado con una cámara de calentamiento para controlar elCO2 y la temperatura. Con el fin de garantizar una cuantificación coherente, representativa de la distribución del quiste en todo el volumen de hidrogel, los quistes se visualizan mediante microscopía de contraste de fase e imágenes en serie (pilas Z), con parámetros predefinidos a lo largo de diferentes puntos de tiempo.- Tome una pila Z a lo largo de la profundidad del hidrogel para cada punto de tiempo(Figura 1C, D). En este ejemplo, las pilas Z se toman en los días 1, 2, 4, 7 y 10.
NOTA: Compruebe que la distribución celular inicial es uniforme en el hidrogel para garantizar la aplicabilidad de este método.- Con un microscopio de contraste de fase equipado con un software de adquisición de imágenes, seleccione el aumento objetivo 10x en la ventana manual de la almohadilla de la boquilla(Figura 2B(1)).
- Encienda la lámpara blanca y seleccione la opción de imagen de campo brillante.
- Encienda la cámara seleccionando el botón"Reproducir"en el submenú de la barra. Concéntrese en un campo de quistes y establezca el tiempo de exposición (Figura 2B(2)). Abra la ventana Carpeta de captura automática para guardar automáticamente las imágenes(Figura 2B(3)).
- Abra la ventana de la serie Z de captura y defina con el tornillo Z los planos superior e inferior de la pila Z (las mismas coordenadas XY pero diferentes de pantalla Z). Ajuste el paso Z dependiendo del objetivo, el nivel de resolución y pulse el botón"Ejecutar ahora"para iniciar la adquisición(Figura 2B(4)).
NOTA: En este ejemplo, los quistes se distribuyen sobre un espesor de hidrogel de 520 m. 26 imágenes se adquieren a lo largo de la profundidad del hidrogel con un intervalo de paso Z de 20 m. Dependiendo del objetivo, el paso Z debe ajustarse para no perder ningún quiste y para asegurar la detección de células y agregados individuales. - Tome al menos 3 pilas Z no superpuestas por pozo.
NOTA: Este muestreo es necesario cuando, como en este ejemplo, los quistes son más numerosos en la profundidad del gel que en los bordes debido a las heterogeneidades en la polimerización de hidrogel. - Para tener un conjunto de datos representativo repita el paso 2.1.1.5. para 3 pozos en total.
NOTA: La distribución heterogénea de los quistes depende del tipo de hidrogel, su polimerización y la línea celular. Teniendo en cuenta tres pilas Z por pozo y tres pozos por experimento, se obtiene una imagen de un mínimo de 200 quistes sobre nueve pilas Z para caracterizar la formación de quistes y el crecimiento del quiste en cada momento.
- Tome una pila Z a lo largo de la profundidad del hidrogel para cada punto de tiempo(Figura 1C, D). En este ejemplo, las pilas Z se toman en los días 1, 2, 4, 7 y 10.
- Procesamiento de imágenes
NOTA: En un hidrogel, los NRC se pueden encontrar como células individuales, quistes o agregados. Los quistes se identifican por la presencia de una cáscara celular redonda y delgada en contraste que encierra un lumen, mientras que los agregados celulares presentan una forma irregular y no tienen un lumen. Los agregados y las células individuales tienen una apariencia densa y contrastada(Figura 3B(4)).- Abra el software de Fiji, abra la pila Z y vaya al menú de Fiji y haga clic en Archivo , Abrir (Figuracomplementaria 1). Seleccione la pila Z que desea analizar. Si es necesario, seleccione la opción "Virtual Stack" y haga clic en "Yes" para abrir (Figura 3A(1)).
- Duplicar la pila a través de La imagen Duplicado. Haga clic en el cuadro "Pila duplicada" y haga clic en "OK" ( Figuracomplementaria 2).
NOTA: En este ejemplo, las pilas Z están en formato de archivo .nd2 codificado en 16 bits. - Cree una proyección de intensidad mínima a partir de la pila duplicada. Ir al menú Imagen ? Pilas ? Proyecto Z. Seleccione el tipo de proyección "Intensidad mínima" y haga clic en "OK" (Figura 3A(2)) (Figura complementaria 3).
- Restar el fondo de la proyección. Ir al menú Proceso . Restar fondo. Escriba 500,0 píxeles de radio de bola rodante y haga clic en "fondo de luz"para representar los quistes más contrastados que el fondo (Figura 3A(3)) (Figura complementaria 4).
NOTA: El radio de la bola rodante define el tamaño de la región en la que se opera la resta de fondo. Este parámetro debe establecerse en el tamaño del objeto más grande que se va a identificar. - Si es necesario mejorar el contraste, vaya al menú Imagen . Ajustar ? Brillo/Contraste ? Automático ? Aplicar. Fiji optimizará automáticamente el brillo y el contraste. En (Figura 3A(3)), los valores de gris inferior y superior se establecieron en 49702 y 65452, respectivamente (Figura complementaria 5).
NOTA: Si la proyección no está calibrada, vaya al menú Analizar . Establezca la escala y escriba la relación de calibración correspondiente(Figura complementaria 6).
- Mediciones de recuento de quistes y tamaño de quiste
- Para medir el diámetro aproximado del quiste, seleccione la herramienta Línea recta en el menú Fiji y dibuje una línea a través del diámetro de cada quiste en la proyección final (Figura 3B(4)). Agregue la nueva región de interés (ROI) creada para cada quiste al gestor de ROI: presione el atajo "t" en el teclado para un conteo y apertura más rápidos del administrador de ROI. Haga clic en "Mostrar todo" para ver los quistes contados(Figura complementaria 7)
- Compruebe que no se ha dejado ningún quiste sin contar superponiendo los ROI establecidos de la proyección en la pila Z. Para ello, haga clic en la ventana Z-stack para seleccionarla. En el Administrador de ROI, haga clic en"Mostrar todo"y mueva el cursor a lo largo de la pila Z para comprobar que la imagen por imagen, se han contado todos los quistes(Figura complementaria 8).
- Una vez que se hayan contado nuevos quistes y añadido ROI en el paso 2.3.1., seleccione el conjunto de ROI y guárdelo a través de la ventana administrador de ROI haciendo clic en Más . Guardar (Figura complementaria 9).
- Seleccione todos los ROI en el Administrador de ROI y haga clic en"Medir"en el Administrador de ROI para obtener el tamaño de cada quiste. Esto abrirá una nueva ventana de medidas denominada"Resultados"numerando cada quiste y su tamaño estimado. A continuación, guarde en formato .csv haciendo clic en la ventana "Resultados" y a través del menú: Archivo ? Guardar como (figura complementaria 10).
NOTA: Se puede crear una macro para procesar pilas semiautomáticamente, estimar el número/tamaños de quiste de las proyecciones y almacenar los datos para un procedimiento de conteo más rápido. Para ello, seleccione la herramienta"Grabar"en el menú de la barra, haciendo clic en Plugins . Macros ? Grabar.
- Cuantificación de la eficiencia de la formación de quistes
- Cuente el número de quistes en el día Y,
 en una proyección (Y-1, 2, 4, 7 o 10).
en una proyección (Y-1, 2, 4, 7 o 10). - Para calcular la eficiencia de formación de quistes para 1000 células al día Y, divida el número de quistes contados en ese momento por el número de células sembradas en el día 0 inferidas del volumen de hidrogel y multiplique por 1000(Figura 3C, Figura 4).
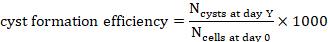
- Cuente el número de quistes en el día Y,
3. Viabilidad celular
- Preparar una solución en stock de diacetato de fluoresceína (FDA) a 5 mg/ml disolviendo 5 mg de FDA en acetona de 1 ml y almacenar a -20 oC.
- Preparar una solución en stock de yoduro de propídico (PI) a una concentración de 2 mg/ml en agua desionizada (dH2O) y conservar a 4oC.
- Preparar el medio NRC sin suero de ternera fetal (FCS).
- Para preparar la solución de tinción FDA/PI, añada 4 ml de solución de stock de la FDA (concentración final de 8 g/ml) y 25 ml de solución de PI en stock (concentración final de 20 g/ml) en 2,5 ml de medio NRC sin FCS.
- Retire el medio del portaobjetos de la cámara, añada 250 l de solución de tinción en cada pocóyo e incubar 4-5 min en la oscuridad a 37 oC, 5% CO2. Pipetear la solución de tinción cuidadosamente y lavar una vez con 250 l de 1x PBS.
- Agregue cuidadosamente 250 l de medio NRC completo a cada poc y tome fotografías usando un microscopio de fluorescencia invertido con filtros de isotiocianato de fluoresceína y rojo de Texas (FITC). Las celdas vivas serán verdes y las celdas muertas serán rojas(Figura 5A).
NOTA: Para cuantificar las células vivas/muertas, tome pilas Z a través del volumen de hidrogel siguiendo el paso 2 y adapte el método de procesamiento de imágenes para la fluorescencia.
4. Actividad de secreción
NOTA: La actividad de secreción a través de la membrana apical de los colangiocitos se evalúa mediante la secreción de fluoresceína en el lumen. Su especificidad se puede evaluar haciendo la misma prueba con Verapamil, un inhibidor de transporte multirresistente (MDR)24.
- Para preparar una solución de tinción de Hoechst 33258 a 5 g/ml, añada 0,83 l de solución en stock de Hoechst (concentración de 15 mg/ml en dH2O) en 2,5 ml del medio NRC sin FCS.
- Añadir 250 l de solución de Hoechst en cada pocóctil e incubar a 37oC, 5% deCO2 durante 15 min.
- Retire la solución de Hoechst y añada 250 ml de solución de la FDA (concentración final de 8 g/ml) en cada pocópido. Incubar 4-5 min a 37oC, 5% CO2.
NOTA: Tan pronto como las células se exponen a la solución de tinción de la FDA, el seguimiento de la cinética de secreción de fluoresceína podría ser útil para calibrar el tiempo necesario para que los quistes se secretan. Para ello, tome fotografías cada minuto durante 1 hora a través de imágenes de lapso de tiempo. En este ejemplo, el tiempo necesario para observar los quistes secretos NRC en el hidrogel es de aproximadamente 15-20 min. - Tome imágenes con un microscopio de fluorescencia invertido 5 minutos después de enjuagar con un medio sin FCS. Utilice filtros de 4o,6-diamidino-2-phenylindole (DAPI) y FITC para revelar el etiquetado de núcleos y la acumulación de fluoresceína en el lumen (Figura 6A). Para cuantificar el número de quistes secretos, tome pilas Z como en el paso 2 y adapte los pasos de procesamiento de imágenes a imágenes fluorescentes.
NOTA: Para la prueba de Verapamil, preceda al proceso anterior (pasos 4.3. a 4.4.) por una incubación con Verapamil, de acuerdo con las siguientes condiciones: - Preparar una solución de 10 mM de Verapamil en dimetil sulfóxido (DMSO). Para preparar una solución de trabajo de 10 m, mezcle 2,5 l de solución de stock de Verapamil con un medio de cultivo de 2,5 ml sin FCS.
- Para evaluar que la fluorescencia en el lumen resulta de la secreción de MDR, tome otra diapositiva y agregue 250 l de solución de trabajo de Verapamil en cada poc y incubar 20 min a 37oC, 5% CO2
- Retire la solución y agregue 250 ml de solución de la FDA (concentración final de 8 g/ml) en cada pocól. Incubar 4-5 min en la oscuridad a 37oC, 5% CO2. A continuación, lave con 250 l de 1x PBS, antes de la toma de imágenes(Figura 6B, C).
5. Evaluación de la polaridad epitelial por inmunofluorescencia
- Para preparar la solución de fijación, mezclar 4% de formaldehído con 5% de sacarosa, en 1x PBS, pH 7.4 e incubar en un baño de agua precalentado a 37oC.
- Para fijar las células, pipetee suavemente el medio de cultivo del pozo sin dañar la matriz. Añadir lentamente 400 l de la solución de fijación a un lado de los pocós. Incubar durante 20 min a temperatura ambiente (RT).
NOTA: Deje siempre 25 l del líquido por encima de la matriz para evitar su daño. - Retire suavemente la solución de fijación y lave 3x con 400 l de 1x PBS a (RT).
- Pipetear el PBS, añadir 400 l de solución de permeabilización (0,5% Tritón X-100 en 1x PBS) e incubar 10 min en RT.
- Retirar suavemente la solución de permeabilización, seguido de 3 lavados rápidos con 400 l de 1x PBS y un largo paso de lavado de 30 min en RT.
NOTA: En este paso, la diapositiva se puede almacenar a 4 oC durante 2 días. En este caso, selle la corredera con una película de parafina para evitar la evaporación y el secado de la matriz. - Retirar el PBS, añadir 400 l de solución de bloqueo que contenga albúmina sérica bovina al 0,1% (BSA) y 10% de suero de cabra en 1x PBS e incubar durante 60 minutos en RT.
ADVERTENCIA: Las concentraciones de BSA superiores al 0,1% darán lugar a una retracción de lúmenes y un colapso adicional del quiste (ver sección Resultados representativos) (Figura 7A). - Pipetear la solución de bloqueo y lavar una vez con 400 ml de PBS/0.05% Tween 20 y desechar.
- Añadir 150 l de la solución de anticuerpos, p. ej., anticuerpo de e-cadherina diluido 1:400 y Phalloidin 568 (concentración final de 16,2 nM) en 1x PBS e incubar durante 90 min a RT.
NOTA: Esta dilución de E-cadherina es la misma utilizada que en un protocolo de inmunofluorescencia 2D estándar. - Lavar la muestra con 400 s de PBS/0,05% Tween 20, 3x; cada vez incubando la muestra durante 10 minutos en RT.
- Añadir 150 l del anticuerpo secundario (cabra anti-conejo IgG Alexa Fluor Plus 647), diluido 1:500 en 1x PBS e incubar 60 min en RT.
- Lavar 3x con 400 ml de PBS/0.05% Tween 20, cada vez incubando la muestra durante 10 min a RT.
- Lavar 3x con 400 l de 1x PBS, cada vez incubando la muestra durante 10 min a RT.
- Deseche el PBS del último lavado y prepare el portaobjetos de la cámara para su visualización mediante microscopía confocal siguiendo una de las dos opciones siguientes.
- Añadir 400 l de 1x PBS y 50 l de DAPI por pozo. Las muestras se pueden examinar a través de la parte inferior del pozo sin necesidad de montar con un cubreobjetos(Figura 7B).
- Añadir 100 l por pociza de reactivo antifada que contenga DAPI y dejar que la diapositiva seque O/N en RT.
Access restricted. Please log in or start a trial to view this content.
Resultados
Formación y caracterización de quistes
Los sistemas de cultivo celular 3D son una herramienta importante para estudiar la organogénesis y el modelado de enfermedades25. Desafortunadamente, la mayoría de estos métodos son cualitativos o utilizan la cuantificación interna realizada en un solo plano comparando el número de quistes frente a los no quistes, en volúmenes variables y a menudo no especificados, evitando cualquier comparación en términos de eficiencia de forma...
Access restricted. Please log in or start a trial to view this content.
Discusión
Con el fin de estudiar la organogénesis y el mantenimiento de estructuras celulares 3D, se han modelado diversos tejidos, utilizando diferentes orígenes celulares, pero también diferentes tipos de matrices extracelulares incluyendo hidrogeles sintéticos8,9,10,21. Sin embargo, debido a la falta de análisis cuantitativo 3D que permite comparaciones entre métodos en términos de formación d...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos al Dr. Nicholas LaRusso (Clínica Mayo, Rochester, Minnesota, Estados Unidos), quien amablemente proporcionó la línea celular NRC.
Este trabajo recibió el apoyo financiero tanto del programa iLite RHU (concesión ANR-16-RHUS-0005) como del DHU Hepatinov.
Agradecemos a Isabelle Garcin y Réseau d'Imagerie Cellulaire Paris Saclay por su apoyo a la imagen.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 10 µl- Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000020 | |
| 100 µl - Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000046 | |
| 1000 µl - Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000062 | |
| 1X PBS | Thermo Fisher Scientific | 14190-094 | |
| 200 µl - Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000054 | |
| 3,3′,5-Triiodo-L-thyronine sodium salt | Sigma-Aldrich | T5516 | NRC complete medium final concentration = 3.4 µg/mL |
| Acetic acid | VWR | 20104-298 | 0.02N final |
| Aerosol barrier pipettes tips 10 µl (Fisherbrand) | Thermo Fisher Scientific | 2707439 | |
| Aerosol barrier pipettes tips 1000 µl (Fisherbrand) | Thermo Fisher Scientific | 2707404 | |
| Aerosol barrier pipettes tips 200 µl (Fisherbrand) | Thermo Fisher Scientific | 2707430 | |
| Antibiotic Antimicotic Solution (100X) | Sigma-Aldrich | A5955 | NRC complete medium final concentration = 1:100 dilution |
| Bovine pituitary extract | Thermo Fisher Scientific | 13028-014 | NRC complete medium final concentration = 30 µg/mL |
| Bovine serum albumin | Sigma-Aldrich | A2153 | 1:1000 dilution |
| Chemically Defined Lipid Concentrate (100X) | Thermo Fisher Scientific | 11905-031 | NRC complete medium final concentration = 1:100 dilution |
| Collagen high concentration, rat tail | Thermo Fisher Scientific | 354249 | 50 µg/mL final concentration |
| Dexamethasone | Sigma-Aldrich | D4902 | NRC complete medium final concentration = 0.393 µg/mL |
| DMEM F12 | Thermo Fisher Scientific | 21331-020 | NRC complete medium final concentration = 1X |
| E-cadherin Rabbit anti-Human, Rat, Polyclonal | Thermo Fisher Scientific | PA5-32178 | 1:400 dilution |
| Eclipse TE300 inverted microscope | Nikon | imaging | |
| Ethanolamine | Sigma-Aldrich | E9508 | NRC complete medium final concentration = 0.32 mM |
| Fetal calf serum | Thermo Fisher Scientific | 10270-106 | NRC complete medium final concentration = 5:100 dilution |
| Fluoroshield with DAPI (Mounting medium) | Sigma-Aldrich | F6057 | |
| Formaldehyde 16% (W/V) | Thermo Fisher Scientific | 28906 | 4% (W/V) |
| Goat serum | Thermo Fisher Scientific | 16210-064 | 1:10 dilution |
| Hamamatsu camera (Digital camera C11440 ORCA - flash 4.OLT) | Hamamatsu | imaging | |
| Hoechst 33258 | Sigma-Aldrich | B1155 | 5 µg/mL final concentration |
| IgG (H+L) Highly Cross-Adsorbed Goat anti-Rabbit, Alexa Fluor Plus 647 | Thermo Fisher Scientific | A32733 | 1:500 dilution |
| ImageJ version 2.0.0-rc-69/1.52n | Open source image processing software | ||
| Insulin-Transferrin-Selenium (100X) | Thermo Fisher Scientific | 51300-044 | NRC complete medium final concentration = 1:100 dilution |
| L-Glutamine (100X) | Thermo Fisher Scientific | 25030-024 | NRC complete medium final concentration = 1:100 dilution |
| Matrigel GFR (stock concentration 9.7 mg/mL) | Thermo Fisher Scientific | 356231 | 4:10 dilution |
| NIS Elements software version 4.50.00 | Nikon | image acquisition and display | |
| Non-Essential-Amino-Acids-Solution (100X) | Thermo Fisher Scientific | 11140-035 | NRC complete medium final concentration = 1:100 dilution |
| Objective Plan Fluor 10X/0.30 Ph1 DL (∞/1.2 WD 15.2) | Nikon | ||
| Prolong Gold Antifade Reagent | Thermo Fisher Scientific | P36931 | |
| Propidium Iodide (PI) | Sigma-Aldrich | P4170 | 20 µg/mL final concentration |
| Rhodamine Phalloidin | Thermo Fisher Scientific | R415 | 16.2 nM final concentration |
| Sir-Actin / Verapamil kit | Spirochrome | SC001 | 10 µM final concentration |
| Soybean trypsin inhibitor | Thermo Fisher Scientific | 17075-029 | NRC complete medium final concentration = 50 µg/mL |
| Sterile cell strainer 40 µm (Fisherbrand) | Thermo Fisher Scientific | 22363547 | |
| Sterile pipettes 10 mL (Fisherbrand) | Thermo Fisher Scientific | 1367811E | |
| Sterile pipettes 5 mL (Fisherbrand) | Thermo Fisher Scientific | 1367811D | |
| Sterile tubes 1.5 mL (Fisherbrand) | Thermo Fisher Scientific | 11926955 | |
| Sterile tubes 15 mL (Fisherbrand) | Thermo Fisher Scientific | 7200886 | |
| Sterile tubes 50 mL (Fisherbrand) | Thermo Fisher Scientific | 553913 | |
| Sucrose | Sigma-Aldrich | S0389 | 5:100 dilution |
| Tissue culture treated flask 25cm2 (Falcon) | Thermo Fisher Scientific | 353108 | |
| Triton X-100 | Sigma-Aldrich | T8787 | 5:1000 dilution |
| Trypsin-EDTA (0.05%) phenol red | Thermo Fisher Scientific | 25300-054 | 1X |
| Tween-20 | Sigma-Aldrich | P1379 | 5:10000 dilution |
| Vitamin (100X) | Thermo Fisher Scientific | 11120-037 | NRC complete medium final concentration = 1:100 dilution |
| μ-Slide 8 Well ibiTreat, Ibidi | Clinisciences | 80826 |
Referencias
- Edmondson, R., Broglie, J. J., Adcock, F., Yang, L. Three-Dimensional Cell Culture Systems and Their Applications in Drug Discovery and Cell-Based Biosensors. ASSAY and Drug Development Technologies. 12 (4), 207-218 (2014).
- Martín-Belmonte, F., et al. Cell-polarity dynamics controls the mechanism of lumen formation in epithelial morphogenesis. Current Biology. 18, 507-513 (2008).
- Debnath, J., Muthuswamy, S. K., Brugge, J. Morphogenesis and oncogenesis of MCF-10A mammary epithelial acini grown in three-dimensional basement membrane cultures. Methods. 30 (3), 256-268 (2003).
- Artym, V. V., Matsumoto, K. Imaging Cells in Three-Dimensional Collagen Matrix. Current Procotols in Cell Biology. , Chapter 10 (Unit 10) (2010).
- Petersen, O. W., Ronnov-Jessen, L., Howlett, A. R., Bisell, M. J. Interaction with basement membrane serves to rapidly distinguish growth and differentiation pattern of normal and malignant human breast epithelial cells. Proceedings of the National Academy of Sciences of the United States of America. 89, 9064-9068 (1992).
- Kim, S. P., Lee, D. H., Park, J. K. Development of hepatocyte spheroids immobilization technique using alternative encapsulation method. Biotechnology and Bioprocess Engineering. 3, 96-102 (1998).
- Lorent, K., et al. Identification of a plant isoflavonoid that causes biliary atresia. Science Translational Medicine. 7 (286), 67(2015).
- Nowak, M., Freudenberga, U., Tsurkana, M. V., Wernera, C., Levental, K. R. Modular GAG-matrices to promote mammary epithelial morphogenesis in vitro. Biomaterials. 112, 20-30 (2017).
- Miroshnikova, Y. A., et al. Engineering Strategies to Recapitulate Epithelial Morphogenesis Within Synthetic Three-Dimensional Extracellular Matrix With Tunable Mechanical Properties. Physical Biology. 8 (2), 026013(2011).
- Ozdemir, T., et al. Tuning Hydrogel Properties to Promote the Assembly of Salivary Gland Spheroids in 3D. ACS Biomaterials Science & Engineering. 2 (12), 2217-2230 (2016).
- Dolega, M. E., Abeille, F., Picollet-D'hahan, N., Gidrol, X. Controlled 3D culture in Matrigel microbeads to analyze clonal acinar development. Biomaterials. 52, 347-357 (2015).
- Laperrousaz, B., et al. Direct transfection of clonal organoids in Matrigel microbeads: a promising approach toward organoid-based genetic screens. Nucleic Acids Research. 46 (12), 70(2018).
- Lazaridis, K. N., LaRusso, N. F. The Cholangiopathies. Mayo Clinic Proceedings. 90 (6), 791-800 (2015).
- Tam, P. K., Yiua, R. S., Lendahl, U., Andersson, E. R. Cholangiopathies - Towards a molecular understanding. EBioMedicine. 35, 381-393 (2018).
- Loarca, L., et al. Development and characterization of cholangioids from normal and diseased human cholangiocytes as an in vitro model to study primary sclerosing cholangitis. Laboratory Investigation. 97, 1385-1396 (2017).
- De Assuncao, T. M., Jalan-Sakrikar, N., Huebert, R. C. Regenerative medicine and the biliary tree. Seminars in Liver Disease. 37, 17-27 (2017).
- Dianat, N. H., et al. Generation of functional cholangiocyte-like cells from human pluripotent stem cells and HepaRG cells. Hepatology. 60, 700-714 (2014).
- Masyuk, A. I., et al. Cholangiocyte autophagy contributes to hepatic cystogenesis in polycystic liver disease and represents a potential therapeutic target. Hepatology. 67 (3), 1088-1108 (2018).
- Sampaziotis, F., Cardoso, M., Madrigal, P., Bertero, A., Saeb-Parsy, K., et al. Cholangiocytes derived from human induced pluripotent stem cells for disease modeling and drug validation. Nature Biotechnology. 33 (8), 845-852 (2015).
- Soroka, J. C., et al. Bile-Derived Organoids From Patients With Primary Sclerosing Cholangitis Recapitulate Their Inflammatory Immune Profile. Hepatology. 70 (3), 871-882 (2019).
- Funfak, F., et al. Biophysical Control of Bile Duct Epithelial Morphogenesis in Natural and Synthetic Scaffolds. Frontiers in Bioengineering and Biotechnology. 7 (417), 417(2019).
- Du, Y., et al. Bile Duct-on-a-Chip With Organ-Level Functions. Hepatology. 0 (0), (2019).
- Shiota, J. M., Mohamad Zaki, N. H., Merchant, J. L., Samuelson, L. C., Razumilava, N. Generation of Organoids from Mouse Extrahepatic Bile Ducts. Journal of Visualized Experiments. (146), e59544(2019).
- Bircsak, K. M., Richardson, J. R., Aleksunes, L. M. Inhibition of Human MDR1 and BCRP Transporter ATPase Activity by Organochlorine and Pyrethroid Insecticides. Journal of Biochemical and Molecular Toxicology. 27 (2), 157-164 (2013).
- Fennema, E., Rivron, N., Rouwkema, J., Blitterswijk, C., Boer, J. Spheroid culture as a tool for creating 3D complex tissues. Trends in Biotechnology. 31 (2), 108-115 (2013).
- Kanade, S., Nataraj, G., Ubale, M., Mehta, P. Fluorescein Diacetate Vital Staining for Detecting Viability of Acid-Fast Bacilli in Patients on Antituberculosis Treatment. International Journal of Mycobacteriology. 5 (3), 294-298 (2016).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide Apoptosis Assay For Accurate Assessment of Cell Death. Journal of Visualized Experiments. (50), e2597(2011).
- Tabibian, J. H., Masyuk, A., Masyuk, T. V., O'Hara, S. P., LaRusso, N. F. Physiology of Cholangiocytes. Comprehensive Physiology. 3 (1), (2013).
- Spirlì, C., et al. Functional polarity of Na+/H+ and Cl-/HCO3- exchangers in a rat cholangiocyte cell line. American Journal Physiology. 275, 1236-1245 (1998).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados