Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Generierung und quantitative Charakterisierung funktioneller und polarisierter Gallenepithelzysten
In diesem Artikel
Zusammenfassung
Dreidimensionale (3D) zelluläre Systeme sind relevante Modelle zur Untersuchung der Organogenese. Eine hydrogelbasierte Methode für die Produktion von Gallenzysten und deren Charakterisierung wird vorgeschlagen. Dieses Protokoll entschlüsselt die Barrieren der 3D-Charakterisierung, mit einer einfachen und zuverlässigen Methode, um die Effizienz der Zystenbildung und ihre Funktionalität zu bewerten.
Zusammenfassung
Cholangiozyten, die Epithelzellen, die die Gallengänge in der Leber aneinanderreihen, bewachen die Gallenbildung und -modifikation. In den letzten zwanzig Jahren sind im Zusammenhang mit Lebererkrankungen dreidimensionale (3D) Modelle entstanden, die auf Cholangiozyten basieren, wie Zysten, Sphäroide oder röhrenartige Strukturen, um Gewebetopologie für Organogenese, Krankheitsmodellierung und Arzneimittelscreening-Studien nachzuahmen. Diese Strukturen wurden hauptsächlich durch Einbettung von Cholangiozyten in ein Hydrogel gewonnen. Der Hauptzweck bestand darin, die Selbstorganisation zu untersuchen, indem epitheliale Polarität, funktionelle und morphologische Eigenschaften angegangen wurden. Jedoch, nur sehr wenige Studien konzentrieren sich auf Zystenbildung Effizienz. Wenn dies der Fall ist, wird die Effizienz oft anhand von Bildern einer einzelnen Ebene quantifiziert. Funktionelle Assays und Strukturanalysen werden durchgeführt, ohne die potenzielle Heterogenität der Zystenverteilung darzustellen, die sich aus Hydrogelpolymerisationsheterogenitäten und Nebenwirkungen ergibt. Daher kann die quantitative Analyse, wenn sie durchgeführt wird, nicht für den Vergleich von einem Artikel zum anderen verwendet werden. Darüber hinaus erlaubt diese Methode keine Vergleiche des 3D-Wachstumspotenzials verschiedener Matrizen und Zelltypen. Darüber hinaus wird die experimentelle Fehlerbehebung für immunstainierende Zysten nicht erwähnt. In diesem Artikel bieten wir eine zuverlässige und universelle Methode, um zu zeigen, dass die anfängliche Zellverteilung mit der heterogenen vertikalen Verteilung der Zystenbildung zusammenhängt. Cholangiozytenzellen, die in Hydrogel eingebettet sind, werden mit Z-Stacks-Analysen entlang der Hydrogeltiefe im Laufe von 10 Tagen verfolgt. Mit dieser Methode wird eine robuste Kinetik der Zystenbildung Effizienz und Wachstum erhalten. Wir präsentieren auch Methoden zur Bewertung der Zystenpolarität und der Sekretorialfunktion. Schließlich werden zusätzliche Tipps zur Optimierung von Immunstaining-Protokollen zur Verfügung gestellt, um den Zystenkollaps für die Bildgebung zu begrenzen. Dieser Ansatz kann auf andere 3D-Zellkulturstudien angewendet werden, wodurch die Möglichkeiten eröffnet werden, ein System mit einem anderen zu vergleichen.
Einleitung
In den letzten drei Jahrzehnten hat sich der Bereich der In-vitro-Forschung zu 3D-Kultursystemen entwickelt. Eine Reihe von Protokollen sind für die Kultivierung von Zellen in 3D als Sphäroide oder Aggregate in Gegenwart oder Abwesenheit eines Gerüstes/Matrix, in einem Tropfen, in Deragitation, in mikrofluidischen Geräten oder schwimmend1entstanden. Die Verwendung von 3D-Kulturmethoden hat sich gegenüber 2-dimensionalen (2D) Kulturen bewährt, insbesondere bei Epithelzellen, die sich nachweislich in 3D-Strukturen, zysten oder acini, selbst organisieren. In diesem Fall bilden die Zellen eine Monoschicht, die ein Lumen umgibt, wo Zellen ihren vollen epitheliaalen Phänotyp mit verbesserten physiologischen spezifischen Funktionen erwerben2.
Zahlreiche Studien haben zur Entwicklung von Methoden zur Bildung dieser epitheliale Organoide in natürlichen Matrizen beigetragen. Dies hat es ermöglicht, in vivo Zell-Zell- und Zell-Mikroumgebung-Wechselwirkungen zu rekapitulieren, um die Etablierung und Stabilität des epithelialen Phänotyps3,4,5,6,7zu erhalten. Kürzlich, insbesondere mit dem Ziel, transplantierbare Organoide zu entwickeln und die Anforderung der Mikroumgebung für die Orchestrierung des Epithelprogramms zu entschlüsseln, wurden synthetische Hydrogele entwickelt, um die Bildung von epithelialen Acini8,9,10zu verbessern. Leider berichten diese Studien über qualitative Daten oder präsentieren Berechnungsmethoden mit internen Referenzen wie das Verhältnis von Zysten zu Nichtzysten in einer 2D-Ebene8,9,10. Dies schließt einen Vergleich zwischen verschiedenen Studien in Bezug auf Effizienz, Stabilität oder morphologische und physiologische Charakterisierung der Epithelorganoide aus.
Die Mikroverkapselung von Epithelzellen in Perlen mit mikrofluidischen Geräten hat realistischere quantitative und vergleichende Ergebnisse ermöglicht. Mit dieser Technologie wurden Organoide aus verschiedenen Zelltypen gebildet und anhand der Morphologie zwischen verschiedenen 3D-Zellstrukturen11,12unterschieden. Diese Technologie ist jedoch nicht einfach zu bedienen und erfordert die Verwendung von Reinräumen, um die mikrofluidischen Geräte herzustellen. Diese Technologie wurde für einige Arten von Hydrogelen etabliert, erfordert aber technische Anpassungen, um auf andere Hydrogele angewendet zu werden, was ihre Vielseitigkeit einschränkt. Daher basieren die meisten Studien, die epitheliale Organoide entwickeln sollen, auf der Einbettung von Epithelzellen in eine Hydrogelmasse. Bei diesen Methoden wird die hohe Heterogenität der Gelstrukturierung und Zellverteilung innerhalb der gesamten 3D-Kultur oft vernachlässigt. Daher beziehen sich die meisten Analysen auf einzelne 2D-Bilder, die nur sehr grob die Verteilung der verschiedenen zellulären Objekte im gesamten 3D-Volumen darstellen.
Krankheiten, die Gallengänge beeinflussen, wie Cholangiokarzinom, Gallenatresie, primäre sklerosierende Cholangitis, unter anderem, sind eine der Hauptursachen für Sterblichkeit und Morbidität. Mit Ausnahme der Lebertransplantation gibt es keine wirksamen Behandlungen für diese Bedingungen13. Bemühungen zur Untersuchung der Gallengangbildung, der Krankheitsursachen und des Fortschreitens werden die Entwicklung neuartiger Therapien ermöglichen14.
Biliäre organotypische Modelle von Zysten, Sphäroiden oder röhrenähnlichen Strukturen mit normalen oder patientenabgeleiteten, differenzierten oder von Vorläufern abgeleiteten Cholangiozytenzelllinien wurden15,16,17,18,19,20entwickelt. Verschiedene Studien haben cholangiozyte Polarität, Expression von Cholangiozytenmarkern, Vorhandensein von Zilien, Cholangiocyte sekretot und reabsortive Fähigkeit, und Lumenbildung und Obstruktion rekapituliert; alle stellen wichtige Merkmale des Cholangiocyten-Phänotyps, der Morphologie und der Funktion15,17,19dar. Andere haben berichtet, dass die Aufrechterhaltung von patientenabgeleiteten Gallenorganoiden über einen längeren Zeitraum20berichtet hat. Kürzlich haben wir die Rolle biochemischer und biophysikalischer Hinweise auf gallenzysische Organogeneseuntersucht 21. Wichtig ist, dass die Pathogenese der Gallenatresie in Gallensphäriden und Röhren untersucht wurde7,22. Darüber hinaus wurden Schlüsselmerkmale der primären sklerosierenden Cholangitis wie Cholangiozytenszenz, Sekretion von proinflammatorischen Zytokinen sowie Makrophagenrekrutierung erfolgreich mit Gallensphäriden15,20untersucht. Reproduzierbare in vitro 3D-quantitative Modelle, die physiologisch modulierencholangioyten Phänotyp, Physiologie und Mikroumgebung, in denen diese Fragen behandelt werden können, sind jedoch noch erforderlich. Darüber hinaus haben nur wenige Veröffentlichungen zystenbildung Effizienzberichtet 21,23. Dies ist ein wichtiger Punkt, um festzustellen, vor allem bei der Untersuchung der Organogenese, Krankheitsursache, und Korrelation der Arzneimittelreaktionen mit Cholangigiocyte Funktion und Polarisation. Darüber hinaus ist es schwierig, mit Unterschieden in Gerüst/Matrix, die von Protokoll zu Protokoll verwendet werden, zwischen Systemen zu vergleichen. Um diese Probleme zu lösen, schlagen wir eine quantitative, zuverlässige und universelle Methode vor, um Gallenzysten zu erzeugen, die Lumenbildung, Cholangiozytenpolarisation und Cholangiozytensekretoriennachlungimitat imitieren. Wichtig ist, dass wir eine systematische Analyse entlang der Z-Achse über das 3D-Gel hinweg präsentieren, wenn wir die Effizienz der Zystenbildung, Größe, Lebensfähigkeit, Polarisation und Funktionalität im Zeitverlauf bewerten. Darüber hinaus verwendeten wir ein natürliches Hydrogel und normale Rattencholangiocyten (NRC)s, als Beispiel für das Protokoll, aber andere natürliche oder synthetische Hydrogele, sowie Epithelzellen könnten für die Bildung von 3D-zystischen Strukturen verwendet werden.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Erzeugung von Zysten
HINWEIS: Dieses Protokoll kann mit jeder Art von Hydrogel durchgeführt werden, wenn die Gegebung das Einbetten von Zellen zulässt.
- Hydrogelbeschichtung
HINWEIS: Die richtige Hydrogelbeschichtung des Kammerschlittens ist ein kritischer Schritt, um die Bildung von 2D-Zellschichten auf der Unterseite des Brunnens zu vermeiden, die die nachfolgende Zystenbildgebung stören und die Berechnung der Zystenbildungseffizienz beeinträchtigen könnten.- Um die Homogenität der Gellösung zu gewährleisten, das Hydrogel über Nacht (O/N) bei 4 °C auftauen.
- Vorcoolpipettenspitzen auf Eis oder O/N bei -20 °C und ein 8-Well-Kammerschlitten bei -20 °C O/N.
- Legen Sie das Hydrogel und die 8-Well-Kammerrutsche auf einen eisbedeckten Eiskübel.
- In einem 15 ml konischen Rohr eine Lösung mit 40% Hydrogel (V/V) in kaltem NRC-Gesamtmedium (siehe Materialtabelle)vorbereiten und die Röhre auf Eis legen.
- Um eine Kammerrutsche mit kalten Pipettenspitzen zu beschichten, fügen Sie 50 L Hydrogellösung in der Mitte jedes Brunnens hinzu und verteilen sie über die gesamte Oberfläche mit einer Pipettenspitze, während Sie die Kammerrutsche auf Eis halten (Abbildung 1A).
HINWEIS: Verteilen Sie die Hydrogellösung so gleichmäßig wie möglich, um Blasen zu vermeiden. - Um das Hydrogel zu polymerisieren, inkubieren Sie den Kammerschlitten mindestens 15 min bei 37 °C, 5%CO2.
- Zellvorbereitung
- Erwärmen Sie NRC-Komplettmedium, phosphatgepufferte Salzsäure (PBS) und Trypsin-Ethylendiamin-Tetraessigsäure (Trypsin-EDTA) in einem auf 37 °C vorgeheizten Wasserbad.
- Während das Hydrogel polymerisiert, stellen Sie sicher, dass NRCs in einem T-25cm2 Kollagenkolben21auf 70% Konfluenz angebaut werden. Waschen Sie die Zellen einmal mit vorgeheiztem 1x PBS.
- Inkubieren Sie die NRCs mit 5 ml vorgeheiztem 1x PBS (für einen T-25 cm2 Kolben) für 20 min bei 37 °C, 5%CO2.
HINWEIS: Dieser Schritt, der die Inkubationszeit mit trypsin-EDTA verkürzt, ist entscheidend, um die selbstorganisierenden Eigenschaften von Zellen beizubehalten. - Entsorgen Sie die PBS, fügen Sie 1 ml Trypsin-EDTA hinzu und brüten Sie 5-10 min bei 37 °C, 5%CO2.
- Neutralisieren Sie mit 4 ml vorgewärmten kompletten NRC Medium. Sammeln und übertragen Sie die Zellsuspension in ein 15 ml konisches Rohr und drehen Sie bei 150 x g für 4 min.
- Entsorgen Sie das Medium und setzen Sie das Zellpellet in 5 ml vorgeheiztem Medium wieder auf.
- Filtern Sie die Zelllösung mit einem 40 mL-Zellsieb in ein 50 ml konisches Rohr und zählen die Zellen.
ANMERKUNG: Das Übergeben der Zellen durch ein Sieb ist ein entscheidender Schritt, damit die quantitativen Ergebnisse reproduzierbar sind, d. h. dass fast ähnliche Größe von Zellaggregaten eingebettet werden soll.
- Einbettung der Zellsuspension in Hydrogellösung
- Bereiten Sie eine Lösung von 1.600 l 80% Hydrogel (V/V) in kaltem komplettem NRC-Medium (Rohr 1); im Eis zu halten. 5 x 105 Zellen/ml in 1.600 l kaltem NRC-Medium (Tube 2) verdünnen und im Eis halten.
HINWEIS: Dieser Schritt muss schnell durchgeführt werden, um eine Polymerisation des Hydrogels beim Mischen mit der Zellsuspension zu vermeiden und die Zelllebensfähigkeit zu erhalten. - Zur Herstellung einer Zellsaatlösung von 2,5 x 105 Zellen/ml in 40% Hydrogel (V/V), Mischen Vonrohr 1 und Tube 2. Fügen Sie 400 l der Zelllösung in jede Bohrung des hydrogelbeschichteten Kammerschlittens ein, um Blasen zu vermeiden (Abbildung 1B).
- Halten Sie die Kammerrutsche in einem Inkubator bei 37 °C mit 5%CO2, bis sich die Medien ändern.
- Nach 2 Tagen in der Kultur, entfernen Sie 250 l des Mediums aus einer Ecke jedes Brunnens, achten Sie darauf, das Hydrogel nicht auszurohren. Fügen Sie dann langsam 250 l des Kulturmediums hinzu. Ändern Sie das Medium alle 2 Tage.
HINWEIS: Minimieren Sie die Bewegung der Kammerrutsche, insbesondere während der Zysteninitiation.
- Bereiten Sie eine Lösung von 1.600 l 80% Hydrogel (V/V) in kaltem komplettem NRC-Medium (Rohr 1); im Eis zu halten. 5 x 105 Zellen/ml in 1.600 l kaltem NRC-Medium (Tube 2) verdünnen und im Eis halten.
2. Zysenquantifizierung
- Zystenbildgebung
HINWEIS: Dieser Abschnitt sollte schnell durchgeführt werden, um die Zelllebensfähigkeit nicht zu beeinträchtigen, wenn das Mikroskop nicht mit einer Heizkammer zur Kontrolle von CO2 und Temperatur ausgestattet ist. Um eine konsistente Quantifizierung zu gewährleisten, die für die Zystenverteilung im gesamten Hydrogelvolumen repräsentativ ist, werden Zysten mittels Phasenkontrastmikroskopie und serieller Bildgebung (Z-Stacks) mit vordefinierten Parametern über verschiedene Zeitpunkte abgebildet.- Nehmen Sie einen Z-Stack entlang der Tiefe des Hydrogels für jeden Zeitpunkt (Abbildung 1C, D). In diesem Beispiel werden Z-Stacks an den Tagen 1, 2, 4, 7 und 10 genommen.
HINWEIS: Überprüfen Sie, ob die anfängliche Zellverteilung im Hydrogel einheitlich ist, um die Anwendbarkeit dieser Methode zu gewährleisten.- Mit einem Phasenkontrastmikroskop, das mit einer Bildaufnahmesoftware ausgestattet ist, wählen Sie die 10-fache Objektivvergrößerung im manuellen Nasenstück-Pad-Fenster aus (Abbildung 2B(1)).
- Schalten Sie die weiße Lampe ein, und wählen Sie die Hellfeld-Bildgebungsoption aus.
- Schalten Sie die Kamera ein, indem Sie die Schaltfläche"Wiedergabe" im Untermenü der Leiste auswählen. Konzentrieren Sie sich auf ein Zystenfeld und legen Sie die Belichtungszeit fest (Abbildung 2B(2)). Öffnen Sie das Fenster Auto Capture Folder für ein automatisches Speichern von Bildern ( Abbildung2B(3)).
- Öffnen Sie das Fenster der Erfassungs-Z-Serie und definieren Sie mit der Z-Schraube die obere und untere Ebene des Z-Stacks (gleiche XY-Koordinaten, aber unterschiedlich Z geschirmt). Passen Sie den Z-Schritt abhängig vom Objektiv, der Auflösungsstufe an und drücken Sie die Taste"Jetzt ausführen", um die Erfassung zu starten (Abbildung 2B(4)).
HINWEIS: In diesem Beispiel werden Zysten über eine Hydrogeldicke von 520 m verteilt. 26 Bilder werden entlang der Hydrogeltiefe mit einem Z-Schritt-Intervall von 20 m aufgenommen. Je nach Ziel sollte der Z-Schritt so eingestellt werden, dass keine Zyste verpasst wird und der Nachweis einzelner Zellen und Aggregate sichergestellt wird. - Nehmen Sie mindestens 3 nicht überlappende Z-Stacks pro Brunnen.
HINWEIS: Diese Probenahme ist notwendig, wenn, wie in diesem Beispiel, Zysten in der Tiefe des Gels zahlreicher sind als an den Rändern aufgrund von Heterogenitäten in der Hydrogelpolymerisation. - Um ein repräsentatives Dataset zu haben, wiederholen Sie Schritt 2.1.1.5. für insgesamt 3 Brunnen.
HINWEIS: Die heterogene Verteilung der Zysten hängt von der Art des Hydrogels, seiner Polymerisation und der Zelllinie ab. Unter Berücksichtigung von drei Z-Stacks pro Brunnen und drei Brunnen pro Experiment werden mindestens 200 Zysten über neun Z-Stacks abgebildet, um die Zystenbildung und das Zystenwachstum zu jedem Zeitpunkt zu charakterisieren.
- Nehmen Sie einen Z-Stack entlang der Tiefe des Hydrogels für jeden Zeitpunkt (Abbildung 1C, D). In diesem Beispiel werden Z-Stacks an den Tagen 1, 2, 4, 7 und 10 genommen.
- Bildverarbeitung
HINWEIS: In einem Hydrogel können NRCs als einzelne Zellen, Zysten oder Aggregate gefunden werden. Zysten werden durch das Vorhandensein einer runden und dünnen kontrastierten Zellhülle identifiziert, die ein Lumen umschließt, während Zellaggregate eine unregelmäßige Form darstellen und kein Lumen haben. Aggregate und einzelne Zellen haben ein dichtes und kontrastiertes Erscheinungsbild (Abbildung 3B(4)).- Öffnen Sie die Fidschi-Software, öffnen Sie den Z-Stack und gehen Sie zum Fidschi-Menü und klicken Sie auf Datei | Öffnen (Ergänzende Abbildung 1). Wählen Sie den zu analysierenden Z-Stack aus. Wählen Sie bei Bedarf die Option"Virtueller Stapel" und klicken Sie zum Öffnen auf "Ja" (Abbildung 3A(1)).
- Duplizieren Sie den Stapel über Image | Duplikat. Klicken Sie auf das Feld "Duplicate Stack" und klicken Sie auf "OK" (Ergänzende Abbildung 2).
HINWEIS: In diesem Beispiel sind Z-Stacks im .nd2-Dateiformat in 16 Bit codiert. - Erstellen Sie eine minimale Intensitätsprojektion aus dem duplizierten Stapel. Gehen Sie zum Bildmenü | Stapel | Z-Projekt. Wählen Sie Projektionstyp "Min Intensität" und klicken Sie auf "OK" (Abbildung 3A(2)) (Ergänzende Abbildung 3).
- Subtrahieren Sie den Hintergrund von der Projektion. Gehen Sie zum Menü Prozess | Hintergrund subtrahieren. Geben Sie 500,0 Pixel rollenden Kugelradius ein und klicken Sie auf"Lichthintergrund", um Zysten kontrastreicher als der Hintergrund zu rendern (Abbildung 3A(3)) (Ergänzende Abbildung 4).
HINWEIS: Der Rollenkugelradius definiert die Größe des Bereichs, auf dem die Hintergrundsubtraktion betrieben wird. Dieser Parameter muss auf die Größe des größten zu identifizierenden Objekts festgelegt werden. - Wenn eine Kontrastverbesserung erforderlich ist, gehen Sie zum Menü Bild | Anpassen | Helligkeit/Kontrast | Auto | Bewerben Sie sich. Fidschi wird automatisch Helligkeit und Kontrast optimieren. In (Abbildung 3A(3)) wurden die unteren und oberen Grauwerte auf 49702 bzw. 65452 (Ergänzende Abbildung 5) gesetzt.
ANMERKUNG: Wenn die Projektion nicht kalibriert ist, gehen Sie zum Menü Analysieren | Setzen Sie die Skala und geben Sie das entsprechende Kalibrierverhältnis von m/Pixel ein (Zusatzabbildung 6).
- Zystenzählung und Zystengrößenmessungen
- Um den ungefähren Zystendurchmesser zu messen, wählen Sie das Straight-Line-Werkzeug im Fidschi-Menü aus und zeichnen Sie eine Linie über den Durchmesser jeder Zyste auf der endigen Projektion (Abbildung 3B(4)). Fügen Sie dem ROI-Manager die neue Region von Interesse (ROI) hinzu, die für jede Zyste erstellt wurde: Drücken Sie die "t"-Verknüpfung" auf der Tastatur, um schneller zu zählen und den ROI-Manager zu öffnen. Klicken Sie auf "Alle anzeigen ", um die gezählten Zysten zu sehen (Ergänzende Abbildung 7)
- Stellen Sie fest, dass keine Zyste ohne Zählung übrig geblieben ist, indem Sie die eingestellten ROIs aus der Projektion auf dem Z-Stack überlagern. Klicken Sie dazu auf das Z-Stack-Fenster, um es auszuwählen. Klicken Sie im ROI-Manager auf"Alle anzeigen" und bewegen Sie den Cursor entlang des Z-Stacks, um dieses Bild pro Bild zu überprüfen, alle Zysten wurden gezählt (Ergänzende Abbildung 8).
- Sobald neue Zysten gezählt und ROIs in Schritt 2.3.1 hinzugefügt wurden, wählen Sie den ROI-Satz aus und speichern Sie ihn über das FENSTER ROI Manager, indem Sie auf Mehr | Speichern (Zusatzabbildung 9).
- Wählen Sie alle ROIs im ROI Manager aus und klicken Sie im ROI Manager auf "Messen", um die Größe jeder Zyste zu erhalten. Dadurch wird ein neues Zeitfenster mit dem Namen "Ergebnisse" geöffnet, das jede Zyste und ihre geschätzte Größe nummeriert. Speichern Sie dann im .csv Format, indem Sie auf das Fenster"Ergebnisse"und über das Menü: Datei | Save As (Zusatzabbildung 10).
HINWEIS: Ein Makro kann erstellt werden, um halbautomatisch Stapel zu verarbeiten, die Anzahl/Größen der Zysten aus den Projektionen zu schätzen und die Daten für eine schnellere Zählung zu speichern. Wählen Sie dazu im Balkenmenü das Tool "Record" aus, indem Sie auf Plugins | Makros | Datensatz.
- Quantifizierung der Effizienz der Zystenbildung
- Zählen Sie die Anzahl der Zysten an Tag Y
 auf einer Projektion (Y=1, 2, 4, 7 oder 10).
auf einer Projektion (Y=1, 2, 4, 7 oder 10). - Um die Zystenbildungseffizienz für 1000 Zellen am Tag Y zu berechnen, dividieren Sie die Anzahl der zu diesem Zeitpunkt gezählten Zysten durch die Anzahl der Zellen, die am Tag 0 aus dem Hydrogelvolumen abgeleitet wurden, und multiplizieren Sie mit 1000 (Abbildung 3C, Abbildung 4).
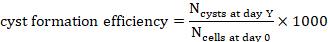
- Zählen Sie die Anzahl der Zysten an Tag Y
3. Zelllebensfähigkeit
- Bereiten Sie eine Stammlösung von Fluorescein-Diacetat (FDA) bei 5 mg/ml vor, indem Sie 5 mg FDA in 1 ml Aceton auflösen und bei -20 °C lagern.
- Bereiten Sie eine Lagerlösung mit Propidiumjodid (PI) in einer Konzentration von 2 mg/ml in entionisiertem Wasser (dH2O) vor und lagern Sie sie bei 4 °C.
- Vorbereiten NRC medium ohne fetales Wadenserum (FCS).
- Zur Vorbereitung der FDA/PI-Färbelösung fügen Sie 4 l FDA-Stammlösung (8 g/ml Endkonzentration) und 25 l PI-Lagerlösung (20 g/ml Endkonzentration) in 2,5 ml NRC-Medium ohne FCS ein.
- Entfernen Sie das Medium aus dem Kammerschlitten, fügen Sie 250 L Färbung Lösung in jeden Brunnen und inkubieren 4-5 min im Dunkeln bei 37 °C, 5%CO2. Die Färbelösung sorgfältig austeilen und einmal mit 250 l 1x PBS waschen.
- Fügen Sie vorsichtig 250 L komplettes NRC-Medium zu jedem Brunnen hinzu und fotografieren Sie mit einem invertierten Fluoreszenzmikroskop mit Texas-Rot- und Fluorescein-Isothiocyanat-Filtern (FITC). Lebende Zellen werden grün und abgestorbene Zellen rot sein (Abbildung 5A).
HINWEIS: Um lebende/tote Zellen zu quantifizieren, nehmen Sie Z-Stacks über das Hydrogelvolumen nach Schritt 2 und passen Sie die Bildverarbeitungsmethode für die Fluoreszenz an.
4. Sekretionsaktivität
HINWEIS: Die Sekretionsaktivität durch die apikale Membran der Cholangiozyten wird durch die Sekretion von Fluorescein im Lumen beurteilt. Seine Spezifität kann durch den gleichen Test mit Verapamil, einem multiresistenten (MDR) Transporterhemmer(24)bewertet werden.
- Um eine Färbelösung von Hoechst 33258 bei 5 g/ml vorzubereiten, fügen Sie 0,83 l Hoechst-Stammlösung (15 mg/ml Lagerkonzentration in dH2O) in 2,5 ml NRC-Medium ohne FCS hinzu.
- Fügen Sie in jedem Brunnen 250 L der Hoechst-Lösung hinzu und brüten Sie bei 37 °C, 5%CO2 für 15 min.
- Entfernen Sie die Hoechst-Lösung, und fügen Sie in jedem Brunnen 250 l FDA-Lösung (8 g/ml Endkonzentration) hinzu. 4-5 min bei 37 °C, 5%CO2inkubieren.
HINWEIS: Sobald Zellen der FDA-Färbelösung ausgesetzt sind, könnte die Nachverfolgung der Fluorescein-Sekretionskinetik nützlich sein, um die Zeit zu kalibrieren, die für das Absepernieren von Zysten benötigt wird. Nehmen Sie dazu jede min für 1 h per Zeitraffer-Bildgebung Bilder auf. In diesem Beispiel beträgt die Zeit, die benötigt wird, um NRC-Sekretionszysten im Hydrogel zu beobachten, etwa 15-20 min. - Nehmen Sie Bilder mit einem invertierten Fluoreszenzmikroskop 5 min nach dem Spülen mit Medium ohne FCS auf. Verwenden Sie 4,6-Diamidino-2-Phenylindole (DAPI) und FITC-Filter, um Kerne Kennzeichnung und Fluorescein-Akkumulation im Lumen zu offenbaren (Abbildung 6A). Um die Anzahl der sezernierenden Zysten zu quantifizieren, nehmen Sie Z-Stacks wie in Schritt 2 und passen Sie die Bildverarbeitungsschritte an fluoreszierende Bilder an.
HINWEIS: Für den Verapamil-Test vor dem vorherigen Prozess (Schritte 4.3. bis 4.4.) durch eine Inkubation mit Verapamil, gemäß den folgenden Bedingungen: - Bereiten Sie eine Stammlösung von 10 mM Verapamil in Dimethylsulfoxid (DMSO) vor. Um eine Arbeitslösung mit 10 m zu erstellen, mischen Sie 2,5 l Verapamil-Lagerlösung mit einem 2,5 ml-Kulturmedium ohne FCS.
- Um zu beurteilen, dass die Fluoreszenz im Lumen aus der MDR-Sekretion resultiert, nehmen Sie einen weiteren Schlitten und fügen Sie 250 L Verapamil Arbeitslösung in jedem Brunnen hinzu und brüten 20 min bei 37° C, 5%CO2
- Entfernen Sie die Lösung und fügen Sie jedem Bohrgut 250 l FDA-Lösung (8 g/ml Endkonzentration) hinzu. 4-5 min im Dunkeln bei 37 °C, 5%CO2inkubieren. Dann waschen Sie mit 250 l 1x PBS, bevor Sie bildgebungsbilden (Abbildung 6B, C).
5. Epitheliale Polaritätsbewertung durch Immunfluoreszenz
- Zur Herstellung der Befestigungslösung 4% Formaldehyd mit 5% Saccharose in 1x PBS, pH 7,4 mischen und in einem bei 37 °C vorgeheizten Wasserbad inkubieren.
- Um die Zellen zu fixieren, pipette das Kulturmedium vorsichtig aus dem Brunnen heraus, ohne die Matrix zu beschädigen. Fügen Sie langsam 400 l der Befestigungslösung an der Seite der Brunnen hinzu. Inkubieren Sie für 20 min bei Raumtemperatur (RT).
HINWEIS: Lassen Sie immer 25 l der Flüssigkeit über der Matrix, um schäden zu verhindern. - Entfernen Sie die Befestigungslösung vorsichtig und waschen Sie 3x mit 400 l 1x PBS bei (RT).
- Pipetten Sie die PBS heraus, fügen Sie 400 L Permeabilisationslösung (0,5% Triton X-100 in 1x PBS) und inkubieren 10 min bei RT.
- Entfernen Sie vorsichtig die Permeabilisationslösung, gefolgt von 3 Schnellwäschen mit 400 l 1x PBS und einem langen Waschschritt von 30 min bei RT.
HINWEIS: In diesem Schritt kann der Schlitten bei 4 °C für 2 Tage gelagert werden. Versiegeln Sie in diesem Fall das Dia mit einem Paraffinfilm, um Verdunstung und Matrixtrocknung zu verhindern. - Entfernen Sie die PBS, fügen Sie 400 L der blockierenden Lösung, die 0,1% Rinderserumalbumin (BSA) und 10% Ziegenserum in 1x PBS enthält, und brüten für 60 min bei RT.
VORSICHT: Konzentrationen von BSA über 0,1% führen zu Lumenrückzug und weiterem Zystenkollaps (siehe Abschnitt Repräsentative Ergebnisse) (Abbildung 7A). - Die Sperrlösung auspfeifen und einmal mit 400 l PBS/0,05% Tween 20 waschen und entsorgen.
- Fügen Sie 150 l der Antikörperlösung hinzu, z. B. E-Cadherin-Antikörper verdünnt 1:400 und Phalloidin 568 (16,2 nM Endkonzentration) in 1x PBS und inkubieren sie 90 min bei RT.
HINWEIS: Diese Verdünnung von E-Cadherin ist die gleiche, die wie in einem Standard-2D-Immunfluoreszenzprotokoll verwendet wird. - Waschen Sie die Probe mit 400 l PBS/0,05% Tween 20, 3x; jedes Mal, wenn die Probe für 10 min bei RT inkubiert wird.
- Fügen Sie 150 l des sekundären Antikörpers (Ziegenanti-Kaninchen IgG Alexa Fluor Plus 647), verdünnt 1:500 in 1x PBS und inkubieren 60 min bei RT.
- Waschen Sie 3x mit 400 l PBS/0,05% Tween 20, jedes Mal inkubieren die Probe für 10 min bei RT.
- Waschen Sie 3x mit 400 l 1x PBS, wobei die Probe jedes Mal 10 min bei RT inkubiert wird.
- Entsorgen Sie die PBS der letzten Wäsche und bereiten Sie die Kammerrutsche nach einer der beiden folgenden Optionen zur Visualisierung mittels konfokaler Mikroskopie vor.
- Fügen Sie 400 l 1x PBS und 50 l DAPI pro Brunnen hinzu. Die Proben können durch den Boden des Brunnens untersucht werden, ohne dass eine Montage mit einem Deckschein erforderlich ist (Abbildung 7B).
- Fügen Sie 100 l pro Bohrung antifade Reagenz enthaltenDAPI und lassen Sie die Folie trocknen O/ N bei RT.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Bildung und Charakterisierung von Zysten
3D-Zellkultursysteme sind ein wichtiges Werkzeug, um Organogenese und Krankheitsmodellierung zu studieren25. Leider sind die meisten dieser Methoden qualitativ oder verwenden interne Quantifizierung auf einer einzigen Ebene durch den Vergleich der Anzahl der Zysten im Vergleich zu Nicht-Zysten, in variablen und oft nicht spezifizierten Volumen, verhindert jeden Vergleich in Bezug auf Zystenbildung Effizienz zwischen den verschiedenen Stu...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Um die Organogenese und die Erhaltung von 3D-Zellstrukturen zu untersuchen, wurden verschiedene Gewebe modelliert, mit unterschiedlichen zellulären Ursprüngen, aber auch verschiedenen Arten von extrazellulären Matrizen einschließlich synthetischer Hydrogele8,9,10,21. Aufgrund des Fehlens einer quantitativen 3D-Analyse, die Vergleiche zwischen Methoden in Bezug auf Organoidbildung oder Funkt...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken Dr. Nicholas LaRusso (Mayo Clinic, Rochester, Minnesota, Vereinigte Staaten), der freundlicherweise die NRC-Zelllinie zur Verfügung gestellt hat.
Diese Arbeit wurde sowohl vom iLite RHU-Programm (Grant ANR-16-RHUS-0005) als auch von der DHU Hepatinov finanziell unterstützt.
Wir danken Isabelle Garcin und Réseau d'Imagerie Cellulaire Paris Saclay für ihre Unterstützung bei der Bildgebung.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 10 µl- Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000020 | |
| 100 µl - Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000046 | |
| 1000 µl - Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000062 | |
| 1X PBS | Thermo Fisher Scientific | 14190-094 | |
| 200 µl - Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000054 | |
| 3,3′,5-Triiodo-L-thyronine sodium salt | Sigma-Aldrich | T5516 | NRC complete medium final concentration = 3.4 µg/mL |
| Acetic acid | VWR | 20104-298 | 0.02N final |
| Aerosol barrier pipettes tips 10 µl (Fisherbrand) | Thermo Fisher Scientific | 2707439 | |
| Aerosol barrier pipettes tips 1000 µl (Fisherbrand) | Thermo Fisher Scientific | 2707404 | |
| Aerosol barrier pipettes tips 200 µl (Fisherbrand) | Thermo Fisher Scientific | 2707430 | |
| Antibiotic Antimicotic Solution (100X) | Sigma-Aldrich | A5955 | NRC complete medium final concentration = 1:100 dilution |
| Bovine pituitary extract | Thermo Fisher Scientific | 13028-014 | NRC complete medium final concentration = 30 µg/mL |
| Bovine serum albumin | Sigma-Aldrich | A2153 | 1:1000 dilution |
| Chemically Defined Lipid Concentrate (100X) | Thermo Fisher Scientific | 11905-031 | NRC complete medium final concentration = 1:100 dilution |
| Collagen high concentration, rat tail | Thermo Fisher Scientific | 354249 | 50 µg/mL final concentration |
| Dexamethasone | Sigma-Aldrich | D4902 | NRC complete medium final concentration = 0.393 µg/mL |
| DMEM F12 | Thermo Fisher Scientific | 21331-020 | NRC complete medium final concentration = 1X |
| E-cadherin Rabbit anti-Human, Rat, Polyclonal | Thermo Fisher Scientific | PA5-32178 | 1:400 dilution |
| Eclipse TE300 inverted microscope | Nikon | imaging | |
| Ethanolamine | Sigma-Aldrich | E9508 | NRC complete medium final concentration = 0.32 mM |
| Fetal calf serum | Thermo Fisher Scientific | 10270-106 | NRC complete medium final concentration = 5:100 dilution |
| Fluoroshield with DAPI (Mounting medium) | Sigma-Aldrich | F6057 | |
| Formaldehyde 16% (W/V) | Thermo Fisher Scientific | 28906 | 4% (W/V) |
| Goat serum | Thermo Fisher Scientific | 16210-064 | 1:10 dilution |
| Hamamatsu camera (Digital camera C11440 ORCA - flash 4.OLT) | Hamamatsu | imaging | |
| Hoechst 33258 | Sigma-Aldrich | B1155 | 5 µg/mL final concentration |
| IgG (H+L) Highly Cross-Adsorbed Goat anti-Rabbit, Alexa Fluor Plus 647 | Thermo Fisher Scientific | A32733 | 1:500 dilution |
| ImageJ version 2.0.0-rc-69/1.52n | Open source image processing software | ||
| Insulin-Transferrin-Selenium (100X) | Thermo Fisher Scientific | 51300-044 | NRC complete medium final concentration = 1:100 dilution |
| L-Glutamine (100X) | Thermo Fisher Scientific | 25030-024 | NRC complete medium final concentration = 1:100 dilution |
| Matrigel GFR (stock concentration 9.7 mg/mL) | Thermo Fisher Scientific | 356231 | 4:10 dilution |
| NIS Elements software version 4.50.00 | Nikon | image acquisition and display | |
| Non-Essential-Amino-Acids-Solution (100X) | Thermo Fisher Scientific | 11140-035 | NRC complete medium final concentration = 1:100 dilution |
| Objective Plan Fluor 10X/0.30 Ph1 DL (∞/1.2 WD 15.2) | Nikon | ||
| Prolong Gold Antifade Reagent | Thermo Fisher Scientific | P36931 | |
| Propidium Iodide (PI) | Sigma-Aldrich | P4170 | 20 µg/mL final concentration |
| Rhodamine Phalloidin | Thermo Fisher Scientific | R415 | 16.2 nM final concentration |
| Sir-Actin / Verapamil kit | Spirochrome | SC001 | 10 µM final concentration |
| Soybean trypsin inhibitor | Thermo Fisher Scientific | 17075-029 | NRC complete medium final concentration = 50 µg/mL |
| Sterile cell strainer 40 µm (Fisherbrand) | Thermo Fisher Scientific | 22363547 | |
| Sterile pipettes 10 mL (Fisherbrand) | Thermo Fisher Scientific | 1367811E | |
| Sterile pipettes 5 mL (Fisherbrand) | Thermo Fisher Scientific | 1367811D | |
| Sterile tubes 1.5 mL (Fisherbrand) | Thermo Fisher Scientific | 11926955 | |
| Sterile tubes 15 mL (Fisherbrand) | Thermo Fisher Scientific | 7200886 | |
| Sterile tubes 50 mL (Fisherbrand) | Thermo Fisher Scientific | 553913 | |
| Sucrose | Sigma-Aldrich | S0389 | 5:100 dilution |
| Tissue culture treated flask 25cm2 (Falcon) | Thermo Fisher Scientific | 353108 | |
| Triton X-100 | Sigma-Aldrich | T8787 | 5:1000 dilution |
| Trypsin-EDTA (0.05%) phenol red | Thermo Fisher Scientific | 25300-054 | 1X |
| Tween-20 | Sigma-Aldrich | P1379 | 5:10000 dilution |
| Vitamin (100X) | Thermo Fisher Scientific | 11120-037 | NRC complete medium final concentration = 1:100 dilution |
| μ-Slide 8 Well ibiTreat, Ibidi | Clinisciences | 80826 |
Referenzen
- Edmondson, R., Broglie, J. J., Adcock, F., Yang, L. Three-Dimensional Cell Culture Systems and Their Applications in Drug Discovery and Cell-Based Biosensors. ASSAY and Drug Development Technologies. 12 (4), 207-218 (2014).
- Martín-Belmonte, F., et al. Cell-polarity dynamics controls the mechanism of lumen formation in epithelial morphogenesis. Current Biology. 18, 507-513 (2008).
- Debnath, J., Muthuswamy, S. K., Brugge, J. Morphogenesis and oncogenesis of MCF-10A mammary epithelial acini grown in three-dimensional basement membrane cultures. Methods. 30 (3), 256-268 (2003).
- Artym, V. V., Matsumoto, K. Imaging Cells in Three-Dimensional Collagen Matrix. Current Procotols in Cell Biology. , Chapter 10 (Unit 10) (2010).
- Petersen, O. W., Ronnov-Jessen, L., Howlett, A. R., Bisell, M. J. Interaction with basement membrane serves to rapidly distinguish growth and differentiation pattern of normal and malignant human breast epithelial cells. Proceedings of the National Academy of Sciences of the United States of America. 89, 9064-9068 (1992).
- Kim, S. P., Lee, D. H., Park, J. K. Development of hepatocyte spheroids immobilization technique using alternative encapsulation method. Biotechnology and Bioprocess Engineering. 3, 96-102 (1998).
- Lorent, K., et al. Identification of a plant isoflavonoid that causes biliary atresia. Science Translational Medicine. 7 (286), 67(2015).
- Nowak, M., Freudenberga, U., Tsurkana, M. V., Wernera, C., Levental, K. R. Modular GAG-matrices to promote mammary epithelial morphogenesis in vitro. Biomaterials. 112, 20-30 (2017).
- Miroshnikova, Y. A., et al. Engineering Strategies to Recapitulate Epithelial Morphogenesis Within Synthetic Three-Dimensional Extracellular Matrix With Tunable Mechanical Properties. Physical Biology. 8 (2), 026013(2011).
- Ozdemir, T., et al. Tuning Hydrogel Properties to Promote the Assembly of Salivary Gland Spheroids in 3D. ACS Biomaterials Science & Engineering. 2 (12), 2217-2230 (2016).
- Dolega, M. E., Abeille, F., Picollet-D'hahan, N., Gidrol, X. Controlled 3D culture in Matrigel microbeads to analyze clonal acinar development. Biomaterials. 52, 347-357 (2015).
- Laperrousaz, B., et al. Direct transfection of clonal organoids in Matrigel microbeads: a promising approach toward organoid-based genetic screens. Nucleic Acids Research. 46 (12), 70(2018).
- Lazaridis, K. N., LaRusso, N. F. The Cholangiopathies. Mayo Clinic Proceedings. 90 (6), 791-800 (2015).
- Tam, P. K., Yiua, R. S., Lendahl, U., Andersson, E. R. Cholangiopathies - Towards a molecular understanding. EBioMedicine. 35, 381-393 (2018).
- Loarca, L., et al. Development and characterization of cholangioids from normal and diseased human cholangiocytes as an in vitro model to study primary sclerosing cholangitis. Laboratory Investigation. 97, 1385-1396 (2017).
- De Assuncao, T. M., Jalan-Sakrikar, N., Huebert, R. C. Regenerative medicine and the biliary tree. Seminars in Liver Disease. 37, 17-27 (2017).
- Dianat, N. H., et al. Generation of functional cholangiocyte-like cells from human pluripotent stem cells and HepaRG cells. Hepatology. 60, 700-714 (2014).
- Masyuk, A. I., et al. Cholangiocyte autophagy contributes to hepatic cystogenesis in polycystic liver disease and represents a potential therapeutic target. Hepatology. 67 (3), 1088-1108 (2018).
- Sampaziotis, F., Cardoso, M., Madrigal, P., Bertero, A., Saeb-Parsy, K., et al. Cholangiocytes derived from human induced pluripotent stem cells for disease modeling and drug validation. Nature Biotechnology. 33 (8), 845-852 (2015).
- Soroka, J. C., et al. Bile-Derived Organoids From Patients With Primary Sclerosing Cholangitis Recapitulate Their Inflammatory Immune Profile. Hepatology. 70 (3), 871-882 (2019).
- Funfak, F., et al. Biophysical Control of Bile Duct Epithelial Morphogenesis in Natural and Synthetic Scaffolds. Frontiers in Bioengineering and Biotechnology. 7 (417), 417(2019).
- Du, Y., et al. Bile Duct-on-a-Chip With Organ-Level Functions. Hepatology. 0 (0), (2019).
- Shiota, J. M., Mohamad Zaki, N. H., Merchant, J. L., Samuelson, L. C., Razumilava, N. Generation of Organoids from Mouse Extrahepatic Bile Ducts. Journal of Visualized Experiments. (146), e59544(2019).
- Bircsak, K. M., Richardson, J. R., Aleksunes, L. M. Inhibition of Human MDR1 and BCRP Transporter ATPase Activity by Organochlorine and Pyrethroid Insecticides. Journal of Biochemical and Molecular Toxicology. 27 (2), 157-164 (2013).
- Fennema, E., Rivron, N., Rouwkema, J., Blitterswijk, C., Boer, J. Spheroid culture as a tool for creating 3D complex tissues. Trends in Biotechnology. 31 (2), 108-115 (2013).
- Kanade, S., Nataraj, G., Ubale, M., Mehta, P. Fluorescein Diacetate Vital Staining for Detecting Viability of Acid-Fast Bacilli in Patients on Antituberculosis Treatment. International Journal of Mycobacteriology. 5 (3), 294-298 (2016).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide Apoptosis Assay For Accurate Assessment of Cell Death. Journal of Visualized Experiments. (50), e2597(2011).
- Tabibian, J. H., Masyuk, A., Masyuk, T. V., O'Hara, S. P., LaRusso, N. F. Physiology of Cholangiocytes. Comprehensive Physiology. 3 (1), (2013).
- Spirlì, C., et al. Functional polarity of Na+/H+ and Cl-/HCO3- exchangers in a rat cholangiocyte cell line. American Journal Physiology. 275, 1236-1245 (1998).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten