É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Geração e Caracterização Quantitativa de Cistos Epiteliais Biliar funcionais e polarizados
Neste Artigo
Resumo
Sistemas celulares tridimensionais (3D) são modelos relevantes para investigar organogênese. Propõe-se um método à base de hidrogel para a produção de cistos biliares e sua caracterização. Este protocolo desvenda as barreiras da caracterização 3D, com um método simples e confiável para avaliar a eficiência, tamanhos e testar sua funcionalidade.
Resumo
Cholangiocytes, as células epiteliais que alinham os dutos biliares no fígado, supervisionam a formação e modificação da bile. Nos últimos vinte anos, no contexto das doenças hepáticas, surgiram modelos tridimensionais (3D) baseados em cholangiocitos, como cistos, esferoides ou estruturas semelhantes a tubos para imitar topologia tecidual para organogênese, modelagem de doenças e estudos de rastreamento de medicamentos. Essas estruturas foram obtidas principalmente pela incorporação de cholangiocitos em um hidrogel. O objetivo principal foi estudar a auto-organização abordando a polaridade epitelial, propriedades funcionais e morfológicas. No entanto, pouquíssimos estudos se concentram na eficiência da formação de cistos. Quando este é o caso, a eficiência é muitas vezes quantificada a partir de imagens de um único plano. Ensaios funcionais e análises estruturais são realizados sem representar a potencial heterogeneidade da distribuição de cisto surgida de heterogeneidades de polimerização de hidrogel e efeitos colaterais. Portanto, a análise quantitativa, quando feita, não pode ser utilizada para comparação de um artigo com outro. Além disso, essa metodologia não permite comparações do potencial de crescimento 3D de diferentes matrizes e tipos de células. Além disso, não há menção à solução experimental de problemas para cistos imunossínetos. Neste artigo, fornecemos um método confiável e universal para mostrar que a distribuição celular inicial está relacionada à distribuição vertical heterogênea da formação de cisto. As células de colangiocitos embutidas em hidrogel são seguidas com a análise de pilhas de Z ao longo da profundidade do hidrogel ao longo do tempo de 10 dias. Com este método, obtém-se uma cinética robusta da eficiência e crescimento da formação de cistos. Também apresentamos métodos para avaliar a polaridade do cisto e a função secreta. Finalmente, dicas adicionais para otimizar protocolos de imunossuagem são fornecidas a fim de limitar o colapso do cisto para imagens. Essa abordagem pode ser aplicada a outros estudos de cultura celular 3D, abrindo assim as possibilidades de comparar um sistema com outro.
Introdução
Nas últimas três décadas, o campo da pesquisa in vitro avançou em direção aos sistemas de cultura 3D. Uma série de protocolos surgiram para a cultura de células em 3D como esferoides ou agregados na presença ou ausência de um andaime/matriz, em uma gota, em agitação, em dispositivos microfluidos, ou flutuante1. O uso de métodos de cultura 3D provou suas vantagens sobre culturas bidimensionais (2D), particularmente para células epiteliais, que foram demonstradas para auto-organizar em estruturas 3D, chamadas cistos ou acini. Neste caso, as células formam uma monocamada circundando um lúmen, onde as células adquirem seu fenótipo epitelial completo com funções específicas fisiológicas melhoradas2.
Inúmeros estudos têm contribuído para o desenvolvimento de métodos para a formação desses organoides epiteliais em matrizes naturais. Isso permitiu recapitular interações in vivo célula-célula e microambiente celular, para obter o estabelecimento e a estabilidade do fenótipo epitelial3,4,5,6,7. Recentemente, e em particular com o objetivo de desenvolver organoides transplantáveis e decifrar a exigência do microambiente para orquestrar o programa epitelial, hidrogéis sintéticos foram desenvolvidos para melhorar a formação de acini epitelial8,9,10. Infelizmente, esses estudos relatam dados qualitativos, ou apresentam métodos de cálculo utilizando referências internas, como a razão de cistos sobre não cistos em um plano 2D8,9,10. Isso exclui qualquer comparação entre diferentes estudos em termos de eficiência, estabilidade ou caracterização morfológica e fisiológica dos organoides epiteliais.
A microencapsulação de células epiteliais em contas usando dispositivos microfluidos permitiu resultados quantitativos e comparativos mais realistas. Utilizando essa tecnologia, organoides de vários tipos celulares foram formados e diferenciados com base na morfologia entre diferentes estruturas celulares 3D11,12. No entanto, essa tecnologia não é fácil de trabalhar e requer o uso de salas limpas para produzir os dispositivos microfluidos. Esta tecnologia foi estabelecida para alguns tipos de hidrogéis, mas requer adaptação técnica para ser aplicada a outros hidrogéis, restringindo sua versatilidade. Portanto, a maioria dos estudos destinados a desenvolver organoides epiteliais dependem da incorporação de células epiteliais em um granel de hidrogel. Nesses métodos, a alta heterogeneidade da estruturação em gel e distribuição celular dentro de toda a cultura 3D é muitas vezes negligenciada. Portanto, a maioria das análises diz respeito a imagens 2D únicas, que representam apenas muito aproximadamente a distribuição dos vários objetos celulares em todo o volume 3D.
Doenças que afetam os ductos biliares, como o colangiocarcinoma, atresia biliar, cholangite esclerosante primária, entre outras, são uma das principais causas de mortalidade e morbidade. Com exceção do transplante de fígado, não há tratamentos eficazes para essas condições13. Os esforços para investigar a formação de dutos biliares, as causas da doença e a progressão permitirão o desenvolvimento de novas terapias14.
Foram desenvolvidos modelos organotipicos biliares de cistos, esferoides ou estruturas semelhantes a tubos utilizando linhas celulares de cholangiocitos normais ou derivadas do paciente, diferenciadas ou progenitoras,15,16,17,18,19,20. Vários estudos têm polarização de colangiocito recapitulado, expressão de marcadores de cholangiocito, presença de cílios, collangiocitos secretos e capacidade reabsortiva, e formação e obstrução de lúmen; todas elas representam características importantes do fenótipo de colangiocito, morfologia e função15,17,19. Outros relataram a manutenção de organoides biliar derivados do paciente por longos períodos detempo 20. Recentemente, investigamos o papel das pistas bioquímicas e biofísicas sobre cistos biliares organogênese21. Importante ressaltar que a patogênese da atresia biliar foi estudada em esferoides biliares e tubos7,22. Além disso, características-chave da colangite esclerosante primária, como senescência de colangiocito, secreção de citocinas pró-inflamatórias, bem como o recrutamento de macrófagos foram estudados com sucesso usando esferoides biliares15,20. No entanto, modelos quantitativos in vitro in vitro 3D que modulam fisiologicamente fenótipo de collangiocito, fisiologia e microambiente onde essas questões podem ser abordadas ainda são necessários. Além disso, apenas poucas publicações relataram eficiência de formação de cisto21,23. Este é um ponto importante a se estabelecer, particularmente ao investigar organogênese, causa da doença e correlação de respostas medicamentosas com função de cholangiocito e polarização. Além disso, com diferenças no andaime/matriz utilizadas do protocolo ao protocolo, é difícil comparar entre sistemas. Para resolver essas questões, propomos um método quantitativo, confiável e universal para gerar cistos biliares imitando a formação de lúmen, a polarização do cholangiocyte e a função secreta do cholangiocito. É importante ressaltar que apresentamos uma análise sistemática realizada ao longo do eixo Z ao longo do gel 3D ao avaliar ao longo do tempo, eficiência de formação de cisto, tamanho, viabilidade, polarização e funcionalidade. Além disso, utilizamos um hidrogel natural e collangiocitos normais de ratos (NRC)s, como exemplo para o protocolo, mas outros hidrogéis naturais ou sintéticos, bem como células epiteliais poderiam ser usados para a formação de estruturas císticas 3D.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Geração de cistos
NOTA: Este protocolo pode ser realizado com qualquer tipo de hidrogel, se a gelação permitir a incorporação de células.
- Revestimento de hidrogel
NOTA: O revestimento adequado do hidrogel do escorregador da câmara é um passo crítico para evitar a formação de camadas de células 2D na parte inferior do poço, que podem interferir com a imagem subsequente do cisto e prejudicar o cálculo da eficiência da formação de cisto.- Para garantir a homogeneidade da solução de gel, descongele o hidrogel a 4 °C durante a noite (O/N).
- Dicas de pipeta pré-mistura no gelo ou O/N a -20 °C e um deslizamento de câmara de 8 poços a -20 °C O/N.
- Coloque o hidrogel e o deslizamento de câmara de 8 poços em um balde de gelo cheio de gelo.
- Em um tubo cônico de 15 mL, prepare uma solução contendo 40% de hidrogel (V/V) em meio completo NRC frio (ver Tabela de Materiais) e coloque o tubo no gelo.
- Para revestir um escorregador de câmara, utilizando pontas de pipeta fria, adicione 50 μL de solução de hidrogel no centro de cada poço, e espalhe por toda a superfície usando uma ponta de pipeta, mantendo o deslizamento da câmara no gelo(Figura 1A).
NOTA: Espalhe a solução de hidrogel o mais uniforme possível evitando bolhas. - Para polimerizar o hidrogel, incubar o escorregador de câmara por pelo menos 15 min a 37 °C, 5% DE CO2.
- Preparação celular
- Aqueça o soro fisiológico tamponado nrc completo, fosfato (PBS), e o ácido tetraácático de trippsin-etilenodiamina (trypsin-EDTA) em um banho de água pré-aquecido a 37 °C.
- Enquanto o hidrogel estiver polimerizando, certifique-se de que as NRCs sejam cultivadas até 70% de confluência em um frasco t-25 cm2 revestido de colágeno21. Lave as células uma vez com 1x PBS pré-aquecido.
- Incubar os NRCs com 5 mL de PBS pré-aquecido 1x (para um frasco T-25 cm2) por 20 min a 37 °C, 5% CO2.
NOTA: Esta etapa, que reduz o tempo de incubação com trypsin-EDTA, é fundamental para manter as propriedades auto-organizadoras das células. - Descarte o PBS, adicione 1 mL de trippsina-EDTA e incubar por 5-10 min a 37 °C, 5% CO2.
- Neutralizar com 4 mL de meio NRC completo pré-aquecido. Colete e transfira a suspensão da célula em um tubo cônico de 15 mL e gire a 150 x g por 4 min.
- Descarte o meio e resuspenque a pelota de célula em 5 mL de meio pré-aquecido.
- Usando um coador de células de 40 μm, filtrar a solução celular em um tubo cônico de 50 mL e contar as células.
NOTA: Passar as células através de um coador é um passo crítico para que os resultados quantitativos sejam reprodutíveis, ou seja, para obter tamanho quase semelhante de agregados celulares a serem incorporados.
- Incorporação de suspensão celular em solução de hidrogel
- Preparar uma solução de 1.600 μL de 80% de hidrogel (V/V) em meio NRC completo a frio (tubo 1); manter no gelo. Diluir 5 x 105 células/mL em 1.600 μL de meio NRC completo frio (tubo 2) e manter no gelo.
NOTA: Esta etapa deve ser realizada rapidamente para evitar a polimerização do hidrogel, ao mesmo tempo em que a mistura com a suspensão celular e para manter a viabilidade celular. - Para preparar uma solução de semeadura celular de 2,5 x 105 células/mL em 40% de hidrogel (V/V), misture tubo 1 e tubo 2. Adicione 400 μL da solução celular em cada poço do slide de câmara revestido de hidrogel evitando bolhas(Figura 1B).
- Mantenha o deslizamento da câmara em uma incubadora a 37 °C com 5% de CO2 até que a mídia mude.
- Após 2 dias na cultura, remova 250 μL do meio de um canto de cada poço, tenha cuidado para não pipeta o hidrogel. Em seguida, adicione lentamente 250 μL do meio de cultura. Troque o meio a cada 2 dias.
NOTA: Minimize o movimento do deslizamento da câmara, particularmente durante a iniciação do cisto.
- Preparar uma solução de 1.600 μL de 80% de hidrogel (V/V) em meio NRC completo a frio (tubo 1); manter no gelo. Diluir 5 x 105 células/mL em 1.600 μL de meio NRC completo frio (tubo 2) e manter no gelo.
2. Quantificação de cisto
- Imagem de cisto
NOTA: Esta seção deve ser realizada rapidamente para não comprometer a viabilidade celular se o microscópio não estiver equipado com uma câmara de aquecimento para controlar o CO2 e a temperatura. Para garantir uma quantificação consistente, representativa da distribuição de cistos no volume total de hidrogel, os cistos são imagens via microscopia de contraste de fase e imagem serial (Z-stacks), com parâmetros pré-definidos em diferentes pontos de tempo.- Pegue uma pilha Z ao longo da profundidade do hidrogel para cada ponto de tempo(Figura 1C, D). Neste exemplo, as pilhas Z são tomadas nos dias 1, 2, 4, 7 e 10.
NOTA: Verifique se a distribuição inicial da célula é uniforme no hidrogel para garantir a aplicabilidade deste método.- Com um microscópio de contraste de fase equipado com um software de aquisição de imagens, selecione a ampliação objetiva de 10x na janela manual da almofada de peça do nariz(Figura 2B(1).
- Ligue a lâmpada branca e selecione a opção de imagem brightfield.
- Ligue a câmera selecionando o botão "Reproduzir" no submenu da barra. Concentre-se em um campo de cistos e ajuste o tempo de exposição(Figura 2B(2)). Abra a janela Pasta de Captura Automática para uma economia automática de imagens(Figura 2B(3)).
- Abra a janela da série Z de captura e defina com o parafuso Z os planos superior e inferior da pilha Z (as mesmas coordenadas XY, mas diferentes telas Z). Ajuste a etapa Z dependendo do objetivo, do nível de resolução e pressione o botão "Executar agora" para iniciar a aquisição(Figura 2B(4)).
NOTA: Neste exemplo, os cistos são espalhados sobre uma espessura de hidrogel de 520 μm. 26 imagens são adquiridas ao longo da profundidade do hidrogel com um intervalo de 20 μm de passo Z. Dependendo do objetivo, a etapa Z deve ser ajustada para não perder nenhum cisto e garantir a detecção de células e agregados únicos. - Tome pelo menos 3 pilhas Z não sobrepostas por poço.
NOTA: Esta amostragem é necessária quando, como neste exemplo, os cistos são mais numerosos na profundidade do gel do que nas bordas devido às heterogeneidades na polimerização do hidrogel. - Para que um conjunto de dados representativo repita a etapa 2.1.1.5. para 3 poços no total.
NOTA: A distribuição heterogênea dos cistos depende do tipo de hidrogel, sua polimerização e da linha celular. Considerando três pilhas de Z por poço e três poços por experimento, um mínimo de 200 cistos são imagens ao longo de nove pilhas Z para caracterizar a formação de cisto e o crescimento de cistos em cada ponto de tempo.
- Pegue uma pilha Z ao longo da profundidade do hidrogel para cada ponto de tempo(Figura 1C, D). Neste exemplo, as pilhas Z são tomadas nos dias 1, 2, 4, 7 e 10.
- Processamento de imagens
NOTA: Em um hidrogel, as NRCs podem ser encontradas como células únicas, cistos ou agregados. Os cistos são identificados pela presença de uma concha celular redonda e fina contrastada que envolve um lúmen, enquanto os agregados celulares apresentam uma forma irregular e não possuem lúmen. Agregados e células individuais têm aparência densa e contrastada(Figura 3B(4)).- Abra o software Fiji, abra a pilha Z e vá para o menu Fiji e clique em Arquivo | Aberto (FiguraSuplementar 1). Selecione a pilha Z para analisar. Se necessário, selecione "Virtual Stack" e clique em "Sim" para abertura (Figura 3A(1)).
- Duplicar a pilha via Imagem | Duplicata. Clique na caixa "Duplicar stack" e clique em "OK"(Figura Suplementar 2).
NOTA: Neste exemplo, as pilhas Z estão em formato de arquivo .nd2 codificado em 16 bits. - Crie uma projeção de intensidade mínima a partir da pilha duplicada. Vá para o menu Imagem | Pilhas | Projeto Z. Selecione o tipo de projeção "Intensidade mínima" e clique em "OK"(Figura 3A(2)(Figura Suplementar 3).
- Subtraia o fundo da projeção. Vá para o menu Processo | Subtrair fundo. Digite 500,0 pixels de raio de bola rolando e clique em "fundo de luz" para tornar os cistos mais contrastados do que o fundo(Figura 3A(3)(Figura Suplementar 4).
NOTA: O raio da bola rolante define o tamanho da região em que a subtração de fundo é operada. Este parâmetro deve ser definido para o tamanho do maior objeto a ser identificado. - Se for necessário aprimoramento de contraste, vá para o menu Imagem | Ajuste | Brilho/Contraste | Auto | Inscreva-se. Fiji otimizará automaticamente o brilho e o contraste. Em (Figura 3A(3)), os valores cinza inferior e superior foram definidos para 49702 e 65452, respectivamente (Figura Suplementar 5).
NOTA: Se a projeção não estiver calibrada, vá para o menu Analisar | Defina a escala e digite a razão de calibração correspondente μm/pixel(Figura Suplementar 6).
- Contagem de cisto e medidas de tamanho de cisto
- Para medir o diâmetro aproximado do cisto, selecione a ferramenta linha reta no menu Fiji e desenhe uma linha através do diâmetro de cada cisto na projeção final(Figura 3B(4)). Adicione a nova região de interesse (ROI) criada para cada cisto ao gerenciador de ROI: pressione o atalho "t" no teclado para uma contagem mais rápida e abertura do gerenciador de ROI. Clique em "Mostrar Tudo" para ver os cistos contados(Figura Suplementar 7)
- Verifique se nenhum cisto foi deixado sem contar sobrepondo os ROIs definidos da projeção na pilha Z. Para isso, clique na janela Z-stack para selecioná-la. No ROI Manager, clique em "Mostrar Tudo" e mova o cursor ao longo da pilha Z para verificar essa imagem por imagem, todos os cistos foram contados(Figura Suplementar 8).
- Uma vez que novos cistos tenham sido contados e ROIs adicionados na etapa 2.3.1., selecione o conjunto ROI e salve-o através da janela ROI Manager clicando mais | Poupança (Figura Suplementar 9).
- Selecione todos os ROIs no GERENCIADOr de ROI e clique em "Medida" no Gerenciador de ROI para obter o tamanho de cada cisto. Isso abrirá uma nova janela de medições chamada "Resultados" numerando cada cisto e seu tamanho estimado. Em seguida, salve em .csv formato clicando na janela "Resultados" e através do menu: Arquivo | Salvar As (Figura Suplementar 10).
NOTA: Uma macro pode ser criada para processar pilhas semiautomáticas, estimar números/tamanhos de cisto das projeções e armazenar os dados para um procedimento de contagem mais rápido. Para isso, selecione a ferramenta "Gravar" no menu da barra, clicando em Plugins | Macros | Recorde.
- Quantificação da eficiência da formação de cisto
- Conte o número de cistos no dia Y,
 em uma projeção (Y=1, 2, 4, 7 ou 10).
em uma projeção (Y=1, 2, 4, 7 ou 10). - Para calcular a eficiência de formação de cisto para 1000 células no dia Y, divida o número de cistos contados nesse ponto de tempo pelo número de células semeadas no dia 0 inferidas do volume de hidrogel e multiplique por 1000 (Figura 3C, Figura 4).
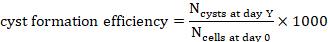
- Conte o número de cistos no dia Y,
3. Viabilidade celular
- Prepare uma solução de estoque de diacetato de fluoresceína (FDA) a 5 mg/mL dissolvendo 5 mg de FDA em acetona de 1 mL e armazene a -20 °C.
- Prepare uma solução de estoque de iodeto de propídio (PI) a uma concentração de 2 mg/mL em água deionizada (dH2O) e armazene a 4 °C.
- Prepare o meio NRC sem soro de bezerro fetal (FCS).
- Para preparar a solução de coloração FDA/PI, adicione 4 μL de solução de estoque FDA (concentração final de 8 μg/mL) e 25 μL de solução de estoque PI (20 μg/mL de concentração final) em 2,5 mL de meio NRC sem FCS.
- Retire o meio do slide da câmara, adicione 250 μL de solução de coloração em cada poço e incuba 4-5 min no escuro a 37 °C, 5% DE CO2. Pipeta a solução de coloração cuidadosamente e lave uma vez com 250 μL de 1x PBS.
- Adicione cuidadosamente 250 μL de meio NRC completo a cada poço e tire fotos usando um microscópio de fluorescência invertido com filtros de isocitonato vermelho e fluoresceína do Texas (FITC). As células vivas serão verdes e as células mortas serão vermelhas(Figura 5A).
NOTA: Para quantificar as células vivas/mortas, pegue pilhas de Z através do volume de hidrogel após o passo 2 e adapte o método de processamento de imagem para fluorescência.
4. Atividade de secreção
NOTA: A atividade de secreção através da membrana apical dos colangiocitos é avaliada pela secreção de fluoresceína no lúmen. Sua especificidade pode ser avaliada fazendo o mesmo teste com o Verapamil, um inibidor de transporte multi-resistente (MDR)24.
- Para preparar uma solução de coloração de Hoechst 33258 a 5 μg/mL, adicione 0,83 μL de solução de estoque Hoechst (concentração de estoque de 15 mg/mL em dH2O) em 2,5 mL de meio NRC sem FCS.
- Adicione 250 μL de solução Hoechst em cada poço e incubar a 37 °C, 5% de CO2 por 15 min.
- Remova a solução Hoechst e adicione 250 μL de solução FDA (concentração final de 8 μg/mL) em cada poço. Incubar 4-5 min a 37 °C, 5% CO2.
NOTA: Assim que as células forem expostas à solução de coloração da FDA, o acompanhamento da cinética de secreção de fluoresceína pode ser útil para calibrar o tempo necessário para que os cistos se secretem. Para isso, tire fotos a cada minuto por 1h através de imagens de lapso de tempo. Neste exemplo, o tempo necessário para observar cistos secretos do NRC no hidrogel é de cerca de 15-20 min. - Tire imagens usando um microscópio de fluorescência invertida 5 minutos após enxaguar com meio sem FCS. Use filtros 4′,6-diamidino-2-fenilôs (DAPI) e FITC para revelar rotulagem de núcleos e acúmulo de fluoresceína no lúmen(Figura 6A). Para quantificar o número de cistos secretos, pegue pilhas Z como na etapa 2 e adapte as etapas de processamento de imagem a imagens fluorescentes.
NOTA: Para o teste de Verapamil, preceder o processo anterior (etapas 4.3. a 4.4.) por uma incubação com Verapamil, de acordo com as seguintes condições: - Prepare uma solução de estoque de 10 mM Verapamil em sulfóxido de dimetila (DMSO). Para preparar a solução de trabalho de 10 μM, misture 2,5 μL de solução de estoque Verapamil com meio de cultura de 2,5 mL sem FCS.
- Para avaliar que a fluorescência no lúmen resulta da secreção de MDR, tome outro slide e adicione 250 μL de solução de trabalho Verapamil em cada poço e incubar 20 min a 37° C, 5% CO2
- Remova a solução e adicione 250 μL de solução FDA (concentração final de 8 μg/mL) em cada poço. Incubar 4-5 min no escuro a 37 °C, 5% CO2. Em seguida, lave com 250 μL de 1x PBS, antes da imagem(Figura 6B, C).
5. Avaliação da polaridade epitelial por imunofluorescência
- Para preparar a solução de fixação, misture 4% de formaldeído com 5% de sacarose, em 1x PBS, pH 7.4 e incubar em banho-maria pré-aquecido a 37 °C.
- Para fixar as células, pipeta suavemente o meio de cultura do poço sem danificar a matriz. Adicione lentamente 400 μL da solução de fixação ao lado dos poços. Incubar por 20 min em temperatura ambiente (RT).
NOTA: Deixe sempre 25 μL do líquido acima da matriz para evitar seu dano. - Remova suavemente a solução de fixação e lave 3x com 400 μL de 1x PBS at (RT).
- Pipeta para fora do PBS, adicione 400 μL de solução de permeabilização (0,5% Triton X-100 em 1x PBS) e incubar 10 min na RT.
- Remova suavemente a solução de permeabilização, seguida de 3 lavagens rápidas com 400 μL de 1x PBS e uma longa etapa de lavagem de 30 min na RT.
NOTA: Nesta etapa, o slide pode ser armazenado a 4 °C por 2 dias. Neste caso, sele o slide com um filme de parafina para evitar a evaporação e a secagem da matriz. - Remova o PBS, adicione 400 μL de solução de bloqueio contendo 0,1% de albumina de soro bovino (BSA) e 10% de soro de cabra em 1x PBS e incubar por 60 min na RT.
ATENÇÃO: Concentrações de BSA superiores a 0,1% resultarão em retração do lúmen e novo colapso de cisto (ver Seção resultados representativos) (Figura 7A). - Pipeta a solução de bloqueio e lave uma vez com 400 μL de PBS/0,05% Interpol 20 e descarte.
- Adicione 150 μL da solução de anticorpos, por exemplo, anticorpo E-cadherin diluído 1:400 e Phalloidin 568 (concentração final de 16,2 nM) em 1x PBS e incubar por 90 min na RT.
NOTA: Esta diluição de E-cadherin é a mesma usada em um protocolo padrão de imunofluorescência 2D. - Lave a amostra com 400 μL de PBS/0,05% Tween 20, 3x; cada vez incubando a amostra por 10 minutos no RT.
- Adicione 150 μL do anticorpo secundário (anti-coelho de cabra IgG Alexa Fluor Plus 647), diluído 1:500 em 1x PBS e incubar 60 min na RT.
- Lave 3x com 400 μL de PBS/0,05% Tween 20, cada vez incubando a amostra por 10 min na RT.
- Lave 3x com 400 μL de 1x PBS, cada vez incubando a amostra por 10 min na RT.
- Descarte o PBS da última lavagem e prepare o slide da câmara para visualização via microscopia confocal seguindo uma das duas opções abaixo.
- Adicione 400 μL de 1x PBS e 50 μL de DAPI por poço. As amostras podem ser examinadas através da parte inferior do poço sem a necessidade de montagem com um deslizamento de tampas (Figura 7B).
- Adicione 100 μL por poço de reagente antifade contendo DAPI e deixe o slide secando O/N na RT.
Access restricted. Please log in or start a trial to view this content.
Resultados
Formação e caracterização de cistos
Os sistemas de cultura celular 3D são uma importante ferramenta para estudar organogênese e modelagem de doenças25. Infelizmente, a maioria desses métodos são qualitativos ou utilizam quantificação interna realizada em um único plano, comparando o número de cistos versus não cistos, em volumes variáveis e muitas vezes não especificados, impedindo qualquer comparação em termos de eficiência de formação de cisto entre os di...
Access restricted. Please log in or start a trial to view this content.
Discussão
Para estudar a organogênese e a manutenção de estruturas celulares 3D, vários tecidos foram modelados, utilizando diferentes origens celulares, mas também diferentes tipos de matrizes extracelulares, incluindo hidrogéis sintéticos8,9,10,21. No entanto, devido à falta de análise quantitativa 3D que permita comparações entre métodos em termos de formação ou funcionalidade organoides...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos ao Dr. Nicholas LaRusso (Mayo Clinic, Rochester, Minnesota, Estados Unidos), que gentilmente forneceu a linha celular NRC.
Este trabalho recebeu o apoio financeiro tanto do programa iLite RHU (conceder ANR-16-RHUS-0005) quanto do DHU Hepatinov.
Agradecemos a Isabelle Garcin e Réseau d'Imagerie Cellulaire Paris Saclay pelo apoio à imagem.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 10 µl- Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000020 | |
| 100 µl - Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000046 | |
| 1000 µl - Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000062 | |
| 1X PBS | Thermo Fisher Scientific | 14190-094 | |
| 200 µl - Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000054 | |
| 3,3′,5-Triiodo-L-thyronine sodium salt | Sigma-Aldrich | T5516 | NRC complete medium final concentration = 3.4 µg/mL |
| Acetic acid | VWR | 20104-298 | 0.02N final |
| Aerosol barrier pipettes tips 10 µl (Fisherbrand) | Thermo Fisher Scientific | 2707439 | |
| Aerosol barrier pipettes tips 1000 µl (Fisherbrand) | Thermo Fisher Scientific | 2707404 | |
| Aerosol barrier pipettes tips 200 µl (Fisherbrand) | Thermo Fisher Scientific | 2707430 | |
| Antibiotic Antimicotic Solution (100X) | Sigma-Aldrich | A5955 | NRC complete medium final concentration = 1:100 dilution |
| Bovine pituitary extract | Thermo Fisher Scientific | 13028-014 | NRC complete medium final concentration = 30 µg/mL |
| Bovine serum albumin | Sigma-Aldrich | A2153 | 1:1000 dilution |
| Chemically Defined Lipid Concentrate (100X) | Thermo Fisher Scientific | 11905-031 | NRC complete medium final concentration = 1:100 dilution |
| Collagen high concentration, rat tail | Thermo Fisher Scientific | 354249 | 50 µg/mL final concentration |
| Dexamethasone | Sigma-Aldrich | D4902 | NRC complete medium final concentration = 0.393 µg/mL |
| DMEM F12 | Thermo Fisher Scientific | 21331-020 | NRC complete medium final concentration = 1X |
| E-cadherin Rabbit anti-Human, Rat, Polyclonal | Thermo Fisher Scientific | PA5-32178 | 1:400 dilution |
| Eclipse TE300 inverted microscope | Nikon | imaging | |
| Ethanolamine | Sigma-Aldrich | E9508 | NRC complete medium final concentration = 0.32 mM |
| Fetal calf serum | Thermo Fisher Scientific | 10270-106 | NRC complete medium final concentration = 5:100 dilution |
| Fluoroshield with DAPI (Mounting medium) | Sigma-Aldrich | F6057 | |
| Formaldehyde 16% (W/V) | Thermo Fisher Scientific | 28906 | 4% (W/V) |
| Goat serum | Thermo Fisher Scientific | 16210-064 | 1:10 dilution |
| Hamamatsu camera (Digital camera C11440 ORCA - flash 4.OLT) | Hamamatsu | imaging | |
| Hoechst 33258 | Sigma-Aldrich | B1155 | 5 µg/mL final concentration |
| IgG (H+L) Highly Cross-Adsorbed Goat anti-Rabbit, Alexa Fluor Plus 647 | Thermo Fisher Scientific | A32733 | 1:500 dilution |
| ImageJ version 2.0.0-rc-69/1.52n | Open source image processing software | ||
| Insulin-Transferrin-Selenium (100X) | Thermo Fisher Scientific | 51300-044 | NRC complete medium final concentration = 1:100 dilution |
| L-Glutamine (100X) | Thermo Fisher Scientific | 25030-024 | NRC complete medium final concentration = 1:100 dilution |
| Matrigel GFR (stock concentration 9.7 mg/mL) | Thermo Fisher Scientific | 356231 | 4:10 dilution |
| NIS Elements software version 4.50.00 | Nikon | image acquisition and display | |
| Non-Essential-Amino-Acids-Solution (100X) | Thermo Fisher Scientific | 11140-035 | NRC complete medium final concentration = 1:100 dilution |
| Objective Plan Fluor 10X/0.30 Ph1 DL (∞/1.2 WD 15.2) | Nikon | ||
| Prolong Gold Antifade Reagent | Thermo Fisher Scientific | P36931 | |
| Propidium Iodide (PI) | Sigma-Aldrich | P4170 | 20 µg/mL final concentration |
| Rhodamine Phalloidin | Thermo Fisher Scientific | R415 | 16.2 nM final concentration |
| Sir-Actin / Verapamil kit | Spirochrome | SC001 | 10 µM final concentration |
| Soybean trypsin inhibitor | Thermo Fisher Scientific | 17075-029 | NRC complete medium final concentration = 50 µg/mL |
| Sterile cell strainer 40 µm (Fisherbrand) | Thermo Fisher Scientific | 22363547 | |
| Sterile pipettes 10 mL (Fisherbrand) | Thermo Fisher Scientific | 1367811E | |
| Sterile pipettes 5 mL (Fisherbrand) | Thermo Fisher Scientific | 1367811D | |
| Sterile tubes 1.5 mL (Fisherbrand) | Thermo Fisher Scientific | 11926955 | |
| Sterile tubes 15 mL (Fisherbrand) | Thermo Fisher Scientific | 7200886 | |
| Sterile tubes 50 mL (Fisherbrand) | Thermo Fisher Scientific | 553913 | |
| Sucrose | Sigma-Aldrich | S0389 | 5:100 dilution |
| Tissue culture treated flask 25cm2 (Falcon) | Thermo Fisher Scientific | 353108 | |
| Triton X-100 | Sigma-Aldrich | T8787 | 5:1000 dilution |
| Trypsin-EDTA (0.05%) phenol red | Thermo Fisher Scientific | 25300-054 | 1X |
| Tween-20 | Sigma-Aldrich | P1379 | 5:10000 dilution |
| Vitamin (100X) | Thermo Fisher Scientific | 11120-037 | NRC complete medium final concentration = 1:100 dilution |
| μ-Slide 8 Well ibiTreat, Ibidi | Clinisciences | 80826 |
Referências
- Edmondson, R., Broglie, J. J., Adcock, F., Yang, L. Three-Dimensional Cell Culture Systems and Their Applications in Drug Discovery and Cell-Based Biosensors. ASSAY and Drug Development Technologies. 12 (4), 207-218 (2014).
- Martín-Belmonte, F., et al. Cell-polarity dynamics controls the mechanism of lumen formation in epithelial morphogenesis. Current Biology. 18, 507-513 (2008).
- Debnath, J., Muthuswamy, S. K., Brugge, J. Morphogenesis and oncogenesis of MCF-10A mammary epithelial acini grown in three-dimensional basement membrane cultures. Methods. 30 (3), 256-268 (2003).
- Artym, V. V., Matsumoto, K. Imaging Cells in Three-Dimensional Collagen Matrix. Current Procotols in Cell Biology. , Chapter 10 (Unit 10) (2010).
- Petersen, O. W., Ronnov-Jessen, L., Howlett, A. R., Bisell, M. J. Interaction with basement membrane serves to rapidly distinguish growth and differentiation pattern of normal and malignant human breast epithelial cells. Proceedings of the National Academy of Sciences of the United States of America. 89, 9064-9068 (1992).
- Kim, S. P., Lee, D. H., Park, J. K. Development of hepatocyte spheroids immobilization technique using alternative encapsulation method. Biotechnology and Bioprocess Engineering. 3, 96-102 (1998).
- Lorent, K., et al. Identification of a plant isoflavonoid that causes biliary atresia. Science Translational Medicine. 7 (286), 67(2015).
- Nowak, M., Freudenberga, U., Tsurkana, M. V., Wernera, C., Levental, K. R. Modular GAG-matrices to promote mammary epithelial morphogenesis in vitro. Biomaterials. 112, 20-30 (2017).
- Miroshnikova, Y. A., et al. Engineering Strategies to Recapitulate Epithelial Morphogenesis Within Synthetic Three-Dimensional Extracellular Matrix With Tunable Mechanical Properties. Physical Biology. 8 (2), 026013(2011).
- Ozdemir, T., et al. Tuning Hydrogel Properties to Promote the Assembly of Salivary Gland Spheroids in 3D. ACS Biomaterials Science & Engineering. 2 (12), 2217-2230 (2016).
- Dolega, M. E., Abeille, F., Picollet-D'hahan, N., Gidrol, X. Controlled 3D culture in Matrigel microbeads to analyze clonal acinar development. Biomaterials. 52, 347-357 (2015).
- Laperrousaz, B., et al. Direct transfection of clonal organoids in Matrigel microbeads: a promising approach toward organoid-based genetic screens. Nucleic Acids Research. 46 (12), 70(2018).
- Lazaridis, K. N., LaRusso, N. F. The Cholangiopathies. Mayo Clinic Proceedings. 90 (6), 791-800 (2015).
- Tam, P. K., Yiua, R. S., Lendahl, U., Andersson, E. R. Cholangiopathies - Towards a molecular understanding. EBioMedicine. 35, 381-393 (2018).
- Loarca, L., et al. Development and characterization of cholangioids from normal and diseased human cholangiocytes as an in vitro model to study primary sclerosing cholangitis. Laboratory Investigation. 97, 1385-1396 (2017).
- De Assuncao, T. M., Jalan-Sakrikar, N., Huebert, R. C. Regenerative medicine and the biliary tree. Seminars in Liver Disease. 37, 17-27 (2017).
- Dianat, N. H., et al. Generation of functional cholangiocyte-like cells from human pluripotent stem cells and HepaRG cells. Hepatology. 60, 700-714 (2014).
- Masyuk, A. I., et al. Cholangiocyte autophagy contributes to hepatic cystogenesis in polycystic liver disease and represents a potential therapeutic target. Hepatology. 67 (3), 1088-1108 (2018).
- Sampaziotis, F., Cardoso, M., Madrigal, P., Bertero, A., Saeb-Parsy, K., et al. Cholangiocytes derived from human induced pluripotent stem cells for disease modeling and drug validation. Nature Biotechnology. 33 (8), 845-852 (2015).
- Soroka, J. C., et al. Bile-Derived Organoids From Patients With Primary Sclerosing Cholangitis Recapitulate Their Inflammatory Immune Profile. Hepatology. 70 (3), 871-882 (2019).
- Funfak, F., et al. Biophysical Control of Bile Duct Epithelial Morphogenesis in Natural and Synthetic Scaffolds. Frontiers in Bioengineering and Biotechnology. 7 (417), 417(2019).
- Du, Y., et al. Bile Duct-on-a-Chip With Organ-Level Functions. Hepatology. 0 (0), (2019).
- Shiota, J. M., Mohamad Zaki, N. H., Merchant, J. L., Samuelson, L. C., Razumilava, N. Generation of Organoids from Mouse Extrahepatic Bile Ducts. Journal of Visualized Experiments. (146), e59544(2019).
- Bircsak, K. M., Richardson, J. R., Aleksunes, L. M. Inhibition of Human MDR1 and BCRP Transporter ATPase Activity by Organochlorine and Pyrethroid Insecticides. Journal of Biochemical and Molecular Toxicology. 27 (2), 157-164 (2013).
- Fennema, E., Rivron, N., Rouwkema, J., Blitterswijk, C., Boer, J. Spheroid culture as a tool for creating 3D complex tissues. Trends in Biotechnology. 31 (2), 108-115 (2013).
- Kanade, S., Nataraj, G., Ubale, M., Mehta, P. Fluorescein Diacetate Vital Staining for Detecting Viability of Acid-Fast Bacilli in Patients on Antituberculosis Treatment. International Journal of Mycobacteriology. 5 (3), 294-298 (2016).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide Apoptosis Assay For Accurate Assessment of Cell Death. Journal of Visualized Experiments. (50), e2597(2011).
- Tabibian, J. H., Masyuk, A., Masyuk, T. V., O'Hara, S. P., LaRusso, N. F. Physiology of Cholangiocytes. Comprehensive Physiology. 3 (1), (2013).
- Spirlì, C., et al. Functional polarity of Na+/H+ and Cl-/HCO3- exchangers in a rat cholangiocyte cell line. American Journal Physiology. 275, 1236-1245 (1998).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados