È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Generazione e caratterizzazione quantitativa di cisti epiteliali biliari funzionali e polarizzate
In questo articolo
Riepilogo
I sistemi cellulari tridimensionali (3D) sono modelli rilevanti per lo studio dell'organogenesi. Viene proposto un metodo a base di idrogel per la produzione di cisti biliari e la loro caratterizzazione. Questo protocollo svela le barriere della caratterizzazione 3D, con un metodo semplice e affidabile per valutare l'efficienza di formazione delle cisti, le dimensioni e testarne la funzionalità.
Abstract
I colangiociti, le cellule epiteliali che allineano i dotti biliari nel fegato, supervisionano la formazione e la modifica della bile. Negli ultimi vent'anni, nel contesto delle malattie del fegato, sono emersi modelli tridimensionali (3D) basati su coriagiociti come cisti, sferoidi o strutture tube-like per imitare la topologia dei tessuti per l'organogenesi, la modellazione delle malattie e gli studi di screening farmacologico. Queste strutture sono state ottenute principalmente incorporando coriagiociti in un idrogel. Lo scopo principale era quello di studiare l'auto-organizzazione affrontando la polarità epiteliale, le proprietà funzionali e morfologiche. Tuttavia, pochissimi studi si concentrano sull'efficienza della formazione della cisti. In questo caso, l'efficienza viene spesso quantificata dalle immagini di un singolo piano. I test funzionali e l'analisi strutturale vengono eseguiti senza rappresentare la potenziale eterogeneità della distribuzione della cisti derivante da eterogeneità di polimerizzazione dell'idrogel ed effetti collaterali. Pertanto, l'analisi quantitativa, al termine, non può essere utilizzata per il confronto da un articolo all'altro. Inoltre, questa metodologia non consente confronti del potenziale di crescita 3D di diverse matrici e tipi di cellule. Inoltre, non si fa menzione della risoluzione dei problemi sperimentale per le cisti immunosotteninti. In questo articolo, forniamo un metodo affidabile e universale per dimostrare che la distribuzione iniziale delle cellule è correlata alla distribuzione verticale eterogenea della formazione di cisti. Le cellule di conlangiociti incorporate nell'idrogel sono seguite con l'analisi z-stack lungo la profondità dell'idrogel nel corso del tempo di 10 giorni. Con questo metodo, si ottiene una robusta cinetica di efficienza e crescita della formazione di cisti. Presentiamo anche metodi per valutare la polarità della cisti e la funzione secretoria. Infine, vengono forniti ulteriori suggerimenti per ottimizzare i protocolli di immunosottenzione al fine di limitare il collasso della cisti per l'imaging. Questo approccio può essere applicato ad altri studi di coltura cellulare 3D, aprendo così le possibilità di confrontare un sistema con un altro.
Introduzione
Negli ultimi tre decenni, il campo della ricerca in vitro è avanzato verso i sistemi di coltura 3D. Un certo numero di protocolli sono emersi per coltivare cellule in 3D come sferoidi o aggregati in presenza o assenza di un'impalcatura / matrice, in una goccia, in agitazione, in dispositivi microfluidici ogalleggianti 1. L'uso di metodi di coltura 3D ha dimostrato i suoi vantaggi rispetto alle colture bidimensionali (2D), in particolare per le cellule epiteliali, che hanno dimostrato di auto-organizzarsi in strutture 3D, chiamate cisti o acini. In questo caso, le cellule formano un monostrato che circonda un lume, dove le cellule acquisiscono il loro fenotipo epiteliale completo con migliori funzioni fisiologichespecifiche 2.
Numerosi studi hanno contribuito allo sviluppo di metodi per formare questi organoidi epiteliali in matrici naturali. Ciò ha permesso di ricapitolare le interazioni cellula-cellula e cellula-microambiente in vivo, per ottenere la creazione e la stabilità del fenotipo epiteliale3,4,5,6,7. Recentemente, e in particolare con l'obiettivo di sviluppare organoidi trapiantabili e decifrare il requisito del microambiente per orchestrare il programma epiteliale, sono stati sviluppati idrogel sintetici per migliorare la formazione di acini epiteliali8,9,10. Sfortunatamente, questi studi riportano dati qualitativi o presentano metodi di calcolo utilizzando riferimenti interni come il rapporto tra cisti e non cisti in un piano 2D8,9,10. Ciò impedisce qualsiasi confronto tra diversi studi in termini di efficienza, stabilità o caratterizzazione morfologica e fisiologica degli organoidi epiteliali.
La microincapsulazione delle cellule epiteliali nelle perline che utilizzano dispositivi microfluidici ha permesso risultati quantitativi e comparativi più realistici. Utilizzando questa tecnologia, gli organoidi di vari tipi di cellule sono stati formati e differenziati in base alla morfologia tra le diverse strutture cellulari 3D11,12. Tuttavia, questa tecnologia non è facile da lavorare e richiede l'uso di camere pulite per produrre i dispositivi microfluidici. Questa tecnologia è stata stabilita per alcuni tipi di idrogel, ma richiede l'applicazione dell'adattamento tecnico ad altri idrogel, limitandone la versatilità. Pertanto, la maggior parte degli studi destinati a sviluppare organoidi epiteliali si basano sull'incorporamento di cellule epiteliali in una massa di idrogel. In questi metodi, l'elevata eterogeneità della strutturazione del gel e della distribuzione cellulare all'interno dell'intera coltura 3D è spesso trascurata. Pertanto, la maggior parte delle analisi si riferisce a singole immagini 2D, che rappresentano solo molto approssimativamente la distribuzione dei vari oggetti cellulari nell'intero volume 3D.
Le malattie che colpiscono i dotti biliari, come il colanocarcinoma, l'atresia biliare, la colangite sclerosi primaria, tra gli altri, sono una delle principali cause di mortalità e morbilità. Ad eccezione del trapianto di fegato, non ci sono trattamenti efficaci per queste condizioni13. Gli sforzi per indagare la formazione del condotto biliare, le cause della malattia e la progressione consentiranno lo sviluppo di nuoveterapie 14.
Sono stati sviluppati modelli organotipici biliari di cisti, sferoidi o strutture tube-like utilizzando linee cellulari conlangiociti normali o derivate dal paziente, differenziate o derivate da progenitore15,16,17,18,19,20. Vari studi hanno riassunto la polarità dei corilangiociti, l'espressione dei marcatori di conlangiociti, la presenza di ciglia, la secrezione di coriandola e capacità riassorbitiva, la formazione e l'ostruzione del lume; che rappresentano tutte importanti caratteristiche del fenotipo, della morfologia e della funzione15,17,19. Altri hanno riportato il mantenimento di organoidi biliari derivati dal paziente per lunghi periodi di tempo20. Recentemente, abbiamo studiato il ruolo degli spunti biochimici e biofisici sulla cisti biliare organogenesi21. È importante sottolineare che la patogenesi dell'atresia biliare è stata studiata in sferoidi biliarie tubi 7,22. Inoltre, le caratteristiche chiave della colangite sclerosante primaria come la senescenza dei corilangiociti, la secrezione di citochine pro-infiammatorie e il reclutamento di macrofagi sono state studiate con successo utilizzando sferoidi biliari15,20. Tuttavia, sono ancora necessari modelli quantitativi 3D riproducibili in vitro che modulano fisiologicamente il fenotipo, la fisiologia e il microambiente del conlangiocita in vitro in cui queste domande possono essere affrontate. Inoltre, solo poche pubblicazioni hanno riportato l'efficienza della formazione dicisti 21,23. Questo è un punto importante da stabilire, in particolare quando si studia l'organogenesi, la causa della malattia e la correlazione delle risposte dei farmaci con la funzione e la polarizzazione dei conlangiociti. Inoltre, con differenze di impalcatura/matrice utilizzate da protocollo a protocollo, è difficile da confrontare tra i sistemi. Per risolvere questi problemi, proponiamo un metodo quantitativo, affidabile e universale per generare cisti biliari che imitano la formazione di lume, la polarizzazione dei colinogiociti e la funzione secretoria dei conlangiociti. È importante sottolineare che presentiamo un'analisi sistematica eseguita lungo l'asse Z attraverso il gel 3D quando si valuta nel tempo, efficienza di formazione della cisti, dimensioni, vitalità, polarizzazione e funzionalità. Inoltre, abbiamo usato un idrogel naturale e normali conlangiociti di ratto (NRC), come esempio per il protocollo, ma altri idrogel naturali o sintetici, così come le cellule epiteliali potrebbero essere utilizzati per la formazione di strutture cistiche 3D.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Generazione di cisti
NOTA: Questo protocollo può essere eseguito con qualsiasi tipo di idrogel, se la gelazione consente l'incorporamento di cellule.
- Rivestimento idrogel
NOTA: Un corretto rivestimento idrogel dello scivolo della camera è un passaggio critico per evitare la formazione di strati cellulari 2D sul fondo del pozzo, che potrebbe interferire con la successiva imaging della cisti e compromettere il calcolo dell'efficienza di formazione della cisti.- Per garantire l'omogeneità della soluzione di gel, scongelare l'idrogel a 4 °C durante la notte (O/N).
- Punte di pipetta precool su ghiaccio o O/N a -20 °C e uno scivolo a camera a 8 pozzi a -20 °C O/N.
- Posizionare l'idrogel e lo scivolo da camera a 8 pozzi su un secchio di ghiaccio pieno di ghiaccio.
- In un tubo conico da 15 ml, preparare una soluzione contenente il 40% di idrogel (V/V) in mezzo completo NRC freddo (vedi Tabella dei materiali)e posizionare il tubo sul ghiaccio.
- Per rivestire uno scivolo a camera, utilizzando punte di pipetta fredda, aggiungere 50 μL di soluzione di idrogel al centro di ogni pozzo e stendere su tutta la superficie utilizzando una punta di pipetta, tenendo la camera scivolare sul ghiaccio(Figura 1A).
NOTA: Stendere la soluzione di idrogel nel modo più uniforme possibile evitando bolle. - Per polimerizzare l'idrogel, incubare lo scivolo della camera per almeno 15 min a 37 °C, 5% CO2.
- Preparazione cellulare
- Riscaldare il mezzo completo NRC, la salina tamponata dal fosfato (PBS) e l'acido tetraacetico tripside-etilenediammina (tripsina-EDTA) in un bagno d'acqua preriscaldato a 37 °C.
- Mentre l'idrogel è polimerizzante, assicurarsi che i NFC vengano coltivati al 70% diconfluenza in un pallone rivestito di collagene T-25 cm21 . Lavare le celle una volta con 1x PBS preriscaldato.
- Incubare gli NFC con 5 ml di PBS 1x preriscaldato (per un pallone T-25 cm2) per 20 min a 37 °C, 5% CO2.
NOTA: Questo passaggio, che riduce il tempo di incubazione con tripina-EDTA, è fondamentale per mantenere le proprietà auto-organizzanti delle cellule. - Scartare il PBS, aggiungere 1 mL di tripside-EDTA e incubare per 5-10 min a 37 °C, 5% CO2.
- Neutralizzare con 4 mL di mezzo NRC completo preriscaldato. Raccogliere e trasferire la sospensione cellulare in un tubo conico da 15 ml e ruotare a 150 x g per 4 min.
- Scartare il mezzo e rimescolare il pellet cellulare in 5 mL di mezzo preriscaldato.
- Utilizzando un filtro cellulare da 40 μm, filtrare la soluzione cellulare in un tubo conico da 50 ml e contare le cellule.
NOTA: Passare le cellule attraverso un colino è un passaggio critico affinché i risultati quantitativi siano riproducibili, cioè per ottenere dimensioni quasi simili di aggregati cellulari da incastre.
- Incorporamento della sospensione cellulare in soluzione di idrogel
- Preparare una soluzione di 1.600 μL di idrogel all'80% (V/V) in mezzo NRC completo a freddo (tubo 1); tenere nel ghiaccio. Diluire 5 x 105 celle/ml in 1.600 μL di mezzo NRC completo a freddo (tubo 2) e tenerlo nel ghiaccio.
NOTA: Questo passaggio deve essere eseguito rapidamente per evitare la polimerizzazione dell'idrogel mescolandolo con la sospensione cellulare e per mantenere la vitalità cellulare. - Per preparare una soluzione di semina cellulare di 2,5 x 105 celle/ml in idrogel al 40% (V/V), mescolare il tubo 1 e il tubo 2. Aggiungere 400 μL della soluzione cellulare in ogni pozzo dello scivolo della camera rivestito di idrogel evitando bolle(Figura 1B).
- Tenere la camera scivolare in un incubatore a 37 °C con 5% DI CO2 fino a quando i supporti cambiano.
- Dopo 2 giorni in coltura, rimuovere 250 μL del mezzo da un angolo di ogni pozzo, fare attenzione a non pipettare l'idrogel. Quindi, aggiungere lentamente 250 μL del mezzo di coltura. Cambia il mezzo ogni 2 giorni.
NOTA: Ridurre al minimo il movimento dello scivolo della camera, in particolare durante l'inizio della cisti.
- Preparare una soluzione di 1.600 μL di idrogel all'80% (V/V) in mezzo NRC completo a freddo (tubo 1); tenere nel ghiaccio. Diluire 5 x 105 celle/ml in 1.600 μL di mezzo NRC completo a freddo (tubo 2) e tenerlo nel ghiaccio.
2. Quantificazione delle cisti
- Imaging cisti
NOTA: Questa sezione deve essere eseguita rapidamente per non compromettere la vitalità cellulare se il microscopio non è dotato di una camera di riscaldamento per controllare la CO2 e la temperatura. Al fine di garantire una quantificazione coerente, rappresentativa della distribuzione della cisti nell'intero volume dell'idrogel, le cisti sono immagini tramite microscopia a contrasto di fase e imaging seriale (Z-stack), con parametri predefiniti in diversi punti di tempo.- Prendere una pila Z lungo la profondità dell'idrogel per ogni punto di tempo (Figura 1C, D). In questo esempio, gli stack Z vengono presi ai giorni 1, 2, 4, 7 e 10.
NOTA: Verificare che la distribuzione iniziale delle cellule sia uniforme nell'idrogel per garantire l'applicabilità di questo metodo.- Con un microscopio a contrasto di fase dotato di un software di acquisizione di immagini, selezionare l'ingrandimento obiettivo 10x nella finestra manuale del nasello(Figura 2B(1)).
- Accendere la lampada bianca e selezionare l'opzione di imaging brightfield.
- Accendere la fotocamera selezionando il pulsante "Riproduci" nel sottomenu della barra. Concentrarsi su un campo di cisti e impostare il tempo di esposizione (Figura 2B(2)). Aprire la finestra Cartella acquisizione automatica per un salvataggio automatico delle immagini ( Figura2B(3)).
- Aprite la finestra della serie Z di acquisizione e definite con la vite Z i piani superiore e inferiore dello Z-stack (stesse coordinate XY ma diverse Z schermate). Regolare il passo Z in base all'obiettivo, al livello di risoluzione e premere il pulsante "Esegui ora" per avviare l'acquisizione (Figura 2B(4)).
NOTA: In questo esempio, le cisti sono distribuite su uno spessore dell'idrogel di 520 μm. A seconda dell'obiettivo, il passo Z deve essere regolato per non perdere alcuna cisti e per garantire il rilevamento di singole cellule e aggregati. - Prendi almeno 3 Z-stack non sovrapposti per pozzo.
NOTA: Questo campionamento è necessario quando, come in questo esempio, le cisti sono più numerose nella profondità del gel che sui bordi a causa di eterogeneità nella polimerizzazione dell'idrogel. - Per fare in modo che un set di dati rappresentativo ripeta il passaggio 2.1.1.5. per 3 pozzi in totale.
NOTA: La distribuzione eterogenea delle cisti dipende dal tipo di idrogel, dalla sua polimerizzazione e dalla linea cellulare. Considerando tre Z-stack per pozzo e tre pozzi per esperimento, un minimo di 200 cisti sono immagini su nove Z-stack per caratterizzare la formazione di cisti e la crescita della cisti in ogni punto di tempo.
- Prendere una pila Z lungo la profondità dell'idrogel per ogni punto di tempo (Figura 1C, D). In questo esempio, gli stack Z vengono presi ai giorni 1, 2, 4, 7 e 10.
- Elaborazione delle immagini
NOTA: In un idrogel, gli NFC possono essere trovati come singole cellule, cisti o aggregati. Le cisti sono identificate dalla presenza di un guscio di cella rotondo e sottile a contrasto che racchiude un lume, mentre gli aggregati cellulari presentano una forma irregolare e non hanno un lume. Gli aggregati e le singole cellule hanno un aspetto denso e contrastato (Figura 3B(4)).- Aprire il software Fiji, aprire lo Z-stack e scegliere File | Apri (Figura complementare 1) dal menu Figi. Selezionare lo Z-stack da analizzare. Se necessario, selezionare l'opzione "Stack virtuale" e fare clic su "Sì" per l'apertura (Figura 3A(1)).
- Duplicare lo stack tramite Image | Duplicato. Fare clic sulla casella "Stack duplicato" e scegliere "OK" (Supplementary Figure 2).
NOTA: in questo esempio, gli stack Z sono in formato nd2 codificati in 16 bit. - Create una proiezione di intensità minima dalla pila duplicata. Passare al menu Immagine | Proprietà Stacks | Progetto Z. Selezionate il tipo di proiezione "Intensità min" e fate clic su "OK" (Figura 3A(2)) (Figura complementare 3).
- Sottrarre lo sfondo dalla proiezione. Passare al menu Processo | Sottrarre lo sfondo. Digitare 500,0 pixel di raggio della palla rotolante e fare clic su "sfondo chiaro" per rendere le cisti più contrastate rispetto allo sfondo (Figura 3A(3)) (Figura complementare 4).
NOTA: Il raggio della palla rotolante definisce la dimensione della regione su cui viene operata la sottrazione di fondo. Questo parametro deve essere impostato sulle dimensioni dell'oggetto più grande da identificare. - Se è necessario un miglioramento del contrasto, passare al menu Immagine | Regola | Luminosità/Contrasto | Proprietà Auto | Applicare. Fiji ottimizzerà automaticamente luminosità e contrasto. Nella(figura 3A, paragrafo 3),i valori grigio inferiore e superiore sono stati impostati rispettivamente su 49702 e 65452(figura complementare 5).
NOTA: se la proiezione non è calibrata, vai al menu Analizza | Impostare la scala e digitare il rapporto μm/pixel di calibrazione corrispondente (Figura complementare 6).
- Misurazione del conteggio delle cisti e delle dimensioni delle cisti
- Per misurare il diametro approssimativo della cisti, selezionate lo strumento Linea retta (Straight-line) nel menu Figi e disegnate una linea attraverso il diametro di ciascuna cisti sulla proiezione finale (Figura 3B(4)). Aggiungi la nuova regione di interesse (ROI) creata per ogni cisti al gestore del ROI: premi lascorciatoia" t " sulla tastiera per un conteggio e un'apertura più rapidi del ROI manager. Fate clicsu " Mostratutto " per visualizzare le cisti contate (Figura complementare 7)
- Verificare che non sia stata lasciata alcuna cisti senza contare sovrapponendo le ROM impostate dalla proiezione sullo stack Z. A tal fine, fare clic sulla finestra Z-stack per selezionarla. In Gestione ROI fare clic su "Mostra tutto" e spostare il cursore lungo lo Z-stack per verificare che l'immagine per immagine, tutte le cisti siano state conteggiate (Figura complementare 8).
- Una volta contate le nuove cisti e aggiunte le ROI al passaggio 2.3.1., selezionare il set di ROI e salvarlo tramite la finestra gestione ROI facendo clic su Altro | Risparmio (figura complementare 9).
- Selezionate tutte le ROI in Roi Manager e fate clic su "Misura" in Roi Manager per ottenere le dimensioni di ogni cisti. Questo aprirà una nuova finestra di misurazioni chiamata "Risultati" numerando ogni cisti e le sue dimensioni stimate. Quindi salvare in .csv formato facendo clic sulla finestra "Risultati" e tramite il menu: File | Salva con nome (figura complementare 10).
NOTA: è possibile creare una macro per elaborare in modo semi-automatico gli stack, stimare il numero/le dimensioni della cisti dalle proiezioni e archiviare i dati per una procedura di conteggio più rapida. A tal fine, selezionare lo strumento "Registra" nel menu della barra, facendo clic su Plugin | Macro | Record.
- Quantificazione dell'efficienza di formazione delle cisti
- Contare il numero di cisti al giorno Y,
 su una proiezione (Y=1, 2, 4, 7 o 10).
su una proiezione (Y=1, 2, 4, 7 o 10). - Per calcolare l'efficienza di formazione della cisti per 1000 cellule al giorno Y, dividere il numero di cisti contate in quel momento per il numero di cellule sementi al giorno 0 dedotte dal volume dell'idrogel e moltiplicare per 1000 (Figura 3C, Figura 4).
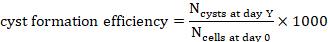
- Contare il numero di cisti al giorno Y,
3. Vitalità cellulare
- Preparare una soluzione stock di diacetato di fluoresceina (FDA) a 5 mg/mL sciogliendo 5 mg di FDA in acetone da 1 mL e conservare a -20 °C.
- Preparare una soluzione stock di ioduro di propidio (PI) ad una concentrazione di 2 mg/mL in acqua deionizzata (dH2O) e conservare a 4 °C.
- Preparare il mezzo NRC senza siero fetale di vitello (FCS).
- Per preparare la soluzione di colorazione FDA/PI, aggiungere 4 μL di soluzione stock FDA (concentrazione finale di 8 μg/mL) e 25 μL di soluzione di serie PI (concentrazione finale di 20 μg/mL) in 2,5 mL di mezzo NRC senza FCS.
- Rimuovere il mezzo dallo scivolo della camera, aggiungere 250 μL di soluzione di colorazione in ogni pozzo e incubare 4-5 min al buio a 37 °C, 5% CO2. Pipettare accuratamente la soluzione di colorazione e lavare una volta con 250 μL di 1x PBS.
- Aggiungere con cura 250 μL di mezzo NRC completo a ciascun pozzo e scattare foto utilizzando un microscopio a fluorescenza invertito con filtri FITC (Texas red and fluorescein isothiocyanate). Le cellule vive saranno verdi e le cellule morte saranno rosse (Figura 5A).
NOTA: Per quantificare le cellule vive/morte, prendere le pile Z attraverso il volume dell'idrogel dopo il passaggio 2 e adattare il metodo di elaborazione delle immagini per la fluorescenza.
4. Attività di secrezione
NOTA: L'attività di secrezione attraverso la membrana apicale dei conlangiociti è valutata dalla secrezione di fluoresceina nel lume. La sua specificità può essere valutata facendo lo stesso test con Verapamil, un inibitore del trasportatore multi-farmaco (MDR)24.
- Per preparare una soluzione di colorazione di Hoechst 33258 a 5 μg/mL, aggiungere 0,83 μL di soluzione di stock di Hoechst (concentrazione di 15 mg/mL in dH2O) in 2,5 mL di mezzo NRC senza FCS.
- Aggiungere 250 μL di soluzione di Hoechst in ogni pozzo e incubare a 37 °C, 5% CO2 per 15 min.
- Rimuovere la soluzione hoechst e aggiungere 250 μL di soluzione FDA (concentrazione finale di 8 μg/mL) in ogni pozzo. Incubare 4-5 min a 37 °C, 5% CO2.
NOTA: Non appena le cellule sono esposte alla soluzione di colorazione FDA, il follow-up della cinetica di secrezione di fluoresceina potrebbe essere utile per calibrare il tempo necessario affinché le cisti si secernono. Per fare ciò, scatta foto ogni minuto per 1 h tramite l'imaging time-lapse. In questo esempio, il tempo necessario per osservare le cisti secrezioni NRC nell'idrogel è di circa 15-20 minuti. - Scatta immagini utilizzando un microscopio a fluorescenza invertito 5 minuti dopo il risciacquo con mezzo senza FCS. Utilizzare filtri 4′,6-diamidino-2-fenilindolo (DAPI) e FITC per rivelare l'etichettatura dei nuclei e l'accumulo di fluoresceina nel lume(Figura 6A). Per quantificare il numero di cisti di secerti, prendi gli stack Z come nel passaggio 2 e adatta i passaggi di elaborazione delle immagini alle immagini fluorescenti.
NOTA: Per il test verapamil, precedere il processo precedente (fasi da 4.3. a 4.4.) con un'incubazione con Verapamil, secondo le seguenti condizioni: - Preparare una soluzione stock di Verapamil da 10 mM in solfossido di dimetile (DMSO). Per preparare una soluzione di lavoro da 10 μM, mescolare 2,5 μL di soluzione stock verapamil con mezzo di coltura da 2,5 mL senza FCS.
- Per valutare che la fluorescenza nel lume è il risultato della secrezione di MDR, prendere un altro scivolo e aggiungere 250 μL di soluzione di lavoro Verapamil in ogni pozzo e incubare 20 min a 37°C, 5% CO2
- Rimuovere la soluzione e aggiungere 250 μL di soluzione FDA (concentrazione finale di 8 μg/mL) in ogni pozzo. Incubare 4-5 min al buio a 37 °C, 5% CO2. Quindi lavare con 250 μL di 1x PBS, prima dell'imaging(Figura 6B, C).
5. Valutazione della polarità epiteliale mediante immunofluorescenza
- Per preparare la soluzione di fissaggio, mescolare il 4% di formaldeide con il 5% di saccarosio, in 1x PBS, pH 7,4 e incubare in un bagno d'acqua preriscaldato a 37 °C.
- Per fissare le cellule, pipettare delicatamente il mezzo di coltura dal pozzo senza danneggiare la matrice. Aggiungere lentamente 400 μL della soluzione di fissaggio sul lato dei pozzi. Incubare per 20 minuti a temperatura ambiente (RT).
NOTA: Lasciare sempre 25 μL del liquido sopra la matrice per prevenirne il danno. - Rimuovere delicatamente la soluzione di fissaggio e lavare 3 volte con 400 μL di 1x PBS a (RT).
- Pipettare il PBS, aggiungere 400 μL di soluzione di permeabilizzazione (0,5% Tritone X-100 in 1x PBS) e incubare 10 min a RT.
- Rimuovere delicatamente la soluzione di permeabilità, seguita da 3 lavaggi rapidi con 400 μL di 1x PBS e una lunga fase di lavaggio di 30 min a RT.
NOTA: In questo passaggio, la diapositiva può essere conservata a 4 °C per 2 giorni. In questo caso, sigillare lo scivolo con un film di paraffina per evitare l'evaporazione e l'essiccazione a matrice. - Rimuovere il PBS, aggiungere 400 μL di soluzione di blocco contenente lo 0,1% di albumina di siero bovino (BSA) e il 10% di siero di capra in 1x PBS e incubare per 60 minuti a RT.
ATTENZIONE: Concentrazioni di BSA superiori a 0,1% si tradurranno in retrazione del lume e ulteriore collasso della cisti (cfr. sezione Risultati rappresentativi)(Figura 7A). - Pipettare la soluzione di blocco e lavare una volta con 400 μL di PBS/0,05% Tween 20 e scartare.
- Aggiungere 150 μL della soluzione anticorpale, ad esempio anticorpo E-cadherina diluito 1:400 e Phalloidin 568 (concentrazione finale di 16,2 nM) in 1x PBS e incubare per 90 minuti a RT.
NOTA: Questa diluizione di E-cadherina è la stessa utilizzata in un protocollo standard di immunofluorescenza 2D. - Lavare il campione con 400 μL di PBS/0,05% Tween 20, 3x; ogni volta che si incuba il campione per 10 minuti a RT.
- Aggiungere 150 μL dell'anticorpo secondario (capra anti-coniglio IgG Alexa Fluor Plus 647), diluito 1:500 in 1x PBS e incubare 60 min a RT.
- Lavare 3 volte con 400 μL di PBS/0,05% Tween 20, ogni volta incubando il campione per 10 minuti a RT.
- Lavare 3 volte con 400 μL di 1x PBS, ogni volta incubando il campione per 10 minuti a RT.
- Scartare il PBS dell'ultimo lavaggio e preparare la diapositiva della camera per la visualizzazione tramite microscopia confocale seguendo una delle due opzioni seguenti.
- Aggiungere 400 μL di 1x PBS e 50 μL di DAPI per pozzo. I campioni possono essere esaminati attraverso il fondo del pozzo senza la necessità di montare con un coverslip (Figura 7B).
- Aggiungere 100 μL per pozzo di reagente antifade contenente DAPI e lasciare asciugare lo scivolo O/N a RT.
Access restricted. Please log in or start a trial to view this content.
Risultati
Formazione e caratterizzazione delle cisti
I sistemi di coltura cellulare 3D sono uno strumento importante per studiare l'organogenesi e la modellazione dellemalattie 25. Sfortunatamente, la maggior parte di questi metodi sono qualitativi o utilizzano la quantificazione interna eseguita su un singolo piano confrontando il numero di cisti rispetto alle non cisti, in volumi variabili e spesso non specificati, impedendo qualsiasi confronto in termini di efficienza di formazione de...
Access restricted. Please log in or start a trial to view this content.
Discussione
Al fine di studiare l'organogenesi e il mantenimento delle strutture cellulari 3D, sono stati modellati vari tessuti, utilizzando diverse origini cellulari ma anche diversi tipi di matrici extracellulari tra cui idrogel sintetici8,9,10,21. Tuttavia, a causa della mancanza di analisi quantitativa 3D che consente confronti tra metodi in termini di formazione di organoidi o funzionalità
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo il Dr. Nicholas LaRusso (Mayo Clinic, Rochester, Minnesota, Stati Uniti), che ha gentilmente fornito la linea cellulare NRC.
Questo lavoro ricevette il sostegno finanziario sia del programma iLite RHU (sovvenzione ANR-16-RHUS-0005) che del DHU Hepatinov.
Ringraziamo Isabelle Garcin e Réseau d'Imagerie Cellulaire Paris Saclay per il loro supporto all'imaging.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 10 µl- Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000020 | |
| 100 µl - Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000046 | |
| 1000 µl - Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000062 | |
| 1X PBS | Thermo Fisher Scientific | 14190-094 | |
| 200 µl - Pipette Eppendorf Research Plus | Thermo Fisher Scientific | 3120000054 | |
| 3,3′,5-Triiodo-L-thyronine sodium salt | Sigma-Aldrich | T5516 | NRC complete medium final concentration = 3.4 µg/mL |
| Acetic acid | VWR | 20104-298 | 0.02N final |
| Aerosol barrier pipettes tips 10 µl (Fisherbrand) | Thermo Fisher Scientific | 2707439 | |
| Aerosol barrier pipettes tips 1000 µl (Fisherbrand) | Thermo Fisher Scientific | 2707404 | |
| Aerosol barrier pipettes tips 200 µl (Fisherbrand) | Thermo Fisher Scientific | 2707430 | |
| Antibiotic Antimicotic Solution (100X) | Sigma-Aldrich | A5955 | NRC complete medium final concentration = 1:100 dilution |
| Bovine pituitary extract | Thermo Fisher Scientific | 13028-014 | NRC complete medium final concentration = 30 µg/mL |
| Bovine serum albumin | Sigma-Aldrich | A2153 | 1:1000 dilution |
| Chemically Defined Lipid Concentrate (100X) | Thermo Fisher Scientific | 11905-031 | NRC complete medium final concentration = 1:100 dilution |
| Collagen high concentration, rat tail | Thermo Fisher Scientific | 354249 | 50 µg/mL final concentration |
| Dexamethasone | Sigma-Aldrich | D4902 | NRC complete medium final concentration = 0.393 µg/mL |
| DMEM F12 | Thermo Fisher Scientific | 21331-020 | NRC complete medium final concentration = 1X |
| E-cadherin Rabbit anti-Human, Rat, Polyclonal | Thermo Fisher Scientific | PA5-32178 | 1:400 dilution |
| Eclipse TE300 inverted microscope | Nikon | imaging | |
| Ethanolamine | Sigma-Aldrich | E9508 | NRC complete medium final concentration = 0.32 mM |
| Fetal calf serum | Thermo Fisher Scientific | 10270-106 | NRC complete medium final concentration = 5:100 dilution |
| Fluoroshield with DAPI (Mounting medium) | Sigma-Aldrich | F6057 | |
| Formaldehyde 16% (W/V) | Thermo Fisher Scientific | 28906 | 4% (W/V) |
| Goat serum | Thermo Fisher Scientific | 16210-064 | 1:10 dilution |
| Hamamatsu camera (Digital camera C11440 ORCA - flash 4.OLT) | Hamamatsu | imaging | |
| Hoechst 33258 | Sigma-Aldrich | B1155 | 5 µg/mL final concentration |
| IgG (H+L) Highly Cross-Adsorbed Goat anti-Rabbit, Alexa Fluor Plus 647 | Thermo Fisher Scientific | A32733 | 1:500 dilution |
| ImageJ version 2.0.0-rc-69/1.52n | Open source image processing software | ||
| Insulin-Transferrin-Selenium (100X) | Thermo Fisher Scientific | 51300-044 | NRC complete medium final concentration = 1:100 dilution |
| L-Glutamine (100X) | Thermo Fisher Scientific | 25030-024 | NRC complete medium final concentration = 1:100 dilution |
| Matrigel GFR (stock concentration 9.7 mg/mL) | Thermo Fisher Scientific | 356231 | 4:10 dilution |
| NIS Elements software version 4.50.00 | Nikon | image acquisition and display | |
| Non-Essential-Amino-Acids-Solution (100X) | Thermo Fisher Scientific | 11140-035 | NRC complete medium final concentration = 1:100 dilution |
| Objective Plan Fluor 10X/0.30 Ph1 DL (∞/1.2 WD 15.2) | Nikon | ||
| Prolong Gold Antifade Reagent | Thermo Fisher Scientific | P36931 | |
| Propidium Iodide (PI) | Sigma-Aldrich | P4170 | 20 µg/mL final concentration |
| Rhodamine Phalloidin | Thermo Fisher Scientific | R415 | 16.2 nM final concentration |
| Sir-Actin / Verapamil kit | Spirochrome | SC001 | 10 µM final concentration |
| Soybean trypsin inhibitor | Thermo Fisher Scientific | 17075-029 | NRC complete medium final concentration = 50 µg/mL |
| Sterile cell strainer 40 µm (Fisherbrand) | Thermo Fisher Scientific | 22363547 | |
| Sterile pipettes 10 mL (Fisherbrand) | Thermo Fisher Scientific | 1367811E | |
| Sterile pipettes 5 mL (Fisherbrand) | Thermo Fisher Scientific | 1367811D | |
| Sterile tubes 1.5 mL (Fisherbrand) | Thermo Fisher Scientific | 11926955 | |
| Sterile tubes 15 mL (Fisherbrand) | Thermo Fisher Scientific | 7200886 | |
| Sterile tubes 50 mL (Fisherbrand) | Thermo Fisher Scientific | 553913 | |
| Sucrose | Sigma-Aldrich | S0389 | 5:100 dilution |
| Tissue culture treated flask 25cm2 (Falcon) | Thermo Fisher Scientific | 353108 | |
| Triton X-100 | Sigma-Aldrich | T8787 | 5:1000 dilution |
| Trypsin-EDTA (0.05%) phenol red | Thermo Fisher Scientific | 25300-054 | 1X |
| Tween-20 | Sigma-Aldrich | P1379 | 5:10000 dilution |
| Vitamin (100X) | Thermo Fisher Scientific | 11120-037 | NRC complete medium final concentration = 1:100 dilution |
| μ-Slide 8 Well ibiTreat, Ibidi | Clinisciences | 80826 |
Riferimenti
- Edmondson, R., Broglie, J. J., Adcock, F., Yang, L. Three-Dimensional Cell Culture Systems and Their Applications in Drug Discovery and Cell-Based Biosensors. ASSAY and Drug Development Technologies. 12 (4), 207-218 (2014).
- Martín-Belmonte, F., et al. Cell-polarity dynamics controls the mechanism of lumen formation in epithelial morphogenesis. Current Biology. 18, 507-513 (2008).
- Debnath, J., Muthuswamy, S. K., Brugge, J. Morphogenesis and oncogenesis of MCF-10A mammary epithelial acini grown in three-dimensional basement membrane cultures. Methods. 30 (3), 256-268 (2003).
- Artym, V. V., Matsumoto, K. Imaging Cells in Three-Dimensional Collagen Matrix. Current Procotols in Cell Biology. , Chapter 10 (Unit 10) (2010).
- Petersen, O. W., Ronnov-Jessen, L., Howlett, A. R., Bisell, M. J. Interaction with basement membrane serves to rapidly distinguish growth and differentiation pattern of normal and malignant human breast epithelial cells. Proceedings of the National Academy of Sciences of the United States of America. 89, 9064-9068 (1992).
- Kim, S. P., Lee, D. H., Park, J. K. Development of hepatocyte spheroids immobilization technique using alternative encapsulation method. Biotechnology and Bioprocess Engineering. 3, 96-102 (1998).
- Lorent, K., et al. Identification of a plant isoflavonoid that causes biliary atresia. Science Translational Medicine. 7 (286), 67(2015).
- Nowak, M., Freudenberga, U., Tsurkana, M. V., Wernera, C., Levental, K. R. Modular GAG-matrices to promote mammary epithelial morphogenesis in vitro. Biomaterials. 112, 20-30 (2017).
- Miroshnikova, Y. A., et al. Engineering Strategies to Recapitulate Epithelial Morphogenesis Within Synthetic Three-Dimensional Extracellular Matrix With Tunable Mechanical Properties. Physical Biology. 8 (2), 026013(2011).
- Ozdemir, T., et al. Tuning Hydrogel Properties to Promote the Assembly of Salivary Gland Spheroids in 3D. ACS Biomaterials Science & Engineering. 2 (12), 2217-2230 (2016).
- Dolega, M. E., Abeille, F., Picollet-D'hahan, N., Gidrol, X. Controlled 3D culture in Matrigel microbeads to analyze clonal acinar development. Biomaterials. 52, 347-357 (2015).
- Laperrousaz, B., et al. Direct transfection of clonal organoids in Matrigel microbeads: a promising approach toward organoid-based genetic screens. Nucleic Acids Research. 46 (12), 70(2018).
- Lazaridis, K. N., LaRusso, N. F. The Cholangiopathies. Mayo Clinic Proceedings. 90 (6), 791-800 (2015).
- Tam, P. K., Yiua, R. S., Lendahl, U., Andersson, E. R. Cholangiopathies - Towards a molecular understanding. EBioMedicine. 35, 381-393 (2018).
- Loarca, L., et al. Development and characterization of cholangioids from normal and diseased human cholangiocytes as an in vitro model to study primary sclerosing cholangitis. Laboratory Investigation. 97, 1385-1396 (2017).
- De Assuncao, T. M., Jalan-Sakrikar, N., Huebert, R. C. Regenerative medicine and the biliary tree. Seminars in Liver Disease. 37, 17-27 (2017).
- Dianat, N. H., et al. Generation of functional cholangiocyte-like cells from human pluripotent stem cells and HepaRG cells. Hepatology. 60, 700-714 (2014).
- Masyuk, A. I., et al. Cholangiocyte autophagy contributes to hepatic cystogenesis in polycystic liver disease and represents a potential therapeutic target. Hepatology. 67 (3), 1088-1108 (2018).
- Sampaziotis, F., Cardoso, M., Madrigal, P., Bertero, A., Saeb-Parsy, K., et al. Cholangiocytes derived from human induced pluripotent stem cells for disease modeling and drug validation. Nature Biotechnology. 33 (8), 845-852 (2015).
- Soroka, J. C., et al. Bile-Derived Organoids From Patients With Primary Sclerosing Cholangitis Recapitulate Their Inflammatory Immune Profile. Hepatology. 70 (3), 871-882 (2019).
- Funfak, F., et al. Biophysical Control of Bile Duct Epithelial Morphogenesis in Natural and Synthetic Scaffolds. Frontiers in Bioengineering and Biotechnology. 7 (417), 417(2019).
- Du, Y., et al. Bile Duct-on-a-Chip With Organ-Level Functions. Hepatology. 0 (0), (2019).
- Shiota, J. M., Mohamad Zaki, N. H., Merchant, J. L., Samuelson, L. C., Razumilava, N. Generation of Organoids from Mouse Extrahepatic Bile Ducts. Journal of Visualized Experiments. (146), e59544(2019).
- Bircsak, K. M., Richardson, J. R., Aleksunes, L. M. Inhibition of Human MDR1 and BCRP Transporter ATPase Activity by Organochlorine and Pyrethroid Insecticides. Journal of Biochemical and Molecular Toxicology. 27 (2), 157-164 (2013).
- Fennema, E., Rivron, N., Rouwkema, J., Blitterswijk, C., Boer, J. Spheroid culture as a tool for creating 3D complex tissues. Trends in Biotechnology. 31 (2), 108-115 (2013).
- Kanade, S., Nataraj, G., Ubale, M., Mehta, P. Fluorescein Diacetate Vital Staining for Detecting Viability of Acid-Fast Bacilli in Patients on Antituberculosis Treatment. International Journal of Mycobacteriology. 5 (3), 294-298 (2016).
- Rieger, A. M., Nelson, K. L., Konowalchuk, J. D., Barreda, D. R. Modified Annexin V/Propidium Iodide Apoptosis Assay For Accurate Assessment of Cell Death. Journal of Visualized Experiments. (50), e2597(2011).
- Tabibian, J. H., Masyuk, A., Masyuk, T. V., O'Hara, S. P., LaRusso, N. F. Physiology of Cholangiocytes. Comprehensive Physiology. 3 (1), (2013).
- Spirlì, C., et al. Functional polarity of Na+/H+ and Cl-/HCO3- exchangers in a rat cholangiocyte cell line. American Journal Physiology. 275, 1236-1245 (1998).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon