Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Metilización Específica Multiplex Droplet PCR usando el Dispositivo Generador de Gotas de Polímero para Diagnóstico Hematológico
En este artículo
Resumen
Los marcadores epigenéticos se utilizan para la subtipificación de glóbulos blancos (CMB) mediante la cuantificación de los patrones de metilación del ADN. Este protocolo presenta un método de reacción en cadena de la polimerasa de gotas múltiples (mdPCR) utilizando un dispositivo microfluídico basado en elastómero termoplástico (TPE) para la generación de gotas que permite la cuantificación objetivo precisa y múltiplex específica de metilación de los recuentos diferenciales del CMB.
Resumen
Se describe un flujo de trabajo de PCR de gotas multiplexadas (mdPCR) y un protocolo detallado para determinar el recuento diferencial de glóbulos blancos basados en epigenéticos (WBC), junto con un dispositivo de generación de gotas microfluídicas de elastómero termoplástico (TPE). Los marcadores epigenéticos se utilizan para la subtipificación del CMB que tiene un importante valor pronóstico en diferentes enfermedades. Esto se logra mediante la cuantificación de los patrones de metilación del ADN de regiones específicas ricas en CG en el genoma (CpG loci). En este artículo, el ADN tratado con bisulfito de células mononucleares de sangre periférica (PBMC) se encapsula en gotas con reactivos mdPCR, incluyendo imprimaciones y sondas fluorescentes de hidrólisis específicas para loci CpG que se correlacionan con subpoblaciones WBC. El enfoque multiplex permite el interrogatorio de muchos loci de CpG sin necesidad de reacciones de mdPCR separadas, lo que permite una determinación paramétrica más precisa de las subpúmedas del CMB mediante el análisis epigenético de sitios de metilación. Esta cuantificación precisa se puede extender a diferentes aplicaciones y destaca los beneficios para el diagnóstico clínico y el pronóstico posterior.
Introducción
El análisis de la composición de los glóbulos blancos (CMB) se encuentra entre las pruebas de laboratorio solicitadas con mayor frecuencia en diagnóstico hematológico. El recuento diferencial de leucocitos sirve como un indicador para un espectro de enfermedades incluyendo infección, inflamación, anemia, y leucemia, y está bajo investigación como un biomarcador de pronóstico temprano para varias otras condiciones, así. El estándar de oro en la subtipificación del CMB implica inmunostaining y/o citometría de flujo, que requieren costosos anticuerpos fluorescentes propensos a la inestabilidad y a menudo dependen en gran medida de la competencia del operador en la preparación de muestras. Además, este método sólo se aplica a muestras de sangre fresca, de forma que las muestras no puedan congelarse para su envío o análisis posteriores.
Los marcadores epigenéticos han surgido recientemente como potentes herramientas analíticas para el estudio de las variaciones fenotípicas. Posteriormente, se ha demostrado que las poblaciones de leucocitos humanos tienen patrones de metilación del ADN del linaje celular que permiten la caracterización precisa de subconjuntos del CMB. El subtipo a base de marcadores epigenéticos proporciona una alternativa prometedora que no depende de la recolección de muestras de sangre fresca o anticuerpos caros y puede ser explotado como un biomarcador para el inicio de la enfermedad y la susceptibilidad1,2,3,4,5.
Se han realizado estudios en todo el genoma para el mapeo extensivo de regiones específicas ricas en CG metiladas en el genoma (islas CpG) en subtipos de leucocitos para identificar marcadores epigenéticos candidatos específicos de subtipos de leucocitos. Los protocolos PCR se han desarrollado debido a esta razón para las regiones genéticas metiladas, por ejemplo, CD3Z y FOXP3, correspondientes a CD3+ células T y CD4+ CD25+ células T reguladoras (T-Regs), respectivamente. Wiencke y otros han demostrado la utilidad de la PCR de gotas dúplex para el subtipado epigenético de células T, produciendo resultados que se correlacionan altamente con el análisis de clasificación celular activada por flujo (FACS)6. Este método de análisis genético cuantitativo se basa en la partición de las moléculas de ácido nucleico de la plantilla y los reactivos de PCR en miles de gotas discretas, definidas volumétricamente, de tamaño subnilóter que contienen cero, una o más copias de ácido nucleico objetivo, utilizando emulsiones de agua en aceite habilitadas por microfluídicos7,,8. La amplificación de PCR se realiza dentro de cada gota individual y se mide la intensidad de fluorescencia final de cada gota, lo que permite la cuantificación absoluta de los objetivos presentes en la muestra. Se ha establecido que la PCR de gotas es más precisa, precisa y técnicamente más simple que la qPCR estándar, lo que lo convierte en un método basado en metilación de ADN más favorable para la evaluación clínica de células T. Aunque se carece de una metodología de subtipado de rápida aparición, se carece de análisis epigenético multiplexado para sondear varias regiones de CpG metiladas simultáneamente. Esto es necesario para los recuentos diferenciales de leucocitos de rutina.
Aquí, se presenta un dispositivo microfluídico de gota de elastómero termoplástico (TPE) y se emplea para la PCR de gota multiplexa específica de metilación (mdPCR). La tecnología se ha utilizado para delinear subtipos específicos de leucocitos, CD3+ células T y CD4+ CD25+ T-Regs, basados en patrones de metilación del ADN de linaje celular, es decir, variación epigenética de las regiones CD3Z y FOXP3 CpG, respectivamente. Un protocolo detallado para la extracción de ADN, la conversión de bisulfito y mdPCR se describe en conjunto con un método de fabricación para un dispositivo de generación de gotas TPE. Los resultados representativos del método se comparan con los de la tinción de inmunofluorescencia que resalta la utilidad del enfoque propuesto.
Protocolo
Todos los experimentos realizados en este estudio con muestras humanas fueron aprobados por la Junta de ética del NRC y se realizaron de acuerdo con las políticas de NRC que rigen a los sujetos humanos que siguen las pautas de investigación aplicables y cumplen con las leyes de Quebec, Canadá.
1. Preparación celular
- Descongelar las células mononucleares de sangre periférica humana congelada (PBMC) inmediatamente colocando el criovial en un baño de agua a 37 oC durante 5 min.
- Invierta el criovial dos veces para resuspender suavemente las células y el uso de una pipeta de 1 ml transfiera la suspensión celular a un tubo cónico de 15 ml.
- Añadir 10 ml de medio de crecimiento precalentó (37 oC) complementado con suero bovino fetal al 10% (RPMI-1640 + 10% FBS) al tubo de 15 ml que contiene los PBMC.
- Centrifugar la suspensión celular en una centrífuga de cucharón oscilante a temperatura ambiente a una velocidad de 330 x g durante 10 minutos con aceleración rápida y el freno en alto.
- Una vez que el giro ha terminado, decantar cuidadosamente el sobrenadante. Resuspender el gránulo celular en 3 ml de solución salina tamponada con fosfato (PBS) pH 7.2, que contiene 2 mM de ácido etilendiaminetetraacético (EDTA) tocando el lado de los tubos.
- Mezcle las células invirtiendo el tubo con la tapa bien cerrada.
- Preparar dos microtubos de 1,5 ml y utilizar una pipeta alícuota de 1,5 ml de la suspensión celular en cada tubo, uno de los cuales se utiliza para la posterior tinción de inmunofluorescencia y otro para la extracción de ADN.
2. Protocolo de tinción por inmunofluorescencia e imágenes
- Células resuspendidas (de 1,7) en 200 ml de tampón PBS que contienen 0,1% de azida sódica y 2% FBS y ajustar la concentración final de la suspensión celular a un máximo de 2 x 107 células/ml.
- Divida la suspensión celular pipeteando un volumen de 100 l en dos microtubos separados de 1,5 ml.
- Añadir un volumen de 20 l de anta hu CD3/CD4 conjugado con isotiocianato de fluoresceína (FITC) y ficoerytrina (PE) a un tubo y anti-Hu CD4/CD25 conjugado con FITC y PE (ver Tabla de materiales)al segundo tubo, respectivamente.
- Agregue 1 gota de mancha de celda viva fluorescente azul (ver Tabla de materiales)a cada tubo.
- Incubar a temperatura ambiente con un rotador de tubo durante 2 h. Proteger de la luz.
- Centrifugar la suspensión celular a temperatura ambiente a 330 x g durante 10 minutos con aceleración rápida y el freno en alto.
- Decantar el sobrenadante y resuspender cuidadosamente el pellet celular tocando el tubo. Añadir 1 mL de PBS, pH 7.2 que contenga 2 mM EDTA. Asegurarse de que la tapa está bien cerrada, mezcle las células invirtiendo el tubo 2x.
- Repita los pasos 2.6 y 2.7 tres veces.
- Células resuspendidas en 20 l de pH PBS 7,2 que contienen 2 mM EDTA.
- Pipetear 10 l gota de la suspensión celular en un portaobjetos de borosilicato y esperar 2 minutos para que las células se sedimenten lentamente hasta el fondo de la gota.
- Coloque cuidadosamente un resbalón de cubierta de vidrio en la parte superior de la corredera del microscopio y coloque la diapositiva en el escenario de un microscopio invertido.
- Grabe imágenes de las células utilizando un objetivo de 10x y una cámara EMCCD conectada al microscopio para cada uno de los fluoróforos para ambas muestras de suspensión celular.
- Recuento manual de celdas con etiquetas fluorescentes (consulte Información adicional para datos sin procesar).
- Tome la proporción de células etiquetadas anti-Hu CD3 y anti-Hu CD4/CD25 a células manchadas por DAPI para obtener las proporciones de CD3+ células T y CD4+ CD25+ T-Regs a leucocitos totales.
3. Extracción de ADN y conversión de bisulfito
- Extracción de ADN
NOTA: Extraiga ADN de PBMC preparado en la sección 1 utilizando un kit de purificación de ADN magnético (ver Tabla de Materiales)siguiendo los procedimientos proporcionados por el fabricante.- En un tubo de 1,5 ml, suspenda las células en 100 l de PBS y añada 20 l de Proteinasa K y 400 l de lysis/buffer de unión. Mezclar pipeteando hacia arriba 10x, luego realizar la incubación a temperatura ambiente durante 5 min.
- Capture el complejo de dna-bead colocando el tubo en un bastidor magnético durante 1-2 min, luego retire cuidadosamente y deseche el sobrenadante.
- Retire el tubo que contiene el complejo de perlas de ADN del bastidor magnético y resuspender las perlas en 600 l de Wash Buffer #1 de lavar cualquier unión no específica.
- Coloque el tubo de nuevo en el bastidor magnético y retire y deseche cuidadosamente el sobrenadante.
- Repita los pasos 3.1.3 y 3.1.4 con 600 s de #2 de tampón de lavado.
- Deje el tubo abierto para secarlo al aire durante 1 min.
- Retire el tubo del bastidor magnético y elute el ADN dispensando 100 l de tampón de elución y pipeteando el complejo de ADN/perla hacia arriba y hacia abajo 20x.
- Coloque el tubo que contiene el ADN eluido de nuevo en el bastidor magnético e incubar durante 1-2 min para separar las perlas magnéticas del ADN eluido.
- Transfiera la solución de ADN purificado eluido a un nuevo tubo limpio.
- Evaluar la concentración de la muestra de ADN purificado midiendo la absorbancia a 260 nm utilizando un espectrofotómetro.
- Conversión de Bisulfite
NOTA: Realice la conversión de bisulfito en el ADN purificado utilizando un kit de metilación (ver Tabla de Materiales)siguiendo los procedimientos proporcionados por el fabricante.- A 20 ml de muestra de ADN (200-500 ng) en un tubo de PCR agregue 130 l de reactivo de conversión. Mezcle bien y gire brevemente.
- Transfiera el tubo de PCR a un ciclor térmico y realice el protocolo de ciclismo de la siguiente manera: 98 oC durante 8 min; 54oC durante 60 min y sujetar a 4oC.
- Agregue 600 l del búfer de enlace a una columna de cromatografía iónica (IC) colocada en un tubo de recogida.
- Agregue la muestra de ADN a la columna IC que contiene el búfer de unión y mezcle invirtiendo el tubo varias veces. Centrífuga para 30 s a toda velocidad. Deseche el flujo acumulado.
- Ahora a la columna agregue 100 l de zona de influencia de lavado. Realice la centrifugación como se describe en el paso 3.2.4 y deseche el flujo a través.
- Añadir 200 l de Tampón de Desulfonación y realizar la incubación a temperatura ambiente durante 15-20 min. Centrifugar y desechar el flujo a través como se describe en los pasos anteriores.
- Agregue 200 l de tampón de lavado a la columna. Centrifugar a toda velocidad durante 30 s y deseche el flujo a través.
- Repita el paso 3.2.7.
- Transfiera la columna a un nuevo tubo de recogida de 1,5 ml y añada 100 ml de agua de grado PCR en la membrana de la columna. Centrífuga a toda velocidad durante 1 min para eluir el ADN.
- Evaluar la concentración de la muestra de ADN convertida en bisulfito midiendo la absorbancia a 260 nm utilizando un espectrofotómetro.
NOTA: Utilice un valor de 40 g/ml para la absorción a 260 nm a 1,0. - Almacene el ADN a -20 oC para su almacenamiento a corto plazo o a -70 oC para su almacenamiento a largo plazo.
4. Fabricación de dispositivos de generación de gotas
NOTA: Un dispositivo microfluídico utilizado para la generación de gotas (archivo CAD proporcionado en la Información Suplementaria)se fabricó en un entorno de sala limpia (clase 1.000) en elastómero termoplástico (ver Tabla de Materiales)utilizando el relieve en caliente generado por el siguienteprotocolo.
- Fabricación de moho SU-8
- Prepare un molde SU-8 en una oblea de silicio de 6" utilizando fotolitografía estándar como se detalla a continuación.
- Limpie una oblea de silicio de 6" con plasma de oxígeno a 500 W durante 10 s.
- La capa giratoria SU-8 resiste en la oblea de silicio a 900 rpm durante 40 s para lograr un espesor total de película de 100 m.
- Coloque la oblea en una placa caliente y pre-hornee durante 15 minutos a 65 oC, seguido de 2 h a 95 oC.
- Exponer a la luz UV a 365 nm (Hg i-line) a través de una fotomasca de transparencia de alta definición utilizando una dosis de exposición de 1.000 mJ/cm2.
- Coloque la oblea en un plato caliente y cocinera durante 15 minutos a 65 oC, seguido de 40 min a 95 oC.
- Desarrollar mediante la supresión en acetato de éter monometil de propilenglicol (PGMEA) durante 5 min.
- Enjuagar con PGMEA e isopropanol y secar con una corriente de gas nitrógeno.
- Coloque la oblea en un plato caliente y hornee duro durante 2 horas a 135 oC.
- Silanizar la oblea en el desecador de vacío que contiene una gota de agente silanizante (tricholoro perfluorooctyl silane) colocado en un portaobjetos de microscopio de vidrio adyacente durante 2 h.
NOTA: Esto se hace para hacer que los sillanes formen una monocapa en la superficie del maestro SU-8. Tricholoro perfluorooctyl silane debe manipularse siempre en la campana de humos y mantenerse alejado de las fuentes de agua.
- Réplica de polidimetilsiloxano (PDMS)
- Preparar prepolímeros líquidos de PDMS (ver Tabla de materiales) en una relación de 10:1 con base de elastómero a agente de curado en una taza de plástico.
- Coloque la copa en un mezclador de vacío centrífugo planetario para mezclar y desgasar la mezcla PDMS.
- Vierta la mezcla PDMS en el molde colocado en el soporte de metal personalizado que evita fugas de resina y cura a 65 oC durante 2 horas.
- Con pinzas, despegue cuidadosamente el molde PDMS del maestro SU-8.
- Moho epoxi
NOTA: Se fabricó un molde epoxi a partir del maestro SU-8/silicon mediante un proceso de replicación intermedio con PDMS.- Preparar la resina epoxi (ver Tabla de Materiales) utilizando una relación 100/83 w/w de resina/endurecedor.
- Desgasar la mezcla a presión reducida utilizando un horno de secado al vacío durante 30 min.
- Vierta la resina sobre la réplica de PDMS y cure a 80 oC durante 12 h.
- Retire el molde epoxi curado de la réplica de PDMS, colóquelo en una placa caliente y hornee duro durante 2 horas a 120 oC.
- Dispositivo TPE
- Extruir pellets de TPE (ver Tabla de Materiales) a 165oC en 2,0 mm de espesor y 7" de ancho y almacenarlos como rollo para uso futuro.
- Corte la hoja de TPE del rollo usando tijeras en un cuadrado de 7".
- Coloque la hoja de TPE entre el molde epoxi y una oblea de silicio silanizado no modelado (consulte el paso 4.1.10 para el procedimiento de silanización de obleas).
- Realizar el grabado en caliente a una temperatura de 125 oC, una fuerza aplicada de 10 kN y una presión de 10–2 mbar durante 10 min.
- Demold cuidadosamente a temperatura ambiente usando spray de metanol para separar el TPE en relieve de la oblea de silicio y el molde epoxi.
- Corte otra hoja cuadrada de 7" de TPE y colóquela entre dos obleas de silicio silanizado no modeladas.
- Realizar el grabado en caliente a una temperatura de 140 oC, una fuerza aplicada de 10 kN y una presión de 10–2 mbar durante 10 minutos para formar una superficie plana para cerrar los canales y sellar el dispositivo.
- Demold cuidadosamente a temperatura ambiente usando spray de metanol para separar el TPE en relieve de las dos obleas de silicio.
- Corte cada una de las hojas de TPE en relieve al tamaño del dispositivo utilizando una cuchilla del médico.
- Golpee los orificios de acceso para los canales de entrada y salida en el dispositivo estructurado utilizando una aguja perforadora de biopsia de 1 mm con un émbolo.
- Encierre los canales colocando un dispositivo TPE plano en contacto directo con los canales a temperatura ambiente.
- Opcionalmente, colocar en un horno a 70 oC durante 2 horas para promover la unión del dispositivo.
- Ajuste los orificios de acceso con un tubo fluido desechable (I.D. 0,25 mm, D.O. 0,8 mm). Selle las juntas con un pegamento epoxi para garantizar la manipulación a prueba de fugas.
5. Generación de gotas y PCR
NOTA: La Tabla 1 describe la información sobre las imprimaciones hacia adelante y hacia atrás junto con las sondas de hidrólisis de doble enfriamiento para los genes C-LESS, CD3Z y Foxp3, que son necesarias para la amplificación multiplex de objetivos genéticos desmetilados.
- Prepare la mezcla maestra como se describe en la Tabla 2.
- Descongelar todos los componentes de la mezcla maestra excepto la mezcla enzimática. Mezclar el maestro mezclar a fondo pipeteando hacia arriba hacia abajo y girar hacia abajo brevemente.
- Agregue el volumen adecuado (1 l) de ADN convertido de bisulfito (de la sección 3.2) a la mezcla maestra en un tubo pcR. Mezclar la reacción pipeteando hacia arriba hacia abajo y girar hacia abajo brevemente.
- Conecte tubos fluidos desechables (I.D. 0,25 mm, D.O. 1,6 mm) a dos jeringas de vidrio de precisión (volumen de 250 oL) utilizando accesorios PEEK.
- Rellene previamente una jeringa de vidrio de precisión con 250 ml de aceite portador que contenga un 5% de fluoro-surfactante.
- Rellene previamente otra jeringa de vidrio de precisión con 50 ml de aceite portador antes de cargar 100 ml de la mezcla de PCR para asegurar la dosificación de todo el volumen de la muestra durante la emulsión.
- Configure un dispositivo microfluídico de gotas en una etapa de un microscopio de luz vertical equipado con una cámara de alta velocidad para observar y grabar la formación de gotas en tiempo real.
- Coloque las jeringas precargadas en la bomba de jeringa programable y utilizando la unión PEEK con accesorios (ver Tabla de materiales),conecte el tubo de las jeringas al tubo de los canales de entrada respectivos del dispositivo microfluídico de gotas.
- Coloque el tubo de la salida del generador de gotas dentro de un tubo PCR de 0,5 ml.
- Ajuste el caudal de la bomba de la jeringa a 2 l/min y permita que el tamaño de la gota se estabilice antes de recoger la emulsión resultante.
- Recoger la emulsión y transferir 75 l a un tubo PCR de 0,2 ml para el ciclo térmico.
- Asegúrese de que el contenido de aceite en el tubo PCR coincida estrechamente con el volumen de la fase dispersa para evitar la carbonescencia de las gotas durante el ciclo térmico.
- Colocar el tubo PCR de 0,2 ml en el ciclo térmico y realizar el protocolo de ciclismo de la siguiente manera: precalentamiento a 95 oC durante 5 min, luego 45 ciclos de desnaturalización a 95 oC durante 15 s y recocido/extensión a 60 oC durante 30 s.
- Utilice la emulsión restante para rellenar un tubo capilar de borosilicato (100 m de profundidad) con un perfil rectangular para crear imágenes de gotas y evaluar el diámetro de las gotas.
- Coloque el tubo de borosilicato lleno de la emulsión en un portaobjetos. Utilice un microscopio invertido equipado con una cámara EMCCD y un objetivo de 10x para grabar imágenes de campo brillante de las gotas.
- Mida el diámetro de la gota utilizando un software de análisis de imágenes como se detalla a continuación.
- Establezca la escala de la distancia conocida en micras correspondiente al número de píxeles de la imagen seleccionando el botón 'Analizar' y luego 'Establecer escala'.
- Convierta la imagen a escala de grises seleccionando el botón 'Imagen' y luego 'Escriba'. Ajuste el brillo y el contraste si es necesario. Establezca un umbral manual para delinear y rellenar los círculos seleccionando el botón 'Imagen' y luego 'Ajustar umbral'.
- Analice las partículas seleccionando el botón'Analizar partículas'y ajustando la circularidad a 0.75 – 1.
- Obtenga el área resultante y el diámetro de las gotas medidas que se muestran automáticamente en el software.
- Calcular el diámetro medio de la gota y suponiendo una gota esférica, estimar el volumen de partición, que se utilizará para calcular la concentración objetivo absoluta.
- Asegúrese de que las gotas son monodispersas mediante el análisis del coeficiente de variación (CV) que se toma como la relación de las desviaciones estándar a los valores medios (k - 1), para el diámetro de gota (< 3%).
6. Imágenes de fluorescencia y análisis de imágenes
- Imágenes de fluorescencia
- Después de la amplificación, transfiera la emulsión PCR al tubo capilar de borosilicato (profundidad de 50 m) con un perfil rectangular para colocar las gotas en una monocapa de paquete cercano para la toma de imágenes.
- Fije los capilares rellenos en un portaobjetos del microscopio y selle ambos lados usando un adhesivo acrílico UV. Aplique una fuente de luz UV sobre el adhesivo UV teniendo cuidado de no iluminar la emulsión para evitar blanquear la muestra.
- Cargue el portaobjetos de vidrio en un microscopio invertido equipado con una cámara EMCCD y un objetivo de 10x.
- Usando el software de imágenes de microscopio, seleccione Adquirir . En vivo - Rápido para iniciar la adquisición de la cámara en tiempo real, observar la muestra, y asegurarse de que se captura la anchura del capilar.
- Ajuste la lámpara de campo brillante para la iluminación diascópica a 3,5 V.
- Ajuste la lámpara fluorescente LED de amplio espectro para la iluminación episcópica a una intensidad del 20%.
- Ajuste manualmente la configuración de captura para todas las longitudes de onda (campo brillante, FAM, HEX y Cy5) seleccionando la calibración . Comando Configuraciones ópticas en el software de imágenes. Para cada fluoróforo, el cubo de filtro de fluorescencia correspondiente debe cambiarse manualmente. Se debe utilizar un cubo de filtro para las imágenes de campo brillante para mantener la misma ruta óptica.
- Ajuste el tiempo de exposición manualmente antes de la adquisición de la imagen utilizando Adquirir . Configuración de la cámara para cada fluoróforo como se resume en la Tabla 3. Ajuste el modo de lectura EM ganancia 17 MHz a 16 bits con un multiplicador de ganancia de 100 en el menú 'Ajustesde captura ' del software.
- En la ventana LUT del software, establezca la escala de LUTs de modo que la señal de transmisión se engloba dentro del rango establecido. En este experimento, la escala se estableció de 500 a 12.000 aproximadamente.
- Automatice la captura utilizando un programa de adquisición multipunto utilizando el escaneo XY y múltiples opciones de longitud de onda en ese orden específico. Asegúrese de que la etapa se mueve a la posición inicial, capture todas las longitudes de onda diferentes y, a continuación, proceda a la siguiente posición.
- En primer lugar, configure el escaneo XY personalizando el perfil de escaneo XY en el menú de escaneo XY del software. En 'Custom Multipoint Definition', elija el cuadro de definición de imagen grande y establézcalo en 40,0 x 1,0 mm aproximadamente (la longitud del llenado de la emulsión). Utilice la superposición del 1%.
- Active la opción'Usar superficie de enfoque'y configure la curva de superficie de enfoque ajustando el plano de enfoque en diferentes puntos de la muestra utilizando la perilla de enfoque del microscopio.
- En segundo lugar, configure el escaneo de longitud de onda múltiple seleccionando la pestaña de escaneo. Agregue cada una de las configuraciones ópticas creadas en el paso 6.1.7 para cada longitud de onda.
- Haga clic en la opción para cerrar el obturador activo durante el movimiento de la etapa y durante el cambio de filtro para evitar el blanqueo de la muestra y ejecutar la adquisición pulsando el botón 'Iniciar ejecución'. Exporte cada fotograma de adquisición en archivos tiff y divida cada canal en un archivo separado utilizando la opción dividir varios archivos y aplicando la configuración de LUTs guardadas. Utilice la opción de nombre de punto y nombre de canal para diferenciar fácilmente cada archivo de imagen.
- Análisis de imágenes
- Ordene todas las imágenes adquiridas por filtros de campo brillante y fluorescencia para cargarlas en un software de análisis de imágenes de código abierto.
- Utilice el software de análisis de imágenes para crear una canalización con el fin de identificar todas las gotas utilizando imágenes de campo brillante y, a continuación, medir la intensidad de las gotas fluorescentes asociadas.
- Para canalizar, cargue imágenes tiff de campo brillante y fluorescencia y, a continuación, agregue módulos 'ColorToGray', 'IdentifyPrimaryObjects', 'MeasureObjectIntensity' y 'ExportToSpreadsheet'. Utilice imágenes de gotas de campo brillante para identificar objetos y, a continuación, utilice objetos como máscara para medir la intensidad de las imágenes fluorescentes.
- Ejecute la canalización con imágenes tiff seleccionadas para extraer la intensidad media de fluorescencia de las imágenes de gotas de fluorescencia. Llevar a cabo el experimento en triplicado, con cada conjunto que consta de 5.000 gotas para el análisis.
- Aplique el algoritmo 'definetherain' (http://definetherain.org.uk/) para identificar los clústeres de gotas positivos y negativos. Los positivos deben estar dentro de 3 desviaciones estándar de la media. Esto determina la intensidad umbral de las gotas positivas que se utilizarán para el recuento.
- De nuevo, utilice el software de análisis de imágenes para implementar una nueva canalización en la que el umbral de fluorescencia para cada objetivo de gen se establezca como se define en el paso anterior.
- Cargue imágenes fluorescentes y de campo brillante en la tubería. Añadir módulos 'ColorToGray', 'RescaleIntensity', 'Umbral', 'IdentifyPrimaryObjects', 'MeasureObjectSizeShape', 'FilterObjects', y 'ExportToSpreadsheet'. Cree módulos únicos para imágenes de campo brillante y cada filtro de fluorescencia para gotas.
- Cambie la escala de intensidad de la imagen de 0 a 1 para cada grupo de imágenes de fluorescencia. A continuación, establezca el umbral para identificar y contar los objetos por encima del umbral establecido. Si es necesario, agregue el módulo 'ExpandOrShrinkObjects'para reducir objetos para facilitar el recuento y la identificación de gotas en brightfield.
- Identifique únicamente los objetos dentro del tamaño seleccionado de 20-30 píxeles y filtre los objetos contados para conservar solo aquellos objetos con un diámetro específico (es decir, gotas en el rango de diámetro de 75 m) y una excentricidad esférica redonda de 0,5 y por debajo.
- Exporte los resultados a una tabla que muestre el recuento total de gotas de imágenes de campo brillante, así como los recuentos de gotas de todos los canales de fluorescencia utilizados; a saber, Cy5 para el gen C-LESS, HEX para el gen CD3Z metilado y FAM para el gen MEtilado FOXP3 (véase Información complementaria para los datos experimentales en bruto obtenidos para el ensayo mdPCR presentado).
- Calcular la relación de gotas negativas para cada objetivo genético y aplicar la distribución de Poisson para obtener las copias respectivas por gota (CPD) utilizando la Ecuación 1:
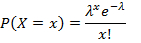
donde x representa el número de gotas que contienen 0, 1, 2 o más moléculas, y representa el valor de CPD. - Calcular, la concentración objetivo absoluta tomando la relación del valor CPD y el volumen de gotas obtenido en el paso 5.20 con la Ecuación 2:
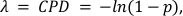
donde el valor (1-p) representa la fracción de gotas negativas. - Calcule el porcentaje de CD3+ células T y CD4+ CD25+ T-Regs dividiendo los valores CPD respectivos de los genes MEtilados CD3Z y FOXP3 por el valor C-LESS – o célula total – CPD (consulte Información complementaria para los cálculos de CPD a partir de datos sin procesar).
- Compare estos valores porcentuales con los obtenidos a partir de imágenes de inmunofluorescencia utilizando anticuerpos para el recuento de células TCD3 + T y CD4+ CD25+ T-Reg.
Resultados
El dispositivo generador de gotas microfluídicas basado en TPE se fabricó utilizando el protocolo descrito como se muestra en la Figura 1. Se utilizó una máscara de transparencia en la fotolitografía para obtener el maestro del silicio (Si). La litografía suave se realizó para obtener una réplica inversa de PDMS del maestro Si que luego se utilizó para fabricar el molde epoxi. El precursor de epoxi se vertió en el PDMS y se curó para reticularse y endurecerse. Este molde, que repr...
Discusión
El protocolo experimental y los métodos presentados permiten el mdPCR interno utilizando un generador de gotas TPE fabricado, un ciclor térmico y un microscopio de fluorescencia. El dispositivo fabricado que utiliza TPE suave a la unión TPE ofrece propiedades de superficie hidrófoba que son uniformes en todas las paredes del canal, de manera que el dispositivo final no requiere ningún tratamiento superficial para su uso posterior como generador de gotas. Este material se ha empleado rutinariamente en plataformas de ...
Divulgaciones
No hay conflictos que declarar.
Agradecimientos
Los autores reconocen el apoyo financiero del Consejo Nacional de Investigación de Canadá.
Materiales
| Name | Company | Catalog Number | Comments |
| Bio-Rad, Mississauga, ON | TFI0201 | PCR tube | |
| RAN Biotechnologies, Beverly, MA | 008-FluoroSurfactant | Fluoro-surfactant | |
| Silicon Quest International, Santa Clara, CA | |||
| Oxford Instruments, Abingdon, UK | EMCCD camera | ||
| Thermo Fisher Scientific, Waltham, MA | MA5-16728 | ||
| Thermo Fisher Scientific, Waltham, MA | 22-8425-71 | ||
| CellProfiler | Used for fluorescence image analysis | ||
| Nikon, Japan | 10x objective | ||
| American Type Culture Collection (ATCC), Manassas, VA | PCS-800-011 | ||
| Ramé-Hart Instrument Co. (Netcong, NJ) | p/n 200-U1 | ||
| Fisher, Canada | |||
| Vitrocom, NJ, USA | 5015 and 5010 | Borosilicate capilary tube | |
| (http://definetherain.org.uk/) | |||
| Hamamatsu, Japan | LC-L1V5 | DEL UV light source | |
| Dolomite | 3200063 | Disposable fluidic tubing | |
| Dolomite | 3200302 | Disposable fluidic tubing | |
| IDT, Coralville, IA | |||
| Nikon, Melville, NY | Upright light microscope | ||
| Cytec Industries, Woodland Park, NJ | |||
| EV Group, Schärding, Austria | |||
| Zymo Research, Irvine, CA | D5030 | ||
| Photron, San Diego, CA | |||
| IDT, Coralville, IA | |||
| Gersteltec, Pully, Switzerland | SU-8 photoresist | ||
| Fineline Imaging, Colorado Springs, CO | |||
| Qiagen, Hilden, Germany | 203603 | ||
| Image J | Used to assess droplet diameter | ||
| Anachemia, Montreal, QC | |||
| Excelitas, MA, USA | Broad-spectrum LED fluorescent lamp | ||
| Galenvs Sciences Inc., Montreal, QC | DE1010 | ||
| Hexpol TPE, Åmål, Sweden | Thermoplastic elastomer (TPE) | ||
| Thermo Fisher Scientific, Waltham, MA | 13-400-518 | ||
| Nikon, Japan | Used for image acquisition | ||
| 3M, St Paul, MN | Carrier Oil | ||
| Thermo Fisher Scientific, Waltham, MA | R37605 | Blue fluorescent live cell stain (DAPI) | |
| IDEX Health & Science, Oak Harbor, WA | P-881 | PEEK fittings | |
| Sigma-Aldrich, Oakville, ON | 806552 | ||
| Dow Corning, Midland, MI | |||
| ThinkyUSA, CA, USA | ARV 310 | ||
| Ihc world, Maryland, USA | IW-125-0 | ||
| Zinsser NA, Northridge, CA | 2607808 | ||
| Cetoni GmbH, Korbussen, Germany | |||
| Sigma-Aldrich, Oakville, ON | 484431 | ||
| Bio-Rad, Mississauga, ON | 1861096 | ||
| Hitachi High-Technologies, Mississauga, ON | |||
| Nikon, Melville, NY | Inverted microscope | ||
| Nikon, Japan | |||
| Loctite | AA 352 |
Referencias
- Teitell, M., Richardson, B. DNA methylation in the immune system. Clinical Immunology. 109 (1), 2-5 (2003).
- Suarez-Alvarez, B., Rodriguez, R. M., Fraga, M. F., López-Larrea, C. DNA methylation: A promising landscape for immune system-related diseases. Trends in Genetics. 28 (10), 506-514 (2012).
- Suárez-Álvarez, B., Raneros, A. B., Ortega, F., López-Larrea, C. Epigenetic modulation of the immune function: A potential target for tolerance. Epigenetics. 8 (7), 694-702 (2013).
- Kondilis-Mangum, H. D., Wade, P. A. Epigenetics and the adaptive immune response. Molecular Aspects of Medicine. 34 (4), 813-825 (2013).
- Zouali, M. . The Autoimmune Diseases. , (2014).
- Wiencke, J. K., et al. A comparison of DNA methylation specific droplet digital PCR (mdPCR) and real time qPCR with flow cytometry in characterizing human T cells in peripheral blood. Epigenetics. 9 (10), 1360-1365 (2014).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Analytical Chemistry. 83 (22), 8604-8610 (2011).
- Pinheiro, L. B., et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification. Analytical Chemistry. 84 (2), 1003-1011 (2012).
- Roy, E., Galas, J. C., Veres, T. Thermoplastic elastomers for microfluidics: Towards a high-throughput fabrication method of multilayered microfluidic devices. Lab on a Chip. 11 (18), 3193-3196 (2011).
- Roy, E., et al. From cellular lysis to microarray detection, an integrated thermoplastic elastomer (TPE) point of care Lab on a Disc. Lab on a Chip. 15 (2), 406-416 (2015).
- Malic, L., et al. Epigenetic subtyping of white blood cells using a thermoplastic elastomer-based microfluidic emulsification device for multiplexed, methylation-specific digital droplet PCR. Analyst. 44 (22), 6541-6553 (2019).
- Malic, L., et al. Polymer-based microfluidic chip for rapid and efficient immunomagnetic capture and release of Listeria monocytogenes. Lab Chip. 15 (20), 3994-4007 (2015).
- Malic, L., Morton, K., Clime, L., Veres, T. All-thermoplastic nanoplasmonic microfluidic device for transmission SPR biosensing. Lab Chip. 13 (5), 798-810 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados