Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Methylierung spezifische Multiplex Droplet PCR mit Polymer Droplet Generator Gerät für die hämatologische Diagnostik
In diesem Artikel
Zusammenfassung
Epigenetische Marker werden für die Untertypisierung von weißen Blutkörperchen (WBC) durch die Quantifizierung von DNA-Methylierungsmustern verwendet. Dieses Protokoll stellt ein Multiplex-Droplet-Polymerase-Kettenreaktionsverfahren (mdPCR) unter Verwendung eines thermoplastischen Elastomer(TPE)-basierten mikrofluidischen Geräts zur Tröpfchenerzeugung dar, das eine präzise und Multiplexmethylierungsspezifische Zielquantifizierung von WBC-Differentialzählungen ermöglicht.
Zusammenfassung
Ein Multiplex-Droplet-PCR-Workflow (mdPCR) und ein detailliertes Protokoll zur Bestimmung der epigenetischen Anzahl von weißen Blutkörperchen (WBC) werden zusammen mit einem thermoplastischen Elastomer (TPE) mikrofluidischen Tröpfchenerzeugungsgerät beschrieben. Epigenetische Marker werden für die WBC-Subtypisierung verwendet, die bei verschiedenen Krankheiten von wichtigem prognostischen Wert ist. Dies wird durch die Quantifizierung von DNA-Methylierungsmustern bestimmter CG-reicher Regionen im Genom (CpG loci) erreicht. In diesem Beitrag wird bisulfitbehandelte DNA aus peripheren mononukleären Blutzellen (PBMCs) in Tröpfchen mit mdPCR-Reagenzien, einschließlich Primern und hydrolysefluoreszierenden Sonden, die speziell für CpG-Loci sind, die mit WBC-Subpopulationen korrelieren, eingekapselt. Der Multiplex-Ansatz ermöglicht die Abfrage vieler CpG-Loci ohne separate mdPCR-Reaktionen, was eine genauere parametrische Bestimmung von WBC-Subpopulationen mithilfe epigenetischer Analysen von Methylierungsstellen ermöglicht. Diese präzise Quantifizierung kann auf verschiedene Anwendungen ausgedehnt werden und hebt die Vorteile für die klinische Diagnose und die anschließende Prognose hervor.
Einleitung
Die Analyse der Zusammensetzung weißer Blutkörperchen (WBCs) gehört zu den am häufigsten nachgefragten Labortests in der hämatologischen Diagnostik. Differential-Leukozyten-Zahl dient als Indikator für ein Spektrum von Krankheiten einschließlich Infektion, Entzündung, Anämie, und Leukämie, und wird als frühe prognostische Biomarker für mehrere andere Bedingungen sowie untersucht. Der Goldstandard in der WBC-Subtypisierung beinhaltet Immunfärbung und/oder Durchflusszytometrie, die beide kostspielige, instabil-anfällige fluoreszierende Antikörper erfordern und oft stark von der Befähigung des Bedieners in der Probenvorbereitung abhängig sind. Darüber hinaus gilt diese Methode nur für frische Blutproben, so dass die Proben nicht für den Versand oder die spätere Analyse eingefroren werden können.
Epigenetische Marker haben sich in letzter Zeit als leistungsfähige Analysewerkzeuge für die Untersuchung von phänotypischen Variationen herausgestellt. In der Folge wurde gezeigt, dass menschliche Leukozytenpopulationen DNA-Methylierungsmuster für Zelllinien aufweisen, die eine präzise Charakterisierung von WBC-Teilmengen ermöglichen. Subtypisierung auf Basis epigenetischer Marker bietet eine vielversprechende Alternative, die nicht von der Entnahme von frischer Blutprobe oder teuren Antikörpern abhängt und als Biomarker für Krankheitsbeginn und Anfälligkeit1,2,3,4,5" genutzt werden kann.
Genomweite Studien wurden für die umfassende Kartierung methylierter spezifischer CG-reicher Regionen im Genom (CpG-Inseln) in Leukozytensubtypen durchgeführt, um epigenetische Kandidatenmarker zu identifizieren, die für Leukozytensubtypen spezifisch sind. PCR-Protokolle wurden aus diesem Grund für methylierte Genregionen entwickelt, z.B. CD3Z und FOXP3, entsprechend CD3+ T-Cells und CD4+ CD25+ Regulatory T-Cells (T-Regs). Wiencke et al. haben die Nützlichkeit der Duplex-Tropfen-PCR für die epigenetische Subtypisierung von T-Zellen nachgewiesen und ergebnissegebracht, die in hohem Maße mit der durchflussaktivierten Zellsortierung (FACS) Korpusanalyse korrelieren6. Diese quantitative genetische Analysemethode beruht auf der Aufteilung der Schablonennukleinsäuremoleküle und PCR-Reagenzien in Tausende von diskreten, volumetrisch definierten Tröpfchen mit Null, einer oder mehreren Zielnukleinsäurekopien, mit Wasser-in-Öl-Emulsionen, die durch Mikrofluidika aktiviert werden7,8. Die PCR-Verstärkung wird in jedem einzelnen Tröpfchen durchgeführt und die Endpunktfluoreszenzintensität jedes Tröpfchens wird gemessen, so dass eine absolute Quantifizierung der in der Probe vorhandenen Ziele möglich ist. Droplet PCR wurde etabliert, um präziser, genauer und technisch einfacher als Standard qPCR zu sein, was es zu einer günstigeren DNA-Methylierungs-basierten Methode für die klinische Bewertung von T-Cells macht. Obwohl eine sich rasch entwickelnde Subtypisierungsmethode fehlt, fehlt es an multiplexed epigenetischen Analysen, um verschiedene methylierte CpG-Regionen gleichzeitig zu untersuchen. Dies ist für routinemäßige Leukozytendifferenzzählungen notwendig.
Hierbei wird ein thermoplastisches Elastomer (TPE) Tröpfchen-Mikrofluid-Gerät vorgestellt und für methylierungsspezifische Multiplex-Tröpfchen-PCR (mdPCR) eingesetzt. Die Technologie wurde verwendet, um spezifische Leukozyten-Subtypen, CD3+ T-Cells und CD4+ CD25+ T-Regs, basierend auf Zelllinien-DNA-Methylierungsmustern, d.h. epigenetischen Variationen von CD3Z- bzw. FOXP3 CpG-Regionen, zu definieren. Ein detailliertes Protokoll zur DNA-Extraktion, Bisulfitumwandlung und mdPCR wird in Abstimmung mit einer Herstellungsmethode für ein TPE-Tröpfchenerzeugungsgerät beschrieben. Repräsentative Ergebnisse der Methode werden mit denen der Immunfluoreszenzfärbung verglichen, die den Nutzen des vorgeschlagenen Ansatzes unterstreicht.
Protokoll
Alle Experimente, die in dieser Studie mit menschlichen Proben durchgeführt wurden, wurden vom NRC Ethics Board genehmigt und wurden gemäß der NRC-Politik für menschliche Probanden durchgeführt, die den geltenden Forschungsrichtlinien folgen und den Gesetzen in Québec, Kanada, entsprechen.
1. Zellvorbereitung
- Die gefrorenen menschlichen peripheren mononukleären Blutzellen (PBMCs) sofort auftauen, indem Sie das Kryovial in einem Wasserbad bei 37 °C für 5 min platzieren.
- Invertieren Sie die Kryovial zweimal, um die Zellen sanft wieder zu suspendieren und mit einer 1 ml Pipette übertragen Sie die Zellsuspension in ein 15 ml konisches Rohr.
- Fügen Sie 10 ml vorgewärmtes (37 °C) Wachstumsmedium, ergänzt mit 10% fetalem Rinderserum (RPMI-1640 + 10% FBS), in das 15 ml-Rohr mit den PBMCs ein.
- Zentrifugieren Sie die Zellaufhängung in einer schwingenden Schaufelzentrifuge bei Raumtemperatur bei einer Geschwindigkeit von 330 x g für 10 min mit schneller Beschleunigung und der Bremse hoch.
- Sobald die Drehung vorbei ist, dekanieren Sie vorsichtig den Überstand. Setzen Sie das Zellpellet in 3 ml phosphatgepufferter Saline (PBS) pH 7,2 wieder auf, die 2 mM Ethylendiamintetraessigsäure (EDTA) enthält, indem Sie auf die Seite der Rohre tippen.
- Mischen Sie die Zellen, indem Sie das Rohr mit der Kappe fest geschlossen.
- Bereiten Sie zwei 1,5 ml Mikroröhren vor und verwenden Sie eine Pipette aliquot 1,5 ml der Zellsuspension in jedem Rohr, von denen eines für die nachfolgende Immunfluoreszenzfärbung und eine für die DNA-Extraktion verwendet wird.
2. Immunfluoreszenz-Färbungs- und Bildgebungsprotokoll
- Resuspend Zellen (von 1,7) in 200 l PBS-Puffer mit 0,1% Natriumazid und 2% FBS und stellen Sie die Endkonzentration der Zellsuspension auf maximal 2 x 107 Zellen/ml.
- Teilen Sie die Zellsuspension, indem Sie 100 l Volumen in zwei separate 1,5 ml Mikroröhren pipetieren.
- Fügen Sie dem zweiten Röhrchen 20 L Anti-Hu CD3/CD4 konjugiert mit Fluorescein-Isothiocyanat (FITC) und Phycoerythrin (PE) und Anti-Hu CD4/CD25 konjugiert mit FITC und PE (siehe Materialtabelle)hinzu.
- Fügen Sie jedem Rohr einen Tropfen blaufluoreszierenden Lebendzellenfleck (siehe Materialtabelle)hinzu.
- Inkubieren bei Raumtemperatur mit einem Rohrrotator für 2 h. Vor Licht schützen.
- Zentrifugieren Sie die Zellaufhängung bei Raumtemperatur bei 330 x g für 10 min mit schneller Beschleunigung und der Bremse hoch.
- Dekantieren Sie den Überstand und setzen Sie das Zellpellet vorsichtig wieder auf, indem Sie auf das Rohr tippen. 1 ml PBS hinzufügen, pH 7,2 mit 2 mM EDTA. Um sicherzustellen, dass die Kappe fest geschlossen ist, mischen Sie die Zellen, indem Sie das Rohr 2x invertieren.
- Wiederholen Sie die Schritte 2.6 und 2.7 dreimal.
- Resuspend-Zellen in 20 l PBS pH 7,2 mit 2 mM EDTA.
- Pipette 10 l Tropfen der Zellsuspension auf ein Borosilikat-Mikroskop-Dia und warten Sie 2 min, bis die Zellen langsam Sediment auf den Boden des Tropfens.
- Legen Sie vorsichtig einen Glasdeckel auf die Oberseite des Mikroskopschlittens und legen Sie das Dia auf die Bühne eines invertierten Mikroskops.
- Zeichnen Sie Bilder der Zellen mit einem 10-fachen Objektiv und einer MIT dem Mikroskop verbundenen EMVD-Kamera für jedes fluorophor e.B. für beide Zellsuspensionsproben auf.
- Zählen Sie fluoreszierend markierte Zellen manuell an (siehe Ergänzende Informationen für Rohdaten).
- Nehmen Sie das Verhältnis von Anti-Hu CD3 und Anti-Hu CD4/CD25-markierten Zellen zu DAPI-gefleckten Zellen, um die Anteile von CD3+ T-Cells und CD4+ CD25+ T-Regs zu Gesamtleukozyten zu erhalten.
3. DNA-Extraktion und Bisulfit-Umwandlung
- DNA-Extraktion
HINWEIS: Extrahieren Sie DNA aus PBMCs, die in Abschnitt 1 mit einem magnetischen DNA-Reinigungskit (siehe Tabelle der Materialien)nach den vom Hersteller bereitgestellten Verfahren hergestellt wurden.- In einem 1,5 ml-Rohr suspendieren Zellen in 100 l PBS und fügen Sie 20 l Proteinase K und 400 l Lysis/Binding Puffer hinzu. Mischen Sie durch Pipettier nach oben 10x, dann durchführen Inkubation bei Raumtemperatur für 5 min.
- Erfassen Sie den DNA-Perlenkomplex, indem Sie die Röhre für 1-2 min auf ein magnetisches Rack legen, dann den Überstand vorsichtig entfernen und entsorgen.
- Entfernen Sie die Tube, die den DNA-Perlenkomplex enthält, aus dem magnetischen Rack und setzen Sie die Perlen in 600 l Waschpuffer #1, um unspezifische Bindungen wegzuwaschen.
- Legen Sie das Rohr wieder auf das magnetische Rack und entfernen und entsorgen Sie den Überstand sorgfältig.
- Wiederholen Sie die Schritte 3.1.3 und 3.1.4 mit 600 l Wash Buffer #2.
- Lassen Sie das Rohr 1 min lufttrocken.
- Entfernen Sie das Rohr aus dem magnetischen Rack und heben Sie die DNA ab, indem Sie 100 l Elution Buffer ausgibt und den DNA/Perlenkomplex 20x nach oben und unten pipetieren.
- Legen Sie die Röhre mit der vereisten DNA wieder auf das magnetische Rack und brüten Sie 1-2 min, um die magnetischen Perlen von der vereisten DNA zu trennen.
- Übertragen Sie die eluierte gereinigte DNA-Lösung in ein neues Röhrchen.
- Bewerten Sie die Konzentration der gereinigten DNA-Probe, indem Sie die Absorption bei 260 nm mit einem Spektralphotometer messen.
- Bisulfit-Umwandlung
HINWEIS: Führen Sie die Bisulfitumwandlung auf gereinigte DNA mit einem Methylierungskit (siehe Materialtabelle) nach den vom Hersteller bereitgestellten Verfahren durch.- Bis zu 20 l DNA-Probe (200-500 ng) in einem PCR-Röhrchen 130 l Konversionsreagenz hinzufügen. Gut mischen und kurz nach unten drehen.
- Übertragen Sie das PCR-Rohr auf einen thermischen Cycler und führen Sie das Zyklusprotokoll wie folgt aus: 98 °C für 8 min; 54 °C für 60 min und halten bei 4 °C.
- Fügen Sie 600 L des Bindungspuffers zu einer Ionchromatographie (IC)-Säule hinzu, die in ein Sammelrohr gelegt wird.
- Fügen Sie die DNA-Probe in die IC-Säule ein, die den Bindungspuffer enthält, und mischen Sie sie, indem Sie die Röhre mehrmals invertieren. Zentrifuge für 30 s bei voller Geschwindigkeit. Entsorgen Sie den gesammelten Durchfluss.
- Zur Spalte fügen Sie nun 100 L Waschpuffer hinzu. Zentrifugation wie in Schritt 3.2.4 beschrieben durchführen und den Durchfluss verwerfen.
- Fügen Sie 200 l Desulfonationspuffer hinzu und führen Sie eine Inkubation bei Raumtemperatur für 15-20 min durch. Zentrifuge und entsorgen Sie den Durchfluss, wie in den oben beschriebenen Schritten beschrieben.
- Fügen Sie der Spalte 200 L Waschpuffer hinzu. Zentrifugieren Sie mit voller Geschwindigkeit für 30 s und entsorgen Sie den Durchfluss.
- Wiederholen Sie Schritt 3.2.7.
- Übertragen Sie die Säule in ein neues 1,5 ml Sammelrohr und fügen Sie 100 l PCR-Wasser auf die Membran der Säule. Zentrifuge bei voller Geschwindigkeit für 1 min, um die DNA zu elute.
- Bewerten Sie die Konzentration der bisulfitum-konvertierten DNA-Probe, indem Sie die Absorption bei 260 nm mit einem Spektralphotometer messen.
ANMERKUNG: Verwenden Sie einen Wert von 40 g/ml für die Absorption bei 260 nm = 1,0. - Speichern Sie DIE DNA bei -20 °C für die kurzfristige Lagerung oder bei -70 °C für die Langzeitlagerung.
4. Droplet-Generierung Gerätefertigung
HINWEIS: Ein mikrofluidisches Gerät, das für die Tröpfchenerzeugung verwendet wird (CAD-Datei, die in den Ergänzenden Informationenbereitgestellt wird), wurde in einer Reinraumumgebung (Klasse 1.000) in thermoplastischem Elastomer (siehe Tabelle der Materialien) unter Verwendung von Heißprägungen hergestellt, die durch das folgende Protokoll erzeugtwerden.
- SU-8 Formenfertigung
- Bereiten Sie eine SU-8-Form auf einem 6"-Siliziumwafer mit Standard-Photolithographie vor, wie unten beschrieben.
- Reinigen Sie einen 6" Siliziumwafer mit Sauerstoffplasma bei 500 W für 10 s.
- Spin-Coat SU-8 widerstehen auf dem Silizium-Wafer bei 900 U/min für 40 s, um eine Gesamtschichtdicke von 100 m zu erreichen.
- Den Wafer auf eine Kochplatte legen und 15 min bei 65 °C vorbacken, gefolgt von 2 h bei 95 °C.
- UV-Licht bei 365 nm (Hg i-line) durch eine hochauflösende Transparenz-Fotomaske mit einer Belichtungsdosis von 1.000 mJ/cm2aussetzen.
- Den Wafer auf eine Kochplatte legen und 15 min bei 65 °C nachbacken, gefolgt von 40 min bei 95 °C.
- Entwickeln Sie durch Eintauchen in Propylenglykolmonomethyletheracetat (PGMEA) für 5 min.
- Mit PGMEA und Isopropanol abspülen und mit einem Stickstoffgasstrom trocknen.
- Den Wafer auf eine Kochplatte legen und 2 h bei 135 °C hart backen.
- Silanisieren Sie den Wafer im Vakuum-Destflutor, der einen Tropfen Silanisatisierungsmittel (Tricholoro perfluorooctylsilan) enthält, der 2 h lang auf einem angrenzenden Glasmikroskopschlitten platziert ist.
HINWEIS: Dies geschieht, um die Silane zu einer Monoschicht auf der Oberfläche des SU-8-Masters zu machen. Tricholoro Perfluoroctylsilan sollte immer in der Dunstabzugshaube behandelt und von Wasserquellen ferngehalten werden.
- Polydimethylsiloxan (PDMS) Replik
- Bereiten Sie flüssige Prepolymere von PDMS (siehe Materialtabelle) mit einem Verhältnis von 10:1 w/w Elastomerbasis zu Härtungsmittel in einer Kunststofftasse vor.
- Legen Sie den Becher in einen planetarischen Zentrifugalvakuummischer, um das PDMS-Gemisch zu mischen und zu entgasen.
- Gießen Sie PDMS-Mischung auf die Form in den benutzerdefinierten Metallhalter, die Harz Leckage verhindert und aushärten bei 65 °C für 2 h.
- Mit einer Pinzette die PDMS-Form vorsichtig vom SU-8-Master abziehen.
- Epoxy-Form
HINWEIS: Eine Epoxidform wurde aus dem SU-8/Silizium-Master unter Verwendung eines Zwischenreplikationsprozesses mit PDMS hergestellt.- Bereiten Sie das Epoxidharz (siehe Materialtabelle) mit einem Verhältnis von 100/83 w/w harz/härter vor.
- Entgasen Sie das Gemisch unter reduziertem Druck mit einem Vakuumtrockner für 30 min.
- Gießen Sie das Harz über das PDMS-Replika und härten Sie bei 80 °C für 12 h aus.
- Die ausgehärtete Epoxidform aus dem PDMS-Replika entfernen, auf eine Kochplatte legen und 2 h bei 120 °C hart backen.
- TPE-Gerät
- Pellets aus TPE (siehe Materialtabelle)bei 165 °C in 2,0 mm Dicke und 7" breite Blätter mit einer Länge von mehreren Metern extrudieren und als Rolle für die zukünftige Verwendung lagern.
- Schneiden Sie das TPE-Blatt von der Rolle mit einer Schere in ein 7" Quadrat.
- Legen Sie das TPE-Blatt zwischen die Epoxidform und einen nicht gemusterten silanisierten Siliziumwafer (siehe Schritt 4.1.10 für Wafer-Silanisierungsverfahren).
- Führen Sie Heißprägungbei bei einer Temperatur von 125 °C, einer aufgebrachten Kraft von 10 kN und einem Druck von 10-2 mbar für 10 min.
- Demold vorsichtig bei Raumtemperatur mit Methanolspray, um das geprägte TPE vom Siliziumwafer und der Epoxidform zu trennen.
- Schneiden Sie ein weiteres 7" quadratisches Blatt TPE und legen Sie es zwischen zwei nicht gemusterten silanisierten Siliziumwafern.
- Führen Sie eine Heißprägung bei einer Temperatur von 140 °C, einer aufgebrachten Kraft von 10 kN und einem Druck von 10–2 mbar für 10 min durch, um eine planare Oberfläche zum Schließen der Kanäle und zum Abdichten des Geräts zu bilden.
- Demold vorsichtig bei Raumtemperatur mit Methanolspray, um das geprägte TPE von den beiden Siliziumwafern zu trennen.
- Schneiden Sie jede der geprägten TPE-Platten mit einer Schaberklinge auf Gerätegröße.
- Stanzen Sie die Zugangslöcher für die Ein- und Auslasskanäle im strukturierten Gerät mit einer 1 mm Biopsie-Stanznadel mit Kolben.
- Schließen Sie die Kanäle ein, indem Sie ein planares TPE-Gerät bei Raumtemperatur in direkten Kontakt mit den Kanälen setzen.
- Optional in einen Ofen bei 70 °C für 2 h stellen, um die Gerätebindung zu fördern.
- Befestigung der Zugangslöcher mit einem Einweg-Fluidschlauch (I.D. 0,25 mm, O.D. 0,8 mm). Versiegeln Sie die Verbindungen mit einem Epoxidkleber, um eine leckagesichere Manipulation zu gewährleisten.
5. Tröpfchenerzeugung und PCR
ANMERKUNG: Tabelle 1 enthält Informationen über die Vorwärts- und Rückwärtsgrundierung zusammen mit den doppelt gelöschten Hydrolysesonden für C-LESS-, CD3Z- und Foxp3-Gene, die für die Multiplex-Amplifikation von demethylierten Genzielen erforderlich sind.
- Bereiten Sie den Mastermix wie in Tabelle 2beschrieben vor.
- Alle Komponenten des Master-Mix mit Ausnahme des Enzymmixes auftauen. Mischen Sie den Master-Mix gründlich, indem Sie nach oben und kurz nach unten drehen.
- Fügen Sie das entsprechende Volumen (1 l) bisulfitkonvertierte DNA (ab Abschnitt 3.2) in eine MASTER-Mischung in einem PCR-Rohr hinzu. Mischen Sie die Reaktion, indem Sie nach oben und drehen Sie kurz nach unten.
- Einweg-Fluidrohre (I.D. 0,25 mm, O.D. 1,6 mm) mit zwei Präzisionsglasspritzen (250 l Volumen) mit PEEK-Fittings verbinden.
- Füllen Sie eine Präzisionsglasspritze mit 250 l Trägeröl, das 5% Fluor-Tensid enthält.
- Befüllen Sie eine weitere Präzisionsglasspritze mit 50 l Trägeröl vor dem Beladen von 100 l des PCR-Mixes, um die Dosierung des gesamten Probenvolumens während der Emulgierung sicherzustellen.
- Richten Sie ein mikrofluidisches Tröpfchengerät auf einer Bühne eines aufrechten Lichtmikroskops ein, das mit einer Hochgeschwindigkeitskamera ausgestattet ist, um die Tröpfchenbildung in Echtzeit zu beobachten und aufzuzeichnen.
- Legen Sie die Fertigspritzen auf die programmierbare Spritzenpumpe und verbinden Sie die Vorfertigspritzen mit PEEK-Union mit Armaturen (siehe Materialtabelle),verbinden Sie die Schläuche der Spritzen mit den Schläuchen der jeweiligen Einlasskanäle des Tröpfchen-Mikrofluid-Geräts.
- Legen Sie die Schläuche aus dem Auslass des Tröpfchengenerators in ein 0,5 ml PCR-Rohr.
- Stellen Sie den Durchfluss der Spritzenpumpe auf 2 l/min ein und lassen Sie die Tröpfchengröße stabilisieren, bevor die resultierende Emulsion gesammelt wird.
- Sammeln Sie die Emulsion und übertragen Sie 75 l auf ein 0,2 ml PCR-Rohr für thermisches Radfahren.
- Stellen Sie sicher, dass der Ölgehalt im PCR-Rohr eng mit dem Volumen der dispergierten Phase übereinstimmt, um eine Koaleszenz der Tröpfchen während des thermischen Zyklus zu verhindern.
- Legen Sie das 0,2 ml PCR-Rohr in den thermischen Cycler und führen Sie das Zyklusprotokoll wie folgt aus: Vorwärmung bei 95 °C für 5 min, dann 45 Denaturierungszyklen bei 95 °C für 15 s und Glühen/Verlängerung bei 60 °C für 30 s.
- Verwenden Sie die verbleibende Emulsion, um ein Borosilikatkapillarrohr (100 m Tiefe) mit einem rechteckigen Profil zu füllen, um Tröpfchen abzubilden und den Tröpfchendurchmesser zu bewerten.
- Legen Sie das mit der Emulsion gefüllte Borosilikatrohr auf ein Mikroskopschlitten. Verwenden Sie ein invertiertes Mikroskop mit einer EMCCD-Kamera und einem 10-fachen Objektiv, um helle Feldbilder der Tröpfchen aufzuzeichnen.
- Messen Sie den Tröpfchendurchmesser mit einer Bildanalysesoftware, wie unten beschrieben.
- Legen Sie die Skala der bekannten Entfernung in Mikrometern fest, die der Anzahl der Pixel im Bild entspricht, indem Sie die Schaltfläche "Analysieren"und dann "Maßstab festlegen"auswählen.
- Konvertieren Sie das Bild in Graustufen, indem Sie die Schaltfläche 'Bild'und dann 'Typ' auswählen. Passen Sie Helligkeit und Kontrast bei Bedarf an. Legen Sie einen manuellen Schwellenwert fest, um die Kreise zu abgrenzen und zu füllen, indem Sie die Schaltfläche"Bild"und dann den Schwellenwertanpassenauswählen.
- Analysieren Sie die Partikel, indem Sie die Schaltfläche"Partikel analysieren"auswählen und die Zirkularität auf 0,75 – 1 einstellen.
- Erhalten Sie den resultierenden Bereich und den Durchmesser der gemessenen Tröpfchen, der automatisch in der Software angezeigt wird.
- Berechnen Sie den mittleren Tröpfchendurchmesser und gehen Sie von einem kugelförmigen Tröpfchen aus, schätzen Sie das Partitionsvolumen, das zur Berechnung der absoluten Zielkonzentration verwendet wird.
- Stellen Sie sicher, dass die Tröpfchen monodisperse sind, indem Sie den Variationskoeffizienten (CV) analysieren, der als Verhältnis der Standardabweichungen zu den Mittelwerten(k = 1) für den Tröpfchendurchmesser (< 3%) angenommen wird.
6. Fluoreszenzbildgebung und Bildanalyse
- Fluoreszenz-Bildgebung
- Nach der Verstärkung die PCR-Emulsion in ein Borosilikatkapillarrohr (50 m Tiefe) mit einem rechteckigen Profil übertragen, um Tröpfchen zur Bildgebung in eine dicht gepackte Monoschicht zu arrangieren.
- Fixieren Sie die gefüllten Kapillaren auf einem Mikroskopschlitten und versiegeln Sie beide Seiten mit einem UV-Acrylkleber. Tragen Sie eine UV-Lichtquelle auf den UV-Klebstoff auf, wobei Sie darauf achten, die Emulsion nicht zu beleuchten, um ein Bleichen der Probe zu vermeiden.
- Laden Sie die Glasrutsche auf ein invertiertes Mikroskop, das mit einer EMVCD-Kamera und einem 10-fachen Objektiv ausgestattet ist.
- Wählen Sie mit der Mikroskop-Bildgebungssoftware Acquire | Live - Schnelles Starten der Echtzeit-Kameraaufnahme, beobachten Sie die Probe und stellen Sie sicher, dass die Breite der Kapillare erfasst wird.
- Stellen Sie die helle Feldlampe für die diaskopische Beleuchtung auf 3,5 V ein.
- Stellen Sie die Breitspektrum-LED-Leuchtstofflampe für die episkopische Beleuchtung auf eine Intensität von 20 % ein.
- Passen Sie die Aufnahmeeinstellung für alle Wellenlängen (Hellfeld, FAM, HEX und Cy5) manuell an, indem Sie die Kalibrierung | Befehl Optische Konfigurationen in der Imaging-Software. Für jedes Fluorophor muss der entsprechende Fluoreszenzfilterwürfel manuell geschaltet werden. Ein Filterwürfel für die Lichtfeldabbildung muss verwendet werden, um denselben optischen Pfad beizubehalten.
- Passen Sie die Belichtungszeit manuell vor der Bildaufnahme mit Acquire | Befehl Kameraeinstellungen für jedes Fluorophor, wie in Tabelle 3zusammengefasst. Stellen Sie den Auslesemodus EM Gain 17 MHz bei 16-Bit mit einem Gain-Multiplikator von 100 im Menü 'Capture Settings' der Software ein.
- Stellen Sie im LUT-Fenster der Software die LUTs-Skala so ein, dass das Übertragungssignal innerhalb des eingestellten Bereichs eingeschlossen ist. In diesem Experiment wurde die Skala von 500 bis etwa 12.000 festgelegt.
- Automatisieren Sie die Erfassung mit einem Multipoint-Erfassungsprogramm mit XY-Scan- und mehreren Wellenlängenoptionen in dieser bestimmten Reihenfolge. Stellen Sie sicher, dass sich die Bühne in die Ausgangsposition bewegt, erfassen Sie alle verschiedenen Wellenlängen, und fahren Sie dann mit der nächsten Position fort.
- Richten Sie zunächst den XY-Scan ein, indem Sie das XY-Scanprofil im XY-Scanmenü der Software anpassen. Wählen Sie in 'Custom Multipoint Definition' das große Bilddefinitionsfeld aus und stellen Sie es auf ca. 40,0 x 1,0 mm (die Länge der Emulsionsfüllung). Verwenden Sie 1% Überlappung.
- Aktivieren Sie die Option"Fokusoberflächeverwenden", und richten Sie die Fokusoberflächenkurve ein, indem Sie die Fokusebene an verschiedenen Punkten der Probe mithilfe des Fokusknopfes am Mikroskop anpassen.
- Stellen Sie zweitens den Scan mit mehreren Wellenlängen ein, indem Sie die Registerkarte "Scan" auswählen. Fügen Sie jede der optischen Konfigurationen hinzu, die in Schritt 6.1.7 für jede Wellenlänge erstellt wurden.
- Klicken Sie auf die Option, um den aktiven Verschluss während der Bühnenbewegung und während des Filterwechsels zu schließen, um das Bleichen der Probe zu vermeiden und die Erfassung auszuführen, indem Sie die Taste"Ausführen starten"drücken. Exportieren Sie jeden Erfassungsrahmen in tiff-Dateien und teilen Sie jeden Kanal mithilfe der Option Split Multiple Files und durch Anwenden gespeicherter LUTs-Einstellungen in eine separate Datei auf. Verwenden Sie die Option Punktname und Kanalname, um jede Bilddatei einfach zu unterscheiden.
- Bildanalyse
- Sortieren Sie alle aufgenommenen Bilder nach Hellfeld- und Fluoreszenzfiltern, um sie in eine Open-Source-Bildanalysesoftware hochzuladen.
- Verwenden Sie Bildanalysesoftware, um eine Pipeline zu erstellen, um alle Tröpfchen mithilfe von Hellfeldbildern zu identifizieren und dann die Intensität der zugehörigen fluoreszierenden Tröpfchen zu messen.
- Um Pipeline, laden Sie hellfeld und fluoreszenz tiff Bilder dann Module 'ColorToGray', 'IdentifyPrimaryObjects', 'MeasureObjectIntensity' und 'ExportToSpreadsheet' hinzufügen. Verwenden Sie Hellfeld-Tröpfchenbilder, um Objekte zu identifizieren, und verwenden Sie dann Objekte als Maske, um die Intensität fluoreszierender Bilder zu messen.
- Führen Sie pipeline mit ausgewählten Tiff-Bildern aus, um die durchschnittliche Fluoreszenzintensität von Fluoreszenztröpfchenbildern zu extrahieren. Führen Sie das Experiment in dreifacher Ausfertigung durch, wobei jeder Satz aus 5.000 Tröpfchen für die Analyse besteht.
- Wenden Sie den 'definetherain' -Algorithmus (http://definetherain.org.uk/) an, um die positiven und negativen Tröpfchencluster zu identifizieren. Die positiven Werte sollten innerhalb von 3 Standardabweichungen des Mittelwerts liegen. Dadurch wird die Schwellenintensität positiver Tröpfchen bestimmt, die für die Zählung verwendet werden sollen.
- Verwenden Sie erneut die Bildanalysesoftware, um eine neue Pipeline zu implementieren, in der der Fluoreszenzschwellenwert für jedes Genziel wie im vorherigen Schritt definiert festgelegt wird.
- Laden Sie hellfeld- und fluoreszierende Bilder in die Pipeline hoch. Fügen Sie die Module 'ColorToGray', 'RescaleIntensity', 'Threshold', 'IdentifyPrimaryObjects', 'MeasureObjectSizeShape', 'FilterObjects' und 'ExportToSpreadsheet' hinzu. Erstellen Sie einzigartige Module für Hellfeldbilder und jeden Fluoreszenzfilter für Tröpfchen.
- Skalieren Sie die Bildintensitätsskala von 0 bis 1 für jede Fluoreszenzbildgruppe neu. Legen Sie dann den Schwellenwert fest, um die Objekte über dem festgelegten Schwellenwert zu identifizieren und zu zählen. Fügen Sie bei Bedarf das Modul 'ExpandOrShrinkObjects' hinzu, um Objekte zu verkleinern, um das Zählen und Identifizieren von Tröpfchen im hellfeldigen Zuspiel zu erleichtern.
- Identifizieren Sie nur Objekte innerhalb der ausgewählten Größe von 20-30 Pixeln, und filtern Sie die gezählten Objekte, um nur diese Objekte mit einem bestimmten Durchmesser (d. h. Tröpfchen im Durchmesserbereich von 75 m) und einer runden kugelförmigen Exzentrizität von 0,5 und darunter beizubehalten.
- Exportieren Sie die Ergebnisse in eine Tabelle, in der die Gesamtzahl der Tröpfchen aus Hellfeldbildern sowie die Tröpfchenanzahl aller verwendeten Fluoreszenzkanäle aufgeführt ist. insbesondere Cy5 für C-LESS-Gen, HEX für methyliertes CD3Z-Gen und FAM für methyliertes FOXP3-Gen (siehe Ergänzende Informationen für rohexperimentelle Daten, die für den vorgestellten mdPCR-Test gewonnen wurden).
- Berechnen Sie das Verhältnis der negativen Tröpfchen für jedes Genziel und wenden Sie die Poisson-Verteilung an, um die entsprechenden Kopien pro Tröpfchen (CPD) mit Gleichung 1 zu erhalten:
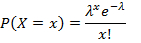
wobei x die Anzahl der Tröpfchen mit 0, 1, 2 oder mehr Molekülen und den CPD-Wert darstellt. - Berechnen Sie die absolute Zielkonzentration unter Berücksichtigung des Verhältnisses zwischen dem CPD-Wert und dem Tröpfchenvolumen in Schritt 5.20 mit Gleichung 2:
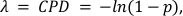
wobei der Wert (1p) den Bruchteil der negativen Tröpfchen darstellt. - Berechnen Sie den Prozentsatz von CD3+ T-Cells und CD4+ CD25+ T-Regs, indem Sie die jeweiligen CPD-Werte der methylierten CD3Z- und FOXP3-Gene durch den C-LESS- oder Gesamtzellen-CPD-Wert dividieren (siehe Ergänzende Informationen für CPD-Berechnungen aus Rohdaten).
- Vergleichen Sie diese Prozentwerte mit denen, die bei der Immunfluoreszenz-Bildgebung unter Verwendung von Antikörpern für CD3+ T-Cell und CD4+ CD25+ T-Reg-Zählung erhalten wurden.
Ergebnisse
Das Mikrofluid-Tröpfchengeneratorgerät tPE wurde mit dem beschriebenen Protokoll hergestellt, wie in Abbildung 1dargestellt. Eine Transparenzmaske wurde in der Photolithographie verwendet, um Silizium (Si) Master zu erhalten. Weiche Lithographie wurde durchgeführt, um eine inverse PDMS-Replik des Si-Meisters zu erhalten, die dann verwendet wurde, um die Epoxidform herzustellen. Epoxid-Vorläufer wurde auf das PDMS gegossen und zu Vernetzung und Aushärten ausgehärtet. Diese Form, die die...
Diskussion
Das vorgestellte experimentelle Protokoll und die vorgestellten Methoden ermöglichen die hauseigene mdPCR mit einem hergestellten TPE-Tröpfchengenerator, einem thermischen Cycler und einem Fluoreszenzmikroskop. Das hergestellte Gerät mit weicher TPE-zu-TPE-Bindung bietet hydrophobe Oberflächeneigenschaften, die über alle Kanalwände gleichmäßig sind, so dass das Endgerät keine Oberflächenbehandlung für die spätere Verwendung als Tröpfchengenerator erfordert. Dieses Material wurde routinemäßig in Point-of-Ca...
Offenlegungen
Es gibt keine Konflikte zu deklarieren.
Danksagungen
Die Autoren würdigen die finanzielle Unterstützung durch den National Research Council of Canada.
Materialien
| Name | Company | Catalog Number | Comments |
| Bio-Rad, Mississauga, ON | TFI0201 | PCR tube | |
| RAN Biotechnologies, Beverly, MA | 008-FluoroSurfactant | Fluoro-surfactant | |
| Silicon Quest International, Santa Clara, CA | |||
| Oxford Instruments, Abingdon, UK | EMCCD camera | ||
| Thermo Fisher Scientific, Waltham, MA | MA5-16728 | ||
| Thermo Fisher Scientific, Waltham, MA | 22-8425-71 | ||
| CellProfiler | Used for fluorescence image analysis | ||
| Nikon, Japan | 10x objective | ||
| American Type Culture Collection (ATCC), Manassas, VA | PCS-800-011 | ||
| Ramé-Hart Instrument Co. (Netcong, NJ) | p/n 200-U1 | ||
| Fisher, Canada | |||
| Vitrocom, NJ, USA | 5015 and 5010 | Borosilicate capilary tube | |
| (http://definetherain.org.uk/) | |||
| Hamamatsu, Japan | LC-L1V5 | DEL UV light source | |
| Dolomite | 3200063 | Disposable fluidic tubing | |
| Dolomite | 3200302 | Disposable fluidic tubing | |
| IDT, Coralville, IA | |||
| Nikon, Melville, NY | Upright light microscope | ||
| Cytec Industries, Woodland Park, NJ | |||
| EV Group, Schärding, Austria | |||
| Zymo Research, Irvine, CA | D5030 | ||
| Photron, San Diego, CA | |||
| IDT, Coralville, IA | |||
| Gersteltec, Pully, Switzerland | SU-8 photoresist | ||
| Fineline Imaging, Colorado Springs, CO | |||
| Qiagen, Hilden, Germany | 203603 | ||
| Image J | Used to assess droplet diameter | ||
| Anachemia, Montreal, QC | |||
| Excelitas, MA, USA | Broad-spectrum LED fluorescent lamp | ||
| Galenvs Sciences Inc., Montreal, QC | DE1010 | ||
| Hexpol TPE, Åmål, Sweden | Thermoplastic elastomer (TPE) | ||
| Thermo Fisher Scientific, Waltham, MA | 13-400-518 | ||
| Nikon, Japan | Used for image acquisition | ||
| 3M, St Paul, MN | Carrier Oil | ||
| Thermo Fisher Scientific, Waltham, MA | R37605 | Blue fluorescent live cell stain (DAPI) | |
| IDEX Health & Science, Oak Harbor, WA | P-881 | PEEK fittings | |
| Sigma-Aldrich, Oakville, ON | 806552 | ||
| Dow Corning, Midland, MI | |||
| ThinkyUSA, CA, USA | ARV 310 | ||
| Ihc world, Maryland, USA | IW-125-0 | ||
| Zinsser NA, Northridge, CA | 2607808 | ||
| Cetoni GmbH, Korbussen, Germany | |||
| Sigma-Aldrich, Oakville, ON | 484431 | ||
| Bio-Rad, Mississauga, ON | 1861096 | ||
| Hitachi High-Technologies, Mississauga, ON | |||
| Nikon, Melville, NY | Inverted microscope | ||
| Nikon, Japan | |||
| Loctite | AA 352 |
Referenzen
- Teitell, M., Richardson, B. DNA methylation in the immune system. Clinical Immunology. 109 (1), 2-5 (2003).
- Suarez-Alvarez, B., Rodriguez, R. M., Fraga, M. F., López-Larrea, C. DNA methylation: A promising landscape for immune system-related diseases. Trends in Genetics. 28 (10), 506-514 (2012).
- Suárez-Álvarez, B., Raneros, A. B., Ortega, F., López-Larrea, C. Epigenetic modulation of the immune function: A potential target for tolerance. Epigenetics. 8 (7), 694-702 (2013).
- Kondilis-Mangum, H. D., Wade, P. A. Epigenetics and the adaptive immune response. Molecular Aspects of Medicine. 34 (4), 813-825 (2013).
- Zouali, M. . The Autoimmune Diseases. , (2014).
- Wiencke, J. K., et al. A comparison of DNA methylation specific droplet digital PCR (mdPCR) and real time qPCR with flow cytometry in characterizing human T cells in peripheral blood. Epigenetics. 9 (10), 1360-1365 (2014).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Analytical Chemistry. 83 (22), 8604-8610 (2011).
- Pinheiro, L. B., et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification. Analytical Chemistry. 84 (2), 1003-1011 (2012).
- Roy, E., Galas, J. C., Veres, T. Thermoplastic elastomers for microfluidics: Towards a high-throughput fabrication method of multilayered microfluidic devices. Lab on a Chip. 11 (18), 3193-3196 (2011).
- Roy, E., et al. From cellular lysis to microarray detection, an integrated thermoplastic elastomer (TPE) point of care Lab on a Disc. Lab on a Chip. 15 (2), 406-416 (2015).
- Malic, L., et al. Epigenetic subtyping of white blood cells using a thermoplastic elastomer-based microfluidic emulsification device for multiplexed, methylation-specific digital droplet PCR. Analyst. 44 (22), 6541-6553 (2019).
- Malic, L., et al. Polymer-based microfluidic chip for rapid and efficient immunomagnetic capture and release of Listeria monocytogenes. Lab Chip. 15 (20), 3994-4007 (2015).
- Malic, L., Morton, K., Clime, L., Veres, T. All-thermoplastic nanoplasmonic microfluidic device for transmission SPR biosensing. Lab Chip. 13 (5), 798-810 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten