È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Metilazione specifica Multiplex Droplet PCR utilizzando Polymer Droplet Generator Device per la diagnostica ematologica
In questo articolo
Riepilogo
Marcatori epigenetici sono utilizzati per sottotipi di globuli bianchi (WBC) attraverso la quantificazione dei modelli di metilazione del DNA. Questo protocollo presenta un metodo di reazione a catena di polimerasi a goccia multiplex (mdPCR) utilizzando un dispositivo microfluidometro termoplastico (TPE) per la generazione di droplet che consente una quantificazione mirata precisa e multiplex specifica della metilazione dei conteggi differenziali WBC.
Abstract
Viene descritto un flusso di lavoro PCR (mdPCR) a goccia multiplexed e un protocollo dettagliato per determinare il numero differenziale dei globuli bianchi a base epigenetica (WBC), insieme a un dispositivo di generazione di goccioline microfluidiche di elastimero termoplastico (TPE). Marcatori epigenetici sono utilizzati per la sottotipazione WBC che è di importante valore prognostico in diverse malattie. Ciò si ottiene attraverso la quantificazione dei modelli di metilazione del DNA di specifiche regioni ricche di CG nel genoma (CpG loci). In questo articolo, il DNA trattato con bisulfite da cellule mononucleari del sangue periferico (PBMC) è incapsulato in goccioline con reagenti mdPCR, tra cui primer e sonde fluorescenti idrolisi specifiche per i loci CpG che sono correlati alle sottopposizioni WBC. L'approccio multiplex consente l'interrogatorio di molti loci CpG senza la necessità di reazioni mdPCR separate, consentendo una determinazione parametrica più accurata delle sottoppole WBC utilizzando l'analisi epigenetica dei siti di metilazione. Questa quantificazione precisa può essere estesa a diverse applicazioni ed evidenzia i benefici per la diagnosi clinica e la successiva prognosi.
Introduzione
L'analisi della composizione dei globuli bianchi (WBC) è tra i test di laboratorio più richiesti nella diagnostica ematologica. Il conteggio differenziale dei leucoc primi è un indicatore per uno spettro di malattie tra cui infezione, infiammazione, anemia e leucemia, ed è sotto indagine come biomarcatore prognostico precoce per diverse altre condizioni. Il Gold Standard nella sottotipizzazione WBC comporta l'immunostaining e/o la citometria di flusso che richiedono anticorpi fluorescenti costosi e soggetti a instabilità e sono spesso fortemente dipendenti dalla competenza dell'operatore nella preparazione del campione. Inoltre, questo metodo è applicabile solo ai campioni di sangue fresco, in modo che i campioni non possano essere congelati per la spedizione o per un'analisi successiva.
I marcatori epigenetici sono recentemente emersi come potenti strumenti analitici per lo studio delle variazioni fenotiptiche. Successivamente, è stato dimostrato che le popolazioni di leucocici umani hanno modelli di metilazione del DNA del lignaggio cellulare che consentono la caratterizzazione precisa dei sottoinsiemi WBC. La sottotipazione basata su marcatori epigenetici fornisce un'alternativa promettente che non dipende dalla raccolta di campioni di sangue fresco o da anticorpi costosi e può essere sfruttata come biomarcatore per l'insorgenza della malattia e la suscettibilità1,2,3,4,5.
Sono stati condotti studi a livello di genoma per un'ampia mappatura di regioni specifiche ricche di CG metilate nel genoma (isole CpG) in sottotipi di leucocideti per identificare marcatori epigenetici candidati specifici per i sottotipi di leucocide. I protocolli PCR sono stati sviluppati per questo motivo per le regioni geniche metilate, ad esempio CD3 e FOXP3, corrispondenti rispettivamente aCD3, cellule T eCD4, CD25, cellule T regolamentari (T-Regs).+ Wiencke et al. hanno dimostrato l'utilità di gocciolina duplex PCR per la sottotipazione epigenetica di T-Cells, producendo risultati che altamente correlati con l'analisi di ordinamento cellulare attivato dal flusso (FACS)6. Questo metodo di analisi genetica quantitativa si basa sulla partizionamento del modello di molecole di acido nucleico e reagenti PCR in migliaia di goccioline discrete, volumetricamente definite, di dimensioni sub-nanoliter contenenti zero, una o più copie di acido nucleico bersaglio, utilizzando emulsioni acqua-in-olio abilitate da microfluidica7,8. L'amplificazione PCR viene eseguita all'interno di ogni singola goccia e viene misurata l'intensità di fluorescenza del punto finale di ogni goccia, consentendo la quantificazione assoluta degli obiettivi presenti nel campione. Droplet PCR è stato stabilito per essere più preciso, preciso, e tecnicamente più semplice di qPCR standard, rendendolo un metodo più favorevole dna metilazione-based per la valutazione clinica delle cellule T. Sebbene manchi contemporaneamente una metodologia di sottotipazione in rapida crescita, l'analisi epigenetica multiplessa per sondare varie regioni CpG metilate. Ciò è necessario per i conteggi differenziali di leucocenza di routine.
In questo caso, un dispositivo microfluidico a goccia termoplastica elastomero (TPE) viene presentato e impiegato per la gocciolina multiplex PCR (mdPCR) specifica della metilazione. La tecnologia è stata utilizzata per delineare sottotipi specifici di leucocici,CD3 , T-Cells eCD4 , CD25 , T-Regs, sulla base di modelli di metilazione del DNA di linea cellulare, cioè, variazione epigenetica delle regioni CD3 e FOXP3 CpG, rispettivamente. Un protocollo dettagliato per l'estrazione del DNA, la conversione dei bisulfiti e mdPCR è descritto in concerto con un metodo di fabbricazione per un dispositivo di generazione di goccioline TPE. I risultati rappresentativi del metodo vengono confrontati con quelli della colorazione dell'immunofluorescenza evidenziando l'utilità dell'approccio proposto.
Protocollo
Tutti gli esperimenti condotti in questo studio che hanno coinvolto campioni umani sono stati approvati dal Comitato Etico del NRC e sono stati fatti secondo le politiche del NRC che disciplinano i soggetti umani che seguono le linee guida di ricerca applicabili e sono conformi alle leggi del Québec, Canada.
1. Preparazione cellulare
- Scongelare immediatamente le cellule mononucleari del sangue periferico umano (PBFC) congelate mettendo il criovial in un bagno d'acqua a 37 gradi centigradi per 5 minuti.
- Invertire il criovial due volte per risundere delicatamente le cellule e utilizzando una pipetta da 1 mL trasferire la sospensione cellulare in un tubo conico da 15 mL.
- Aggiungere 10 mL di mezzo di crescita pre-riscaldato (37 gradi centigradi) integrato con il 10% di siero bovino fetale (RPMI-1640 - 10% FBS) al tubo da 15 mL contenente i PBMC.
- Centrifugare le sospensioni delle celle in una centrifuga oscillante a temperatura ambiente ad una velocità di 330 x g per 10 min con accelerazione rapida e il freno in alta.
- Una volta che lo spin è finito, decantare con cura il supernatant. Rispendere il pellet cellulare in 3 mL di salina tamponata di fosfati (PBS) pH 7.2, contenente acido etilenediaminetetraacetico (EDTA) da 2 mM toccando il lato dei tubi.
- Mescolare le cellule invertendo il tubo con il tappo strettamente chiuso.
- Preparare due microtubi da 1,5 mL e utilizzare una pipetta aliquot 1,5 mL della sospensione cellulare in ogni tubo, uno dei quali viene utilizzato per la successiva colorazione immunofluorescenza e uno per l'estrazione del DNA.
2. Protocollo di colorazione e imaging dell'immunofluorescenza
- Riesposo delle cellule (da 1,7) in 200 L di buffer PBS contenente 0,1% di azide di sodio e 2% di FBS e regolare la concentrazione finale della sospensione cellulare ad un massimo di 2 x 107 celle/mL.
- Dividere le sospensioni delle celle mediante pipettamento di 100 L in due microtubi separati da 1,5 mL.
- Aggiungere il volume 20 L di anti-Hu CD3/CD4 coniugato con Fluorescein isothiocyanate (FITC) e Phycoerythrin (PE) a un tubo e anti-Hu CD4/CD25 coniugati con FITC e PE (vedi Tabella dei Materiali)al secondo tubo, rispettivamente.
- Aggiungere 1 goccia di macchia cellulare fluorescente blu (vedi Tabella dei materiali) ad ogni tubo.
- Incubare a temperatura ambiente utilizzando un rotatore di tubi per 2 h. Proteggere dalla luce.
- Centrifugare le sospensioni delle celle a temperatura ambiente a 330 x g per 10 min con accelerazione rapida e il freno in alto.
- Decantare il supernatant e rispendere attentamente il pellet cellulare toccando il tubo. Aggiungere 1 mL di PBS, pH 7.2 contenente 2 mM EDTA. Assicurarsi che il tappo sia strettamente chiuso, mescolare le celle invertendo il tubo 2x.
- Ripetere i passaggi 2.6 e 2.7 per tre volte.
- Le celle di riespette in 20 L di PBS pH 7.2 contenenti 2 mM EDTA.
- Pipette 10 caduta di 10 gradi della sospensione cellulare su un microscopio borosilicato e attendere 2 min per le cellule di sedimentarsi lentamente al fondo della goccia.
- Posizionare con attenzione un coperchio di vetro sopra il vetrato e posizionare il vetrato sul palco di un microscopio invertito.
- Registrare le immagini delle cellule utilizzando un obiettivo 10x e una telecamera EMCCD collegata al microscopio per ciascuno dei fluorofori per entrambi i campioni di sospensione cellulare.
- Contare manualmente le celle con etichetta fluorescente (vedere Informazioni supplementari per i dati non elaborati).
- Prendete il rapporto tra le cellule con etichetta anti-Hu CD3 e anti-Hu CD4/CD25 con celle macchiate da DAPI per ottenere le proporzioni di CD3- cellule T e CD4- CD25- T-Regs al leucocato totale.
3. Estrazione del DNA e conversione dei bisulfiti
- Estrazione del DNA
NOTA: Estrarre il DNA dai PBMC preparato nella sezione 1 utilizzando un kit di purificazione del DNA magnetico (vedi Tabella dei materiali) seguendo le procedure fornite dal produttore.- In un tubo da 1,5 mL sospendi le celle in 100 L di PBS e aggiungi 20 L di Proteinase K e 400 L di Lysis/Binding buffer. Mescolare pipettando 10x, quindi eseguire l'incubazione a temperatura ambiente per 5 min.
- Catturare il complesso di perline di DNA posizionando il tubo su un rack magnetico per 1-2 min, quindi rimuovere con attenzione e scartare il supernante.
- Rimuovere il tubo contenente il complesso di perline di DNA dal rack magnetico e risundere le perline in 600 L di Wash Buffer #1 per lavare via qualsiasi legame non specifico.
- Posizionare nuovamente il tubo sul rack magnetico e rimuovere con attenzione e scartare il supernante.
- Ripetere i passaggi 3.1.3 e 3.1.4 con 600 L di Buffer lavaggio #2.
- Lasciare il tubo aperto all'aria per 1 min.
- Rimuovere il tubo dal rack magnetico ed elute il DNA erogando 100 L di Elution Buffer e pipettando il complesso DNA/perline su e giù 20x.
- Posizionare nuovamente il tubo contenente il DNA eldato sul rack magnetico e incubare per 1-2 minuti per separare le perline magnetiche dal DNA eldato.
- Trasferire la soluzione di DNA purificato el pubblicizzato in un nuovo tubo pulito.
- Valutare la concentrazione del campione di DNA purificato misurando l'assorbimento a 260 nm utilizzando uno spettrofotometro.
- Conversione Bisulfite
NOTA: eseguire la conversione del bisulfite sul DNA purificato utilizzando un kit di metilazione (vedere Tabella dei materiali) seguendo le procedure fornite dal produttore.- A 20 L di campione di DNA (200-500 ng) in un tubo PCR aggiungere 130 L di reagente di conversione. Mescolare bene e girare giù brevemente.
- Trasferire il tubo PCR in un ciclo termico ed eseguire il protocollo di ciclismo come segue: 98 gradi centigradi per 8 min; 54 gradi centigradi per 60 minuti e tenere premuto a 4 gradi centigradi.
- Aggiungere 600 L del buffer di rilegatura a una colonna di cromatografia iione (IC) inserita in un tubo di raccolta.
- Aggiungere il campione di DNA alla colonna IC contenente il buffer di rilegatura e mescolare invertendo il tubo più volte. Centrifuga per 30 s a tutta velocità. Eliminare il flusso raccolto.
- Alla colonna ora aggiungere 100 L di Wash Buffer. Eseguire la centrifugazione come descritto al punto 3.2.4 ed eliminare il flusso.
- Aggiungere 200 l di Buffer di Desulfonation ed eseguire l'incubazione a temperatura ambiente per 15-20 min. Centrifugare e scartare il flusso-through come descritto nei passaggi precedenti.
- Aggiungere 200 L di Wash Buffer alla colonna. Centrifuga a tutta velocità per 30 s e scarta il flusso-through.
- Ripetere il passaggio 3.2.7.
- Trasferire la colonna in un nuovo tubo di raccolta da 1,5 mL e aggiungere 100 L di acqua di grado PCR sulla membrana della colonna. Centrifuga a tutta velocità per 1 min per elute il DNA.
- Valutare la concentrazione del campione di DNA convertito in bisulfite misurando l'assorbimento a 260 nm utilizzando uno spettrofotometro.
NOTA: utilizzare un valore di 40 g/mL per l'assorbimento a 260 nm e 1,0. - Conservare il DNA a -20 gradi centigradi per l'immagazzinamento a breve termine o a -70 gradi centigradi per l'immagazzinamento a lungo termine.
4. Fabbricazione del dispositivo di generazione delle goccioline
NOTA: un dispositivo microfluidico utilizzato per la generazione di droplet (file CAD fornito nelle informazioni supplementari) è stato fabbricato in un ambiente di stanza pulita (classe 1.000) in elastomero termoplastico (vedere Tabelladei materiali ) utilizzando il rilievo a caldo generato dal seguente protocollo.
- Fabbricazione di stampi SU-8
- Preparare uno stampo SU-8 su un wafer di silicio da 6" utilizzando la fotolitografia standard come descritto di seguito.
- Pulire un wafer di silicio da 6" utilizzando plasma di ossigeno a 500 W per 10 s.
- Spin-coat SU-8 resistere sul wafer di silicio a 900 rpm per 40 s per ottenere uno spessore totale della pellicola di 100 m.
- Mettere il wafer su una piastra calda e pre-cuocere per 15 min a 65 gradi centigradi, seguito da 2 h a 95 gradi centigradi.
- Esporre alla luce UV a 365 nm (Hg i-line) attraverso una fotomaschera trasparenza ad alta definizione utilizzando una dose di esposizione di 1.000 mJ/cm2.
- Mettere il wafer su un piatto caldo e post-cuocere per 15 minuti a 65 gradi centigradi, seguito da 40 min a 95 gradi centigradi.
- Sviluppare immergendosi in acetato di etere monometile di glilonico propilene (PGMEA) per 5 minuti.
- Risciacquare con PGMEA e isopropanolo e asciugare con un flusso di gas azoto.
- Mettere il wafer su un piatto caldo e cuocere duro per 2 h a 135 gradi centigradi.
- Silanizzare il wafer nel desiccatore sottovuoto contenente una goccia di agente silanizzante (tricholoro perfluorooctyl silane) posto su un vetro di vetro adiacente per 2 h.
NOTA: Questo viene fatto per rendere le silane formano un monostrato sulla superficie del master SU-8. Tricholoro perfluorooctyl silane deve essere sempre maneggiato nel cappuccio di fumi e tenuto lontano dalle fonti d'acqua.
- Replica Polydimethylsiloxane (PDMS)
- Preparare i prepolimeri liquidi del PDMS (vedere Tabella dei materiali) a un rapporto 10:1 w/w della base elastomero per curare l'agente in una tazza di plastica.
- Mettere la tazza in un mixer a vuoto centrifugo planetario per mescolare e degas la miscela PDMS.
- Versare la miscela PDMS sullo stampo posto nel supporto metallico personalizzato che previene la fuoriuscita di resina e curare a 65 gradi centigradi per 2 h.
- Utilizzando una pinzetta, staccare con cura lo stampo PDMS dal master SU-8.
- Muffa epossina
NOTA: uno stampo epossidy è stato fabbricato dal master SU-8/silicon utilizzando un processo di replica intermedio con PDMS.- Preparare la resina epossidica (vedi Tabella dei Materiali) utilizzando un rapporto 100/83 w/w di resina/induritore.
- Degas la miscela sotto pressione ridotta utilizzando un forno di essiccazione sottovuoto per 30 min.
- Versare la resina sulla replica PDMS e curare a 80 gradi centigradi per 12 h.
- Togliere lo stampo epossico stagionato dalla replica PDMS, posizionarsi su una piastra calda e cuocere duro per 2 h a 120 gradi centigradi.
- Dispositivo TPE
- Pellet di estrusione di TPE (vedi Tabella dei Materiali) a 165 gradi centigradi in fogli spessi 2,0 mm e 7" di lunghezza diversi metri e conservarli come rotolo per un uso futuro.
- Tagliare il foglio TPE dal rotolo utilizzando le forbici in un quadrato di 7".
- Posizionare il foglio TPE tra lo stampo epossidato e un wafer di silanizzato non modellato (vedere il punto 4.1.10 per la procedura di silanizzazione del wafer).
- Eseguire l'in rilievo a caldo ad una temperatura di 125 gradi centigradi, una forza applicata di 10 kN e una pressione di 10–2 mbar per 10 min.
- Demold con attenzione a temperatura ambiente utilizzando spray al metanolo per separare il TPE goffrato dal wafer di silicio e lo stampo epossidy.
- Tagliare un altro foglio quadrato di TPE da 7" e posizionarlo tra due wafer di silanizzato non modellati.
- Eseguire l'in rilievo a caldo ad una temperatura di 140 gradi centigradi, una forza applicata di 10 kN e una pressione di 10–2 mbar per 10 min per formare una superficie planare per chiudere i canali e sigillare il dispositivo.
- Demold con attenzione a temperatura ambiente utilizzando spray al metanolo per separare il TPE in rilievo dai due wafer di silicio.
- Tagliare ciascuno dei fogli TPE in rilievo alle dimensioni del dispositivo utilizzando una lama del medico.
- Punzona i fori di accesso per i canali di ingresso e di uscita nel dispositivo strutturato utilizzando un ago perforato biopsy da 1 mm con uno stantuffo.
- Racchiudere i canali mettendo un dispositivo TPE planare a contatto diretto con i canali a temperatura ambiente.
- Facoltativamente, mettere in forno a 70 gradi centigradi per 2 h per promuovere l'incollaggio del dispositivo.
- Montare i fori di accesso con un tubo fluido usa e getta (I.D. 0,25 mm, O.D. 0.8 mm). Sigillare le articolazioni utilizzando una colla epossiccia per garantire una manipolazione a prova di perdita.
5. Generazione di droplet e PCR
NOTA: la tabella 1 delinea le informazioni sui primer in avanti e inverso insieme alle sonde idrolisi a doppia dissetazione per i geni C-LESS, CD3 e Foxp3, necessarie per l'amplificazione multiplex dei bersagli genici demetilati.
- Preparare il mix principale come descritto nella Tabella 2.
- Scongelare tutti i componenti del mix master ad eccezione del mix di enzimi. Mescolare accuratamente il mix master pipettando verso l'alto e girare brevemente verso il basso.
- Aggiungere il volume appropriato (1 L) di DNA convertito di bisulfite (dalla sezione 3.2) alla miscela principale in un tubo PCR. Mescolare la reazione pipettando verso l'alto verso il basso e girare giù brevemente.
- Collegare tubi fluidici usa e getta (I.D. 0,25 mm, O.D. 1.6 mm) a due siringhe di vetro di precisione (volume 250 L) utilizzando raccordi PEEK.
- Precompilare una siringa di vetro di precisione con 250 L di olio di supporto contenente il 5% di fluoro-surfactant.
- Precompilare un'altra siringa di vetro di precisione con 50 L di olio di supporto prima di caricare 100 L della miscela PCR per garantire l'erogazione dell'intero volume del campione durante l'emulsione.
- Impostare un dispositivo microfluidico a goccioline su un microscopio leggero verticale dotato di una telecamera ad alta velocità per osservare e registrare la formazione di goccioline in tempo reale.
- Posizionare le siringhe precompilate sulla pompa di siringa programmabile e utilizzando l'unione PEEK con raccordi (vedi Tabella dei Materiali), collegare il tubo delle siringhe al tubo dei rispettivi canali di ingresso del dispositivo microfluidico della gocciolina.
- Posizionare il tubo dall'uscita del generatore di goccioline all'interno di un tubo PCR da 0,5 mL.
- Regolare la velocità di flusso della pompa di siringa a 2 L/min e lasciare che la dimensione della gocciolina si stabilizzi prima di raccogliere l'emulsione risultante.
- Raccogliere l'emulsione e trasferire 75 L in un tubo PCR da 0,2 mL per il ciclo termico.
- Assicurarsi che il contenuto di olio nel tubo PCR corrisponda strettamente al volume della fase di dispersione al fine di evitare coalescenza delle goccioline durante il ciclo termico.
- Posizionare il tubo PCR da 0,2 mL nel cycler termico ed eseguire il protocollo di ciclismo come segue: preriscaldamento a 95 gradi centigradi per 5 min, quindi 45 cicli di denaturazione a 95 gradi centigradi per 15 s e annealing/estensione a 60 gradi centigradi per 30 s.
- Utilizzare l'emulsione rimanente per riempire un tubo capillare borosilicato (profondità di 100 m) con un profilo rettangolare al fine di immagini goccioline e valutare il diametro della gocciolina.
- Posizionare il tubo borosilicato riempito con l'emulsione su un faro al microscopio. Utilizzare un microscopio invertito dotato di una fotocamera EMCCD e obiettivo 10x per registrare immagini di campo luminoso delle goccioline.
- Misurare il diametro del droplet utilizzando un software di analisi delle immagini come descritto di seguito.
- Impostare la scala della distanza nota in micron corrispondente al numero di pixel nell'immagine selezionando il pulsante 'Analizza' e quindi 'Imposta scala'.
- Convertire l'immagine in scala di grigi selezionandoilpulsante ' Immagine ' e quindi 'Tipo'. Se necessario, regolare la luminosità e il contrasto. Impostare una soglia manuale per delineare e riempire i cerchi selezionando il pulsante 'Immagine' e quindi 'Regola soglia'.
- Analizzate le particelle selezionando il pulsante 'Analizza particelle' e impostando la circolarità su 0,75 – 1.
- Ottenere l'area risultante e il diametro delle goccioline misurate che viene visualizzato automaticamente nel software.
- Calcolare il diametro medio del goccioline e ipotizzando una goccia sferica, stimare il volume della partizione, che verrà utilizzato per calcolare la concentrazione di destinazione assoluta.
- Assicurarsi che le goccioline siano monodisperse analizzando il coefficiente di variazione (CV) che viene preso come rapporto tra le deviazioni standard e i valorimedio (k - 1), per il diametro della goccia (< 3%).
6. Imaging della fluorescenza e analisi delle immagini
- Imaging della fluorescenza
- Dopo l'amplificazione, trasferire l'emulsione PCR in tubo capillare borosilicate (profondità di 50 m) con un profilo rettangolare al fine di disporre le goccioline in un monostrato ravvicinato per l'imaging.
- Fissare i capillari riempiti su un scivolo al microscopio e sigillare entrambi i lati utilizzando un adesivo acrilico UV. Applicare una sorgente di luce UV sull'adesivo UV facendo attenzione a non illuminare l'emulsione per evitare lo sbiancamento del campione.
- Caricare il vetrato di vetro su un microscopio invertito dotato di una fotocamera EMCCD e di un obiettivo 10x.
- Utilizzando il software di imaging al microscopio, selezionare Acquisisci Dal vivo - Veloce per avviare l'acquisizione della fotocamera in tempo reale, osservare il campione e assicurarsi che la larghezza del capillare sia catturata.
- Impostare la lampada da campo luminoso per l'illuminazione diascopica a 3,5 V.
- Impostare la lampada fluorescente LED ad ampio spettro per l'illuminazione episcopica con un'intensità del 20%.
- Regolare manualmente l'impostazione di acquisizione per tutte le lunghezze d'onda (campo luminoso, FAM, HEX e Cy5) selezionando la casella di controllo Calibrazione Comando Configurazioni ottiche nel software di imaging. Per ogni fluoroforo, il cubo del filtro a fluorescenza corrispondente deve essere commutato manualmente. Per mantenere lo stesso percorso ottico, è necessario utilizzare un cubo di filtro per l'imaging del campo luminoso.
- Regolare manualmente il tempo di esposizione prima dell'acquisizione dell'immagine utilizzando Acquisisci Comando Impostazioni fotocamera per ogni fluoroforo come riepilogato nella Tabella 3. Impostare la modalità di lettura EM guadagno 17 MHz a 16 bit con un moltiplicatore di guadagno di 100 nel menu 'Impostazionidi acquisizione ' del software.
- Nella finestra LUT del software, impostare la scala LTs in modo che il segnale di trasmissione sia racchiuso all'interno dell'intervallo impostato. In questo esperimento, la scala è stata impostata da 500 a 12.000 circa.
- Automatizza l'acquisizione utilizzando un programma di acquisizione multipunto utilizzando sia la scansione XY che le opzioni di lunghezza d'onda multiple in quell'ordine specifico. Assicurarsi che lo stage si muova nella posizione iniziale, catturare tutte le diverse lunghezze d'onda, quindi passare alla posizione successiva.
- In primo luogo, impostare la scansione XY personalizzando il profilo di scansione XY nel menu di scansione XY del software. In 'Custom Multipoint Definition', scegliere la casella di definizione dell'immagine grande e impostarla su 40,0 x 1,0 mm approssimativamente (la lunghezza del riempimento dell'emulsione). Utilizzare la sovrapposizione dell'1%.
- Attivare l'opzione 'Usa superficie di messaa fuoco ' e impostare la curva della superficie di messa a fuoco regolando il piano di messa a fuoco in diversi punti del campione utilizzando la manopola di messa a fuoco sul microscopio.
- In secondo luogo, impostare la scansione a lunghezza d'onda multipla selezionando la scheda di scansione. Aggiungere ciascuna delle configurazioni ottiche create nel punto 6.1.7 per ogni lunghezza d'onda.
- Fare clic sull'opzione per chiuderel'otturatoreattivo durante il movimento dello stage e durante la modifica del filtro per evitare di sbiancare il campione ed eseguire l'acquisizione premendo il pulsante ' Avvia esecuzione '. Esportate ogni fotogramma di acquisizione in file tiff e dividete ogni canale in un file separato utilizzando l'opzione Dividi più file e applicando le impostazioni LUT salvate. Utilizzare il nome del punto e l'opzione del nome del canale per differenziare facilmente ogni file di immagine.
- Analisi delle immagini
- Ordina tutte le immagini acquisite in base ai filtri di fluorescenza e a campo luminoso da caricare su un software di analisi delle immagini open source.
- Utilizzare il software di analisi delle immagini per creare una pipeline al fine di identificare tutte le goccioline utilizzando immagini a campo luminoso, quindi misurare l'intensità delle goccioline fluorescenti associate.
- Per eseguire la pipeline, caricare immagini tiff di campo luminoso e fluorescenza, quindi aggiungere i moduli 'ColorToGray', 'IdentifyPrimaryObjects', 'MeasureObjectIntensity' e 'ExportToSpreadsheet'. Utilizzare le immagini a goccia a campo luminoso per identificare gli oggetti, quindi utilizzare gli oggetti come maschera per misurare l'intensità delle immagini fluorescenti.
- Eseguire la pipeline con le immagini tiff selezionate per estrarre l'intensità media di fluorescenza delle immagini a goccia di fluorescenza. Condurre l'esperimento in tripliceto, con ogni set composto da 5.000 goccioline per l'analisi.
- Applicarel'algoritmo ' definetherain' (http://definetherain.org.uk/) per identificare i cluster di droplet positivi e negativi. Gli aspetti positivi devono essere entro 3 deviazioni standard della media. Questo determina l'intensità di soglia delle goccioline positive da utilizzare per il conteggio.
- Utilizzare nuovamente il software di analisi delle immagini per implementare una nuova pipeline in cui la soglia di fluorescenza per ogni target genico è impostata come definito nel passaggio precedente.
- Carica immagini luminose e fluorescenti sulla pipeline. Aggiungere i moduli 'ColorToGray', 'RescaleIntensity', 'Threshold', 'IdentifyPrimaryObjects', 'MeasureObjectSizeShape', 'FilterObjects' e 'ExportToSpreadsheet'. Crea moduli unici per immagini a campo luminoso e ogni filtro a fluorescenza per le goccioline.
- Ridimensiona la scala dell'intensità dell'immagine da 0 a 1 per ogni gruppo di immagini a fluorescenza. Impostare quindi la soglia per identificare e contare gli oggetti al di sopra della soglia stabilita. Se necessario, aggiungere ilmodulo ' ExpandOrShrinkObjects' per ridurre gli oggetti per facilitare il conteggio e l'identificazione delle goccioline in brightfield.
- Identificare solo gli oggetti entro le dimensioni selezionate di 20-30 pixel e filtrare gli oggetti contati per mantenere solo gli oggetti con un diametro specifico (ad esempio, goccioline nell'intervallo di diametro di 75 m) e un'eccentricità sferica rotonda di 0,5 e inferiore.
- Esportare i risultati in una tabella che elenca il numero totale di droplet da immagini di campo luminoso, nonché i conteggi delle goccioline di tutti i canali di fluorescenza utilizzati; vale a dire Cy5 per il gene C-LESS, HEX per il gene MEtilato CD3 e FAM per il gene FOXP3 metilato (vedi Informazioni supplementari per i dati sperimentali grezzi ottenuti per il test mdPCR presentato).
- Calcolare il rapporto di goccioline negative per ogni bersaglio gene e applicare la distribuzione di Poisson per ottenere le rispettive copie per gocciolina (CPD) utilizzando l'equazione 1:
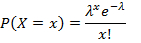
dove x rappresenta il numero di goccioline contenenti 0, 1, 2 o più molecole e λ il valore CPD. - Calcolare, la concentrazione obiettivo assoluta prendendo il rapporto tra il valore CPD e il volume di goccia ottenuto al punto 5.20 con l'equazione 2:
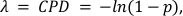
dove il valore (1-p) rappresenta la frazione di goccioline negative. - Calcolare la percentuale di CD3- T-Cells e CD4- CD25- T-Regs dividendo i rispettivi valori CPD dei geni MEtilati CD3 e FOXP3 per il C-LESS – o cellula totale – valore CPD (vedere Informazioni supplementari per i calcoli CPD da dati grezzi).
- Confrontare questi valori di percentuale con quelli ottenuti dall'imaging dell'immunofluorescenza utilizzando anticorpi per CD3- T-Cell e CD4- CD25- conteggio T-Reg.
Risultati
Il dispositivo generatore di goccioline microfluidiche basato su TPE è stato fabbricato utilizzando il protocollo descritto, come illustrato nella Figura 1. Una maschera di trasparenza è stata utilizzata nella fotolithografia per ottenere il maestro del silicio (Si). La litiografia morbida è stata eseguita per ottenere una replica PDMS inversa del maestro Si che è stata poi utilizzata per fabbricare lo stampo epossico. Il precursore dell'epossia è stato versato sul PDMS e curato per inc...
Discussione
Il protocollo e i metodi sperimentali presentati consentono di mdPCR interno utilizzando un generatore di goccioline TPE fabbricato, un ciclo termico e un microscopio a fluorescenza. Il dispositivo fabbricato utilizzando il legame morbido da TPE a TPE offre proprietà di superficie idrofobiche uniformi su tutte le pareti del canale, in modo che il dispositivo finale non richieda alcun trattamento superficiale per un uso successivo come generatore di goccioline. Questo materiale è stato regolarmente impiegato in piattafo...
Divulgazioni
Non ci sono conflitti da dichiarare.
Riconoscimenti
Gli autori riconoscono il sostegno finanziario del Consiglio Nazionale delle Ricerche del Canada.
Materiali
| Name | Company | Catalog Number | Comments |
| Bio-Rad, Mississauga, ON | TFI0201 | PCR tube | |
| RAN Biotechnologies, Beverly, MA | 008-FluoroSurfactant | Fluoro-surfactant | |
| Silicon Quest International, Santa Clara, CA | |||
| Oxford Instruments, Abingdon, UK | EMCCD camera | ||
| Thermo Fisher Scientific, Waltham, MA | MA5-16728 | ||
| Thermo Fisher Scientific, Waltham, MA | 22-8425-71 | ||
| CellProfiler | Used for fluorescence image analysis | ||
| Nikon, Japan | 10x objective | ||
| American Type Culture Collection (ATCC), Manassas, VA | PCS-800-011 | ||
| Ramé-Hart Instrument Co. (Netcong, NJ) | p/n 200-U1 | ||
| Fisher, Canada | |||
| Vitrocom, NJ, USA | 5015 and 5010 | Borosilicate capilary tube | |
| (http://definetherain.org.uk/) | |||
| Hamamatsu, Japan | LC-L1V5 | DEL UV light source | |
| Dolomite | 3200063 | Disposable fluidic tubing | |
| Dolomite | 3200302 | Disposable fluidic tubing | |
| IDT, Coralville, IA | |||
| Nikon, Melville, NY | Upright light microscope | ||
| Cytec Industries, Woodland Park, NJ | |||
| EV Group, Schärding, Austria | |||
| Zymo Research, Irvine, CA | D5030 | ||
| Photron, San Diego, CA | |||
| IDT, Coralville, IA | |||
| Gersteltec, Pully, Switzerland | SU-8 photoresist | ||
| Fineline Imaging, Colorado Springs, CO | |||
| Qiagen, Hilden, Germany | 203603 | ||
| Image J | Used to assess droplet diameter | ||
| Anachemia, Montreal, QC | |||
| Excelitas, MA, USA | Broad-spectrum LED fluorescent lamp | ||
| Galenvs Sciences Inc., Montreal, QC | DE1010 | ||
| Hexpol TPE, Åmål, Sweden | Thermoplastic elastomer (TPE) | ||
| Thermo Fisher Scientific, Waltham, MA | 13-400-518 | ||
| Nikon, Japan | Used for image acquisition | ||
| 3M, St Paul, MN | Carrier Oil | ||
| Thermo Fisher Scientific, Waltham, MA | R37605 | Blue fluorescent live cell stain (DAPI) | |
| IDEX Health & Science, Oak Harbor, WA | P-881 | PEEK fittings | |
| Sigma-Aldrich, Oakville, ON | 806552 | ||
| Dow Corning, Midland, MI | |||
| ThinkyUSA, CA, USA | ARV 310 | ||
| Ihc world, Maryland, USA | IW-125-0 | ||
| Zinsser NA, Northridge, CA | 2607808 | ||
| Cetoni GmbH, Korbussen, Germany | |||
| Sigma-Aldrich, Oakville, ON | 484431 | ||
| Bio-Rad, Mississauga, ON | 1861096 | ||
| Hitachi High-Technologies, Mississauga, ON | |||
| Nikon, Melville, NY | Inverted microscope | ||
| Nikon, Japan | |||
| Loctite | AA 352 |
Riferimenti
- Teitell, M., Richardson, B. DNA methylation in the immune system. Clinical Immunology. 109 (1), 2-5 (2003).
- Suarez-Alvarez, B., Rodriguez, R. M., Fraga, M. F., López-Larrea, C. DNA methylation: A promising landscape for immune system-related diseases. Trends in Genetics. 28 (10), 506-514 (2012).
- Suárez-Álvarez, B., Raneros, A. B., Ortega, F., López-Larrea, C. Epigenetic modulation of the immune function: A potential target for tolerance. Epigenetics. 8 (7), 694-702 (2013).
- Kondilis-Mangum, H. D., Wade, P. A. Epigenetics and the adaptive immune response. Molecular Aspects of Medicine. 34 (4), 813-825 (2013).
- Zouali, M. . The Autoimmune Diseases. , (2014).
- Wiencke, J. K., et al. A comparison of DNA methylation specific droplet digital PCR (mdPCR) and real time qPCR with flow cytometry in characterizing human T cells in peripheral blood. Epigenetics. 9 (10), 1360-1365 (2014).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Analytical Chemistry. 83 (22), 8604-8610 (2011).
- Pinheiro, L. B., et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification. Analytical Chemistry. 84 (2), 1003-1011 (2012).
- Roy, E., Galas, J. C., Veres, T. Thermoplastic elastomers for microfluidics: Towards a high-throughput fabrication method of multilayered microfluidic devices. Lab on a Chip. 11 (18), 3193-3196 (2011).
- Roy, E., et al. From cellular lysis to microarray detection, an integrated thermoplastic elastomer (TPE) point of care Lab on a Disc. Lab on a Chip. 15 (2), 406-416 (2015).
- Malic, L., et al. Epigenetic subtyping of white blood cells using a thermoplastic elastomer-based microfluidic emulsification device for multiplexed, methylation-specific digital droplet PCR. Analyst. 44 (22), 6541-6553 (2019).
- Malic, L., et al. Polymer-based microfluidic chip for rapid and efficient immunomagnetic capture and release of Listeria monocytogenes. Lab Chip. 15 (20), 3994-4007 (2015).
- Malic, L., Morton, K., Clime, L., Veres, T. All-thermoplastic nanoplasmonic microfluidic device for transmission SPR biosensing. Lab Chip. 13 (5), 798-810 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon