Method Article
Cribado de fragmentos basado en RMN en una muestra mínima pero en modo de automatización máxima
En este artículo
Resumen
El cribado basado en fragmentos mediante RMN es un método robusto para identificar rápidamente aglutinantes de moléculas pequeñas a biomacromoléculas (ADN, ARN o proteínas). Se presentan protocolos que describen la preparación de muestras basada en la automatización, los experimentos de RMN y las condiciones de adquisición, y los flujos de trabajo de análisis. La técnica permite la explotación óptima de núcleos activos de RMN de 1H y 19F para su detección.
Resumen
El cribado basado en fragmentos (FBS) es un concepto bien validado y aceptado dentro del proceso de descubrimiento de fármacos tanto en la academia como en la industria. La mayor ventaja del cribado de fragmentos basado en RMN es su capacidad no solo para detectar aglutinantes de más de 7-8 órdenes de magnitud de afinidad, sino también para monitorear la pureza y la calidad química de los fragmentos y, por lo tanto, producir aciertos de alta calidad y falsos positivos o falsos negativos mínimos. Un requisito previo dentro del FBS es realizar un control de calidad inicial y periódico de la biblioteca de fragmentos, determinar la solubilidad y la integridad química de los fragmentos en tampones relevantes y establecer múltiples bibliotecas para cubrir diversos andamios para acomodar varias clases de macromoléculas objetivo (proteínas / ARN / ADN). Además, se requiere una amplia optimización del protocolo de cribado basado en RMN con respecto a las cantidades de muestra, la velocidad de adquisición y el análisis a nivel de constructo/fragmento-espacio biológico, en el espacio de condiciones (tampón, aditivos, iones, pH y temperatura) y en el espacio del ligando (análogos de ligandos, concentración de ligandos). Al menos en el mundo académico, estos esfuerzos de detección hasta ahora se han llevado a cabo manualmente de manera muy limitada, lo que lleva a una disponibilidad limitada de infraestructura de detección no solo en el proceso de desarrollo de medicamentos sino también en el contexto del desarrollo de sondas químicas. Para cumplir con los requisitos de forma económica, se presentan flujos de trabajo avanzados. Aprovechan el último hardware avanzado de última generación, con el que la recolección de muestras líquidas se puede llenar de manera controlada por temperatura en los tubos de RMN de manera automatizada. 1Los espectros basados en ligandos de RMN H/19F se recogen a una temperatura dada. El cambiador de muestras de alto rendimiento (cambiador de muestras HT) puede manejar más de 500 muestras en bloques de temperatura controlada. Esto, junto con herramientas de software avanzadas, acelera la adquisición y el análisis de datos. Además, se describe la aplicación de rutinas de cribado en muestras de proteínas y ARN para conocer los protocolos establecidos para una amplia base de usuarios en la investigación biomacromolecular.
Introducción
El cribado basado en fragmentos es ahora un método comúnmente utilizado para identificar moléculas bastante simples y de bajo peso molecular (MW <250 Da) que muestran una unión débil a objetivos macromoleculares que incluyen proteínas, ADN y ARN. Los golpes iniciales de las pantallas primarias sirven como base para realizar una pantalla secundaria de análogos más grandes disponibles comercialmente de los golpes y luego para utilizar estrategias de crecimiento de fragmentos o enlaces basadas en la química. Para una plataforma exitosa de descubrimiento de fármacos basados en fragmentos (FBDD), en general, se requiere un método biofísico robusto para detectar y caracterizar golpes débiles, una biblioteca de fragmentos, un objetivo biomolecular y una estrategia para la química de seguimiento. Cuatro métodos biofísicos comúnmente aplicados dentro de las campañas de descubrimiento de fármacos son los ensayos de desplazamiento térmico, la resonancia de plasmones de superficie (SPR), la cristalografía y la espectroscopia de resonancia magnética nuclear (RMN).
La espectroscopia de RMN ha mostrado diversos roles dentro de las diferentes etapas del FBDD. Además de garantizar la pureza química y la solubilidad de los fragmentos en una biblioteca de fragmentos disueltos en un sistema tampón optimizado, los experimentos de RMN observados por ligandos pueden detectar la unión de fragmentos a un objetivo con baja afinidad y los experimentos de RMN observados pueden delinear el epítopo de unión del fragmento, lo que permite estudios detallados de la relación estructura-actividad. Dentro del mapeo de epítopos, los cambios de desplazamiento químico basados en RMN no solo pueden identificar los sitios de unión ortostérica, sino también los sitios alostéricos que podrían ser crípticos y solo accesibles en los llamados estados conformacionales excitados del objetivo biomolecular. Si el objetivo biomolecular ya se une a un ligando endógeno, los impactos del fragmento identificados se pueden clasificar fácilmente como alostéricos u ortostéricos mediante la realización de experimentos de competencia basados en RMN. La determinación de la constante de disociación (KD) de la interacción ligando-objetivo es un aspecto importante en el proceso FBDD. Las titulaciones de desplazamiento químico basadas en RMN, ya sea ligando u objetivo observado, se pueden realizar fácilmente para determinar el K D. Una ventaja importante de la RMN es que los estudios de interacción se realizan en solución y cerca de condiciones fisiológicas. Por lo tanto, todos los estados conformacionales para el análisis de la interacción ligando/fragmento con su objetivo pueden ser probados. Además, los enfoques basados en RMN no solo se limitan a la detección de proteínas solubles bien plegadas, sino que también se están aplicando para acomodar un espacio objetivo más grande, incluyendo ADN, ARN, proteínas unidas a membrana e intrínsecamente desordenadas1.
Las bibliotecas de fragmentos son una parte indispensable del proceso FBDD. En general, los fragmentos actúan como los precursores iniciales que eventualmente se convierten en parte (subestructura) del nuevo inhibidor desarrollado para una diana biológica. Se ha informado que varios medicamentos (Venetoclax2, Vemurafenib3, Erdafitinib4, Pexidartnib5) comenzaron como fragmentos y ahora se usan con éxito en las clínicas. Típicamente, los fragmentos son moléculas orgánicas de bajo peso molecular (<250 Da) con una alta solubilidad acuosa y estabilidad. Una biblioteca de fragmentos cuidadosamente elaborada que contiene típicamente unos pocos cientos de fragmentos, ya puede prometer una exploración eficiente del espacio químico. La composición general de las bibliotecas de fragmentos ha evolucionado con el tiempo y la mayoría de las veces se derivaron diseccionando medicamentos conocidos en fragmentos más pequeños o diseñados computacionalmente. Estas diversas bibliotecas de fragmentos contienen principalmente aromáticos planos o heteroátomos y se adhieren a la Regla de Lipinski de 5 6, o a la Regla de tendencia comercial actual de 3 7, pero evitan los grupos reactivos. Algunas bibliotecas de fragmentos también fueron derivadas o compuestas de metabolitos altamente solubles, productos naturales y/o sus derivados8. Un desafío general planteado por la mayoría de las bibliotecas de fragmentos es la facilidad de la química posterior.
El Centro de Resonancia Magnética Biomolecular (BMRZ) de la Universidad Goethe de Frankfurt, es socio del iNEXT-Discovery (Infrastructure for NMR, EM and X-rays for Translational research-Discovery), un consorcio de infraestructuras de investigación estructural para todos los investigadores europeos de todos los campos de la investigación bioquímica y biomédica. Dentro de la iniciativa anterior de iNEXT que finalizó en 2019, se creó una biblioteca de fragmentos compuesta por 768 fragmentos con el objetivo de "fragmentos mínimos y máxima diversidad" que cubren un gran espacio químico. Además, a diferencia de cualquier otra biblioteca de fragmentos, la biblioteca de fragmentos iNEXT también se diseñó en base al concepto de "fragmentos preparados" con el objetivo de facilitar la síntesis posterior de ligandos complejos de alta afinidad y, en adelante, conocida como biblioteca interna (Diamond, Structural Genomic Consortium e iNEXT).
El establecimiento de FBDD por RMN requiere mano de obra, conocimiento e instrumentación. En el BMRZ, se han desarrollado flujos de trabajo optimizados para apoyar la asistencia técnica para el cribado de fragmentos por RMN. Estos incluyen control de calidad y evaluación de solubilidad de la biblioteca de fragmentos 9, optimización del tampón para los objetivos elegidos, cribado basado en ligandos 1D observados en 1H o 19F, experimentos de competencia para diferenciar entre unión ortostérica y alostérica, experimentos de RMN observados en 2D para el mapeo de epítopos y para caracterizar la interacción con el conjunto secundario de derivados de los impactos iniciales del fragmento. BMRZ ha establecido rutinas automatizadas para el análisis, como también se discutió anteriormente en la literatura 10,11, de las interacciones molécula-proteína pequeña y cuenta con toda la infraestructura automatizada necesaria para el cribado de fragmentos basado en RMN. Ha implementado la diferencia de transferencia de saturación RMN (STD-NMR), ligando de agua observado a través de espectroscopia de gradiente (waterLOGSY) y experimentos de relajación basados en Carr-Purcell-Meiboom-Gill (basados en CPMG) para identificar fragmentos dentro de una amplia gama de regímenes de afinidad, así como instrumentación y software de RMN automatizados de última generación para el descubrimiento de fármacos. Si bien la detección de fragmentos basada en RMN está bien establecida para las proteínas, este enfoque se usa con menos frecuencia para encontrar nuevos ligandos que interactúan con el ARN y el ADN. BMRZ ha establecido una prueba de concepto para nuevos protocolos que permiten la identificación de interacciones molécula-ARN/ADN pequeñas. En las siguientes secciones de esta contribución, se informa de la aplicación de rutinas de cribado en muestras de proteínas y ARN para dar a conocer los protocolos establecidos para una amplia base de usuarios en la investigación biomacromolecular.
Protocolo
1. Biblioteca de fragmentos
- Biblioteca de fragmentos interna
NOTA: En el marco de una de las actividades de investigación conjuntas de iNEXT, se desarrolló una biblioteca de fragmentos de primera generación robusta y amigable con la química12 y, posteriormente, se reunió una segunda generación de la biblioteca en colaboración con Enamine y se conoce como la biblioteca de fragmentos preparada para DSI (Diamond-SGC-iNEXT) (en adelante denominada "Biblioteca interna"). Esta biblioteca puede estar disponible en el BMRZ para fines de detección.- Evaluar la integridad y solubilidad de la biblioteca de fragmentos utilizando un protocolo basado en RMN previamente informado9.
NOTA: La biblioteca interna consta de 768 fragmentos con una diversidad química muy alta (>200 Singletons). Realizar el cribado en mezclas de fragmentos puede acelerar significativamente la campaña de cribado; sin embargo, el número de fragmentos en una mezcla es limitado debido a la superposición de señales en el espectro de 1H-RMN. La mayor diversidad química ofrecida por la biblioteca interna permite la preparación de mezclas que contienen 12 fragmentos diferentes sin ninguna superposición significativa de desplazamiento químico en los espectros de RMN observados en 1H. - 103 fragmentos dentro de los 768 fragmentos poseen un átomo de flúor. Para fines de cribado de 19 F, divida los 103fragmentos que poseen un grupo flúor en 5 mezclas basadas en una superposición mínima de 19F de desplazamiento químico. Para minimizar la superposición de señales en el cribado 19F, utilice la información de desplazamiento químico de las mediciones de compuestos individuales para diseñar mezclas con el número máximo de fragmentos y la superposición mínima de señales. Cada mezcla tiene 20-21 fragmentos con distintos cambios químicos de 19F que permiten una asignación inequívoca de fragmentos.
- Evaluar la integridad y solubilidad de la biblioteca de fragmentos utilizando un protocolo basado en RMN previamente informado9.
- Biblioteca de fragmentos definida por el usuario/proporcionada
- Realizar campañas de proyección con la biblioteca de fragmentos definida o proporcionada por el usuario; sin embargo, los siguientes pasos deben preceder a la campaña de detección.
- Si el usuario no lo especifica de antemano, realice un control de calidad basado en RMN de los fragmentos (en el BMRZ, se utilizan herramientas de software avanzadas para esto; 9, capítulo 6.1.1).
- Verifique la solubilidad de los fragmentos en el tampón de elección para el objetivo biomolecular, la integridad de la estructura y la concentración de fragmentos antes de su uso.
- Diseñe la mezcla para disminuir tanto la superposición de señales en los espectros de RMN como el tiempo de medición.
- Diseñar las mezclas según el paso 4.2.
- Filtre fragmentos individuales o un subconjunto de mezclas en lugar de toda la biblioteca.
2. Preparación de la muestra
NOTA: El cribado de alto rendimiento por RMN utiliza un robot pipeteador para la preparación de muestras. Los espectros de RMN, pero también las estabilidades durante varios días de adquisición de señales de proteínas, ARN y ADN son extremadamente sensibles a las fluctuaciones de temperatura y, por lo tanto, los sistemas automatizados controlados por temperatura facilitarán enormemente la estabilidad de las muestras que se pipetean. Para ello, un dispositivo adicional adicional, que funciona entre 4 y 40 °C, está acoplado al robot de pipeteo para el manejo de líquidos de las muestras de RMN en un entorno de temperatura controlada.
- Preparación de la mezcla de ligandos
- Prepare muestras de detección para mediciones de RMN utilizando un robot de preparación de muestras. La configuración flexible del robot permite una amplia gama de aplicaciones (por ejemplo, recuperación de las muestras de tubos de RMN en contenedores de almacenamiento o tareas generales de manejo de líquidos). Se pueden utilizar tubos de RMN con diferentes diámetros (1,7, 2,0, 2,5, 3,0 y 5,0 mm). El sistema de robot de muestra junto con el software de control avanzado lee el código de barras asignado para cada tipo de contenedor y ejecuta el protocolo de llenado de líquidos de manera óptima.
- Para la preparación de las mezclas de ligandos de la biblioteca interna, use viales con código de barras. Los viales con código de barras garantizan el más alto nivel de fiabilidad y una trazabilidad óptima de las muestras.
- Distribuir 768 compuestos en 8 placas de formato de 96 pocillos. La concentración madre de cada fragmento individual es de 50 mM en d6-DMSO/D2O(9:1). En total, prepare 64 mezclas que contengan 12 fragmentos cada una. La concentración final de cada fragmento en una mezcla es de 4,2 mM.
NOTA: El robot de pipeteo puede acomodar una variedad de tipos de contenedores con geometrías variadas (viales de muestreador criogénico o automático, placas de 96 pocillos de profundidad redonda o cuadrada, viales estándar con código de barras, tubos de microcentrífuga) y ayuda a la ejecución eficiente de la transferencia de líquido a una variedad de tubos y bastidores de RMN.

Figura 1: (A) Preparación de muestras de RMN de alto rendimiento y robot de llenado de tubos de RMN instalado en BMRZ. (B) Cambiador de muestras de alto rendimiento con bastidores individuales de temperatura controlada instalados en un espectrómetro de 600 MHz en la instalación BMRZ. Haga clic aquí para ver una versión más grande de esta figura.
- Preparación de la muestra de cribado en blanco (espectro del ligando de referencia) y con el objetivo (ligando en presencia del objetivo)
- Para la preparación de las muestras de cribado de RMN, en presencia de la biomolécula diana (proteína/ARN/ADN) y la mezcla de ligandos, utilice tubos cambiadores de muestras NMR HT de 3 mm seleccionados de la cartera de RMN Bruker de tubos de RMN estándar.
- Transfiera el objetivo biomolecular (por ejemplo, cribado de 1 H: ARN o proteína de 10 μM) en un tampón de cribado definido al tubo de RMN de 3 mm (volumen final de 200 μL) manualmente o utilizando el robot depipeteo.
- Transfiera 10 μL (por ejemplo, cribado de 1H) de la mezcla de ligandos en el siguiente paso utilizando el sistema robótico a los tubos de RMN de 3 mm con código de barras que contienen la biomolécula objetivo y la mezcla utilizando el protocolo incorporado del software de control.
NOTA: El número de código de barras del tubo de RMN se incorpora de forma conveniente y automática en el conjunto de datos de RMN adquirido, lo que garantiza un flujo de trabajo orientado a la identificación sin ninguna confusión. El accesorio de control de temperatura del robot de pipeteo permite mantener las muestras preparadas en los tubos de RMN a temperatura constante.
- Condiciones y parámetros definidos internamente
- Establecer las condiciones óptimas de amortiguación para realizar el cribado de ARN y proteínas contra la biblioteca de fragmentos interna. La siguiente muestra acondicionada se utiliza para el ARN en el BMRZ: 25 mM KPi, 50 mM KCl, pH 6.2. Mg2+ es opcional.
- Las proteínas son extremadamente sensibles para las condiciones de solución; Utilice búferes óptimos para el objetivo de elección. Para cada uno de esos tampones, adquiera espectros de referencia adicionales de los ligandos para que sirvan como espacios en blanco para el análisis.
- Condiciones especificadas por el usuario
NOTA: En los casos en que las condiciones establecidas internamente no sean adecuadas para los objetivos que se seleccionarán de un usuario potencial, se deben implementar los siguientes pasos.- Realizar 1RMN-H del tampón solo para garantizar una interferencia mínima de los componentes del tampón en la realización y análisis de los experimentos de cribado observados por el ligando. Los componentes interferentes podrían reemplazarse adecuadamente con equivalentes deuterados.
- Limitaciones en la producción de muestras (cantidades objetivo)/condiciones y disponibilidad
NOTA: El aislamiento o la producción recombinante de ciertas biomacromoléculas pueden, en ciertos casos, resultar difíciles y dar lugar a una disponibilidad limitada del objetivo para llevar a cabo una campaña exitosa de detección de fármacos. En los casos de disponibilidad limitada o ilimitada de los objetivos, se podrían utilizar las siguientes alternativas para realizar con éxito un cribado de fragmentos basado en RMN.- Si está limitado, use 19exámenes de detección basados en R-F. Los ligandos fluorados típicos tienen una sola señal de 19F; Por lo tanto, use cócteles con 25-30 fragmentos sin ninguna superposición de señal. Hay menos señales para analizar, no hay interferencia de señal de los componentes del búfer y menos señales en las que confiar para la identificación de golpes.
- Si es ilimitado, use pantallas más grandes como 1H-RMN. La biblioteca de fragmentos más grande se puede filtrar. Por lo general, los fragmentos están compuestos de más de un protón, lo que significa más señales en las que confiar para el análisis.
3. Condiciones de adquisición de RMN
- Condiciones internas generalmente definidas
- Espectrómetro equipado con cambiador de muestras HT (Automatización)
- Para el cribado de alto rendimiento, utilice placas de 96 pocillos que solo se pueden medir con un cambiador de muestras HT. El cambiador de muestras HT también ofrece la posibilidad de templar cada bastidor individualmente.
- Para una relación señal-ruido óptima, utilice un espectrómetro con una sonda criogénica refrigerada por helio o nitrógeno. Un módulo automatizado de ajuste y coincidencia (ATM) es necesario para la automatización.
- Conjuntos de parámetros y secuencias de pulsos
NOTA: Muchos experimentos de RMN pueden caracterizar eventos de enlace. La identificación del acierto varía según la configuración experimental. Los siguientes experimentos se utilizan rutinariamente en las campañas de detección de BMRZ. Sin embargo, se pueden realizar cambios para las campañas de proyección definidas por el usuario y de acuerdo con las especificaciones del usuario.- Si utiliza el software TopSpin, incluya el conjunto de parámetros para experimentos basados en ligandos: SCREEN_STD, SCREEN_T1R, SCREEN_T2 SCREEN_WLOGSY. El conjunto de parámetros incluye todos los parámetros necesarios y las secuencias de pulsos: STD: stddiffesgp.3; T1ρ: t1rho_esgp2d; T2: cpmg_esgp2d; y waterLOGSY: ephogsygpno.2.
- Para todos los experimentos enumerados, use la escultura de excitación13 como supresión de agua. Para una referencia, use la escultura de excitación 1D (zgesgp). El número de escaneos depende de la sensibilidad del sistema (intensidad del campo magnético y cabeza de la sonda), la concentración de la muestra y la elección del experimento. Una recomendación es: 1D con NS = 64, T1ρ y T2 con NS = 128, STD con NS = 256 y waterLOGSY con NS = 384 o 512.
- Para el cribado 19 F, use experimentos 1D y T2: 1D: F19CPD (pp=zgig) para el cabezal de sonda 19 F{1 H} y F19(pp=zg) para el cabezal de sonda 19F/1H; SCREEN_19F_T2 (pp = cpmgigsp).
- Utilice un ancho espectral de 220 ppm y una frecuencia de excitación de -140 ppm. El tiempo de experimento es de entre 1 y 5 horas (asegurar la estabilidad a largo plazo de la biomacromolécula) dependiendo del hardware y la concentración de la muestra. Para T2, el tiempo de CPMG debe alternar entre 0 ms y 200 ms.
- Tratamiento
- Registre los experimentos STD, T1ρ y T2 como pseudo 2D. Para procesar los dos espectros 1D individuales, IconNMR utiliza el programa au-proc_std con o sin la opción relax. La primera opción proporciona la referencia 1D y la diferencia de dos espectros. La segunda opción produce dos espectros separados con un tiempo de relajación corto y largo. El waterLOGSY es un solo 1D que debe ser escalonado con un negativo para la señal de disolvente.
- Espectrómetro equipado con cambiador de muestras HT (Automatización)
- Condiciones específicas del usuario
- Adapte cualquiera de los parámetros mencionados anteriormente a las condiciones definidas por el usuario. Por ejemplo, si una proteína proporcionada por el usuario de una instalación no es estable a la temperatura generalmente utilizada, se pueden realizar experimentos de optimización variando la temperatura, la concentración, las condiciones de amortiguación, etc.
4. Análisis de datos
- Control de calidad de la biblioteca de fragmentos (d6-DMSO/buffer específico) y cuantificación
- CMC-q
NOTA: El control de calidad de las bibliotecas de fragmentos es esencial antes del inicio de las campañas de selección. Además, es necesario garantizar la estabilidad a largo plazo de la biblioteca de fragmentos para la aplicación de varias campañas de selección, por lo que se debe realizar una evaluación periódica de la calidad de la biblioteca. Para ello, se utiliza el software integrado CMC-q y CMC-a de TopSpin para la evaluación de la calidad y la cantidad. El CMC-q y CMC-a son módulos de software dentro de Topspin que permiten una adquisición fluida, el análisis incluyendo la verificación de la estructura utilizando 1espectro H-RMN obtenido de pequeñas moléculas orgánicas 9.- Para mayor integridad, prepare muestras de evaluación con una concentración de fragmento de 1 mM en d6-DMSO. Prepare muestras de forma automatizada con un robot de pipeteo llenando la recolección de muestras líquidas en un tubo de RMN de 3 mm.
- Para la evaluación de la solubilidad, utilice una muestra que consiste en 1 mM compuesto en tampón fosfato de sodio de 50 mM a pH 7.4, cloruro de sodio 150 mM, 90%H2O/ 10% D 2 O y 1 mM de sal sódica ácida 3-(trimetilsilil)propiónica-2,2,3,3-d4 (TMSP-Na).
- Recopile espectros de RMN a 298 K o 293 K utilizando un espectrómetro de RMN de 600 MHz equipado con sonda criogénica TCI de 5 mm de triple resonancia y un cambiador de muestras HT, que puede manejar 579 muestras a la vez.
- Para configurar el software CMC-q, siga las instrucciones del manual del usuario, que implementa la creación de un usuario IconNMR, la activación de FastLaneNMR y el cambio del cambiador de muestras HT.
- Calibre el pulso de 90° y guárdelo en la tabla de prosol TopSpin.
- Coloque la placa de pozo de 96 muestras en una de las 5 posiciones de bastidor en el cambiador de muestras HT.
- Para cargar un archivo SDF (archivo de datos de estructura) que debe contener su estructura química propuesta, un identificador único y la posición en el cambiador de muestras HT de cada muestra en un lote, vaya a Examinar en la ventana Configuración de CMC-q y haga clic en Abrir después de seleccionar un archivo que termina en .sdf.
- En la configuración de CMC.q Batch Automation, establezca el tipo de verificación que define el experimento que se medirá, el usuario de IconNMR y defina el disolvente.
- Defina los archivos SDF para la ruta del archivo SDF, el ID de la molécula y la posición de la muestra.
- Comience la adquisición haciendo clic en Iniciar. Haga clic en Iniciar adquisición nuevamente. La configuración de CMC-q también se puede guardar haciendo clic en Guardar.
- Para obtener una descripción detallada de los pasos de configuración de CMC-q, siga las instrucciones del manual del usuario de Bruker.
- CMC-a
- Para CMC-a, utilice el módulo de software dentro de Topspin que permite el análisis, incluida la verificación de la estructura, utilizando el espectro de RMN-1H obtenido de pequeñas moléculas orgánicas9.
- CMC-q
- Diseño de mezcla
NOTA: Un diseño de mezcla adecuado juega un papel importante para la detección utilizando RMN como plataforma. Un alto número de fragmentos por mezcla permite una detección más rápida, pero aumenta el riesgo de falsos positivos y negativos. Un número menor disminuye ese riesgo, pero aumenta el tiempo que lleva realizar la evaluación. En general, debe evitarse una superposición de señales al crear mezclas. Usando la biblioteca interna, esto se puede descuidar para la proyección de 1H, ya que la biblioteca fue diseñada específicamente para ser diversa y mostrar poca superposición de señales mientras mantiene una alta diversidad química. Esto a su vez significa que no se debe someterse a un procedimiento de diseño especial para crear las 64 mezclas.- Como la proyección 19F se basa en los fragmentos de la biblioteca interna que contienen flúor y la biblioteca no se creó para reducir la superposición de señales para estos fragmentos específicos, diseñe una mezcla adecuada.
- Mida espectros compuestos individuales para todos los fragmentos que contengan 19F.
- Tenga en cuenta la información de desplazamiento químico de cada señal.
- De acuerdo con esta información, elija 20-21 fragmentos por mezcla. Esto a su vez da 5 mezclas que contienen cada una de 20-21 fragmentos sin superposición de señal y permite un análisis semiautomático de los datos.
- Realizar la identificación de aciertos dentro de una interacción biomacromolécula-ligando observada por ligando
NOTA: Existen diferentes definiciones de un golpe entre el procedimiento de detección de 19F y 1H. Las siguientes identificaciones de visitas fueron configuradas por nosotros y siguen reglas específicas. El tema de la determinación de aciertos es muy subjetivo y puede diferir de un usuario a otro. Sin embargo, es de suma importancia que las reglas para la identificación de visitas no cambien una vez acordadas para mantener la validación y la credibilidad.- 1Pantalla H
- Para determinar con confianza los golpes, adquiera espectros 1D 1H, experimentos de relajación waterLOGSY y T2 tanto en presencia como en ausencia de objetivo para identificar aglutinantes. Los tres experimentos tienen el potencial de mostrar un evento vinculante. Si un CSP de más de 6 Hz es visible en los espectros de muestra en comparación con los espectros en blanco, esto se considera una indicación de un acierto. Lo mismo ocurre si es visible una fuerte señal positiva en el waterLOGSY, así como una reducción de T2 de más del 30% en los espectros de la muestra. Los eventos de enlace se pueden mostrar en los tres experimentos, al comparar la muestra que contiene espectros con sus respectivos espectros en blanco. Sin embargo, los eventos de enlace pueden no ser visibles en los tres experimentos. Debido a esto, se acordó que al menos dos de los eventos descritos anteriormente deben ocurrir para clasificar un fragmento como un golpe vinculante.
- Utilice la herramienta FBS en TopSpin para definir el estado de los fragmentos en binding, ambiguous, unknown, aggregates y non-binding.
- Cuando termine con una mezcla, apruébela dentro de la herramienta FBS.
- En la pestaña de resumen dentro del proyecto FBS, haga clic en Crear un informe de selección. Esto abrirá una ventana que crea un archivo .xlsx. El usuario puede optar por elegir entre todos los ligandos, ligando de unión solamente, ligando no vinculante solamente y ligandos ambiguos que se informarán en la hoja de cálculo.
- 19Pantalla F
- Para diferenciar entre no aglutinante, cuaderno semanal y aglutinante fuerte, divida el cociente de integración entre la medición objetivo de 200 ms y la medición en blanco de 200 ms por el cociente de la medición objetivo de 0 ms y se utiliza la medición en blanco de 0 ms:
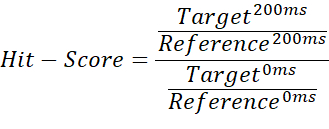
NOTA: Esto proporciona valores que van de 0 a ~1 (la puntuación de aciertos), lo que permite asignar umbrales para cada estado de enlace. - Utilice el promedio de la medición de referencia de 200 ms como umbral de referencia para marcar los casos en que la puntuación de aciertos supera 1. Esto puede ocurrir si las integrales importadas contienen valores negativos o si la medición de referencia es mayor que la medición objetivo. Un puntaje de acierto de ≤ 0.67 se considera un golpe débil, < 0.33 un golpe fuerte, y cualquier cosa > 0.67 como no-hit. En la figura 2 se muestra un ejemplo.
- Para diferenciar entre no aglutinante, cuaderno semanal y aglutinante fuerte, divida el cociente de integración entre la medición objetivo de 200 ms y la medición en blanco de 200 ms por el cociente de la medición objetivo de 0 ms y se utiliza la medición en blanco de 0 ms:
- 1Pantalla H
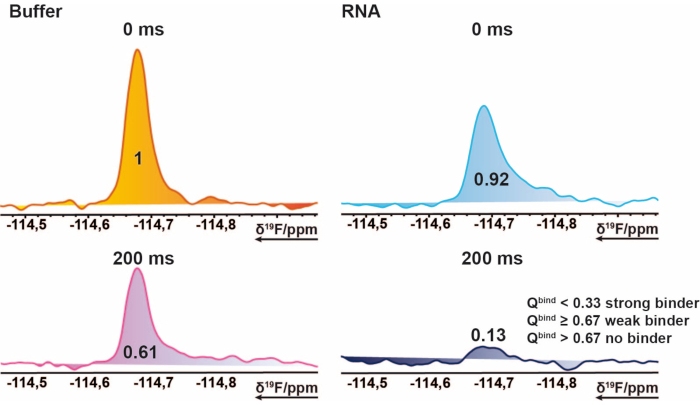
Figura 2: Identificación de aciertos para la evaluación de 19F. Sección de 19espectros de RMN de CPMG F de un compuesto ejemplar. Esta representación pictórica explica las propiedades de un aglutinante. 19Espectros F-CPMG de un compuesto adquirido de muestras de mezcla en presencia y ausencia de ARN. Los valores representan los valores integrales normados del pico correspondiente. Haga clic aquí para ver una versión más grande de esta figura.
- Análisis de datos
- Preparar los datos para el análisis
NOTA: Es importante que los datos adquiridos no tengan defectos visibles. Esto significa que los datos en los que el shimming fue problemático o la supresión del agua fue insuficiente no deben considerarse para el análisis. Más bien, se recomienda registrar los datos nuevamente y asegurarse de que todo esté bien con la muestra (por ejemplo, sin burbujas de aire), con la temperatura, el shimming y la supresión de agua. La corrección de los datos siempre se puede evaluar al comparar las señales DMSO. - 1Cribado H
- Para analizar los datos de detección de 1H, utilice la herramienta FBS (necesita licencia adicional) en TopSpin 4.0.9.
- Siga las instrucciones del manual de la herramienta FBS para comenzar con el análisis de datos. Los pasos siguientes resumen el procedimiento descrito en el manual.
- Almacene los datos de RMN BMRZ de las campañas de detección de modo que cada mezcla de detección diferente tenga su propio directorio en el que un subdirectorio contiene los diferentes experimentos medidos en la muestra.
- Para usar la herramienta FBS, almacene los espectros de referencia que tienen todos los datos guardados de muestras sin el objetivo biomolecular pero con las mezclas, así como el compuesto único medido en diferentes directorios /nmr. Esto es importante ya que la herramienta FBS solicitará la ruta del directorio de cada uno individualmente.
NOTA: La herramienta FBS reconocerá un directorio como un proyecto de cribado si los siguientes conjuntos de datos se almacenaron en el mismo directorio donde se almacenan las mezclas de una muestra de cribado (csv, documentos XML FragmentScreen y archivo BAK). - Cuando utilice TopSpin 4.0.9, cree una ruta directa al directorio que contiene los datos adquiridos, un llamado DIR. Elija el directorio /nmr en el que todas las mezclas deben tener un directorio distinto.
- Para iniciar la herramienta FBS de un ejemplo filtrado, arrastre el símbolo proyecto FBS al centro de la ventana TopSpin. En el directorio elegido, el símbolo del proyecto FBS debe aparecer si dichos conjuntos de datos se copiaron previamente en él.
- La ventana Opciones de filtrado basadas en fragmentos debería abrirse automáticamente al cargar por primera vez un nuevo proyecto de FBS. En esta ventana, elija un archivo de cóctel. El archivo cóctel es un archivo csv que contiene la asignación del nombre de las mezclas, el nombre de cada fragmento y su división en las mezclas. También defina una carpeta de espectros de ligando de referencia que tenga todos los espectros medidos de los fragmentos individuales. Por último, defina una carpeta de experimento en blanco de referencia, que suele ser la carpeta que contiene los conjuntos de datos de las mezclas sin el objetivo investigado.
- Las opciones de cribado basadas en fragmentos tienen una pestaña llamada Tipos de espectros que permite definir los espectros investigados, así como el color para mostrar los espectros. Establezca el Spectype de acuerdo con los datos procesados de antemano. En la pestaña Diseño de pantalla , defina los espectros que se compararán entre sí según sus tipos de especificación.
- Presione Aceptar para iniciar el proyecto FBS.
- Mientras se miran los datos, se abrirá una ventana separada, que resume todas las mezclas de cócteles y todos los ligandos de cada mezcla en una tabla. Al hacer doble clic en una celda, se abrirán los conjuntos de datos respectivos, comparando, por ejemplo,los espectros 1 H 1D Blank con el conjunto de datos que contiene el objetivo.
- Antes de asignar aglutinantes, asegúrese de que los picos de referencia (DMSO de todas las mediciones, así como los compuestos individuales) coincidan entre sí y tengan el mismo cambio químico. Si se observan diferencias, corríjalas utilizando la opción de procesamiento en serie de TopSpin.
- La opción de procesamiento en serie se encuentra en la pestaña Proceso en Avanzado. Aplica cambios a todos los espectros seleccionados de un conjunto de datos. De esta manera, los Spectypes se pueden asignar fácilmente a los números de experimento y todos los espectros se pueden cambiar a la vez para alinearse con la referencia.
- 19F Cribado
- Para el primer análisis de las mezclas de 19F, cree un archivo de integración para cada mezcla. Para definir la región de integración, haga clic en la función Integrar en la pestaña Analizar . Asegúrese de que para cada fragmento de la mezcla se defina una región de integración clara para el 19F-singal correspondiente.
- Utilice el botón Guardar/Exportar regiones de integración para exportar el archivo de integración para su uso futuro. Guarde los archivos de integración usados en C:\Bruker\TopSpin4.0.9\exp\stan\nmr\lists\intrng, o en la ruta correspondiente del directorio de instalación de TopSpin.
- Para 19datos F, abra un conjunto de datos con o sin el objetivo investigado.
- Para cargar el archivo de integración en el espectro actual, abra la pestaña Analizar nuevamente, vaya a Integrar y, utilizando el botón Leer/Importar regiones de integración, cargue el archivo de integración correspondiente. Esto cargará cualquier región definida de ese archivo en el espectro actual.
- Guarde y vuelva para encontrar una lista de todas las regiones integradas en la pestaña Integrales . Copie esto en una hoja de cálculo o cualquier otra herramienta utilizada para el análisis posterior de los datos.
- Repita este procedimiento para cada mezcla, con y sin objetivo.
- Gestión de datos
- Para facilitar el uso y aumentar la productividad, tenga un flujo de trabajo uniforme configurado para el análisis y almacenamiento posterior de los datos adquiridos. Tanto para la proyección 1 H como para la 19F, use una hoja de cálculo diseñada específicamente para cada una.
NOTA: Para el cribado 1H se utilizó exclusivamente para la gestión de datos y para resumir cada objetivo, mientras que para el cribado 19F se utilizó el cociente explicado en el capítulo 4.3 para etiquetar automáticamente cada fragmento como hit/no hit después de que los datos integrales se copiaron en él. Esto reduce el riesgo de error humano durante el análisis, suponiendo que el archivo se configuró correctamente, y facilita el intercambio de información, ya que toda la información importante se recopila en un solo lugar en un archivo que puede ser abierto por prácticamente cualquier persona sin la necesidad de más programas para echar un vistazo inicial a los datos.
- Para facilitar el uso y aumentar la productividad, tenga un flujo de trabajo uniforme configurado para el análisis y almacenamiento posterior de los datos adquiridos. Tanto para la proyección 1 H como para la 19F, use una hoja de cálculo diseñada específicamente para cada una.
- Preparar los datos para el análisis
Resultados
Control de calidad de la biblioteca de fragmentos
Los fragmentos de la biblioteca interna se entregaron como soluciones madre de 50 mM en 90% d6-DMSO y 10% D 2 O (10% de D2O asegura la minimización de la degradación del compuesto debido a ciclos repetidos de congelación-descongelación14). Las muestras de compuestos individuales consistieron en 1 mM de ligando en 50 mM de tampón fosfato (25 mM KPi pH 6.2 + 50 mM KCl + 5 mM MgCl 2), pH 6.0 en 90% H 2 O/9% D2O/1% d6-DMSO. 1Los experimentos de RMN-H de fragmentos de la biblioteca iNEXT se midieron en un espectrómetro de RMN de 500/600 MHz. Estos datos se utilizaron además para identificar los compuestos individuales en campañas de cribado de 1 H utilizando el software CMC-q que permite al usuario adquirir espectros completamente de manera automatizada y se evaluó el agregado de análisis CMC-a la calidad (solubilidad e integridad) delos fragmentos. Los resultados del análisis automatizado de CMC-a se muestran como una salida gráfica similar a la representada en la Figura 3. La salida gráfica muestra una representación de una placa de 96 pocillos. Un círculo de color rojo significa que este fragmento muestra inconsistencia en la estructura o concentración. Los pozos de color verde indican que el fragmento es consistente.
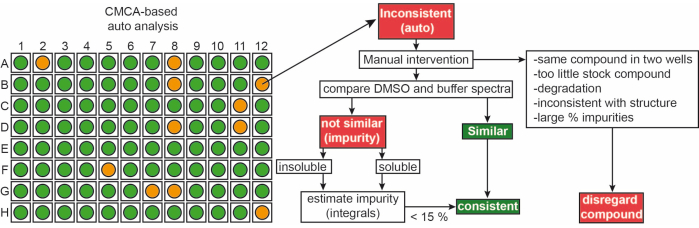
Figura 3: Control de calidad de la biblioteca de fragmentos. Representación esquemática de la salida automatizada basada en CMC-a. Se evalúan las propiedades del fragmento, como la concentración y la integridad estructural. El verde significa consistente, el naranja en este caso significa inconsistente. Los fragmentos incoherentes se revisan manualmente siguiendo el flujo de trabajo mostrado. Haga clic aquí para ver una versión más grande de esta figura.
Aproximadamente, el 65% y el 35% de los fragmentos se clasificaron como consistentes e inconsistentes, respectivamente, tanto en DMSO como en tampón. Además, el 30% de los ligandos clasificados inconsistentes se volvieron consistentes después de una cuidadosa inspección manual de los espectros9.
19F Diseño de mezcla
103 fragmentos que contenían uno o varios grupos de flúor de la biblioteca interna se dividieron en 5 mezclas (A, B, C, D, E). Cada mezcla tiene de 20 a 21 fragmentos. En este caso, las mezclas tuvieron que diseñarse cuidadosamente para evitar la superposición de señales. 19Se midieron experimentos de relajación transversal F para cada mezcla que aplica trenes de pulsos CPMG. Estos experimentos se pueden modificar variando los retrasos de relajación. El desplazamiento químico 19F de las mezclas A-E se puede ver en la Figura 4.
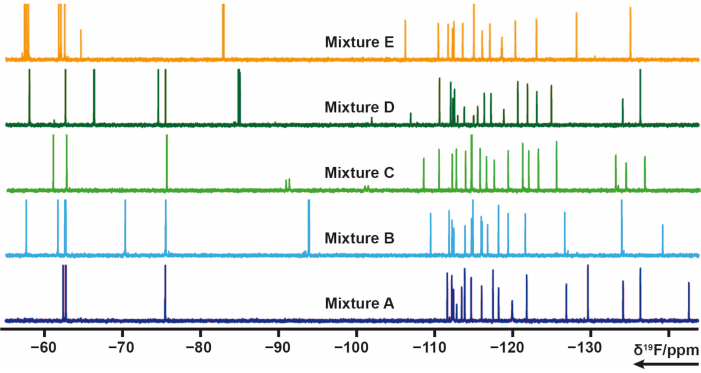
Figura 4: 19espectros F 1D-RMN de muestras de mezcla de la biblioteca interna. Haga clic aquí para ver una versión más grande de esta figura.
Preparación de muestras
La preparación de la muestra en el procedimiento de cribado 19F se realizó manualmente o con pipeteo automatizado utilizando un robot de pipeteo. Los fragmentos en cada mezcla tenían una concentración de 2.5 mM en 90% d6-DMSO y 10%D2O. El volumen final de una muestra de cribado fue de 170 μL con 5% deD2Ocomo agente de bloqueo. Cada mezcla se pipeteó dos veces, una en una solución tampón que contenía (sin objetivo) y otra en una solución tampón que contenía diana. La relación entre el objetivo y el fragmento se estableció en 1:1, lo que dio como resultado una concentración final objetivo/ligando de 50 μM. Además, las muestras de control son la biomolécula objetivo en el tampón de cribado sin mezcla para garantizar la integridad del objetivo, así como una muestra de control con solo tampón y D2O para garantizar la calidad del amortiguador.
Los datos de cribado de RMNde 19 F-1D y 19F-CPMG-T2 fueron mediciones como se describe en la sección 3.1. Por ejemplo, en el caso del ARN se adquirió una secuencia de eco de retorno de salto (pp = zggpjrse,15) para la única muestra diana en tampón.
Análisis de datos
El procedimiento de cribado 19 F se aplicó al riboswitch thiM TPP de E. coli y a la proteína tirosina quinasa (PtkA) de M. tuberculosis entre varios otros objetivos 16. La biblioteca de cribado 19F tiene 103 fragmentos que se dividen en 5 mezclas etiquetadas de Mix A a E. La preparación de muestras de cribado se puede realizar manualmente sin el uso de un robot pipeteador de muestras. Se mezclaron 40 μM de solución que contenía ARN de thiM (condiciones tampón) con 3,2 μL de las mezclas. Se prepararon muestras de control adicionales que consistían en tampón solamente, tampón con 5% de DMSO (previamente asegurar la estabilidad de la biomacromolécula en presencia de la concentración deseada de DMSO) y tampón con ARN. Estas 13 muestras de cribado se prepararon y se transfirieron a tubos de RMN de 3 mm. Los códigos de barras de los tubos de RMN se escanean y cada mezcla en presencia y ausencia de ARN, así como las muestras de control se midieron de acuerdo con los experimentos de RMN 19F mencionados anteriormente realizados a 298 K. El cribado de este ARN contra la biblioteca interna se realizó realizando mediciones deT2 con CPMG de 0 ms y 200 ms para cada muestra diferente. El shimming adecuado y la supresión de agua se monitorearon después de terminar las mediciones comparando todos los picos de DMSO en términos de ensanchamiento de línea y pérdida de intensidad de experimentos 1 H1D medidos adicionalmente para todas las muestras. El procesamiento de los espectros de relajación CPMG T219F obtenidos se realizó utilizando una macro previamente preparada y automatizada en TopSpin, respectivamente. El análisis de los datos se realizó siguiendo las instrucciones de la sección de protocolo. Los datos integrales obtenidos de TopSpin (siguiendo las instrucciones del protocolo) se pueden evaluar rápida y fácilmente utilizando una hoja de cálculo prefabricada o cualquier programa similar, estableciendo las condiciones y umbrales correctos. Como se describió anteriormente, los umbrales son útiles para definir aglutinante, cuaderno débil o no cuaderno. La Figura 5 muestra los resultados típicos de los espectros CPMG de thiM RNA y PtkA, respectivamente. En algunos casos, fue necesario realizar una revisión más por parte de expertos.
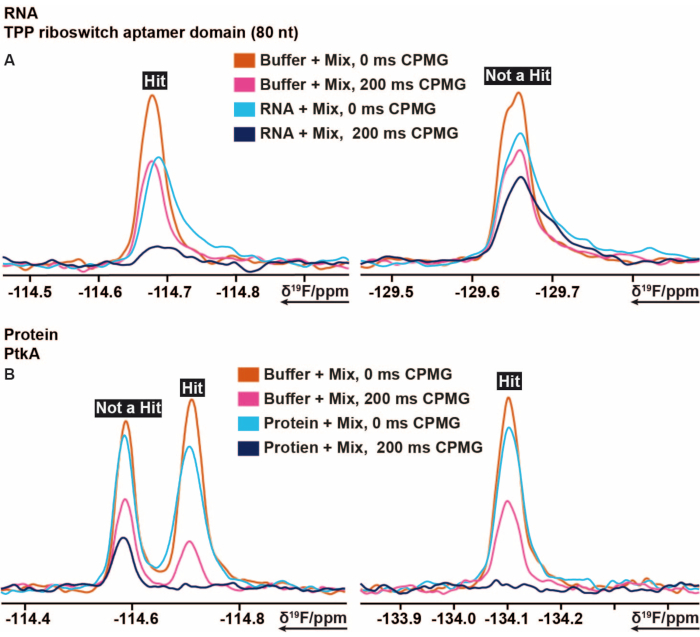
Figura 5: Corte de 19 espectros de RMN de CPMG F que muestran los cambios de intensidad obtenidos de diferentes tiempos de retardo de experimentos basados en CPMG. (A) Representación de un aglutinante (hit) y un no aglutinante en el cribado basado en fragmentos 19F realizado en el riboswitch TPP thiM RNA de E. coli. (B) Representación de un aglutinante y un no aglutinante en el cribado basado en fragmentos 19F realizado en PtkA de M. tuberculosis. Haga clic aquí para ver una versión más grande de esta figura.
1H Proyección
Diseño de mezcla
La biblioteca interna utilizada es tan diversa que para fines de cribado de 1 H no se realizó ningún diseño demezcla. Esto significa que 64 mezclas se prepararon eligiendo al azar 12 para mezclar en una mezcla.
Preparación de muestras
Para el cribado de 1H de un ARN ejemplar del SARS-CoV-2, se realizó un pipeteo automatizado utilizando un robot pipeteador para preparar las muestras. Los fragmentos en cada mezcla tenían una concentración de 4.2 mM en 90% d6-DMSO y 10%D2O. El volumen final de una muestra de cribado fue de 200 μL con 5% deD2Ocomo agente de bloqueo. Se pipetearon 64 muestras cada una con una mezcla diferente en 25 mM KPi, 50 mM KCl a pH 6.2 sin ARN diana. Respectivamente, 64 muestras fueron pipeteadas con ARN diana, cada una conteniendo una mezcla diferente. La relación ARN:Ligando se estableció en 1:20, lo que resultó en una concentración de ARN de 10 μM y una concentración de ligando de 200 μM.
Análisis de datos
Para el análisis de 1H, se utilizó la herramienta FBS en TopSpin. Para determinar si un fragmento es un éxito, se realizaron experimentos de desplazamiento químico 1D, waterLOGSY y relajaciónT2 . Para la relajaciónT2 , una disminución en la intensidad superior al 30% se contó como un golpe, mientras que para el cambio químico un cambio de más de 6 Hz fue el corte. El waterLOGSY tenía que mostrar un cambio de señal significativo (de negativo a positivo en este caso). Si dos de estos tres criterios eran positivos, un fragmento se contaba como un acierto. Dos ejemplos de esto se pueden ver en la Figura 6.
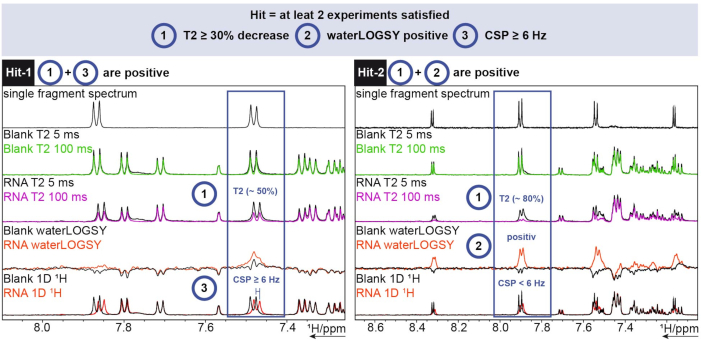
Figura 6: Cribado de 1 H realizado en un ARN ejemplar del SARS-CoV-2 que muestra los criterios de determinación deresultados. Adquisición de tres experimentos diferentes (1 H T2 CPMG (5/100 ms), waterLOGSY y 1D 1H). Haga clic aquí para ver una versión más grande de esta figura.
Hit-1 muestra una disminución de T2 de ~ 50% y un CSP ≥ 6 Hz. El waterLOGSY no muestra un cambio lo suficientemente significativo en la señal como para ser contado como positivo. Como dos de cada tres experimentos son positivos, este fragmento se cuenta como un éxito. Para Hit-2, el T2 muestra una disminución de ~ 80% de intensidad de señal y se puede ver un cambio de señal claro para el waterLOGSY. El CSP no es suficiente en este caso, pero como los dos criterios anteriores son positivos, todavía se cuenta como un éxito.
Discusión
Versatilidad del cribado de fragmentos/fármacos basado en RMN. BMRZ ha implementado con éxito instrumentación automatizada de RMN de última generación, así como STD-RMN, waterLOGSY y experimentos de relajación para identificar fragmentos dentro de una amplia gama de regímenes de afinidad para el descubrimiento de fármacos. El hardware instalado incluye un robot de preparación de muestras de alto rendimiento y un almacenamiento de muestras de alto rendimiento, cambiador y unidad de adquisición de datos asociada a un espectrómetro de 600 MHz. Una sonda criogénica recientemente adquirida para 1 H, 19 F, 13C y 15N garantiza la sensibilidad requerida para las mediciones propuestas y permite un desacoplamiento de 1 H (1) durante la detección de 19F. Esta sonda está conectada a la última generación de consola de RMN que ofrece la posibilidad de utilizar las herramientas de software avanzadas de Bruker, incluyendo CMC-q, CMC-assist, CMC-se y FBS (incluidas en TopSpin). La herramienta de detección basada en fragmentos (FBS) se incluye en la última versión de TopSpin y ayuda a analizar los datos de alto rendimiento que comprenden experimentos de STD, waterLOGSY, T2 / T1 r-relajación. La recolección de muestras líquidas 1D 1H se puede llenar en los tubos de RMN de manera automatizada utilizando el robot de llenado de muestras. Por lo general, un bloque de 96 tubos (3 mm) se llena en aproximadamente dos horas. Los bastidores de placas de 96 pocillos se colocan directamente en el cambiador de muestras HT, que lee el código de barras del bloque y asigna los tubos de RMN a los experimentos controlados por el software de automatización (IconNMR). Se pueden almacenar y programar cinco bastidores de placas de 96 pocillos en el cambiador de muestras HT al mismo tiempo. La temperatura de cada uno de los bastidores individuales se puede controlar y regular por separado. Además, cada muestra individual se puede preacondicionar (precalentamiento y secado en tubo para eliminar la humedad condensada) a la temperatura deseada antes de la medición.
Idoneidad para una amplia gama de aplicaciones. Una de las amplias aplicaciones de este cribado automatizado basado en RMN es identificar y desarrollar nuevos ligandos que se unen a un objetivo biomacromolecular (ADN / ARN / proteínas). Estos ligandos pueden incluir inhibidores ortostéricos y alostéricos que típicamente se unen de forma no covalente. Además, FBDD por RMN se utiliza típicamente como un primer paso para seleccionar compuestos prometedores, los requisitos que deben cumplirse son la disponibilidad del objetivo biomolecular en cantidades suficientes. Este objetivo se divide en dos tareas principales.
La primera tarea es desarrollar y caracterizar una biblioteca de fragmentos interna por las siguientes razones: control de calidad inicial y periódico, caracterización y cuantificación de más de 1000 fragmentos; determinación de la solubilidad de los fragmentos en tampones optimizados para cada diana, en particular para las dianas proteicas; y el establecimiento de varias bibliotecas para dar cabida a diversos andamios y extenderse hacia otras clases de macromoléculas. La segunda tarea es integrar flujos de trabajo para el diseño de fármacos basados en fragmentos (FBDD) mediante RMN utilizando: cribado automatizado observado con ligando 1D (1H y 19F observados); ensayos de reemplazo automatizados (experimentos de competencia con ligando (natural)) para diferenciar la unión ortostérica y alostérica; proyecciones secundarias automatizadas con múltiples fragmentos; cribado automatizado de proteínas 2D y cribado secundario de un conjunto de derivados en torno a un golpe inicial haciendo uso de la biblioteca EU-OPENSCREEN o cualquier otra biblioteca; y volver a perfilar la selección de la biblioteca de la FDA en comparación con los objetivos elegidos.
Además, se puede realizar el metabotipado de varias líneas celulares (relevantes para la enfermedad) para desentrañar los mecanismos reguladores que vinculan el control del ciclo celular y el metabolismo. Además, existe una caracterización funcional de los elementos de regulación de ARN/ADN/proteínas in vivo e in vitro para la optimización de constructo/dominio (optimización de estabilidad para investigaciones estructurales (tampones, pH, temperatura y cribado de sal), y una extensión del cribado de fragmentos basado en RMN a proteínas de membrana y proteínas intrínsecamente desordenadas, que generalmente son inaccesibles para otras técnicas.
Limitaciones. El uso de bibliotecas de fragmentos de 19F y 1H tiene sus pros y sus contras, pocos de los cuales se mencionarán a continuación. El mayor beneficio de las mediciones de 19F frente a 1H es la velocidad tanto del tiempo de medición real como del análisis posterior, ya que las mezclas contienen casi el doble del número de fragmentos y se deben realizar menos experimentos. El análisis de seguimiento también es más fácil para el cribado de 19F, ya que no hay interferencia de los tampones y, además, ofrece un rango de desplazamiento químico más amplio casi sin superposición de señales para una mezcla de fragmentos diseñada de manera óptima. Los espectros mismos están muy simplificados, por lo general sólo tienen una o dos señales por fragmento, dependiendo del número de átomos de flúor. Por lo tanto, el análisis de estos espectros puede automatizarse, reduciendo nuevamente el tiempo. Esto se produce a costa de la diversidad química, al menos para la biblioteca utilizada en este estudio. Como solo ~ 13% de la biblioteca contiene 19 F, pero naturalmente todos ellos son utilizables en la proyección de 1H, la diversidad de los fragmentos de detección de 19F será menor. Esto podría evitarse utilizando bibliotecas 19F diseñadas específicamente con más fragmentos y mayor diversidad química. Otra desventaja para el cribado 19F es el bajo número de señales por fragmento. Los fragmentos generalmente están compuestos de más de un átomo de hidrógeno. Por lo tanto, los experimentos de cribado observados de 1H pueden basarse en diferentes señales para el mismo fragmento para detectar la unión. Esto da un mayor grado de confianza al identificar los aciertos para el cribado de 1H, mientras que el cribado de 19F debe basarse en una o dos señales dadas por fragmento.
Se ha presentado una descripción detallada de la instrumentación automatizada de detección de fragmentos, el software y los métodos y protocolos de análisis automatizados basados en RMN. El hardware instalado incluye un robot de preparación de muestras de alto rendimiento y una unidad de almacenamiento, cambiador y adquisición de datos de muestras de alto rendimiento asociada a un espectrómetro de 600 MHz. Un cabezal de sonda criogénica recientemente instalado para 1 H, 19 F, 13C y 15 N garantiza la sensibilidad requerida para las mediciones propuestas y permite el desacoplamiento de 1H durante la detección de 19F. Además, la última generación de la consola de RMN ofrece la posibilidad de utilizar software analítico avanzado para ayudar a la adquisición y el análisis sobre la marcha. La tecnología discutida anteriormente, los flujos de trabajo y los protocolos descritos deberían fomentar un éxito notable para los usuarios que buscan FBS por RMN.
Divulgaciones
Ninguno.
Agradecimientos
Este trabajo ha sido apoyado por iNEXT-Discovery, proyecto número 871037, financiado por el programa Horizonte 2020 de la Comisión Europea.
Materiales
| Name | Company | Catalog Number | Comments |
| Bruker Avance III HD | Bruker | 600 MHz NMR Spectrometer | |
| Matrix Clear Polypropylene 2D Barcoded Open-Top Storage Tubes | 3731-11 0.75ML V-BOTTOM TUBE/LATCH RACK | ThermoFisher Scientific | Barcoded Tubes |
| Matrix SepraSeal und DuraSeal& | 4463 Cap Mat, SeptraSeal 10/CS | ThermoFisher Scientific | |
| SampleJet | Bruker | HT Sample Changer | |
| SamplePro Tube | Bruker | Pipetting Robot |
Referencias
- Yanamala, N., et al. NMR-Based Screening of Membrane Protein Ligands. Chemical Biology & Drug Design. 75, 237-256 (2010).
- Souers, A. J., et al. ABT-199, a potent and selective BCL-2 inhibitor, achieves antitumor activity while sparing platelets. Nature Medicine. 19, 202-208 (2013).
- Su, M. C., Te Chang, C., Chu, C. H., Tsai, C. H., Chang, K. Y. An atypical RNA pseudoknot stimulator and an upstream attenuation signal for -1 ribosomal frameshifting of SARS coronavirus. Nucleic Acids Research. 33, 4265-4275 (2005).
- Perera, T. P. S., et al. Discovery & pharmacological characterization of JNJ-42756493 (Erdafitinib), a functionally selective small-molecule FGFR family inhibitor. Molecular Cancer Therapeutics. 16, 1010-1020 (2017).
- Zhang, C., et al. Design and pharmacology of a highly specific dual FMS and KIT kinase inhibitor. Proceedings of the National Academy of Sciences of the United States of America. 110, 5689-5694 (2013).
- Lipinski, C. A., Lombardo, F., Dominy, B. W., Feeney, P. J. Experimental and computational approaches to estimate solubility and permeability in drug discovery and development settings. Advanced Drug Delivery Reviews. 23, 3-25 (1997).
- Congreve, M., Carr, R., Murray, C., Jhoti, H. A 'Rule of Three' for fragment-based lead discovery. Drug Discovery Today. 8, 876-877 (2003).
- Chávez-Hernández, A. L., Sánchez-Cruz, N., Medina-Franco, J. L. A Fragment Library of Natural Products and its Comparative Chemoinformatic Characterization. Molecular Informatics. 39, 2000050 (2020).
- Sreeramulu, S., et al. NMR quality control of fragment libraries for screening. Journal of Biomolecular NMR. , 00327-00329 (2020).
- Gao, J., et al. Automated NMR Fragment Based Screening Identified a Novel Interface Blocker to the LARG/RhoA Complex. PLoS One. 9, 88098 (2014).
- Peng, C., et al. Fast and Efficient Fragment-Based Lead Generation by Fully Automated Processing and Analysis of Ligand-Observed NMR Binding Data. Journal of Medicinal Chemistry. 59, 3303-3310 (2016).
- Cox, O. B., et al. A poised fragment library enables rapid synthetic expansion yielding the first reported inhibitors of PHIP(2), an atypical bromodomain. Chemical Science. 7, 2322-2330 (2016).
- Hwang, T. L., Shaka, A. J. Water Suppression That Works. Excitation Sculpting Using Arbitrary Wave-Forms and Pulsed-Field Gradients. Journal of Magnetic Resonance, Series A. 112, 275-279 (1995).
- Gossert, A. D., Jahnke, W. NMR in drug discovery: A practical guide to identification and validation of ligands interacting with biological macromolecules. Progress in Nuclear Magnetic Resonance Spectroscopy. 97, 82-125 (2016).
- Sklenar, V., Bax, A. A new water suppression technique for generating pure-phase spectra with equal excitation over a wide bandwidth. Journal of Magnetic Resonance. 75, 378-383 (1987).
- Binas, O., et al. 19F NMR-Based Fragment Screening for 14 Different Biologically Active RNAs and 10 DNA and Protein Counter-Screens. ChemBioChem. , (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados