Method Article
Criblage de fragments basé sur la RMN dans un échantillon minimum mais un mode d’automatisation maximal
Dans cet article
Résumé
Le criblage par fragments par RMN est une méthode robuste pour identifier rapidement les liants de petites molécules aux biomacromolécules (ADN, ARN ou protéines). Des protocoles décrivant la préparation des échantillons basée sur l’automatisation, les expériences RMN et les conditions d’acquisition, ainsi que les flux de travail d’analyse sont présentés. La technique permet une exploitation optimale des noyaux actifs RMN 1H et 19F pour la détection.
Résumé
Le dépistage par fragments (FBS) est un concept bien validé et accepté dans le processus de découverte de médicaments, tant dans le milieu universitaire que dans l’industrie. Le plus grand avantage du criblage de fragments basé sur la RMN est sa capacité non seulement à détecter des liants de plus de 7 à 8 ordres de grandeur d’affinité, mais aussi à surveiller la pureté et la qualité chimique des fragments et donc à produire des résultats positifs de haute qualité et un minimum de faux positifs ou de faux négatifs. Une condition préalable au sein du FBS est d’effectuer un contrôle de qualité initial et périodique de la bibliothèque de fragments, de déterminer la solubilité et l’intégrité chimique des fragments dans les tampons pertinents et d’établir plusieurs bibliothèques pour couvrir divers échafaudages afin d’accueillir diverses classes cibles de macromolécules (protéines / ARN / ADN). En outre, une optimisation approfondie du protocole de criblage basée sur la RMN en ce qui concerne les quantités d’échantillons, la vitesse d’acquisition et d’analyse au niveau de la construction biologique / espace fragmentaire, dans l’espace des conditions (tampon, additifs, ions, pH et température) et dans l’espace des ligands (analogues du ligand, concentration du ligand) est nécessaire. Au moins dans le milieu universitaire, ces efforts de dépistage ont jusqu’à présent été entrepris manuellement de manière très limitée, ce qui a limité la disponibilité de l’infrastructure de dépistage non seulement dans le processus de développement de médicaments, mais aussi dans le contexte du développement de sondes chimiques. Afin de répondre aux exigences de manière économique, des flux de travail avancés sont présentés. Ils tirent parti du dernier matériel avancé de pointe, avec lequel la collecte d’échantillons liquides peut être remplie de manière automatisée dans les tubes RMN de manière contrôlée. 1Les spectres basés sur les ligands RMN H/19F sont ensuite collectés à une température donnée. Le changeur d’échantillons à haut débit (changeur d’échantillons HT) peut traiter plus de 500 échantillons dans des blocs à température contrôlée. Ceci, associé à des outils logiciels avancés, accélère l’acquisition et l’analyse des données. De plus, l’application de routines de dépistage sur des échantillons de protéines et d’ARN est décrite pour faire connaître les protocoles établis pour une vaste base d’utilisateurs dans la recherche biomacromoléculaire.
Introduction
Le criblage par fragments est maintenant une méthode couramment utilisée pour identifier des molécules plutôt simples et de faible poids moléculaire (MW <250 Da) qui montrent une faible liaison aux cibles macromoléculaires, y compris les protéines, l’ADN et l’ARN. Les résultats initiaux des criblages primaires servent de base pour effectuer un criblage secondaire des analogues plus importants des résultats disponibles dans le commerce, puis pour utiliser des stratégies de croissance ou de liaison de fragments basées sur la chimie. Pour une plate-forme de découverte de médicaments basée sur des fragments (FBDD), en général, une méthode biophysique robuste est nécessaire pour détecter et caractériser les résultats faibles, une bibliothèque de fragments, une cible biomoléculaire et une stratégie de chimie de suivi. Quatre méthodes biophysiques couramment appliquées dans les campagnes de découverte de médicaments sont les tests de décalage thermique, la résonance plasmonique de surface (SPR), la cristallographie et la spectroscopie par résonance magnétique nucléaire (RMN).
La spectroscopie RMN a montré des rôles variés au sein des différentes étapes du FBDD. En plus d’assurer la pureté chimique et la solubilité des fragments dans une bibliothèque de fragments dissoute dans un système tampon optimisé, les expériences RMN observées par ligand peuvent détecter la liaison de fragments à une cible à faible affinité et les expériences RMN observées sur la cible peuvent délimiter l’épitope de liaison du fragment, permettant ainsi des études détaillées de la relation structure-activité. Dans la cartographie des épitopes, les changements de déplacement chimique basés sur la RMN permettent non seulement d’identifier les sites de liaison orthostériques, mais aussi les sites allostériques qui pourraient être cryptiques et accessibles uniquement dans les états conformationnels dits excités de la cible biomoléculaire. Si la cible biomoléculaire se lie déjà à un ligand endogène, les fragments identifiés peuvent être facilement classés comme allostériques ou orthostériques en effectuant des expériences de compétition basées sur la RMN. La détermination de la constante de dissociation (KD) de l’interaction ligand-cible est un aspect important du processus FBDD. Les titrages de décalage chimique basés sur la RMN, que ce soit le ligand ou la cible observée, peuvent être facilement effectués pour déterminer le KD. Un avantage majeur de la RMN est que les études d’interaction sont réalisées en solution et à proximité de conditions physiologiques. Ainsi, tous les états conformationnels pour l’analyse de l’interaction ligand/fragment avec sa cible peuvent être sondés. De plus, les approches basées sur la RMN ne se limitent pas seulement au criblage de protéines solubles bien repliées, mais sont également appliquées pour s’adapter à un espace cible plus large, y compris l’ADN, l’ARN, les protéines liées à la membrane et les protéines intrinsèquement désordonnées1.
Les bibliothèques de fragments sont une partie indispensable du processus FBDD. En général, les fragments agissent comme les précurseurs initiaux qui finissent par faire partie (sous-structure) du nouvel inhibiteur développé pour une cible biologique. Plusieurs médicaments (Vénétoclax2, Vemurafenib3, Erdafitinib4, Pexidartnib5) ont commencé sous forme de fragments et sont maintenant utilisés avec succès dans les cliniques. Typiquement, les fragments sont des molécules organiques de faible poids moléculaire (<250 Da) avec une solubilité et une stabilité aqueuses élevées. Une bibliothèque de fragments soigneusement conçue contenant généralement quelques centaines de fragments peut déjà promettre une exploration efficace de l’espace chimique. La composition générale des bibliothèques de fragments a évolué au fil du temps et a le plus souvent été dérivée en disséquant des médicaments connus en fragments plus petits ou conçus par calcul. Ces diverses bibliothèques de fragments contiennent principalement des aromatiques plats ou des hétéroatomes et adhèrent à la règle de Lipinski de 5 6, ou à la règle de tendance commerciale actuelle de 3 à 7, mais évitent les groupes réactifs. Certaines banques de fragments ont également été dérivées ou composées de métabolites hautement solubles, de produits naturels et/ou de leurs dérivés8. Un défi général posé par la plupart des bibliothèques de fragments est la facilité de la chimie en aval.
Le Center for Biomolecular Magnetic Resonance (BMRZ) de l’Université Goethe de Francfort est partenaire de iNEXT-Discovery (Infrastructure for NMR, EM and X-rays for Translational research-Discovery), un consortium d’infrastructures de recherche structurelle pour tous les chercheurs européens de tous les domaines de la recherche biochimique et biomédicale. Dans le cadre de la précédente initiative d’iNEXT qui s’est achevée en 2019, une bibliothèque de fragments comprenant 768 fragments a été conçue dans le but de « fragments minimum et diversité maximale » couvrant un grand espace chimique. En outre, contrairement à toute autre bibliothèque de fragments, la bibliothèque de fragments iNEXT a également été conçue sur la base du concept de « fragments posés » dans le but de faciliter la synthèse en aval de ligands complexes de haute affinité et désormais connue sous le nom de bibliothèque interne (Diamond, Structural Genomic Consortium et iNEXT).
L’établissement de la DFDD par RMN nécessite de la main-d’œuvre, des connaissances et de l’instrumentation. Au BMRZ, des flux de travail optimisés pour soutenir l’assistance technique au criblage des fragments par RMN ont été développés. Il s’agit notamment du contrôle de la qualité et de l’évaluation de la solubilité de la bibliothèque de fragments 9, de l’optimisation du tampon pour les cibles choisies, du criblage basé sur le ligand 1D observé 1 H ou 19F, des expériences de compétition pour différencier la liaison orthostérique et allostérique, desexpériences de RMN observées sur des cibles 2D pour la cartographie des épitopes et pour caractériser l’interaction avec un ensemble secondaire de dérivés des impacts initiaux du fragment. BMRZ a établi des routines automatisées pour l’analyse, comme indiqué précédemment dans la littérature 10,11, des interactions petites molécules-protéines et a mis en place toute l’infrastructure automatisée nécessaire pour le criblage de fragments basé sur la RMN. Il a mis en œuvre la RMN par différence de transfert de saturation (STD-RMN), les ligands de l’eau observés par spectroscopie de gradient (waterLOGSY) et les expériences de relaxation basées sur Carr-Purcell-Meiboom-Gill (basées sur CPMG) pour identifier des fragments dans un large éventail de régimes d’affinité ainsi que des instruments RMN automatisés de pointe et des logiciels pour la découverte de médicaments. Bien que le criblage de fragments basé sur la RMN soit bien établi pour les protéines, cette approche est moins couramment utilisée pour trouver de nouveaux ligands interagissant avec l’ARN et l’ADN. BMRZ a établi une preuve de concept pour de nouveaux protocoles permettant l’identification des interactions petites molécules-ARN/ADN. Dans les sections suivantes de cette contribution, l’application de routines de dépistage sur des échantillons de protéines et d’ARN est signalée pour faire connaître les protocoles établis pour une vaste base d’utilisateurs dans la recherche biomacromoléculaire.
Protocole
1. Bibliothèque de fragments
- Bibliothèque de fragments interne
NOTE: Dans le cadre de l’une des activités de recherche conjointes de l’iNEXT, une bibliothèque de fragments de première génération robuste et adaptée à la chimie en aval a été développée12 et par la suite une deuxième génération de la bibliothèque a été constituée en collaboration avec Enamine et est connue sous le nom de bibliothèque de fragments DSI (Diamond-SGC-iNEXT) (désormais appelée « bibliothèque interne »). Cette bibliothèque peut être mise à disposition au BMRZ à des fins de projection.- Évaluer l’intégrité et la solubilité de la bibliothèque de fragments à l’aide d’un protocole 9 basé sur la RMNprécédemment signalé.
NOTE: La bibliothèque interne se compose de 768 fragments avec une très grande diversité chimique (>200 singletons). La réalisation du criblage dans des mélanges de fragments peut accélérer considérablement la campagne de criblage; cependant, le nombre de fragments dans un mélange est limité en raison du chevauchement du signal dans le spectre 1H-RMN. La plus grande diversité chimique offerte par la bibliothèque interne permet la préparation de mélanges contenant 12 fragments différents sans chevauchement significatif de décalage chimique dans les spectres RMN observés à 1H. - 103 fragments dans les 768 fragments possèdent un atome de fluor. Aux fins du criblage 19 F, diviser les 103 fragments qui possèdent un groupe fluor en 5 mélanges basés sur un chevauchement chimique minimal de 19F. Pour minimiser le chevauchement des signaux dans le criblage 19F, utiliser les informations de décalage chimique provenant de mesures de composés uniques pour concevoir des mélanges avec un nombre maximal de fragments et un chevauchement minimal du signal. Chaque mélange a 20-21 fragments avec des décalages chimiques distincts de 19F permettant une affectation sans ambiguïté des fragments.
- Évaluer l’intégrité et la solubilité de la bibliothèque de fragments à l’aide d’un protocole 9 basé sur la RMNprécédemment signalé.
- Bibliothèque de fragments définie/fournie par l’utilisateur
- Effectuer des campagnes de filtrage avec la bibliothèque de fragments définie ou fournie par l’utilisateur; Cependant, les étapes suivantes doivent précéder la campagne de dépistage.
- Si l’utilisateur ne l’a pas spécifié au préalable, effectuez un contrôle de qualité des fragments basé sur la RMN (au BMRZ, des outils logiciels avancés sont utilisés à cet effet; 9, chapitre 6.1.1).
- Vérifiez la solubilité des fragments dans le tampon de choix pour la cible biomoléculaire, l’intégrité de la structure et la concentration des fragments avant utilisation.
- Concevoir le mélange de manière à réduire à la fois le chevauchement des signaux dans les spectres RMN et le temps de mesure.
- Concevoir les mélanges conformément à l’étape 4.2.
- Filtrez des fragments uniques ou un sous-ensemble de mélanges au lieu de la bibliothèque entière.
2. Préparation des échantillons
REMARQUE: Le criblage à haut débit par RMN utilise un robot de pipetage pour la préparation des échantillons. Les spectres RMN, mais aussi les stabilités sur plusieurs jours d’acquisition du signal des protéines, des ARN et de l’ADN sont extrêmement sensibles aux fluctuations de température et, par conséquent, les systèmes automatisés à température contrôlée faciliteront grandement la stabilité des échantillons pipetés. À cette fin, un dispositif complémentaire supplémentaire, qui fonctionne entre 4 et 40 °C, est couplé au robot de pipetage pour la manipulation liquide des échantillons RMN dans un environnement à température contrôlée.
- Préparation du mélange de ligands
- Préparer des échantillons de dépistage pour les mesures RMN à l’aide d’un robot de préparation d’échantillons. La configuration flexible du robot permet un large éventail d’applications (par exemple, récupération des échantillons des tubes RMN dans des conteneurs de stockage ou tâches générales de manipulation de liquides). Des tubes RMN de différents diamètres (1,7, 2,0, 2,5, 3,0 et 5,0 mm) peuvent être utilisés. Le système d’échantillonnage robotisé ainsi que le logiciel de contrôle avancé lisent le code-barres attribué à chaque type de conteneur et exécutent le protocole de remplissage de liquide de manière optimale.
- Pour la préparation des mélanges de ligands de bibliothèque internes, utilisez des flacons à code-barres. Les flacons à code-barres garantissent le plus haut niveau de fiabilité et une traçabilité optimale des échantillons.
- Répartir 768 composés dans 8 plaques de format 96 puits. La concentration stockaire de chaque fragment individuel est de 50 mM dans d6-DMSO/D2O (9:1). Au total, préparez 64 mélanges contenant chacun 12 fragments. La concentration finale de chaque fragment dans un mélange est de 4,2 mM.
REMARQUE: Le robot de pipetage peut accueillir une variété de types de conteneurs avec des géométries variées (flacons cryo- ou échantillonneurs, plaques de 96 puits de profondeur ronde ou carrée, flacons standard à code-barres, tubes microcentrifuges) et aide à l’exécution efficace du transfert de liquide vers une variété de tubes et de racks RMN.

Figure 1 : (A) Préparation d’échantillons RMN à haut débit et robot de remplissage de tubes RMN installés à BMRZ. (B) Changeur d’échantillons à haut débit avec racks individuels à température contrôlée installés sur un spectromètre de 600 MHz dans l’installation de BMRZ. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Préparation de l’échantillon de criblage à blanc (spectre du ligand de référence) et avec cible (ligand en présence de la cible)
- Pour la préparation des échantillons de criblage RMN, en présence de la biomolécule cible (protéine/ARN/ADN) et du mélange de ligands, utiliser des tubes changeurs d’échantillons RMN HT de 3 mm choisis dans le portefeuille de tubes RMN standard Bruker.
- Transférer la cible biomoléculaire (p. ex., criblage 1H : ARN ou protéine 10 μM) dans un tampon de criblage défini dans le tube RMN de 3 mm (volume final de 200 μL) manuellement ou à l’aide du robot de pipetage.
- Transférer 10 μL (p. ex., criblage de 1H) du mélange de ligands à l’étape suivante à l’aide du système robotique dans les tubes RMN de 3 mm à code à barres contenant la biomolécule cible et mélanger à l’aide du protocole intégré du logiciel de contrôle.
REMARQUE: Le numéro de code-barres du tube RMN est incorporé de manière pratique et automatique dans l’ensemble de données RMN acquis, garantissant ainsi un flux de travail orienté ID sans aucune confusion. L’accessoire de contrôle de température du robot de pipetage permet de maintenir les échantillons préparés dans les tubes RMN sous température constante.
- Conditions et paramètres définis en interne
- Établir des conditions tampons optimales pour effectuer le criblage de l’ARN et des protéines par rapport à la bibliothèque de fragments interne. Les échantillons conditionnés suivants sont utilisés pour l’ARN au BMRZ : 25 mM KPi, 50 mM KCl, pH 6,2. Mg2+ est facultatif.
- Les protéines sont extrêmement sensibles aux conditions de solution; Utilisez des tampons optimaux pour la cible de choix. Pour chacun de ces tampons, acquérir des spectres de référence supplémentaires des ligands pour servir de blanc pour l’analyse.
- Conditions spécifiées par l’utilisateur
REMARQUE : Dans les cas où les conditions établies à l’interne ne conviennent pas aux cibles à filtrer auprès d’un utilisateur potentiel, les étapes suivantes doivent être mises en œuvre.- Effectuer 1H-RMN du tampon seul pour assurer une interférence minimale des composants du tampon dans l’exécution et l’analyse des expériences de criblage observées par le ligand. Les composants interférents pourraient être remplacés de manière appropriée par des équivalents deutérés.
- Limites de la production d’échantillons (quantités cibles)/conditions et disponibilité
REMARQUE : L’isolement ou la production recombinante de certaines biomacromolécules peut, dans certains cas, s’avérer difficile et limiter la disponibilité de la cible pour mener à bien une campagne de sélection de médicaments. En cas de disponibilité limitée ou illimitée des cibles, les solutions de rechange suivantes pourraient être utilisées pour mener à bien un filtrage des fragments basé sur la RMN.- S’il est limité, utilisez 19tests de dépistage basés sur la RMN-F. Les ligands fluorés typiques ont un seul signal 19F; Par conséquent, utilisez des cocktails avec 25-30 fragments sans aucun chevauchement de signal. Il y a moins de signaux à analyser, aucune interférence de signal provenant des composants tampons et moins de signaux sur lesquels s’appuyer pour l’identification des résultats.
- Si illimité, utilisez des écrans plus grands comme 1H-RMN. La plus grande bibliothèque de fragments peut être filtrée. En règle générale, les fragments sont composés de plus d’un proton, ce qui signifie plus de signaux sur lesquels s’appuyer pour l’analyse.
3. Conditions d’acquisition RMN
- Conditions internes généralement définies
- Spectromètre équipé d’un changeur d’échantillons HT (Automation)
- Pour le criblage à haut débit, utilisez des plaques à 96 puits qui ne peuvent être mesurées qu’à l’aide d’un changeur d’échantillons HT. Le changeur d’échantillons HT offre également la possibilité de tempérer chaque rack individuellement.
- Pour un rapport signal/bruit optimal, utilisez un spectromètre avec une sonde cryogénique refroidie à l’hélium ou à l’azote. Un module de réglage et de correspondance automatisé (ATM) est nécessaire pour l’automatisation.
- Jeux de paramètres et séquences d’impulsions
REMARQUE: De nombreuses expériences RMN peuvent caractériser les événements de liaison. L’identification des résultats varie en fonction de la configuration expérimentale. Les expériences suivantes sont couramment utilisées dans les campagnes de dépistage BMRZ. Néanmoins, des modifications peuvent être apportées pour les campagnes de dépistage définies par l’utilisateur et selon les spécifications de l’utilisateur.- Si vous utilisez le logiciel TopSpin, incluez le jeu de paramètres pour les expériences basées sur des ligands : SCREEN_STD, SCREEN_T1R, SCREEN_T2, SCREEN_WLOGSY. L’ensemble de paramètres comprend tous les paramètres nécessaires et les séquences d’impulsions: STD: stddiffesgp.3; T1ρ: t1rho_esgp2d; T2: cpmg_esgp2d; et waterLOGSY: ephogsygpno.2.
- Pour toutes les expériences énumérées, utilisez la sculpture d’excitation13 comme suppression de l’eau. Pour référence, utilisez la sculpture d’excitation 1D (zgesgp). Le nombre de balayages dépend de la sensibilité du système (intensité du champ magnétique et tête de sonde), de la concentration de l’échantillon et du choix de l’expérience. Une recommandation est: 1D avec NS = 64, T1ρ & T2 avec NS = 128, STD avec NS = 256 et waterLOGSY avec NS = 384 ou 512.
- Pour le criblage 19 F, utilisez les expériences 1D et T2 : 1D : F19CPD (pp=zgig) pour 19 F{1 H}-tête de sonde et F19(pp=zg) pour 19F/1tête de sonde H ; SCREEN_19F_T2 (pp = cpmgigsp).
- Utiliser une largeur spectrale de 220 ppm et une fréquence d’excitation à -140 ppm. La durée de l’expérience est comprise entre 1 et 5 heures (assurer la stabilité à long terme de la biomacromolécule) en fonction du matériel et de la concentration de l’échantillon. Pour T2, le temps CPMG devrait alterner entre 0 ms et 200 ms.
- Traitement
- Enregistrer les expériences STD, T1ρ et T2 en pseudo 2D. Pour traiter les deux spectres 1D simples, IconNMR utilise le programme au proc_std avec ou sans l’option relax. La première option fournit la référence 1D et la différence de deux spectres. La deuxième option donne deux spectres distincts avec un temps de relaxation court et long. Le waterLOGSY est un 1D unique qui doit être phasé avec un négatif pour le signal de solvant.
- Spectromètre équipé d’un changeur d’échantillons HT (Automation)
- Conditions spécifiques à l’utilisateur
- Adaptez l’un des paramètres mentionnés précédemment aux conditions définies par l’utilisateur. Par exemple, si une protéine fournie par l’utilisateur d’une installation n’est pas stable à la température généralement utilisée, des expériences d’optimisation peuvent être menées en variant la température, la concentration, les conditions tampons, etc.
4. Analyse des données
- Bibliothèque de fragments QC (d6-DMSO/tampon spécifique) et quantification
- CMC-q
REMARQUE : Le contrôle de la qualité des bibliothèques de fragments est essentiel avant le lancement des campagnes de sélection. En outre, la stabilité à long terme de la bibliothèque de fragments doit être assurée pour l’application de plusieurs campagnes de filtrage, c’est pourquoi une évaluation périodique de la qualité de la bibliothèque doit être effectuée. À cette fin, les logiciels intégrés CMC-q et CMC-a de TopSpin sont utilisés pour l’évaluation de la qualité et de la quantité. Le CMC-q et le CMC-a sont des modules logiciels de Topspin qui permettent une acquisition fluide, une analyse incluant la vérification de la structure à l’aide du spectre RMN 1H obtenu à partir de petites molécules organiques 9.- Pour assurer l’intégrité, préparer des échantillons d’évaluation contenant une concentration de fragments de 1 mM dans du d6-DMSO. Préparez les échantillons de manière automatisée avec un robot de pipetage en remplissant le prélèvement d’échantillons liquides dans un tube RMN de 3 mm.
- Pour l’évaluation de la solubilité, utiliser un échantillon constitué de 1 mM composé dans un tampon de phosphate de sodium de 50 mM à pH 7,4, de chlorure de sodium de 150 mM, de sel de sodium à 90 % H 2O/ 10 % D 2 O et de 1 mM de sel de sodium acide 3-(triméthylsilyl)propionique-2,2,3,3-d4 (TMSP-Na).
- Recueillir des spectres RMN à 298 K ou 293 K à l’aide d’un spectromètre RMN de 600 MHz équipé d’une sonde cryogénique TCI de 5 mm à triple résonance et d’un changeur d’échantillons HT, qui peut traiter 579 échantillons à la fois.
- Pour configurer le logiciel CMC-q, suivez les instructions du manuel de l’utilisateur, qui implémente la création d’un utilisateur IconNMR, l’activation de FastLaneNMR et la modification du changeur d’échantillons HT.
- Calibrez l’impulsion à 90° et enregistrez-la dans la table de prosol TopSpin.
- Placez la plaque de puits d’échantillon 96 dans l’une des 5 positions de rack dans le changeur d’échantillons HT.
- Pour charger un fichier SDF (fichier de données de structure) qui doit contenir sa structure chimique proposée, un identifiant unique et la position dans le changeur d’échantillons HT de chaque échantillon d’un lot, allez dans Parcourir dans la fenêtre de configuration CMC-q et cliquez sur Ouvrir après avoir sélectionné un fichier qui se termine par .sdf.
- Dans les paramètres CMC.q Batch Automation, définissez le type de vérification qui définit l’expérience qui sera mesurée, l’utilisateur IconNMR et définissez le solvant.
- Définissez les fichiers SDF pour le chemin du fichier SDF, l’ID de molécule et la position de l’échantillon.
- Commencez l’acquisition en cliquant sur Démarrer. Cliquez à nouveau sur Démarrer l’acquisition . Le programme d’installation CMC-q peut également être sauvegardé en cliquant sur Enregistrer.
- Pour une description détaillée des étapes de configuration CMC-q, suivez les instructions du manuel d’utilisation de Bruker.
- CMC-a
- Pour CMC-a, utilisez le module logiciel de Topspin qui permet l’analyse, y compris la vérification de la structure à l’aide du spectre RMN 1H obtenu à partir de petites molécules organiques9.
- CMC-q
- Conception du mélange
REMARQUE: Une conception de mélange appropriée joue un rôle important pour le dépistage en utilisant la RMN comme plate-forme. Un nombre élevé de fragments par mélange permet un dépistage plus rapide, mais augmente le risque de faux positifs et négatifs. Un nombre plus faible diminue ce risque, mais augmente le temps nécessaire pour effectuer le dépistage. En général, un chevauchement de signal doit être évité lors de la création de mélanges. En utilisant la bibliothèque interne, cela peut être négligé pour le criblage de 1 H car la bibliothèque a été spécialement conçue pour être diversifiée et montrer peu dechevauchement de signaux tout en maintenant une grande diversité chimique. Cela signifie qu’aucune procédure de conception spéciale ne doit être suivie pour créer les 64 mélanges.- Comme le criblage 19F repose sur les fragments de la bibliothèque interne qui contiennent du fluor et que la bibliothèque n’a pas été créée pour réduire le chevauchement du signal pour ces fragments spécifiques, concevez un mélange approprié.
- Mesurer les spectres de composés simples pour tous les fragments contenant 19F.
- Notez les informations de décalage chimique de chaque signal.
- Selon ces informations, choisissez 20-21 fragments par mélange. Cela donne à son tour 5 mélanges contenant chacun 20-21 fragments sans chevauchement de signal et permet une analyse semi-automatisée des données.
- Effectuer l’identification des coups dans une interaction biomacromolécule-ligand observée
NOTE: Il existe différentes définitions d’un résultat positif entre la procédure de dépistage 19F et 1H. Les identifications de succès suivantes ont été mises en place par nos soins et suivent des règles spécifiques. Le sujet de la détermination des résultats est très subjectif et peut différer d’un utilisateur à l’autre. Néanmoins, il est de la plus haute importance que les règles d’identification des résultats positifs ne changent pas une fois convenues pour maintenir la validation et la crédibilité.- 1Écran H
- Pour déterminer en toute confiance les coups, acquérir des spectres 1D 1H, waterLOGSY et des expériences de relaxation T2 à la fois en présence et en l’absence de cible pour identifier les liants. Les trois expériences ont le potentiel de montrer un événement liant. Si un CSP supérieur à 6 Hz est visible dans les spectres d’échantillon par rapport aux spectres blancs, cela est considéré comme une indication d’un résultat. Il en va de même si un signal positif fort dans l’eauLOGSY ainsi qu’une réduction de plus de 30% T2 dans les spectres de l’échantillon sont visibles. Les événements de liaison peuvent être présentés dans les trois expériences, lors de la comparaison de l’échantillon contenant des spectres avec leurs spectres blancs respectifs. Cependant, les événements contraignants peuvent ne pas être visibles dans les trois expériences. Pour cette raison, il a été convenu qu’au moins deux des événements décrits précédemment doivent se produire afin de classer un fragment comme un résultat contraignant.
- Utilisez l’outil FBS de TopSpin pour définir l’état des fragments en liaison, ambigu, inconnu, agrégats et non liants.
- Lorsque vous avez terminé avec un mélange, approuvez-le dans l’outil FBS.
- Dans l’onglet résumé du projet FBS, cliquez sur Créer un rapport de présélection. Cela ouvrira une fenêtre qui crée un fichier .xlsx. L’utilisateur peut alors choisir entre tous les ligands, ligand liant uniquement, non ligand contraignant uniquement et ligands ambigus à signaler dans la feuille de calcul.
- 19Écran F
- Pour différencier les reliures sans classeur, les classeurs hebdomadaires et les reliures pleins, divisez le quotient d’intégration entre la mesure cible de 200 ms et la mesure d’un blanc de 200 ms par le quotient de la mesure cible de 0 ms et la mesure de blanc de 0 ms est utilisée :
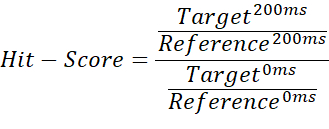
REMARQUE: Cela donne des valeurs allant de 0 à ~1 (le hit-score), ce qui permet d’attribuer des seuils pour chaque état de liaison. - Utiliser la moyenne de la mesure de référence de 200 ms comme seuil de référence pour marquer les cas où le score de réussite dépasse 1. Cela peut se produire si les intégrales importées contiennent des valeurs négatives ou si la mesure de référence est supérieure à la mesure cible. Un score de ≤ 0,67 est considéré comme un coup faible, < 0,33 un coup fort, et tout ce qui > 0,67 comme non-coup. Un exemple est présenté à la figure 2.
- Pour différencier les reliures sans classeur, les classeurs hebdomadaires et les reliures pleins, divisez le quotient d’intégration entre la mesure cible de 200 ms et la mesure d’un blanc de 200 ms par le quotient de la mesure cible de 0 ms et la mesure de blanc de 0 ms est utilisée :
- 1Écran H
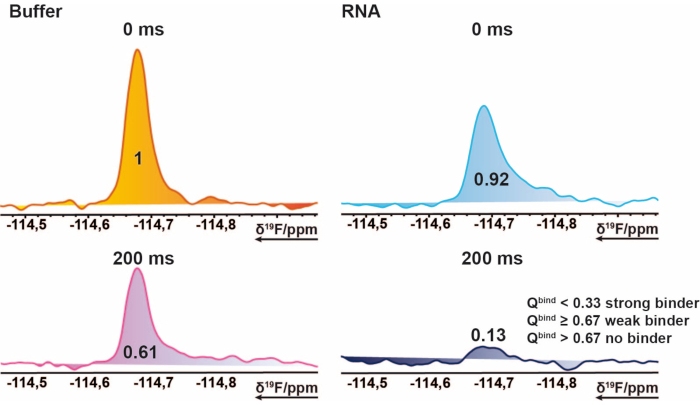
Figure 2 : Identification des résultats positifs pour le dépistage 19F. Section de 19F spectres RMN cpmg d’un composé exemplaire. Cette représentation picturale explique les propriétés d’un liant. 19Spectres F-CPMG d’un composé acquis à partir d’échantillons de mélange en présence et en l’absence d’ARN. Les valeurs représentent les valeurs intégrales normalisées du pic correspondant. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Analyse des données
- Préparer les données pour l’analyse
REMARQUE: Il est important que les données acquises ne présentent pas de défauts visibles. Cela signifie que les données où le calage était problématique ou où la suppression de l’eau était insuffisante ne devraient pas être prises en compte pour l’analyse. Il est plutôt recommandé d’enregistrer à nouveau les données et de s’assurer que tout va bien avec l’échantillon (par exemple, pas de bulles d’air), avec la température, le calage et la suppression de l’eau. L’exactitude des données peut toujours être évaluée lors de la comparaison des signaux DMSO. - 1Dépistage H
- Pour analyser les données de dépistage 1H, utilisez l’outil FBS (nécessite une licence supplémentaire) dans TopSpin 4.0.9.
- Suivez les instructions du manuel de l’outil FBS pour commencer l’analyse des données. Les étapes suivantes résument la procédure décrite dans le manuel.
- Stocker les données RMN BMRZ des campagnes de dépistage de manière à ce que chaque mélange de dépistage ait son propre répertoire dans lequel un sous-répertoire contient les différentes expériences mesurées sur l’échantillon.
- Pour utiliser l’outil FBS, stockez les spectres de référence qui ont toutes les données enregistrées à partir d’échantillons sans la cible biomoléculaire mais avec les mélanges ainsi que le composé unique mesuré dans différents répertoires /nmr. Ceci est important car l’outil FBS demandera le chemin du répertoire de chacun individuellement.
REMARQUE : L’outil FBS reconnaît un répertoire comme projet de sélection si les jeux de données suivants ont été stockés dans le même répertoire que les mélanges d’un échantillon de dépistage (csv, documents XML FragmentScreen et fichier BAK). - Lorsque vous utilisez TopSpin 4.0.9, créez un chemin direct vers le répertoire contenant les données acquises, appelé DIR. Choisissez le répertoire /nmr dans lequel tous les mélanges doivent avoir un répertoire distinct.
- Pour démarrer l’outil FBS d’un échantillon filtré, faites glisser le symbole du projet FBS au milieu de la fenêtre TopSpin. Dans le répertoire choisi, le symbole du projet FBS doit apparaître si les jeux de données précédemment en ont été copiés.
- La fenêtre Options de filtrage basées sur les fragments doit s’ouvrir automatiquement lors du premier chargement d’un nouveau projet FBS. Dans cette fenêtre, choisissez un fichier cocktail. Le fichier cocktail est un fichier csv contenant l’attribution du nom des mélanges, le nom de chaque fragment et leur division en mélanges. Définissez également un dossier de spectres de ligand de référence contenant tous les spectres mesurés des fragments individuels. Enfin, définissez un dossier d’expérience vierge de référence, qui est généralement le dossier contenant les jeux de données des mélanges sans la cible étudiée.
- Les options de criblage basées sur les fragments ont un onglet appelé Types de spectres qui permet de définir les spectres étudiés ainsi que la couleur pour afficher les spectres . Définissez le Spectype en fonction des données préalablement traitées. Dans l’onglet Disposition de l’affichage , définissez les spectres qui seront comparés les uns aux autres en fonction de leurs spécifications.
- Appuyez sur OK pour démarrer le projet FBS.
- Tout en regardant les données, une fenêtre séparée s’ouvrira, résumant tous les mélanges de cocktails et tous les ligands de chaque mélange dans un tableau. En double-cliquant sur une cellule, les jeux de données respectifs s’ouvriront, comparant par exemple les spectres blancs 1H 1D avec le jeu de données contenant la cible.
- Avant d’attribuer des liants, assurez-vous que les pics de référence (DMSO de toutes les mesures ainsi que des composés individuels) correspondent les uns aux autres et ont le même décalage chimique. Si des différences sont observées, corrigez-les en utilisant l’option de traitement série de TopSpin.
- L’option de traitement série se trouve sous l’onglet Processus sous Avancé. Il applique les modifications à tous les spectres sélectionnés à partir d’un jeu de données. De cette façon, les spécifices peuvent facilement être assignées aux numéros d’expérience et tous les spectres peuvent être déplacés en même temps pour s’aligner sur la référence.
- 19F Dépistage
- Pour la première analyse des mélanges 19F, créez un fichier d’intégration pour chaque mélange. Pour définir la région d’intégration, cliquez sur la fonction Intégrer dans l’onglet Analyser . Assurez-vous que pour chaque fragment du mélange, une région d’intégration claire pour les 19F-singal correspondants est définie.
- Utilisez le bouton Enregistrer/Exporter les régions d’intégration pour exporter le fichier d’intégration en vue d’une utilisation ultérieure. Enregistrez tous les fichiers d’intégration utilisés dans C:\Bruker\TopSpin4.0.9\exp\stan\nmr\lists\intrng, ou le chemin correspondant du répertoire d’installation de TopSpin.
- Pour les données 19F, ouvrez un jeu de données avec ou sans la cible étudiée.
- Pour charger le fichier d’intégration dans le spectre actuel, ouvrez à nouveau l’onglet Analyser , allez dans Intégrer et, à l’aide du bouton Lecture/Importation des régions d’intégration, chargez le fichier d’intégration correspondant. Cela chargera toutes les régions définies de ce fichier dans le spectre actuel.
- Enregistrez et revenez pour rechercher une liste de toutes les régions intégrées dans l’onglet Intégrales . Copiez-le dans une feuille de calcul ou tout autre outil utilisé pour l’analyse plus approfondie des données.
- Répétez cette procédure pour chaque mélange, avec et sans cible.
- Gestion des données
- Pour faciliter l’utilisation et la productivité, configurez un flux de travail uniforme pour l’analyse et le stockage ultérieurs des données acquises. Pour le dépistage 1H et 19F, utilisez une feuille de calcul spécialement conçue pour chacun.
NOTE: Pour le criblage 1H, cela a été purement utilisé pour la gestion des données et pour résumer chaque cible, tandis que pour le criblage 19F, il a utilisé le quotient expliqué au chapitre 4.3 pour étiqueter automatiquement chaque fragment comme hit / no hit après que les données intégrales y aient été copiées. Cela réduit le risque d’erreur humaine lors de l’analyse, en supposant que le fichier a été configuré correctement, et facilite le partage des informations, car toutes les informations importantes sont rassemblées en un seul endroit dans un fichier qui peut être ouvert par pratiquement n’importe qui sans avoir besoin d’autres programmes pour examiner les données.
- Pour faciliter l’utilisation et la productivité, configurez un flux de travail uniforme pour l’analyse et le stockage ultérieurs des données acquises. Pour le dépistage 1H et 19F, utilisez une feuille de calcul spécialement conçue pour chacun.
- Préparer les données pour l’analyse
Résultats
Contrôle qualité de la bibliothèque de fragments
Les fragments de la bibliothèque interne ont été livrés sous forme de solutions mères de 50 mM dans 90% de d6-DMSO et 10% deD2O(10% de D2O assure la minimisation de la dégradation des composés due aux cycles répétés de gel-dégel14). Les échantillons composés uniques étaient constitués de 1 mM de ligand dans un tampon phosphate de 50 mM (25 mM KPi pH 6,2 + 50 mM KCl + 5 mM MgCl2), pH 6,0 dans 90% H 2 O/9% D2O/1% d6-DMSO. 1Des expériences de RMN-H de fragments de la bibliothèque iNEXT ont été mesurées sur un spectromètre RMN 500/600 MHz. Ces données ont ensuite été utilisées pour identifier les composés individuels dans les campagnes de criblage de 1 H à l’aide du logiciel CMC-q qui permet à l’utilisateur d’acquérir pleinement des spectres demanière automatisée et l’addon d’analyse CMC-a la qualité (solubilité et intégrité) des fragments a été évaluée. Les résultats de l’analyse automatisée de CMC-a sont présentés sous forme de sortie graphique similaire à ce qui est représenté à la figure 3. La sortie graphique montre une représentation d’une plaque de 96 puits. Un cercle de couleur rouge signifie que ce fragment présente une incohérence dans la structure ou la concentration. Les puits de couleur verte indiquent que le fragment est cohérent.
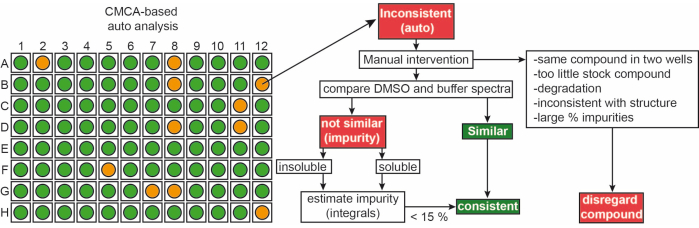
Figure 3 : Contrôle de la qualité de la bibliothèque de fragments. Représentation schématique de la sortie automatisée basée sur CMC-a. Les propriétés des fragments telles que la concentration et l’intégrité structurelle sont évaluées. Le vert signifie cohérent, l’orange dans ce cas signifie incohérent. Les fragments incohérents sont révisés manuellement en suivant le flux de travail affiché. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Environ 65 % et 35 % des fragments ont été classés comme cohérents et incohérents, respectivement, dans le DMSO et le tampon. De plus, 30 % des ligands classifiés incohérents sont devenus cohérents après une inspection manuelle minutieuse des spectres9.
19F Conception du mélange
103 fragments contenant un ou plusieurs groupes fluorés de la bibliothèque interne ont été divisés en 5 mélanges (A, B, C, D, E). Chaque mélange contient de 20 à 21 fragments. Dans ce cas, les mélanges devaient être soigneusement conçus pour éviter le chevauchement des signaux. 19Des expériences de relaxation transversale F ont été mesurées pour chaque mélange qui applique des trains d’impulsions CPMG. Ces expériences peuvent être modifiées en variant les délais de relaxation. Le décalage chimique de 19F des mélanges A-E peut être vu sur la figure 4.
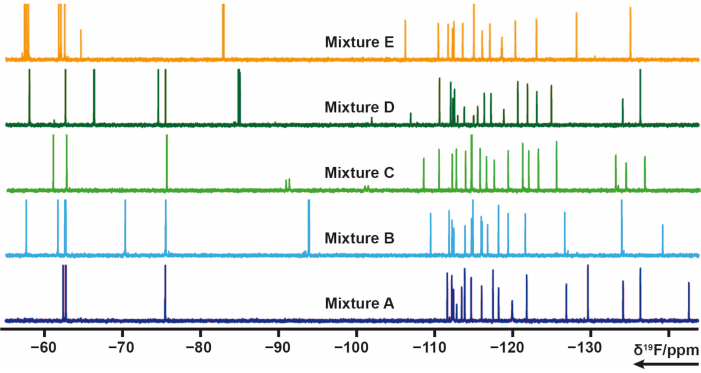
Figure 4 : 19spectres RMN F 1D-RMN d’échantillons de mélange provenant de la bibliothèque interne. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Préparation des échantillons
La préparation de l’échantillon dans la procédure de criblage 19F a été effectuée manuellement ou avec un pipetage automatisé à l’aide d’un robot de pipetage. Les fragments de chaque mélange avaient une concentration de 2,5 mM dans 90% de d6-DMSO et 10% deD2O. Le volume final d’un échantillon de dépistage était de 170 μL avec 5 % deD2Ocomme agent de verrouillage. Chaque mélange a été pipeté deux fois, l’une dans une solution tampon contenant (sans cible) et l’autre dans une solution tampon contenant une cible. Le rapport entre la cible et le fragment a été fixé à 1:1, ce qui a donné une concentration finale cible/ligand de 50 μM. De plus, les échantillons témoins sont la biomolécule cible dans le tampon de criblage sans mélange pour assurer l’intégrité de la cible ainsi qu’un échantillon témoin avec seulement un tampon et D2O pour assurer la qualité du tampon.
Les données de dépistage RMN de 19 F-1Det 19F-CPMG-T2 ont été mesurées comme décrit à la section 3.1. Par exemple, dans le cas de l’ARN, une séquence d’écho de saut-retour (pp = zggpjrse,15) a été acquise pour l’échantillon cible unique en tampon.
Analyse des données
La procédure de dépistage 19 F a été appliquée au riboswitch TPP thiM d’E. coli et à la protéine tyrosine kinase (PtkA) de M. tuberculosis parmi plusieurs autres cibles 16. La bibliothèque de criblage 19F comprend 103 fragments qui sont divisés en 5 mélanges étiquetés du mélange A au mélange. La préparation des échantillons de criblage peut être effectuée manuellement sans l’utilisation d’un robot de pipetage d’échantillons. Une solution contenant 40 μM d’ARN thiM (conditions tampons) a été mélangée avec 3,2 μL provenant des mélanges. D’autres échantillons témoins ont été préparés comprenant uniquement un tampon, un tampon contenant 5 % de DMSO (garantissant auparavant la stabilité de la biomacromolécule en présence de la concentration souhaitée de DMSO) et un tampon contenant de l’ARN. Ces 13 échantillons de dépistage ont été préparés et transférés dans des tubes RMN de 3 mm. Les codes-barres des tubes RMN sont scannés et chaque mélange en présence et en l’absence d’ARN, ainsi que les échantillons témoins, ont été mesurés conformément aux 19expériences RMN F susmentionnées effectuées à 298 K. Le criblage de l’ARN thiM par rapport à la bibliothèque interne a été effectué en effectuant des mesures T2 avec des CPMG de 0 ms et 200 ms pour chaque échantillon différent. Le calage et la suppression de l’eau ont été surveillés après avoir terminé les mesures en comparant tous les pics de DMSO en termes d’élargissement de la ligne et de perte d’intensité d’expériences 1H 1D mesurées supplémentaires pour tous les échantillons. Le traitement des spectres de relaxation CPMG T219F obtenus a été effectué à l’aide d’une macro préalablement préparée et automatisée dans TopSpin, respectivement. L’analyse des données a été effectuée en suivant les instructions de la section protocole. Les données intégrales obtenues à partir de TopSpin (en suivant les instructions du protocole) peuvent être évaluées rapidement et facilement à l’aide d’une feuille de calcul prédéfinie ou de tout programme similaire, en définissant les conditions et les seuils corrects. Comme décrit précédemment, les seuils sont utiles pour définir le classeur, le classeur faible ou le non-classeur. La figure 5 montre les résultats typiques des spectres CPMG de thiM ARN et PtkA, respectivement. Dans certains cas, une révision plus poussée par des experts a été nécessaire.
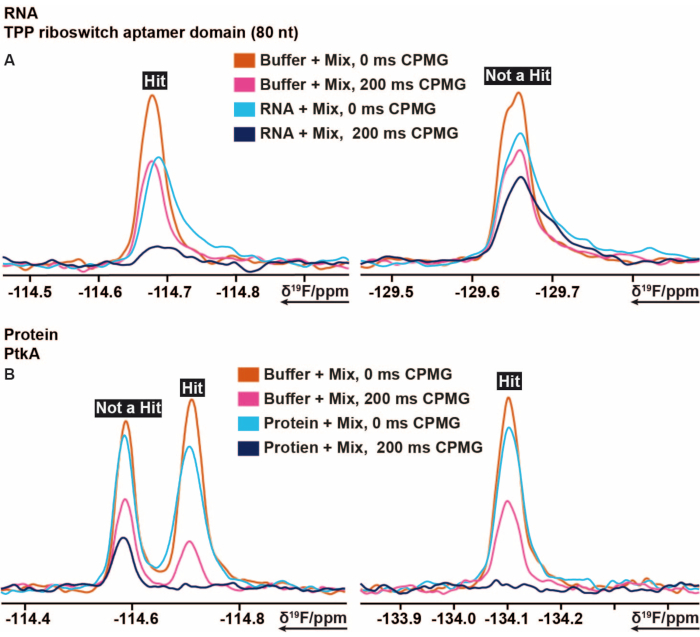
Figure 5 : Coupe de 19 spectres RMN CPMG montrant les changements d’intensité obtenus à partir de différents temps de retard d’expériences basées sur CPMG . (A) Représentation d’un liant (hit) et d’un non-liant dans le criblage à base de fragments 19F effectué sur l’ARN TPP riboswitch thiM d’E. coli. (B) Représentation d’un liant et d’un non-liant dans le criblage à base de fragments 19F effectué sur PtkA de M. tuberculosis. Veuillez cliquer ici pour voir une version agrandie de cette figure.
1h de dépistage
Conception du mélange
La bibliothèque interne utilisée est si diversifiée que pendant 1H, aucune conception de mélange n’a été réalisée. Cela signifie que 64 mélanges ont été préparés en choisissant au hasard 12 mélanges à mélanger dans un mélange.
Préparation des échantillons
Pour le criblage 1H d’un ARN SARS-CoV-2 exemplaire, un pipetage automatisé à l’aide d’un robot de pipetage a été effectué pour préparer les échantillons. Les fragments de chaque mélange présentaient une concentration de 4,2 mM dans 90 % de d6-DMSO et 10 % deD2O. Le volume final d’un échantillon de dépistage était de 200 μL avec 5 % deD2Ocomme agent de verrouillage. 64 échantillons contenant chacun un mélange différent dans 25 mM KPi, 50 mM KCl à pH 6,2 ont été pipetés sans ARN cible. Respectivement, 64 échantillons ont été pipetés avec de l’ARN cible, chacun contenant un mélange différent. Le rapport ARN:ligand a été fixé à 1:20, ce qui a donné une concentration d’ARN de 10 μM et une concentration de ligand de 200 μM.
Analyse des données
Pour l’analyse 1H, l’outil FBS de TopSpin a été utilisé. Pour déterminer si un fragment est un hit, des expériences de décalage chimique 1D, waterLOGSY et relaxation T2 ont été menées. Pour la relaxation T2 , une diminution de l’intensité supérieure à 30% a été considérée comme un résultat, tandis que pour le décalage chimique, un décalage supérieur à 6 Hz était le seuil. Le waterLOGSY devait montrer un changement de signal significatif (de négatif à positif dans ce cas). Si deux de ces trois critères étaient positifs, un fragment était compté comme un résultat. La figure 6 en donne deux exemples.
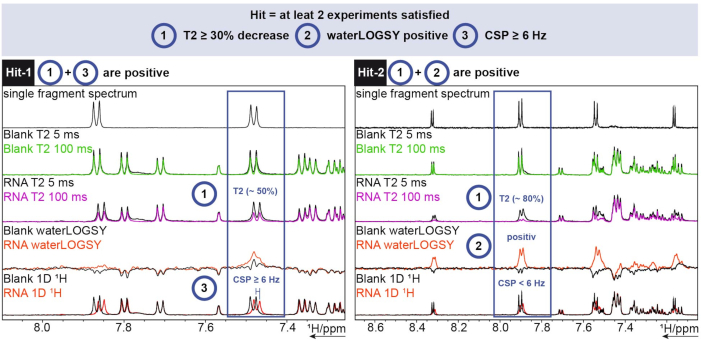
Figure 6 : 1H de dépistage réalisé sur un ARN exemplaire du SARS-CoV-2 présentant des critères de détermination des coups. Acquisition de trois expériences différentes (1 H T2 CPMG (5/100 ms), waterLOGSY, et 1D 1H). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Hit-1 montre une diminution de T2 de ~50% et un CSP ≥ 6 Hz. Le waterLOGSY ne montre pas un changement de signal suffisamment significatif pour être également considéré comme positif. Comme deux expériences sur trois sont positives, ce fragment est compté comme un succès. Pour Hit-2, le T2 montre une diminution de ~80% de l’intensité du signal et un changement de signal clair peut être vu pour le waterLOGSY. Le CSP n’est pas suffisant dans ce cas, mais comme les deux critères précédents sont positifs, il est toujours considéré comme un succès.
Discussion
Polyvalence du dépistage des fragments / drogues basé sur la RMN. BMRZ a mis en œuvre avec succès des instruments RMN automatisés de pointe ainsi que des expériences de RMN-STD, waterLOGSY et de relaxation pour identifier des fragments dans un large éventail de régimes d’affinité pour la découverte de médicaments. Le matériel installé comprend un robot de préparation d’échantillons à haut débit et une unité de stockage d’échantillons à haut débit, de changeur et d’acquisition de données associée à un spectromètre de 600 MHz. Une sonde cryogénique récemment achetée pour 1 H, 19 F, 13C et 15 N assure la sensibilité requise pour les mesures proposées et permet un découplage de1 H (1) lors de la détection 19F. Cette sonde est connectée à la dernière génération de console RMN qui offre la possibilité d’utiliser les outils logiciels avancés de Bruker, notamment CMC-q, CMC-assist, CMC-se et FBS (inclus dans TopSpin). L’outil de criblage basé sur les fragments (FBS) est inclus dans la dernière version de TopSpin et aide à analyser les données à haut débit comprenant les expériences de relaxation RTS, waterLOGSY, T2 / T1. La collecte d’échantillons liquide 1D 1H peut être remplie dans les tubes RMN de manière automatisée à l’aide du robot de remplissage d’échantillons. En règle générale, un bloc de 96 tubes (3 mm) est rempli en environ deux heures. Les racks à plaques de 96 puits sont directement positionnés dans le changeur d’échantillons HT, qui lit le code-barres du bloc et attribue les tubes RMN aux expériences contrôlées par le logiciel d’automatisation (IconNMR). Cinq racks à plaques de 96 puits peuvent être stockés et programmés dans le changeur d’échantillons HT en même temps. La température de chacun des racks individuels peut être contrôlée et réglée séparément. De plus, chaque échantillon individuel peut être préconditionné (préchauffage et séchage du tube pour éliminer l’humidité condensée) à la température souhaitée avant la mesure.
Convient à une large gamme d’applications. L’une des vastes applications de ce criblage automatisé basé sur la RMN est d’identifier et de développer de nouveaux ligands se liant à une cible biomacromoléculaire (ADN / ARN / protéines). Ces ligands peuvent inclure des inhibiteurs orthostériques et allostériques qui se lient généralement de manière non covalente. De plus, le FBDD par RMN est généralement utilisé comme première étape pour sélectionner des composés prometteurs, les exigences à respecter sont la disponibilité de la cible biomoléculaire en quantités suffisantes. Cet objectif est divisé en deux tâches principales.
La première tâche consiste à développer et à caractériser une bibliothèque de fragments interne pour les raisons suivantes: contrôle initial et périodique de la qualité, caractérisation et quantification de plus de 1000 fragments; détermination de la solubilité des fragments dans des tampons optimisés pour chaque cible, en particulier pour les cibles protéiques; et la création de plusieurs bibliothèques pour accueillir divers échafaudages et s’étendre à d’autres classes de macromolécules. La deuxième tâche consiste à intégrer les flux de travail pour la conception de médicaments à base de fragments (FBDD) par RMN en utilisant: le dépistage automatisé observé par ligand 1D (1H et 19F observés); essais de remplacement automatisés (expériences de compétition avec un ligand (naturel)) pour différencier la liaison orthostérique et allostérique; criblages secondaires automatisés avec plusieurs fragments; criblage automatisé des protéines 2D et filtrage secondaire d’un ensemble de dérivés autour d’un résultat initial en utilisant la bibliothèque EU-OPENSCREEN ou toute autre bibliothèque; et le reprofilage de la bibliothèque de la FDA par rapport aux cibles choisies.
De plus, le métabotypage de diverses lignées cellulaires (pertinentes pour la maladie) peut être effectué afin de démêler les mécanismes de régulation qui relient le contrôle du cycle cellulaire et le métabolisme. En outre, il existe une caractérisation fonctionnelle des éléments de régulation de l’ARN / ADN / protéine in vivo et in vitro pour l’optimisation de la construction / du domaine (optimisation de la stabilité pour les études structurelles (tampon, pH, température et criblage de sel), et une extension du criblage de fragments basé sur la RMN aux protéines membranaires et aux protéines intrinsèquement désordonnées, qui sont généralement inaccessibles à d’autres techniques.
Limitations. L’utilisation de bibliothèques de fragments 19F et 1H a ses avantages et ses inconvénients, dont peu seront mentionnés ci-dessous. Le plus grand avantage des mesures 19F par rapport à 1H est la rapidité du temps de mesure réel et de l’analyse ultérieure, car les mélanges contiennent presque le double du nombre de fragments et moins d’expériences doivent être menées. L’analyse de suivi est également plus facile pour le criblage 19F, car il n’y a pas d’interférence des tampons et offre en outre une plage de décalage chimique plus large avec presque aucun chevauchement de signal pour un mélange de fragments conçu de manière optimale. Les spectres eux-mêmes sont grandement simplifiés, n’ayant généralement qu’un ou deux signaux par fragment, en fonction du nombre d’atomes de fluor. L’analyse de ces spectres peut donc être automatisée, ce qui réduit encore le temps. Cela se fait au détriment de la diversité chimique, du moins pour la bibliothèque utilisée dans cette étude. Comme seulement ~13% de la bibliothèque contient 19 F, mais naturellement tous sont utilisables en criblage 1H, la diversité des fragments de criblage 19F sera plus faible. Cela pourrait être contourné en utilisant des bibliothèques 19F spécialement conçues avec plus de fragments et une plus grande diversité chimique. Un autre inconvénient du criblage 19F est le faible nombre de signaux par fragment. Les fragments sont généralement composés de plus d’un atome d’hydrogène. Par conséquent, les expériences de criblage observées à 1H peuvent s’appuyer sur différents signaux pour le même fragment afin de détecter la liaison. Cela donne un degré de confiance plus élevé lors de l’identification des résultats positifs pour le criblage de 1H, alors que le dépistage 19F doit s’appuyer sur un ou deux signaux donnés par fragment.
Un compte rendu détaillé de l’instrumentation moderne automatisée de criblage de fragments, des logiciels et des méthodes et protocoles d’analyse correspondants a été présenté. Le matériel installé comprend un robot de préparation d’échantillons à haut débit et une unité de stockage d’échantillons à haut débit, un changeur et une unité d’acquisition de données associés à un spectromètre de 600 MHz. Une tête de sonde cryogénique récemment installée pour 1 H, 19 F, 13C et 15 N assure la sensibilité requise pour les mesures proposées et permet un découplage de 1H lors dela détection 19F. De plus, la dernière génération de console RMN offre la possibilité d’utiliser un logiciel analytique avancé pour faciliter l’acquisition et l’analyse à la volée. La technologie, les flux de travail et les protocoles décrits ci-dessus devraient favoriser un succès remarquable pour les utilisateurs qui poursuivent FBS par RMN.
Déclarations de divulgation
Aucun.
Remerciements
Ce travail a été soutenu par iNEXT-Discovery, projet numéro 871037, financé par le programme Horizon 2020 de la Commission européenne.
matériels
| Name | Company | Catalog Number | Comments |
| Bruker Avance III HD | Bruker | 600 MHz NMR Spectrometer | |
| Matrix Clear Polypropylene 2D Barcoded Open-Top Storage Tubes | 3731-11 0.75ML V-BOTTOM TUBE/LATCH RACK | ThermoFisher Scientific | Barcoded Tubes |
| Matrix SepraSeal und DuraSeal& | 4463 Cap Mat, SeptraSeal 10/CS | ThermoFisher Scientific | |
| SampleJet | Bruker | HT Sample Changer | |
| SamplePro Tube | Bruker | Pipetting Robot |
Références
- Yanamala, N., et al. NMR-Based Screening of Membrane Protein Ligands. Chemical Biology & Drug Design. 75, 237-256 (2010).
- Souers, A. J., et al. ABT-199, a potent and selective BCL-2 inhibitor, achieves antitumor activity while sparing platelets. Nature Medicine. 19, 202-208 (2013).
- Su, M. C., Te Chang, C., Chu, C. H., Tsai, C. H., Chang, K. Y. An atypical RNA pseudoknot stimulator and an upstream attenuation signal for -1 ribosomal frameshifting of SARS coronavirus. Nucleic Acids Research. 33, 4265-4275 (2005).
- Perera, T. P. S., et al. Discovery & pharmacological characterization of JNJ-42756493 (Erdafitinib), a functionally selective small-molecule FGFR family inhibitor. Molecular Cancer Therapeutics. 16, 1010-1020 (2017).
- Zhang, C., et al. Design and pharmacology of a highly specific dual FMS and KIT kinase inhibitor. Proceedings of the National Academy of Sciences of the United States of America. 110, 5689-5694 (2013).
- Lipinski, C. A., Lombardo, F., Dominy, B. W., Feeney, P. J. Experimental and computational approaches to estimate solubility and permeability in drug discovery and development settings. Advanced Drug Delivery Reviews. 23, 3-25 (1997).
- Congreve, M., Carr, R., Murray, C., Jhoti, H. A 'Rule of Three' for fragment-based lead discovery. Drug Discovery Today. 8, 876-877 (2003).
- Chávez-Hernández, A. L., Sánchez-Cruz, N., Medina-Franco, J. L. A Fragment Library of Natural Products and its Comparative Chemoinformatic Characterization. Molecular Informatics. 39, 2000050(2020).
- Sreeramulu, S., et al. NMR quality control of fragment libraries for screening. Journal of Biomolecular NMR. , 00327-00329 (2020).
- Gao, J., et al. Automated NMR Fragment Based Screening Identified a Novel Interface Blocker to the LARG/RhoA Complex. PLoS One. 9, 88098(2014).
- Peng, C., et al. Fast and Efficient Fragment-Based Lead Generation by Fully Automated Processing and Analysis of Ligand-Observed NMR Binding Data. Journal of Medicinal Chemistry. 59, 3303-3310 (2016).
- Cox, O. B., et al. A poised fragment library enables rapid synthetic expansion yielding the first reported inhibitors of PHIP(2), an atypical bromodomain. Chemical Science. 7, 2322-2330 (2016).
- Hwang, T. L., Shaka, A. J. Water Suppression That Works. Excitation Sculpting Using Arbitrary Wave-Forms and Pulsed-Field Gradients. Journal of Magnetic Resonance, Series A. 112, 275-279 (1995).
- Gossert, A. D., Jahnke, W. NMR in drug discovery: A practical guide to identification and validation of ligands interacting with biological macromolecules. Progress in Nuclear Magnetic Resonance Spectroscopy. 97, 82-125 (2016).
- Sklenar, V., Bax, A. A new water suppression technique for generating pure-phase spectra with equal excitation over a wide bandwidth. Journal of Magnetic Resonance. 75, 378-383 (1987).
- Binas, O., et al. 19F NMR-Based Fragment Screening for 14 Different Biologically Active RNAs and 10 DNA and Protein Counter-Screens. ChemBioChem. , (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon